2013~2016年东部沿海地区甲型H1N1流感病毒分子特征分析*
章惠萍,易文晶,崔大伟(.杭州市肿瘤医院,杭州市第一人民医院吴山院区检验科,杭州 0002;2.江苏大学医学院检验医学系,江苏镇江 220;.浙江大学医学院附属第一医院检验科,浙江省临床体外诊断技术重点实验室,杭州 000)
·调查研究·
2013~2016年东部沿海地区甲型H1N1流感病毒分子特征分析*
章惠萍1,易文晶2,3,崔大伟3
(1.杭州市肿瘤医院,杭州市第一人民医院吴山院区检验科,杭州 310002;2.江苏大学医学院检验医学系,江苏镇江 212013;3.浙江大学医学院附属第一医院检验科,浙江省临床体外诊断技术重点实验室,杭州 310003)
目的 通过分析东部沿海地区(江苏、浙江和上海)甲型H1N1流感病毒[A(H1N1) pdm09]的分子特征,为防控A(H1N1) pdm09流感提供科学依据。方法 从GISAID数据库中获得江浙沪地区2013~2016年分离的A(H1N1) pdm09病毒株和WHO推荐疫苗株的血凝素(HA)和神经氨酸酶(NA)基因序列。运用MEGA6.0软件分析HA和NA的分子特征、关键氨基酸位点和耐药位点的变异情况。结果 进化树分析显示江浙沪地区A(H1N1) pdm09病毒株与WHO参考株的HA、NA基因序列差异较小,与WHO推荐疫苗株A/California/07/2009(H1N1)间的同源性分别为97.0%~99.5%和97.9%~99.6%;与WHO推荐疫苗株A/South_Africa/3626/2013(H1N1)间的同源性分别为98.0%~99.8%和98.6%~99.9%。病毒氨基酸变异分析显示70株A(H1N1)pdm09病毒的HA蛋白均发生83位(P83S)、203位(S203T)和321位(I321V)突变,且2016年分离株出现A195V突变;HA的裂解位点未发生变异,仍为IQSR↓GLF;2013年有2株NA蛋白出现H275Y耐药位点的变异。结论 2013~2016年东部沿海地区A(H1N1) pdm09流感病毒的HA和NA蛋白与WHO推荐疫苗株具有高度同源性,关键的氨基酸位点发生变异与病毒的毒力及对药物的敏感性相关,因此我们要密切关注A(H1N1) pdm09病毒,防止其再次发生暴发。
甲型H1N1流感病毒;分子特征;进化树;变异
2009年3月,墨西哥和美国等地首次发生了甲型H1N1流感病毒[A(H1N1)pdm09]流感大流行,流感病毒在随后的几个月里迅速蔓延到世界各地[1-2],此后受到高度关注。该甲流病毒由人流感病毒、猪流感病毒以及禽流感病毒的基因片段共同重组而成[1,3]。该病毒细胞膜蛋白血凝素(hemagglutinin, HA)和神经氨酸酶(neuraminidase,NA)的基因极易发生抗原漂移,且二者决定的关键的氨基酸位点对病毒的突变和耐药起决定性作用[4-5]。因此,本研究利用生物信息学方法,对2013年至2016年东部沿海地区感染A(H1N1) pdm09病毒患者的基因序列进行分子特征和氨基酸变异情况的分析,进而为东部沿海地区A(H1N1) pdm09流感病毒的防控提供相关参考依据。
1 材料与方法
1.1 材料 从全球禽流感共享数据库(the Global Initiative on Sharing Avian Influenza Data, GISAID)中下载江苏、浙江、上海地区2013~2016年人感染A(H1N1)pdm09病毒的基因序列,同时下载WHO推荐疫苗株A/California/07/2009(H1N1)(作为原始参考株)和A/South_Africa/3626/2013(H1N1)(作为对比参考株)的基因序列。
1.2 方法 采用Molecular Evolutionary Genetics Analysis(MEGA)version 6.0将下载的序列进行比对,用邻接法(Neighbour-Jioning, N-J)构建HA、NA的氨基酸种系进化树,采用Bootstrap法(replication=1 000)进行检验。同时进行病毒氨基酸比对,分析病毒氨基酸重要位点的变异情况。
2 结果
2.1 A(H1N1)pdm09病毒代表株同源性和系统进化树
2.1.1 HA蛋白同源性和系统进化树 用2013~2016年东部沿海地区70株A(H1N1)pdm09病毒代表株和2株WHO推荐参考株构建HA氨基酸系统进化树,见图1。结果显示A/California/07/2009(H1N1)参考株与A/Shanghai-Minxing/SWL11041/2015差异最小,同源性为99.5%,同属组1(Group 1);A/South_Africa/3626/2013(H1N1)参考株与A/Shanghai-Changning/SWL1621/2013差异最小,同源性为99.8%,同属组2(Group 2)。Group 1中,各年份各地区的病毒株均有分布,同源性为98.5%~100%;Group 2中,各年份各地区病毒株均有分布,同源性为99.4%~100%;组3(Group 3)为浙江和上海地区近两年的病毒株,同源性为99.4%~100%。
2.1.2 NA蛋白同源性和系统进化树 构建了A(H1N1)pdm09 病毒代表株的NA蛋白系统进化树,见图2。结果显示A/California/07/2009(H1N1)参考株与A/Shanghai-Minxing/SWL1660/2013差异最小,同源性为99.6%;A/South_Africa/3626/2013(H1N1)参考株与A/Shanghai-Baoshan/SWL1970/2015差异最小,同源性为99.9%,同属组3(Group 3)。组1(Group 1)中,各地区近两年病毒株均有分布,同源性为99.5%~100%;组3(Group 3)中,各年份各地区病毒株均有分布,同源性为99.1%~100%。
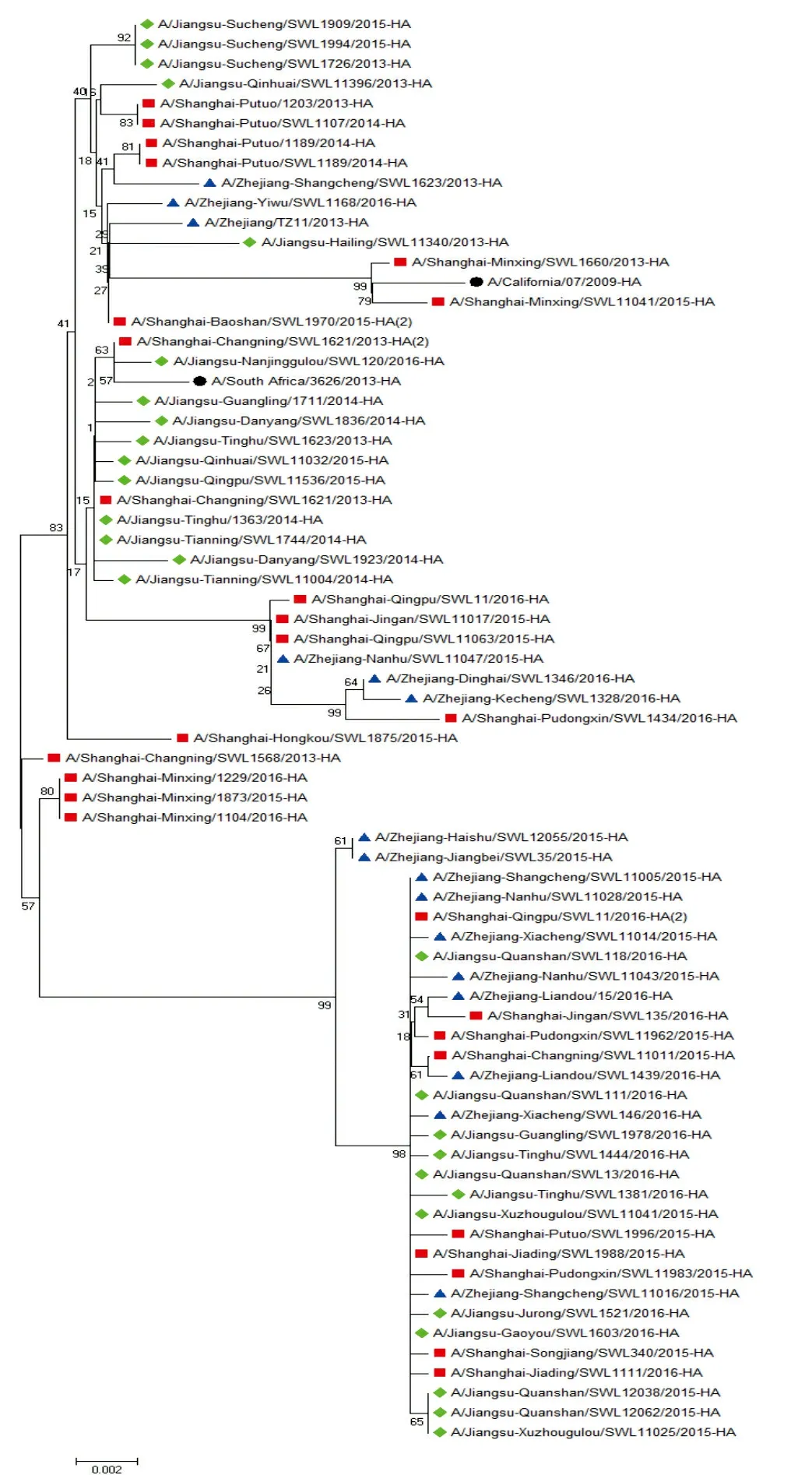
注:●WHO推荐疫苗株;■上海地区病毒株;▲浙江地区病毒株;◆江苏地区病毒株。
图1 A(H1N1)pdm09病毒禽流感HA蛋白系统进化树
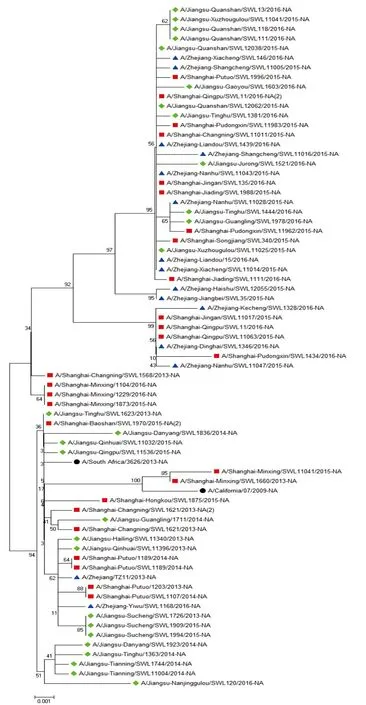
注:●WHO推荐疫苗株;■上海地区病毒株;▲浙江地区病毒株;◆江苏地区病毒株。
图2 A(H1N1)pdm09病毒NA蛋白系统进化树
2.2 HA和NA蛋白关键位点变异情况分析 HA蛋白受体结合关键氨基酸位点分析:将2013~2016年70株代表株和2株人感染A(H1N1)pdm09病毒参考毒株进行对比,70株病毒株在3个关键位点83位、321位、203位均相对于2009年A/California/07/2009(H1N1)参考株发生了P83S、I321V、S203T突变;这3个位点与2013年A/South_Africa/3626/2013(H1N1)参考株突变情况相同。值得注意的是,在195位上A/Shanghai-Minxing/1229/2016病毒株发生A195V突变。东部沿海地区70株A(H1N1) pdm09切割位点序列均为IQSR↓GLF,与WHO推荐疫苗株相比皆无改变。此外,为了探讨70株病毒株对神经氨酸酶抑制剂类药物的敏感性和耐药性,我们对70株A(H1N1)pdm09病毒的NA的氨基酸序列进行分析,发现NA中与耐药有关的氨基酸位点有1株发生H275Y变异,而氨基酸位点S247N均未发生变异。
3 讨论
本研究发现东部沿海地区病毒株的基因序列和WHO推荐的2株疫苗株高度同源;江浙沪三省的病毒株总体来讲各年份各地区散在分布。HA进化树可见病毒株与WHO推荐疫苗株处于同一主要分支,表明东部沿海地区流行的毒株具有代表性,对东部沿海地区的病毒株进行监测可能对及时掌握全国流感流行情况有代表性意义。
流感病毒的致病性与HA抗原决定簇位点的变异最为相关[5-7],但只有在HA区4个以上氨基酸序列中的2~3个抗原决定簇位点发生替换现象,才能说明该变异具有流行病学意义[7]。此次我们用东部沿海地区病毒代表株HA基因组序列与WHO推荐的疫苗株进行比较,发现HA区的抗原决定簇只有1个氨基酸位点发生替换,其中涉及了A与V两个抗原决定簇。有研究显示,HA中氨基酸位点P83S、S203T、1321V突变在流行早期已经成为3个适应性突变,并在以后流行的甲型H1N1流感病毒中稳定遗传[6],本次分析也符合此观点。因此可以推测东部沿海地区70株病毒株的HA基因基本处于比较稳定的状态。
通过比较HA的氨基酸序列发现,分析的70株病毒株在HA切割位点处的氨基酸序列均为IQSR↓GLF,与参考株在HA切割位点处的氨基酸序列相一致。A(H1N1)pdm09流感病毒的组织嗜性由上述关键氨基酸的切割位点决定,该切割位点发生突变更容易导致高致病性病毒的产生,进而引起更高的发病率和致死率,然而这70株病毒株不符合高致病性毒株的分子特征[RXR/KR↓GLT(X为碱性氨基酸)][5, 7-9]。这些结果提示我们东部沿海地区70株A(H1N1)pdm09均为非高致病性毒株。
为了分析东部沿海地区的A(H1N1)pdm09毒株的药物敏感性,我们对病毒耐药相关的关键氨基酸位点进行了比较。在NA蛋白中,氨基酸S247N与H275Y的任一位点突变均可导致A(H1N1)pdm09流感病毒对神经氨酸酶抑制剂类药物(如奥司他韦,扎那米韦)的敏感性下降[5,10,11]。在选取的东部沿海地区流行的70株A(H1N1)pdm09流感病毒中有2株氨基酸位点发生了H275Y变异,而氨基酸位点S247N均未发生突变。分析结果提示,虽然目前东部沿海地区的A(H1N1)pdm09流感病毒对奥司他韦耐药现象仍属罕见,暂不会发生新型耐药性流感的暴发,但是耐药毒株已经出现,需要密切监测病毒变化,防止耐药毒株的进一步扩散和传播。
迄今为止大多数A(H1N1)pdm09病毒毒株的抗原特征仍可以由疫苗预防,但是随着A(H1N1) pdm09流感持续发生,临床在关注该病毒本地流行进化的同时,也应时刻警惕耐药毒株转化为优势株的可能性,由此来指导临床治疗用药,进而有效地预防A(H1N1)pdm09流感的再次暴发。
本研究对GenBank上登录的东部沿海地区甲型流感病毒HA及NA基因的氨基酸序列进行比较,通过进化树、同源性和基因变异的分析,初步探讨甲流病毒H基因与N基因大致的变异趋势,为防控甲流疫情提供科学依据。但因甲流毒株在基因库上登录的资料并不完全,本研究尚存在着一定的局限性,提示我们在甲流防控工作中应加强对病毒基因型变异的监测,以便做更全面准确的分析。
[1]Novel Swine-Origin Influenza A(H1N1) Virus Investigation Team, Dawood FS, Jain S,etal. Emergence of a novel swine-origin influenza A(H1N1) virus in humans[J]. N Engl J Med, 2009, 360(25):2605-2615.
[2]Fang Q, Gao Y, Chen M,etal. Molecular epidemiology and evolution of A(H1N1)pdm09 and H3N2 virus during winter 2012-2013 in Beijing, China[J]. Infect Genet Evol, 2014, 26: 228-240.
[3]Byarugaba DK, Erima B, Millard M,etal. Whole-genome analysis of influenza A(H1N1)pdm09 viruses isolated in Uganda from 2009 to 2011[J]. Influenza Other Respir Viruses, 2016, 10(6):486-492.
[4]Tran DN, Pham TM, Ha MT,etal. Molecular epidemiology of influenza A virus infection among hospitalized children in Vietnam during post-pandemic period[J]. J Med Virol, 2015, 87(6):904-912.
[5]吴英萍,崔大伟,秦志梅,等. 2009~2012年杭州地区甲型H1N1流感病毒HA和NA基因的进化分析[J]. 临床检验杂志, 2013, 31(4): 272-275.
[6]Hong M, Lee PS, Hoffman RM,etal. Antibody recognition of the pandemic H1N1 Influenza virus hemagglutinin receptor binding site[J]. J Virol, 2013, 87(22):12471-80.
[7]Xu R, Ekiert DC, Krause JC,etal. Structural basis of preexisting immunity to the 2009 H1N1 pandemic influenza virus[J]. Science, 2010, 328(5976):357-360.
[8]易丽娴,陶鸿,崔大伟. 江苏省2013至2015年人H7N9流感病毒的分子特征[J]. 临床检验杂志, 2016, 34(7):612-616.
[9]Böttcher-Friebertshäuser E, Freuer C, Sielaff F,etal. Cleavage of influenza virus hemagglutinin by airway proteases TMPRSS2 and HAT differs in subcellular localization and susceptibility to protease inhibitors[J]. J Virol, 2010, 84(11):5605-5614.
[10]Xu H, Yang Y, Wang S,etal. Predicting the mutating distribution at antigenic sites of the influenza virus[J]. Sci Rep, 2016, 6:20239.
[11]Takashita E, Kiso M, Fujisaki S,etal. Characterization of a large cluster of influenza A(H1N1)pdm09 viruses cross-resistant to oseltamivir and peramivir during the 2013-2014 influenza season in Japan[J]. Antimicrob Agents Chemother, 2015, 59(5):2607-2617.
(本文编辑:刘群)
Molecular characteristics analysis of influenza A(H1N1) pdm09 virus in the eastern foreland during 2013-2016
ZHANGHui-ping1,YIWen-jing2,3,CUIDa-wei3
(1.DepartmentofLaboratoryMedicine,HangzhouCancerHospital,WushanDistrictofHangzhouFirstPeople′sHospital,Hangzhou310002,Zhejiang; 2.DepartmentofLaboratoryMedicine,SchoolofMedicine,JiangsuUniversity,Zhenjiang212013,Jiangsu; 3.DepartmentofLaboratoryMedicine,theFirstAffiliatedHospital,CollegeofMedicine,ZhejiangUniversity,KeyLaboratoryofClinicalInVitroDiagnosticTechniquesofZhejiangProvince,Hangzhou310003,Zhejiang,China)
Objective To provide scientific basis for the prevention and control of influenza A(H1N1) pdm09 influenza by analyzing the molecular characteristics of influenza A(H1N1) pdm09 virus in the eastern foreland(Jiangsu, Zhejiang and Shanghai). Methods The gene sequences of hemagglutinin(HA) and neuraminidase(NA) of influenza A(H1N1) pdm09 virus strains isolated from Jiangsu, Zhejiang and Shanghai regions during 2013-2016 and the vaccine strains recommended by WHO were obtained from the GISAID database. The molecular characteristics and variations of key amino acid sites and drug resistant loci of HA and NA were analyzed by the MEGA6.0 software. Results Phylogenetic tree analysis showed that there were little discrepancies of HA and NA gene sequences between influenza A(H1N1) pdm09 virus strains from Jiangsu, Zhejiang and Shanghai regions and reference strains recommended by WHO, and that the homologies between these isolated strains and reference strain A/California/07/2009(H1N1) were 97.0%-99.5% and 97.9%-99.6%, respectively, and 98.0%-99.8% and 98.6%-99.9% between these isolated strains and reference strain A/South_Africa/3626/2013(H1N1). Viral amino acid variation analysis showed that the mutations of HA protein, including P83S, S203T and I321V, existed in 70 influenza A(H1N1) pdm09 virus strains, and that the A195V mutation was found in the isolated strains from 2016. The cleavage site, IQSR↓GLF, of HA remained no change. In addition, the variation of H275Y resistant locus in NA protein was found in 2 strains from 2013. Conclusion The HA and NA proteins of influenza A(H1N1) pdm09 virus strains from the eastern foreland during 2013-2016 are highly homologous with the vaccine strains recommended by WHO. The variations of key amino acid sites are associated with the virulence of viruses and the sensitivity of drugs. Therefore, we should pay closely attention to the variations of influenza A(H1N1) pdm09 viruses so as to prevent its outbreak again.
influenza A(H1N1) pdm09 virus; molecular characteristics; phylogenetic tree; variation
浙江省医药卫生科技项目(2015KYB149);浙江省自然科学基金(LY16H200001);浙江省教育厅项目(Y201534117)。
章惠萍,1985年生,女,技师,大学本科,从事医学微生物检验工作。
崔大伟,副主任技师,博士,E-mail:cuidawei1978@163.com。
10.13602/j.cnki.jcls.2017.04.20
R512.5
A
2017-01-04)

