冷冻原肠浸泡解冻池水细菌多样性分析
仇保丰,宋鸿雁,董蓉莲,高雪梅,蒋荧梅,顾炳泉,胡顺林
(1.扬州大学江苏省动物预防医学重点实验室,江苏扬州 225009;2.南通出入境检验检疫局,江苏南通 226004;3.南通大学实验动物中心,江苏南通 226001;4.江苏省疾病预防控制中心,江苏南京 210009)
冷冻原肠浸泡解冻池水细菌多样性分析
仇保丰1,2,宋鸿雁1,3,董蓉莲4,高雪梅2,蒋荧梅3,顾炳泉2,胡顺林1*
(1.扬州大学江苏省动物预防医学重点实验室,江苏扬州 225009;2.南通出入境检验检疫局,江苏南通 226004;3.南通大学实验动物中心,江苏南通 226001;4.江苏省疾病预防控制中心,江苏南京 210009)
为了研究冷冻原肠浸泡解冻池水的细菌多样性,采集3份水样直接提取细菌基因组总DNA,再分别用PCR技术扩增出细菌16S rDNA并构建基因文库,经PCR鉴定后从3个文库中分别随机挑取50个阳性克隆进行DNA序列测定。分别对3个文库的测序结果进行分析发现,除去24.00%~32.00%为无法鉴定的未培养细菌和未分类细菌以外,其余细菌来自于芽胞杆菌纲、γ-变形菌纲、放线菌纲和黄杆菌纲,分别占各文库克隆总数的38.00%~42.00%、20.00%~26.00%、4.00%~8.00%和0~2.00%。除去44株无法鉴定的细菌,进一步对其他106株细菌进行分析发现,其中2株放线菌目细菌和5株肠杆菌科细菌无法进一步鉴定,其余99个菌株包含了来自19个属的至少30种细菌,且肠球菌属、变形杆菌属和芽胞杆菌属为其中的优势菌属。本研究显示冷冻原肠浸泡解冻池水中细菌多样性较高,其中部分细菌为致病菌和条件致病菌。
冷冻原肠;解冻池;细菌多样性;16S rDNA文库
我国是世界上最大的天然肠衣(natural casings)生产国,年产量占世界总产量的三分之一[1]。由于我国生产的天然肠衣具有加工细致、肠壁韧性适宜、口径分路准确和可用于加工不同类型的灌肠类食品等优点而广受国内外客户的喜爱,目前仅盐渍猪肠衣就已远销至40多个国家和地区。但是,随着近年来人们对食品安全问题的愈加重视,天然肠衣携带微生物的问题已逐渐成为继兽药残留之后的又一个制约肠衣行业大发展的新难题。例如,早在1996年欧洲天然肠衣协会(ENSCA)就为天然肠衣设立了微生物推荐值,且这些限量指标正不断趋于更加严苛[2-3]。不仅如此,ENSCA还将沙门菌、大肠埃希菌和金黄色葡萄球菌等7种细菌列为天然肠衣中有生物危害的细菌,认为可对食品安全形成威胁。
天然肠衣的主要原料是动物原肠(intestine),它是猪、羊等健康牲畜屠宰后去粪、清洗且未经刮制的小肠。因此,天然肠衣携带肠内或外源微生物是无法避免的;不仅如此,清洗、冻藏、运输等环节的温度和卫生条件的控制情况等也是影响微生物污染水平的重要因素[4-5]。但是截至目前,国内外对原肠携带细菌的多样性研究仍严重偏少,且相关报道主要采用细菌人工培养、鉴定的传统方法,研究结论也相差甚远[6-7];在肠衣加工和污水处理等环节有关微生物的监控资料更是严重匮乏,这都不能满足肠衣生产加工中降低生物安全风险和促进肠衣行业健康发展的现实需求。本研究采用非培养法直接从冷冻原肠的浸泡解冻池水中提取细菌基因组DNA,通过PCR扩增构建16S rDNA克隆文库,再根据随机测序结果分析浸泡解冻池水中的细菌多样性,不但可以为研究冷冻原肠携带细菌的菌群结构等提供思路,也可为掌握肠衣加工环节污染情况和提高肠衣污水处理成效等提供资料。
1 材料与方法
1.1 材料
1.1.1 水样 3份水样分别采集自南通地区3家肠衣生产、加工企业的冷冻原肠浸泡解冻池。
1.1.2 主要试剂 酵母浸提物和胰蛋白胨为OXOID公司产品;琼脂粉为中国医药(集团)上海化学试剂公司产品;细菌基因组DNA提取试剂盒为天根生化科技(北京)有限公司产品;pGEM-T easy vector为Promega公司产品;dNTPs(10 mmol/L each)、rTaqDNA聚合酶(5 U/μL)、DNA Marker DL 2 000、DNA胶回收试剂盒和16S rDNA Bacterial Identification PCR Kit均为宝生物工程(大连)有限公司产品。
1.2 方法
1.2.1 水样的采集和处理 水样的采集方法简述如下:企业冷冻原肠浸泡解冻池经过排水、清洗和消毒后,密封排水口。从塑料桶内拿出尼龙编织袋包装的冷冻原肠放入浸泡解冻池,同时重新注入自来水至完全浸没肠衣,每间隔30 min用清洁的带钩长杆充分翻转肠衣包装袋3 min,待所有的原肠基本解冻时,再用长杆翻转肠衣包装袋10 min,接着用灭菌处理过的玻璃器皿,从解冻池的四角和中间分别取水样500 mL,将5个点的水样共同注入一个洁净的采样袋内,充分混匀后取500 mL注入灭菌处理过的玻璃瓶内,密封后放入装有冰袋的保温箱内立即送往实验室,将水样品摇匀后分装入无菌离心管内,4℃、10 000 r/min离心10 min,集中菌体加入1 mL无菌TE缓冲液,置-40℃冻存备用。
1.2.2 细菌基因组DNA的提取 将3份水样收集细菌分别用商品化的细菌基因组DNA提取试剂盒直接提取细菌基因组总DNA,具体操作方法参照试剂盒的说明书。
1.2.3 细菌16S rDNA文库的构建 将1.2.2提取的3份细菌基因组总DNA分别作为模板,用16S rDNA Bacterial Identification PCR Kit扩增肠衣携带细菌的16S rDNA,琼脂糖凝胶电泳后,分别切胶、回收目的条带并克隆入pGEM-T easy vector,转化DH5α后依次涂布AIX选择性琼脂平板,37 ℃培养12 h~16 h,进行蓝白斑筛选。
1.2.4 细菌16S rDNA序列的测定及分析 用灭菌处理过的牙签随机从3份水样的AIX平板上挑取白色菌落,分别接种含氨苄(终浓度100 μg/mL)的LB液体培养基,37 ℃培养12 h~16 h,提取质粒用PCR鉴定出阳性克隆,每份水样随机挑取50个阳性克隆送生工生物工程(上海)股份有限公司进行DNA序列测定,并将所测序列用PDRⅡ中的Chimera Check程序进行检查,以剔除嵌合体。最后将DNA测序结果输入http://www.ncbi.nlm.nih.gov/BLAST/进行网上比对,根据比对结果分析细菌的多样性。
2 结果
2.1 细菌16S rDNA的PCR扩增
对冷冻原肠解冻水池的水样收集细菌,用细菌基因组DNA提取试剂盒直接提取基因组总DNA,接着用16S rDNA Bacterial Identification PCR Kit对细菌16S rDNA进行扩增,琼脂糖电泳结果显示,扩增出了约为1 700 bp的条带,与预期的条带大小一致(图1)。
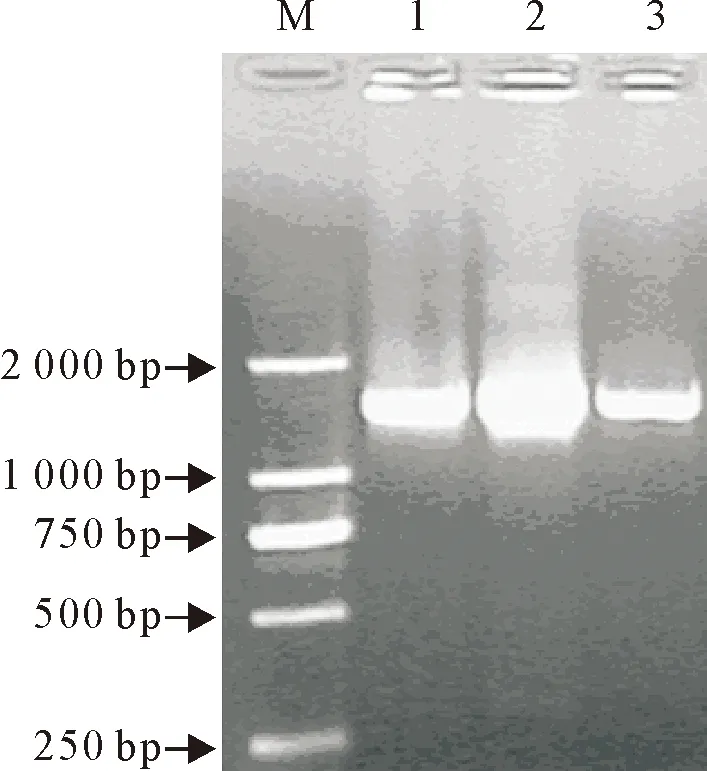
M.DNA标准 DL 2 000; 1~3.细菌16S rDNA的PCR产物
2.2 细菌16S rDNA文库的构建
根据3份原肠细菌16S rDNA的琼脂糖凝胶电泳结果,分别切胶、回收目的条带并克隆入pGEM-T easy vector,转化DH5α后依次涂布AIX选择性琼脂平板,37 ℃培养12 h~16 h,发现AIX选择性琼脂平板上白色菌落较多,蓝色菌落很少(图2),初步判断冷冻原肠细菌16S rDNA文库的构建比较成功。

图2 AIX平板上的重组细菌菌落
2.3 细菌16S rDNA文库的鉴定
从3份水样的AIX平板上分别随机挑取白色菌落,接种含氨苄抗性的LB液体培养基,37 ℃培养12 h~16 h,提取质粒用PCR鉴定出阳性克隆,绝大多数菌落均能扩增出约为1 700 bp条带,与预期大小一致(图3)。从3份水样的阳性克隆中分别选择50个PCR鉴定目的条带较亮的菌落,送生工生物工程(上海)股份有限公司进行DNA序列测定。测序结果经嵌合体检验,均不是嵌合序列,最后将测序结果输入http://www.ncbi.nlm.nih.gov/BLAST/进行网上比对,克隆文库中的序列与NCBI已登录的任一序列同源性最高,且同源性大于或等于97%的归结为同一种细菌;序列同源性小于97%的定义为“未分类细菌(Unclassified bacteria)”,比对结果见表1。

表1 16S rDNA克隆文库分析
注:“-”表示“未确定”;“N”表示菌株名称未见中文翻译。
Note:“-” indicates “unidentified”; “N” indicates the Chinese name of this bacterium is not found.
2.4 细菌16S rDNA文库的分析
本研究测定的150个克隆中,将28株未培养细菌和16株未分类细菌共同定义为“未确定(unidentified)”细菌,剩余106株细菌分别来自4个纲,即放线菌纲(Actinobacteria)8株,γ-变形菌纲(γ-Proteobacteria)37株,芽胞杆菌纲(Bacilli)59株和黄杆菌纲(Flavobacteria)2株。其中芽胞杆菌纲的细菌占克隆总数的比例最高,约为39.33% (59/150),γ-变形菌纲细菌所占比例约为24.67% (37/150),放线菌纲和黄杆菌纲的细菌所占比例较低,而“未确定(unidentified)”细菌所占比例为29.33% (44/150)。再分别对3份水样的克隆文库进行分析发现,芽胞杆菌纲细菌在各文库中所占的比例为38.00%~42.00%;γ-变形菌纲细菌所占的比例20.00%~26.00%,放线菌纲和黄杆菌纲的细菌所占比例分别为4.00%~8.00%和0~2.00%,另外,“未确定”细菌所占的比例为24.00%~32.00%(图4)。由此可见,在冷冻原肠浸泡解冻池水中,芽胞杆菌纲和γ-变形菌纲的细菌为优势菌群。
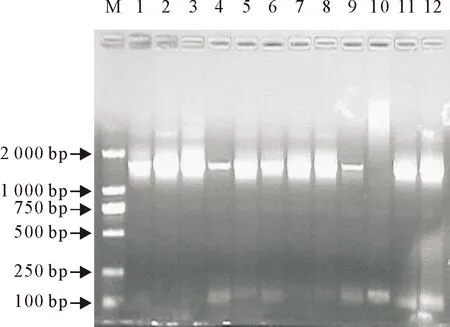
M.DNA标准 DL 2 000; 1~12.重组质粒的PCR产物
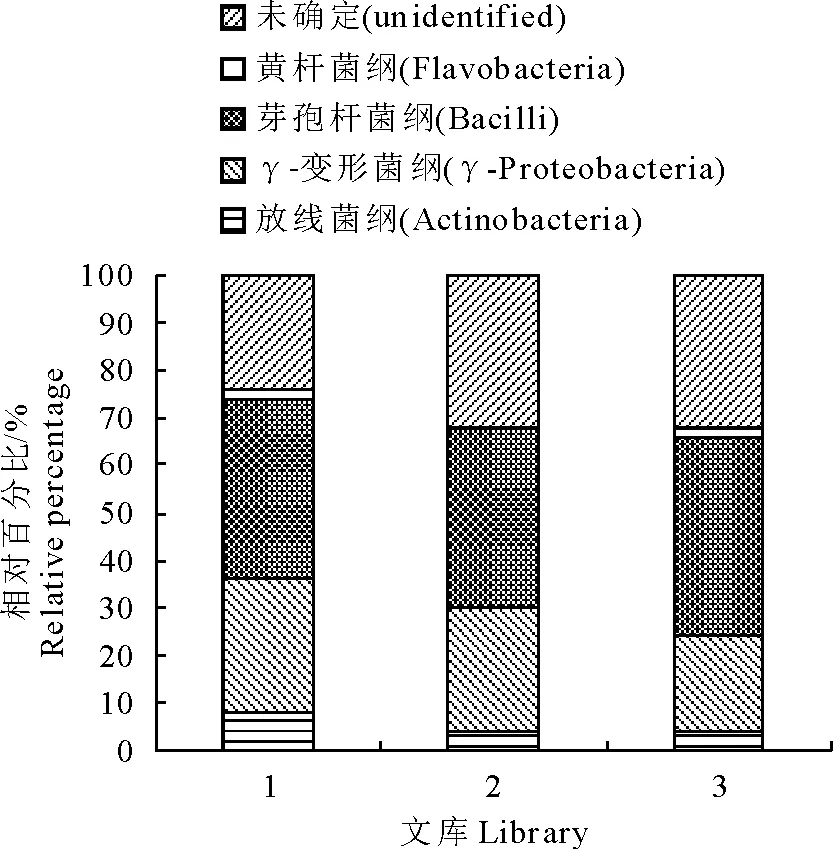
图4 3个16S rDNA克隆文库的细菌群落结构
进一步分析发现,除去44株“未确定”细菌,其余106株细菌中,2株放线菌目细菌和5株肠杆菌科细菌未能鉴定到属(Genus),其余99个菌株分别为来自于19个属的至少30种细菌(94株细菌鉴定到种),其中肠球菌属(Enterococcus)、变形杆菌属(Proteus)和芽胞杆菌属(Bacillus)为优势菌属(表1)。另外,研究中还检出了沙门菌、葡萄球菌、肠球菌和变形杆菌等致病菌或条件致病菌。
3 讨论
研究表明,冷冻原肠携带的细菌量可达到104CFU/g~107CFU/g[4-5,8],有时甚至可能携带包括沙门菌、肠出血性大肠杆菌O157∶H7、摩根氏菌、肠球菌、变形杆菌、志贺菌、链球菌和亚硫酸盐还原梭状芽胞杆菌等在内的多种致病菌或条件性致病菌[6-10]。但由于我国出口的肠衣主要是经过盐渍(腌制)的成品肠衣,且绝大多数细菌在盐渍的高渗环境下经过一段时间就会被杀灭[2,8];再加之出口的肠衣主要是用作灌肠类食品的衣膜并经过再次加工、蒸煮后才供食用,因此,出口成品肠衣携带细菌的生物安全风险相对较低。也正因如此,近年来国外的相关报道主要集中在采用人工添加特定细菌的方法,研究氯化钠[2,5,8]、臭氧[11]和辐射[4,12]等手段对肠衣携带细菌的杀灭效果上,但是对冷冻原肠携带细菌的多样性研究非常少。我国由于肠衣原料需求量大、劳动力成本低廉和加工工艺优良等原因,不但是肠衣出口大国,还是世界上重要的肠衣来料和进料加工大国,每年有大量的来自不同国家和地区的肠衣原料输入我国进行生产加工,其中有相当一部分肠衣原料为冷冻原肠,这给我国畜牧业健康和公共卫生安全带来严峻挑战。但截至目前,国内对原肠携带细菌的多样性研究仍严重偏少,且仅有的少量报道也主要采用传统的细菌人工培法,研究结论更是无法相互印证[6-7]。分析其原因:首先,传统的细菌人工培养法研究细菌多样性具有工作量大、耗时长等不足,从而导致各研究小组鉴定细菌的总数差异较大且总体偏少;其次,即便是同一批冷冻原肠其来源动物的数量也非常庞大,而冷冻原肠携带的肠内或外源微生物的数量和种类,不但易受动物个体屠宰前的健康、饮食和用药等多种因素的影响,而且容易受到动物屠宰后小肠去粪、清洗、保存和运输过程中卫生及温度控制情况的影响,因此小批量采集的原肠样品难以保证其代表性和科学性。再次,由于肠道细菌有60%~80%是无法人工培养的[13],故而采用非培养法研究肠道细菌多样性已被广泛认同和采用,但因为动物原肠是经过去粪、清洗过的小肠,因此既无法通过肠道内容物(粪便)来研究原肠携带细菌的多样性,也由于原肠细菌数量比正常动物肠道大为减少和动物组织细胞等干扰物质过多等原因,从原肠中直接提取细菌基因组总DNA的难度非常大,目前国内尚无报道。
肠衣生产在整个加工链中用水量极大,并且产生大量的有机废水[14]。据统计,我国肠衣加工废水的年排放量约为270万吨,并且发展了生物降解、化学水解、物理混凝和综合利用等技术用于处理高COD、高盐、高氨氮和易腐臭的肠衣污水[14-15]。尽管早在1987年尹振声[16]就报道了肠衣废水的细菌总数可达5.0×104个/mL~3.0×105个/mL。但多年以来,研究人员对肠衣浸泡解冻等加工废水中的细菌多样性问题仍然缺乏关注,对肠衣废水处理的效果评价中也鲜有微生物指标。造成这一局面的原因,可能是目前有关肠衣加工废水中微生物多样性的研究不足,公开的相关数据和报道极度匮乏,未能引起肠衣废水处理研究人员的重视,同时由于肠衣加工后续环节中氯化钠(NaCl)的使用量比较大,人们想当然地认为肠衣废水收集池中的高盐环境会对细菌起到有效的抑制生长和杀灭作用。事实上由于耐盐菌、嗜盐菌和对高盐环境耐受力极强的细菌芽胞的存在,即便是用过饱和的盐卤或干盐充分盐渍的成品肠衣,其菌落总数有时也会超过103CFU/g[3]。由此就不难理解,为何对于富含有机物的肠衣加工废水来说,即便含盐量较高,其细菌总数也非常惊人了。另外,目前国内尚无对肠衣加工废水进行细菌多样性、溯源性和生物安全性的研究报道,这可能会为进一步优化肠衣废水处理工艺和科学设立评价指标等埋下隐患,尤其是当前多种肠衣污水处理工艺因成本、效率和干扰等问题遇到阻碍后,提出将发展方向转为先对各股废水分别进行适当的预处理,后混合进行生化处理的联合治理工艺的形势下[14-15],这一隐患很有可能会变为现实。
本研究采用非培养法直接从冷冻原肠的浸泡解冻池水中提取细菌基因组总DNA,通过PCR扩增构建16S rDNA克隆文库,再根据随机测序结果分析浸泡解冻池水中的细菌多样性,不但可以规避非培养法研究原肠细菌多样性中遇到的很多难题,为研究冷冻原肠携带细菌的菌群结构等提供思路。同时,冷冻原肠冷水浸泡解冻过程也是肠衣加工中发生交叉污染最为严重的环节,对原肠浸泡解冻池水的细菌多样性进行研究,还可为掌握肠衣加工环节污染情况、探索肠衣原料卫生控制途径及肠衣加工污水无害化处理等提供资料。通过研究发现,芽胞杆菌纲细菌在3份水样的细菌16S rDNA克隆文库中所占的比例为38.00%~42.00%,γ-变形菌纲细菌所占的比例为20.00%~26.00%,放线菌纲和黄杆菌纲的细菌所占比例分别为4.00%~8.00%和0~2.00%。除此之外,3个克隆文库中尚有24.00%~32.00%的细菌无法确定种群(图4)。进一步分析发现,除去44个无法确定种群的克隆,其余106个克隆中有2株放线菌目细菌和5株肠杆菌科细菌未能鉴定到属,其余99个菌株包含了来自19个属的至少30种细菌,冷冻原肠浸泡解冻池水中肠球菌属(Enterococcus)、变形杆菌属(Proteus)和芽胞杆菌属(Bacillus)为优势菌属。研究中还检出了沙门菌、葡萄球菌、肠球菌和变形杆菌等致病菌或条件致病菌。目前,国内外的成品肠衣主要采用盐渍(腌制)的方法进行保存,但肠衣的盐渍过程对细菌芽胞的杀灭效果非常有限[3,17]。国外已有研究发现,盐渍肠衣携带的细菌60%以上来自芽胞杆菌属(Bacillus)[11]。考虑到国外曾对包括我国在内的多个国家出口的盐渍肠衣进行过监测,发现仅有来自中国的盐渍肠衣携带还原亚硫酸盐梭状芽胞杆菌(sulphite-reducingClostridium)的芽胞水平超过了ENSCA推荐的可接受指标,部分样品甚至超过了最大限量指标[17]。因此,对于本研究发现原肠浸泡解冻池水中芽胞杆菌属细菌所占比例偏高的问题要给予关注。另外,本课题小组曾用人工培养法对冷冻原肠分离的65株细菌进行过研究,发现肠球菌属和变形杆菌属的细菌所占的比例非常高,这与本研究的结果比较一致[6]。刘芳等[7]用人工培养法从腌制前的肠衣原料中分离到10株细菌,鉴定发现其中4株为肠球菌属细菌。这一方面,说明本课题采用研究冷冻原肠的浸泡解冻池水的细菌多样性用以反映冷冻原肠的细菌多样的思路,在当下的确有一定的参考价值;另一方面,由于近年来肠球菌和变形杆菌导致人和动物发病的报道越来越多,并且它们的耐药性问题也越来越突出,这需要引起肠衣加工企业、肠衣行业和污水处理的行政主管部门的关注。
[1] 王国栋,贺稚非,李洪军,等.不同盐分贮藏条件下天然肠衣的微生物及理化性质变化[J].食品科学,2014,35(4):215-219.
[2] Wijnker J J,Koop G,Lipman L J A.Antimicrobial properties of salt (NaCl) used for the preservation of natural casings[J].Food Microbiol,2006,23(7):657-662.
[3] 仇保丰,宋鸿雁,郭桂萍,等.肠衣携带细菌情况的分析及对策[J].中国动物检疫,2014,31(1):30-33.
[4] Byun M W,Lee J W,Jo C,et al.Quality properties of sausage made with gamma-irradiated natural pork and lamb casing[J].Meat Sci,2001,59 (3):223-228.
[5] Hammou F B,Skali S N,Idaomar M,et al.Combinations of nisin with salt (NaCl) to controlListeriamonocytogeneson sheep natural sausage casings stored at 6℃[J].Afr J Biotechnol,2010,9(8):1190-1195.
[6] 仇保丰,宋鸿雁,蔡宝亮,等.65株冷冻肠衣分离细菌的鉴定[J].中国食品卫生杂志,2013,25(5):458-461.
[7] 刘 芳,张丹彤,王道营,等.3种肠衣中的细菌菌群多样性分析[J].食品科学,2013,34(21):233-236.
[8] Gabis D A,Silliker J H.Salmonellain natural animal casings[J].Appl Microbiol,1974,27(1):66-71.
[9] Chawla S P,Chander R,Sharma A.Safe and shelf-stable natural casing using hurdle technology[J].Food Control,2006,17(2):127-131.
[10] 仇保丰,蔡宝亮,宋鸿雁,等.冷冻原肠中肠出血性大肠杆菌O157:H7的研究[J].中国人兽共患病学报,2012,28(2):191-192.
[11] Benli H,Hafley B S,Keeton J T,et al.Biomechanical and microbiological changes in natural hog casings treated with ozone[J].Meat Sci,2008,79(1):155-162.
[12] Trigo M J,Fraqueza M J.Effect of gamma radiation on microbial population of natural casings[J].Radiat Phys Chem,1998,52(1-6):125-128.
[13] 刘健华,陈杖榴,李 云,等.肠道菌群多样性变性梯度凝胶电泳分析法的建立[J].中国兽医科技,2005,35(6):445-449.
[14] 熊 强,张菲菲,姚 忠.超滤/纳滤技术深度处理肠衣废水的研究[J].食品与机械,2012,28(1):59-61.
[15] 翟 建,姜春华,鲁秀国.肠衣-肝素钠加工废水处理技术进展[J].食品工业,2013,34(8):192-194.
[16] 尹振声.电滤法处理肠衣加工废水[J].重庆环境科学,1987,3:10-13.
[17] Houben J H.A Survey of dry-salted natural casings for the presence ofSalmonellaspp.,Listeriamonocytogenesand sulphite-reducingClostridiumspores[J].Food Microbiol,2005,22(2-3):221-225.
Analysis of Bacterial Diversity in Thawing Pool Water of Frozen Intestines
QIU Bao-feng1,2,SONG Hong-yan1,3,DONG Rong-lian4,GAO Xue-mei2,JIANG Ying-mei3, GU Bing-quan2,HU Shun-lin1
(1.KeyLaboratoryofJiangsuPreventiveVeterinaryMedicine,YangzhouUniversity,Yangzhou,Jiangsu,225009,China; 2.NantongEntry-ExitInspectionandQuarantineBureau,Nantong,Jiangsu,226004,China; 3.LaboratoryAnimalCenterofNantongUniversity,Nantong,Jiangsu,226001,China; 4.JiangsuProvincialCenterforDiseaseControlandPrevention,Nanjing,Jiangsu,210009,China)
The aim of this study was to study the bacterial diversity in water of thawing pool for frozen intestines,three water samples were collected and total bacterial genomic DNA was extracted directly.After the 16S rDNA genes were amplified from the total bacterial genomic DNA by PCR,three clone libraries were constructed separately.Positive clones were identified by PCR and then 50 of them in each library were randomly selected for DNA sequencing.Based on the analyses of DNA sequences,it showed that,except 24.00 to 32.00 percent (%) of the unidentified bacteria which including uncultured and unclassified bacteria,the rest bacteria were affiliated with the classes Bacilli,γ-Proteobacteria,Actinobacteria and Flavobacteria,which accounting for 38.00%-42.00%,20.00%-26.00%,4.00%-8.00% and 0-2.00% of the clones in each library,respectively.Further analysis was conducted on the 106 stains of identified bacteria and no consideration was given to 44 unidentified bacteria,the results showed that,except 2 bacteria belonging to Actinomycetales and 5 bacteria belonging to Enterobacteriaceae which could not be further identified,the remaining 99 bacteria belonged to 19 genera and more than 30 species,and the dominant genera wereEnterococcus,ProteusandBacillus.This study revealed that the bacterial diversity in water of thawing pool for frozen intestines is high,and part of them are pathogenic and opportunistic pathogenic strains.
frozen intestine; thawing pool; bacterial diversity; 16S rDNA library
2016-09-15
江苏省动物预防医学重点实验室开放课题项目(K13044);南通市科技项目(BK2012091和MS12016014);江苏检验检疫局科研项目(2010KJ35)
仇保丰(1978-),男,安徽长丰人,兽医师,博士,主要从事动物及动物产品微生物检测及研究。*通讯作者
S852.6
A
1007-5038(2017)05-0043-06

