禽蛋表面肠杆菌的分布及其喹诺酮耐药基因的检测*
王乐晨 农宇涵 唐婉滢 付 鑫
(北京市十一学校 北京 100039)
喹诺酮类药物广泛应用于临床治疗各种细菌感染。细菌对喹诺酮类的耐药机制主要由染色体介导,不具有水平传播性。随后国外学者发现了质粒介导的喹诺酮类耐药基因[1],其作用机制是其所编码的蛋白质可对喹诺酮类药物靶位点产生保护,从而导致菌株产生耐药。由于耐药基因可在不同菌株和不同菌种之间传播,加重了细菌耐药的传播,临床分离细菌对其耐药性迅速增加。
我国是喹诺酮类药物耐药最严重的国家之一,最近报道发现,在我国动物和禽类市场中的细菌存在多种抗生素耐药基因,并且发现了一种新的对多黏菌素产生高度耐药的基因(MCR-1)[2],广泛存在于取自中国南方的猪和患者肠杆菌科细菌中,包括具有流行可能性的菌株。禽蛋为日常生活中的常用食物,但其表面携带的细菌种类和耐药状况鲜有报道。本实验在北京市几家禽类市场上,采集禽蛋表面涂抹物作为标本,调查了禽蛋表面肠杆菌科细菌的种类,并对细菌携带的喹诺酮耐药基因进行了检测。
1 材料与方法
1.1 标本来源 标本为禽蛋表面涂抹物,收集自2016年8月10—12日间,在北京市海淀区6处禽类市场的在售禽蛋进行采样。6处禽类市场来自于13个不同的养殖场,每个养殖场随机采集5份禽蛋涂抹标本。采集标本时,使用无菌棉签沾取采样管中的LB液体(胰蛋白胨1%,酵母提取物0.5%,NaCl 1%),在3~5个禽蛋表面进行涂抹,然后放入含有LB的采样管中,作为1份标本。标本于4 h内送至实验室,进行菌株分离。
1.2 菌株分离 将标本划线接种至SS培养基平板(沙门菌、志贺菌选择性培养基平板,北京陆桥生物技术公司产品),然后将SS培养基平板放至37℃培养箱中培养24 h,以该平板上菌落形态大小与颜色不同为依据,每个平板上挑选1~7个有差异的单菌落,转移至普通培养基平板,至37℃培养箱培养18 h。
1.3 DNA模板的制备 本研究中DNA模板的制备采用水煮模板法,用无菌牙签挑取普通培养基平板的菌落,至含有200 μL灭菌水的Eppendorf管中,充分混匀后,将Eppendorf管在沸水中煮10 min,即刻取出于0℃冰浴中放置 10 min,然后12 000 rpm离心2 min,上清移至新的Eppendorf中,即为DNA模版待用。
1.4 PCR 细菌种属的鉴定,采用细菌16S r RNA通用引物(见表1),喹诺酮耐药基因的检测,采用3对引物,qnrA、qnrB和qnrS,见表1,引物序列参考文献[3]。每个菌落PCR反应体系体积为50 μL,反应体系组成如下:2×Taq Master mix(TaKaRa公司产品)25.0 μL,正向引物 1.0 μL,反向引物 1.0 μL,ddH2O 21 μL,模版 DNA 2.0 μL。 PCR 条件:94℃10 min;循环 94℃ 45 s,54℃ 45 s,72℃ 45 s。 共 30个循环;最后延伸72℃ 10 min。

表1 本研究用PCR引物的信息
1.5 PCR扩增产物鉴定与测序 PCR扩增产物用1.0%琼脂糖5V/cm电压电泳45~60 min后,用紫外检测仪观察结果。PCR扩增产物送公司进行商业测序,测序结果在GenBank网站(ncbi.nlm.nih.gov/BLASTn)进行序列比对分析。
2 结果
2.1 标本的采集结果:取样于6处采集点、来自于13个不同的养殖场,每个养殖场随机采集5份禽蛋涂抹标本,共收集65份标本,包括鸡蛋(60份)和鹌鹑蛋(5份)。
2.2 菌株的分离 将标本划线至SS培养基上进行培养,在65份标本中,有35份有菌落生长。每份在SS培养基上生长的菌落依据其形态与颜色的不同,挑选1~7个有差异的单菌落,共108个菌落,转移至普通培养基培养。
2.3 菌落的鉴定 将108个单菌落进行细菌16 S rRNA通用引物的PCR扩增,获得了1.5 kb大小的片段(图 1),PCR产物经测序后,序列在Gen-Bank网站经比对后,共鉴定出11个种或属的细菌(表 2)。其中埃希氏菌属46个(42.6%),克雷伯氏菌19个(17.6%),假单胞菌 13个(12.0%),其他还有沙门菌、克吕沃尔菌、柠檬酸菌、假单胞菌、泛菌属、嗜气杆菌、奇异变形杆菌、志贺菌等种属。
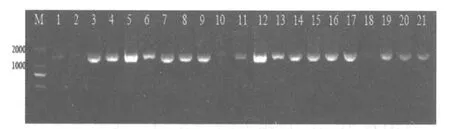
图1 部分细菌16s rRNA的PCR扩增结果
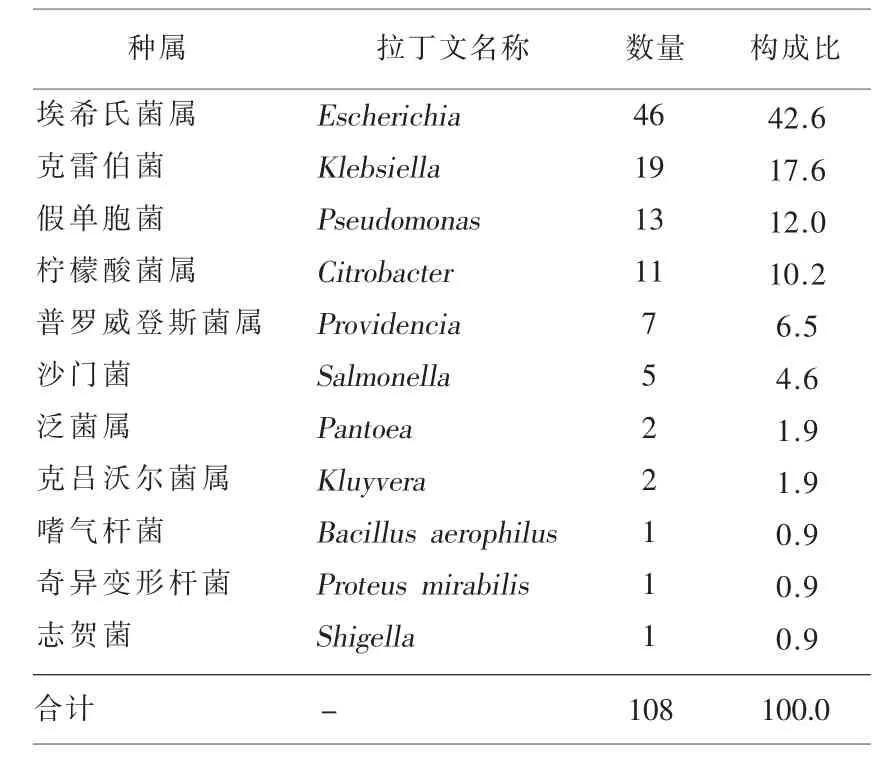
表2 禽蛋表面涂抹物标本中肠杆菌种属的构成
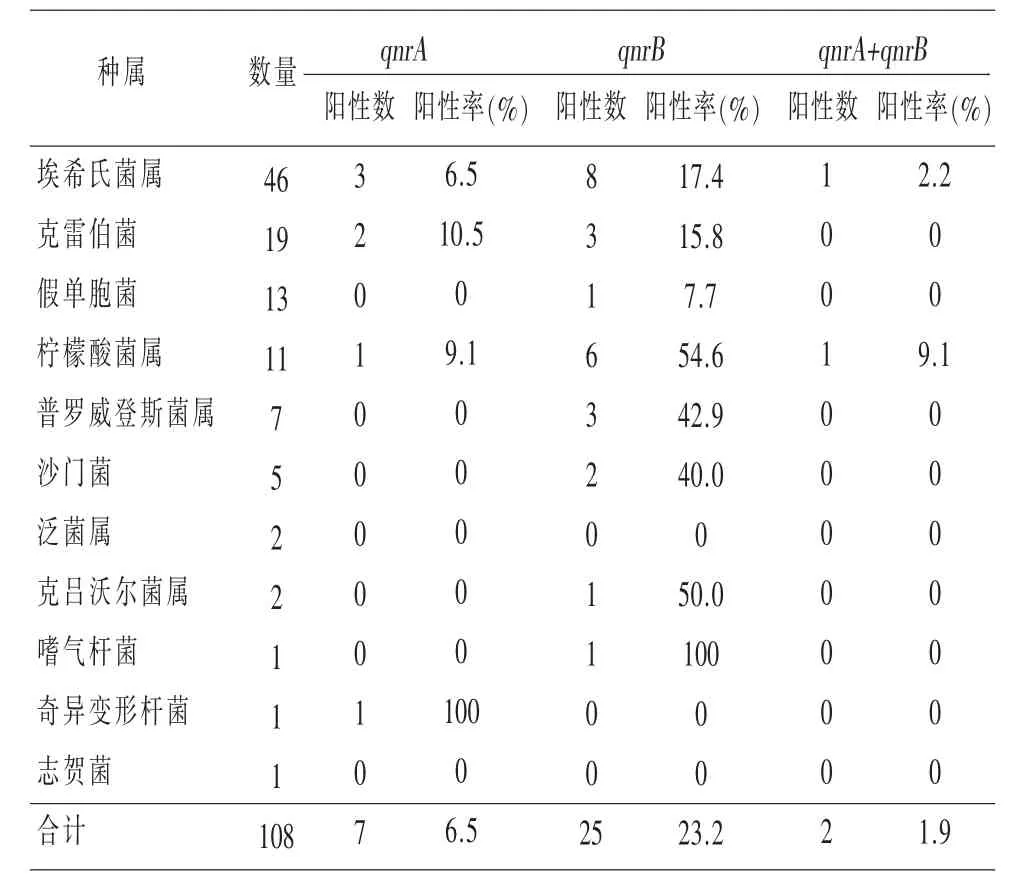
表3 喹诺酮耐药基因在肠杆菌种属中的分布
2.4 耐药基因的PCR检测 在108个菌落、所检测的3种耐药基因中,共30株菌种检测到耐药基因,阳性率为27.8%。其中qnrB阳性率为23.2%,qnrA阳性率为6.5%,但所有菌株中均未检测出qnrS基因;qnrA与qnrB同时呈阳性的有2株菌(见表3)。而根据菌株种属的不同,耐药基因呈阳性的种属,最多的为埃希氏菌属(8株),其次为柠檬酸菌属(6株),以及普罗威登斯菌(3株)和克雷伯菌(3株);其他携带耐药基因的菌属还有假单胞菌、克吕沃尔菌、沙门菌等(见表3)。
3 讨论
喹诺酮类也称为吡酮酸类或吡啶酮酸类药物,是含4-喹诺酮基本结构人工合成抗菌药品。这类抗生素以细菌DNA为靶,阻碍DNA回旋酶的作用,进而造成不可逆的DNA损伤,此其抗菌机理[12]。自1979年诺氟沙星合成后,又有新药问世——含氟的新喹诺酮类药(氟喹诺酮类)。目前在临床上使用频繁的是四代喹诺酮类药物的第三代。其中常用的有诺氟沙星、氟罗沙星、氧氟沙星和环丙沙星等。此类药物针对多种革兰氏阴性菌,常借以治疗泌尿生殖系统和胃肠疾病,也用于抵抗皮肤和呼吸道的革兰氏阴性细菌感染。
本次调查在菌株分离过程中,将标本划线至SS培养基上进行培养,在收集到的65份标本中,仅35份有菌落生长。标本没有菌落生长的原因,可能是由于使用了肠杆菌科的强选择性平板(SS培养基平板)所致,在此平板上,一些肠杆菌科细菌及其他革兰氏阳性细菌的生长可能被抑制而不能筛选。所以,在SS培养基平板上无生长的标本,不代表禽蛋表面没有肠杆菌或其他种类的细菌,如金黄色葡萄球菌等。
本研究发现,北京禽类市场禽蛋表面的肠杆菌种类有11个种或属,构成最多的前3个种属为埃希菌属(42.6%)、克雷伯菌(17.6%)和假单胞菌属(12.0%)。这种构成不同于陈力力等的报道[4],鸡蛋壳表面细菌种类(构成最多的前3个种属为假单胞菌属、埃希氏-志贺氏菌属和不动杆菌属)。究其原因是两者使用的方法不同,陈力力等采用的是菌落总数计数法,获得的是所有可生长的细菌,而本研究使用的是肠杆菌科强选择性培养基,因而获得的仅是肠杆菌科细菌,这有利于筛选出肠杆菌科细菌,进而分析其中的喹诺酮耐药基因。另外,本研究采用16S rRNA序列的网上比对鉴定细菌种属。在细菌基因组中,编码16S rRNA的rDNA基因具有良好的进化保守性,以及与进化距离相匹配的良好变异性,所以成为细菌分子鉴定的标准标识序列[5]。保守序列区域反映了生物物种间的亲缘关系,而高变序列区域则能体现物种间的差异。目前16S rDNA的序列信息已经广泛应用于菌种鉴定和系统发生学研究。
在质粒介导的喹诺酮耐药基因中,首个喹诺酮类耐药基因现命名为qnrAl[6],随后qnrB[7]、qnrS[8]等新型质粒介导的喹诺酮类耐药基因陆续被报道。本研究在检测了禽蛋表面肠杆菌科细菌中的上述3种耐药基因,共30株菌种检测到耐药基因,阳性率为27.8%,其中,不同种类的qnr基因,其阳性率(qnrA为 6.5%,qnrB为 23.2%)明显高于临床来源肠杆菌科的细菌[9],提示禽蛋表面肠杆菌科细菌耐药的严重性。但本研究的所有菌株中,均未检测出qnrS基因,而qnrS基因在食品来源的肠杆菌科细菌时有报道[10]。其原因可能是qnrS基因存在较大变异体[11],本研究使用的qnrS引物无法扩增而产生假阴性,需要进一步实验验证。同时,本研究根据菌株种属的不同,耐药基因呈阳性的种属,最多的为埃希氏菌属、柠檬酸菌属,以及普罗威登斯菌和克雷伯菌等,这些种属的细菌携带喹诺酮耐药基因,也在临床和食品中有过报道[12-14]。本研究还在禽蛋表面分离鉴定到沙门菌和志贺菌这些常见的致病菌,其中沙门菌还携带喹诺酮耐药基因(表3),因此应警惕食源性疾病的发生。同时,本调研结果也表明,应该打破过多依赖喹诺酮类药物的现状,药物多样化开发很有必要。
本研究的不足之处是采集的样本量较少,加之使用的是肠杆菌科的强选择性平板,所以筛选到的肠杆菌科细菌种类有限,并且喹诺酮耐药基因的阳性率不足以做统计学比较。
致谢:本研究由十一学校付鑫老师指导完成。本研究的实验内容在中国疾病预防控制中心传染病预防控制所完成,一并致谢!
主要参考文献
[1]Martinez-Martinez,L A Pascual,G A Jacoby.Quinolone resistance from a transferable plasmid.Lancet,1998,351(9105):797.
[2]Yi Yun Liu,Yang Wang,Timothty R Walshet al.Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China:a microbiological and molecular biological study.The Lancet Infections Diseases,2016,16(2):161.
[3]应华永,徐瑞龙,胡付品,等.肺炎克雷伯菌中质粒介导喹诺酮耐药基因qnr的检测及其耐药特征.中华检验医学杂志,2009,32(3):293.
[4]陈力力,杨伊磊,青文哲,等.鸡蛋壳表面细菌数量及种群多样性分析.现代食品科技,2015,31(10):74.
[5]Coenye T,P Vandamme.Intragenomic heterogeneity between multiple16S ribosomal RNA operons in sequenced bacterial genomes.FEMS Microbiol Lett,2003,228(1):45.
[6]李从荣,黄俊,吕霞,等.肠杆菌科细菌质粒介导喹诺酮耐药基因qnr的研究.中华检验医学杂志,2008,31(12):1348.
[7]George A Jacoby,Kelley E Walsh,Debra M Mills,et al.qnrB,another plasmid-mediated gene for quinolone resistance.Antimicrob Agents Chemother,2006,50(4):1178.
[8]Mami Hata,Masahiro Suzuki,Masakado Matsumoto,et al.Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b.Antimicrob Agents Chemother,2005,49(2):801.
[9]黄丽,高晓坤,张宏.肠杆菌科细菌质粒介导喹诺酮类耐药基因的检测.中国感染与化疗杂志,2014(4):286.
[10]刘贵深,于涛.食源性沙门氏菌耐药性及质粒介导喹诺酮耐药基因检测.生物技术通报,2014,46(8):202.
[11]闫雷,徐海.质粒介导的喹诺酮耐药基因qnr的分类、耐药机制及其在国内的流行状况.微生物学报,2016,56(2):169.
[12]茆海丰,刘洪书,赵勇,等.大肠埃希菌对喹诺酮类抗菌药物耐药机制研究.中华医院感染学杂志,2015,(15):3375.
[13]Kees Veldman,Arie Kant,Cindy Dierikx,et al.Enterobacteriaceae resistant to third-generation cephalosporins and quinolones in fresh culinary herbs imported from Southeast Asia.Intetnational Journal of Food Microbiology,2014(177):72.
[14]Bin Li,Yong Yi,Qi Wang,et al.Analysis of drug resistance determinants in Klebsiella pneumoniae isolates from a tertiarycare hospital in Beijing,China.PLoS One,2012,7(7):e42280.

