菠菜NBS-LRR类抗病基因同源序列的克隆及分析
折红兵 范桂彦 张合龙 王晓武 武 剑 钱 伟* 徐兆生*
〔1中国农业科学院蔬菜花卉研究所,北京 100081;2中蔬种业科技(北京)有限公司,北京 100081〕
菠菜NBS-LRR类抗病基因同源序列的克隆及分析
折红兵1范桂彦1张合龙2王晓武1武 剑1钱 伟2*徐兆生2*
〔1中国农业科学院蔬菜花卉研究所,北京 100081;2中蔬种业科技(北京)有限公司,北京 100081〕
依据已知NBS-LRR类抗病基因的P-loop和GLPL两个保守结构域设计简并引物,对抗菠菜霜霉病商业杂交种RZ51-147、自交系12S3和12S4的基因组DNA进行PCR扩增、克隆和序列测定,共获得23条具有完整开放阅读框且含有NBS结构的抗病基因同源序列(resistance gene analogs,RGAs),编号为SP1~SP17、SP19~SP24,其在NCBI中的登录号为KX914865~KX914887。核苷酸比较分析结果表明,这23条RGAs大多与甜菜中推测的一些抗病蛋白基因或其他相关蛋白基因具有较高的同源性,其中SP1、SP2、SP5、SP6、SP10、SP11、SP12、SP13等8条序列均与甜菜RF45类抗病蛋白有着86%以上的同源性;SP3、SP4、SP8、SP9、SP24等5条序列与甜菜RPP13类抗病蛋白的同源性在85%以上,与向日葵抗霜霉病基因PI8相应区域的氨基酸序列相似性为32.20%~33.52%,且在进化树上聚为一类,推测其可能与抗霜霉病相关。同源进化分析结果表明,除SP7外,其余序列均为non-TIR-NBS-LRR类抗病基因,并与推导的氨基酸序列多重比较结果一致。
菠菜;抗病基因同源序列;NBS-LRR;菠菜霜霉病
菠菜(Spinacia oleraceaL.)别名波斯草、赤根菜,是黎科菠菜属一年生草本植物,二倍体(2n=12)雌雄异株,栽培历史悠久,抗寒性强,适应性广,复种指数高,经济效益好,在我国南北各地广泛种植,是秋、冬、春三季重要的绿叶蔬菜之一(钱伟 等,2014)。菠菜霜霉病是一种主要为害叶部的真菌性病害,是菠菜生产上为害严重的常发性病害,不仅严重影响菠菜产量,而且直接影响菠菜的商品品质和食用品质(李金堂 等,2013)。
在自然界中,植物生长发育的过程中易受到多种病原微生物的侵害,为抵抗这些病原微生物的侵袭,植物体内会形成不同的抗病机制,其中之一便是抗病基因(R基因)介导的防御反应(刘登全 等,2016),病原菌中能够引起特定R蛋白介导响应的分子结构是无毒因子(Avr),特异的R蛋白识别无毒因子后便可触发免疫反应(Soosaar et al.,2005)。据报道,R基因对细菌、病毒、真菌、卵菌甚至线虫和昆虫病原体都有抗性,目前国内外已经克隆了约70个植物R基因,氨基酸序列比对发现R基因的蛋白结构极其保守,其中最大的一类R基因为NBS-LRR类抗病基因,它由高度保守的核苷酸结合位点(Nucletide-binding site,NBS)和富含亮氨酸重复域(Leucine rich repeat,LRR)两个主要部分组成,且含有P-loop、Kinase-2、Kinase-3和GLPL 4个高度保守的结构域(Dangl et al.,2001;丁国华,2004;Harris et al.,2013)。目前菠菜中抗病基因的报道较少,Feng等(2015)构建了1个菠菜BCA文库并鉴定出14个抗病相关基因,其中有9个抗病基因同源序列(resistance gene analogs,RGAs)编码NBS-LRR结构。与菠菜同科的甜菜中已报道了大量的NBS-LRR类抗病基因,Hunger等(2003)利用简并引物从甜菜基因组、甜菜叶和根cDNA以及EST序列中总共得到47个R相关基因,其中有21个RGAs属于NSBLRR类基因;Tian等(2004)同样根据保守域NBS-LRR设计简并引物从甜菜基因组中克隆出12个NBS-LRR类抗病基因;Dohm等(2014)通过对甜菜转录本数据的分析,共预测出715个RGAs,其中仅含有33个NBS-LRR类抗病基因,占总基因数的12.1%。在其他植物基因组中也存在大量的NBS-LRR类抗病基因,Meyers等(2003)研究发现拟南芥基因组序列中包含149个NBS-LRR类抗病基因和58个缺少LRRs的相关基因;Huang等(2009)通过对黄瓜的基因组测序,大量黄瓜R基因也被鉴定出来,共发现61个含有NBS结构的抗性基因且3/4的基因以11个簇分布在黄瓜染色体基因组上;此外,在水稻中也发现了600个NBSLRR类抗病基因,占总基因数(40 000个)的1.5%(Azhar et al.,2011)。根据NBS-LRR类抗病基因广泛存在于植物基因组中且具有高度保守结构域这一特点,设计简并引物,进行同源序列的扩增,成为克隆该类抗病基因的有效途径,目前这种方法已在辣椒(赵敬 等,2012)、小麦(谢欢 等,2016)、大豆(丁海 等,2003)、水稻(郑先武 等,2001)、甜瓜(Zhao & Li,2004)等多种植物中广泛应用。
菠菜霜霉病是菠菜生产中最主要的病害之一,目前国内外有关菠菜抗霜霉病基因克隆的报道较少。本试验依据NBS-LRR类抗病基因保守区域设计简并引物,对不同菠菜材料的基因组DNA进行PCR扩增,获得一定数量的RGAs;并对获得的RGAs进行序列分析和鉴定,旨在为进一步研究菠菜抗霜霉病基因、培育抗病品种提供理论依据。
1 材料与方法
1.1 试验材料
供试材料为菠菜商业杂交种RZ51-147〔抗菠菜霜霉病生理小种1~8,瑞克斯旺(中国)种子有限公司〕,中国农业科学院蔬菜花卉研究所菠菜课题组选育的菠菜自交系12S3和12S4。3份材料均于2016年春在中国农业科学院蔬菜花卉研究所廊坊基地种植。
PCR相关试剂购于北京天根生化科技有限公司,pEASY-T1载体试剂盒和大肠杆菌(E.coli)菌株DH5α购于北京全式金生物技术有限公司。
1.2 试验方法
1.2.1 引物的设计与合成 根据已克隆的植物抗霜霉病基因PI8、RPP1、RPP4、RGC2、RPP8等(http://prgdb.crg.eu/old.php)的NBS-LRR保守区域P-loop和GLPL的核苷酸序列特征,运用Primer 5软件和Codehop(Rose et al.,1998,2003)设计2对简并引物,引物序列见表1,并交由生工生物工程(上海)股份有限公司合成。

表1 用于扩增菠菜中RGAs的简并引物
1.2.2 DNA的提取 5月3日、幼苗5~6叶期,各材料分别取幼嫩叶片2~3片,采用CTAB法(Doyle,1987)提取基因组DNA,通过2%琼脂糖凝胶电泳检测DNA质量。
1.2.3 NBS-LRR序列的PCR扩增 以提取的菠菜基因组DNA为模板,分别采用上述2对简并引物Ploop-1/GLPL-1和Ploop-2/GLPL-2进行常规PCR扩增,反应体系为:10×HIFI bufferⅠ3.2 μL, dNTP(2.5 mmol·L-1)1.6 μL,HIFI酶 0.2 μL,引 物(10 pmol·L-1)各1 μL,DNA模板(100 ng· μL-1)3 μL,加ddH2O补足至20 μL。PCR反应程序为:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃补平10 min。PCR扩增产物采用1.5%琼脂糖凝胶电泳,在凝胶成像分析系统上进行拍照分析。
1.2.4 PCR产物的回收与克隆 PCR产物经1.5%琼脂糖凝胶检测,按照普通琼脂糖凝胶DNA回收试剂盒的使用说明书进行操作。将回收的片段连接到pEASY-T1载体上,接着转化至大肠杆菌菌株DH5α感受态细胞中,而后均匀涂布于含有Kan/ IPTG/X-gal的LB固体培养基平板上,37 ℃培养过夜。挑取白斑,利用M13引物进行PCR扩增来检测T1载体中是否成功插入目的片段,最后将含有目的片段的菌液送上海美吉生物有限公司进行测序。
1.2.5 序列分析、比对及系统发育树构建 将测序结果用NCBI上的Vecscreen程序去除pEASY-T1载体,获得插入片段的核苷酸序列。然后利用NCBI中BLAST工具与GenBank中登记的序列进行同源性比较,并利用ORF Finder搜索开放阅读框;采用MEGA软件进行氨基酸同源性分析、序列比对并和已知NBS-LRR类抗病基因相应区域的氨基酸序列一起构建系统发育树。用于氨基酸序列参比的NBS-LRR类抗病基因有:拟南芥抗霜霉病基因RPP1、RPP4、RPP5、RPP8、RPP13,莴苣抗霜霉病基因RGC2B,向日葵抗霜霉病基因PI8,葡萄抗霜霉病基因MrRPV1,亚麻抗锈病基因M、L6,小麦抗叶锈病基因Lr1、Lr10,其中RPP1、RPP4、RPP5、MrRPV1、M、L6基因属于TIR-NBS-LRR类抗病基因,而RPP8、RPP13、RGC2B、PI8、Lr1和Lr10基因属于non-TIR-NBS-LRR类抗病基因。
2 结果与分析
2.1 菠菜RGAs的扩增、克隆及序列测定
利用2对简并引物Ploop-1/GLPL-1和Ploop-2/ GLPL-2对菠菜材料RZ51-147、12S3、12S4的基因组DNA进行PCR扩增,获得6条500 bp左右的片段(图1),这些片段的大小与已知NBS-LRR类抗病基因中P-loop与GLPL区域之间的距离相一致。目的片段经回收、克隆后得到大量的白斑重组子,从每份材料中随机挑选60个重组子进行PCR检测,共获得130个阳性重组子,每份材料挑取40个阳性重组子进行测序。测序结果比对显示参试材料具有较高比例的重复序列,3份菠菜材料RZ51-147、12S3、12S4中雷同重组子的比例分别为57.5%、54.5%、42.1%。将重复序列合并整理后得到27条序列,初步确认为RGAs;进一步分析后发现27条RGAs中只有23条具有连续的开放阅读框和NBS功能结构域(表2),编号为SP1~SP17、SP19~SP24,GenBank登录号为KX914865~KX914887;其余4条序列中2条在阅读框内存在终止密码子,推测可能为无功能基因,编号为SP18和SP25,另外2条有开放的阅读框但无NBS功能结构域,编号为SP26和SP27。上述结果表明,利用简并引物对菠菜基因组DNA进行扩增是一种获得RGAs的有效途径。

图1 菠菜NBS-LRR类抗病基因PCR产物电泳结果

表2 3份菠菜材料中扩增出23条抗病基因同源序列(RGAs)
2.2 菠菜RGAs氨基酸保守结构域分析
前人研究报道,NBS-LRR类抗病基因包括4个保守区域,依次为P-loop(GMGGVGKTT)、Kinase-2(VVLDDVW)、Kinase-3(GSR/ KILVTTR) 和 GLPL(GLPLALV)(Williamson,1999;丁国华,2004)。通过ORF Finder软件分析,本试验获得的23条RGAs结构域相当保守,除SP20没有P-loop保守结构域外,剩余序列均具有NBS-LRR保守结构域(图2);少数序列保守结构域中发生氨基酸突变,如序列SP12和SP13的GLPL结构域由GLPLALV分别变为GPTFSSR和GTSTSSR。上述结果表明,本试验获得的23条序列均为NBS-LRR类RGAs。
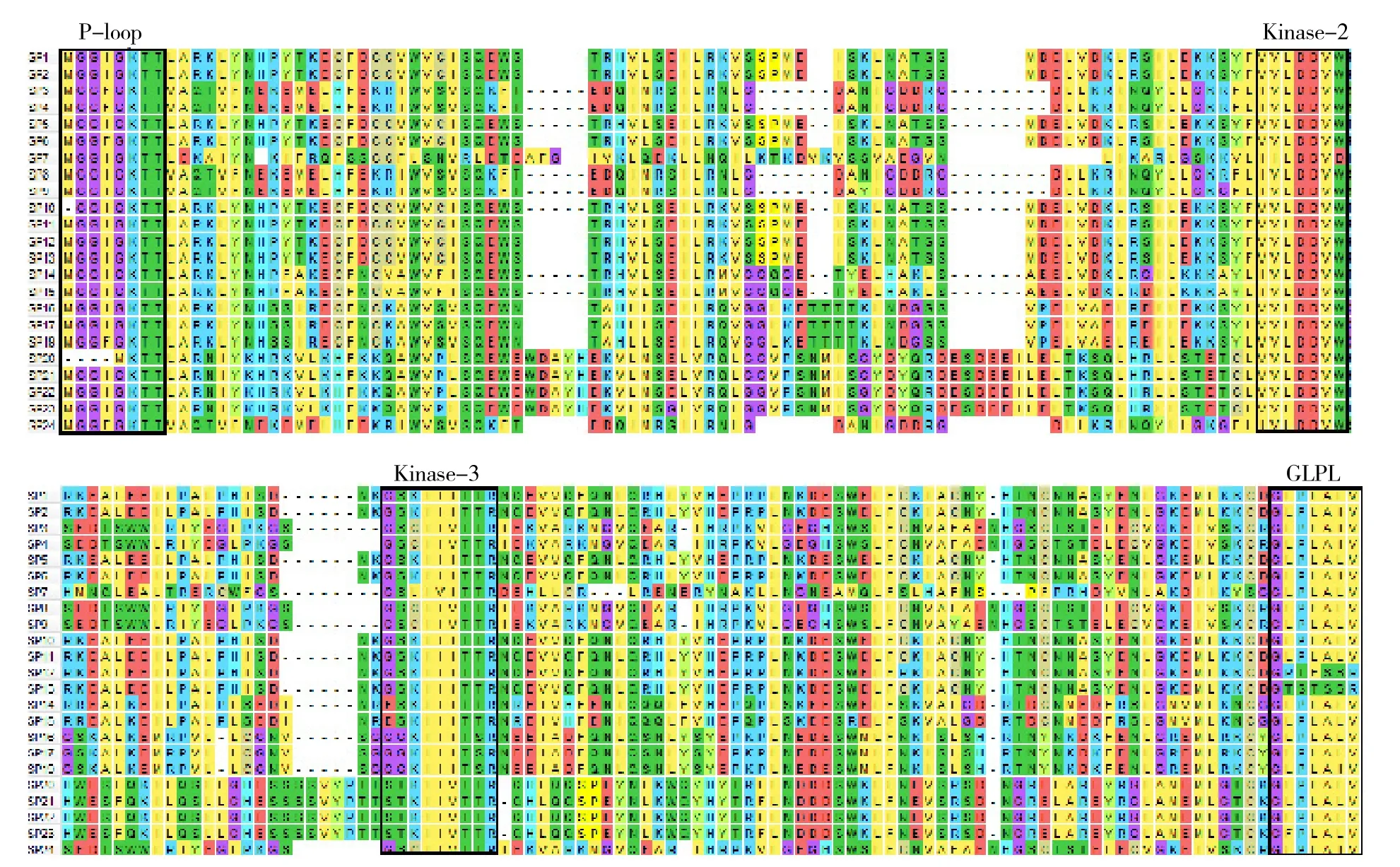
图2 菠菜RGAs氨基酸序列NBS-LRR区域多重比较结果
在 SpinachDB网 站 中(http://222.73.98.124/ spinachdb/)运用Blastn程序对这23条RGAs进行同源搜索,结果显示除SP7、SP14和SP15外,其余RGAs都可以锚定在相应的菠菜scaffold上。这些RGAs位于scaffold2266、scaffold8025、scaffold52304以及scaffold13200这4条scaffold上。其 中SP1、SP2、SP5、SP6、SP10、SP11、SP12、SP13位 于 scaffold2266上 33 000~33 530区 间内;SP3、SP4、SP8、SP9、SP24位于scaffold8025上 17 937~18 458区 间 内;SP16、SP17、SP19位 于 scaffold52304上 33 536~34 077区 间 内;SP20、SP21、SP22、SP23位 于 scaffold13200上11 378~11 957区间内。以上结果表明,菠菜NBS-LRR类抗病基因成簇出现,可能大量存在于 scaffold2266、scaffold8025、scaffold52304以 及scaffold13200这4条scaffold上。
研究表明,按N-端的结构特点可以把NBSLRR类抗病基因细分为TIR-NBS-LRR和non-TIRNBS-LRR两大类。保守结构域Kinase-2末端的氨基酸是区分这两类抗病基因的重要特征之一,若Kinase-2区域末端氨基酸为天冬氨酸(D),则是TIR-NBS-LRR类抗病基因;若Kinase-2区域末端氨基酸为色氨酸(W),则是non-TIR-NBSLRR类抗病基因(Meyers et al.,1999,2003)。从图2可以看出,除SP7外,其余RGAs氨基酸序列中Kinase-2区域域末端的氨基酸均为色氨酸(W),表明菠菜RGAs主要属于non-TIR-NBS-LRR类型。
2.3 菠菜RGAs核苷酸序列同源性分析
通过NCBI Blastn程序对获得的23条RGAs序列进行同源搜索,除SP20、SP21、SP22、SP23序列在GenBank中没有找到同源序列外,其余19条RGAs序列均匹配到与之具有高同源性的序列(表3)。这些基因序列大多来自与菠菜同科的甜菜,还有1个来自辣椒;大部分为假定的抗病蛋白,少部分为功能未知的蛋白。这19条RGAs序 列 中,SP1、SP2、SP5、SP6、SP10、SP11、SP12以及SP13之间核苷酸序列相似性高达95.81%~99.01%,且这8条序列均与甜菜RF45类(LOC104888057)抗病蛋白有着86%以上的同源性;SP3、SP4、SP8、SP9和SP24之间核苷酸序列相似性为96.39%~99.62%,这5条序列与甜菜RPP13类(LOC104902631)抗病蛋白的同源性在85%以上。
2.4 菠菜RGAs氨基酸序列聚类分析
应用MEGA软件将获得的23条RGAs氨基酸序列与亚麻M、L6、拟南芥RPP1、RPP4、RPP5、RPP8、RPP13、莴苣RGC2B、向日葵PI8、葡萄MrRPV1以及小麦Lr1、Lr10基因的相应区域氨基酸序列进行相似性比较。结果显示(图3),供试序列共聚成两大类,SP7与亚麻M、L6、葡萄MrRPV1和拟南芥RPP1、RPP4、RPP5基因聚成一大类,为TIR-NBS-LRR类抗病基因,SP7与葡萄抗霜霉病基因MrRPV1相应区域的氨基酸序列相似性为41.44%;其余22条RGAs与拟南芥RPP8、RPP13、莴苣RGC2B、小麦Lr1、Lr10以及向日葵PI8基因聚成另一大类,为non-TIR-NBS-LRR类抗病基因,这与前面推导的氨基酸序列比较结果是一致的。其中non-TIR-NBS-LRR类型又可细分为5小类,SP1、SP2、SP5、SP6、SP10、SP11、SP12、SP13聚为一类(Ⅰ类),它们的氨基酸序列相似性高达98.83%,推测它们属于同一基因簇;SP20、SP21、SP22、SP23、RPP8聚为一类(Ⅳ类),其中菠菜RGAs氨基酸序列的相似性高达99.12%,推测它们处于同一基因簇,并且与拟南芥RPP8的相似 性 为27.27%~29.21%;SP3、SP4、SP8、SP9、SP24与PI8、RGC2B和Lr1聚为一类(Ⅴ类),这
5条菠菜RGAs氨基酸序列相似性为99.08%,且与向日葵抗霜霉病基因PI8相应区域氨基酸相似性为32.20%~33.52%,故推测这5条RGAs同样属于同一基因簇;菠菜23条RGAs大部分都聚在这3类中。Ⅰ、Ⅱ、Ⅲ类没有已知的抗病基因与其聚在一起,可能是用于聚类的抗病基因较少,也可能是它们属于新的抗病基因类型。
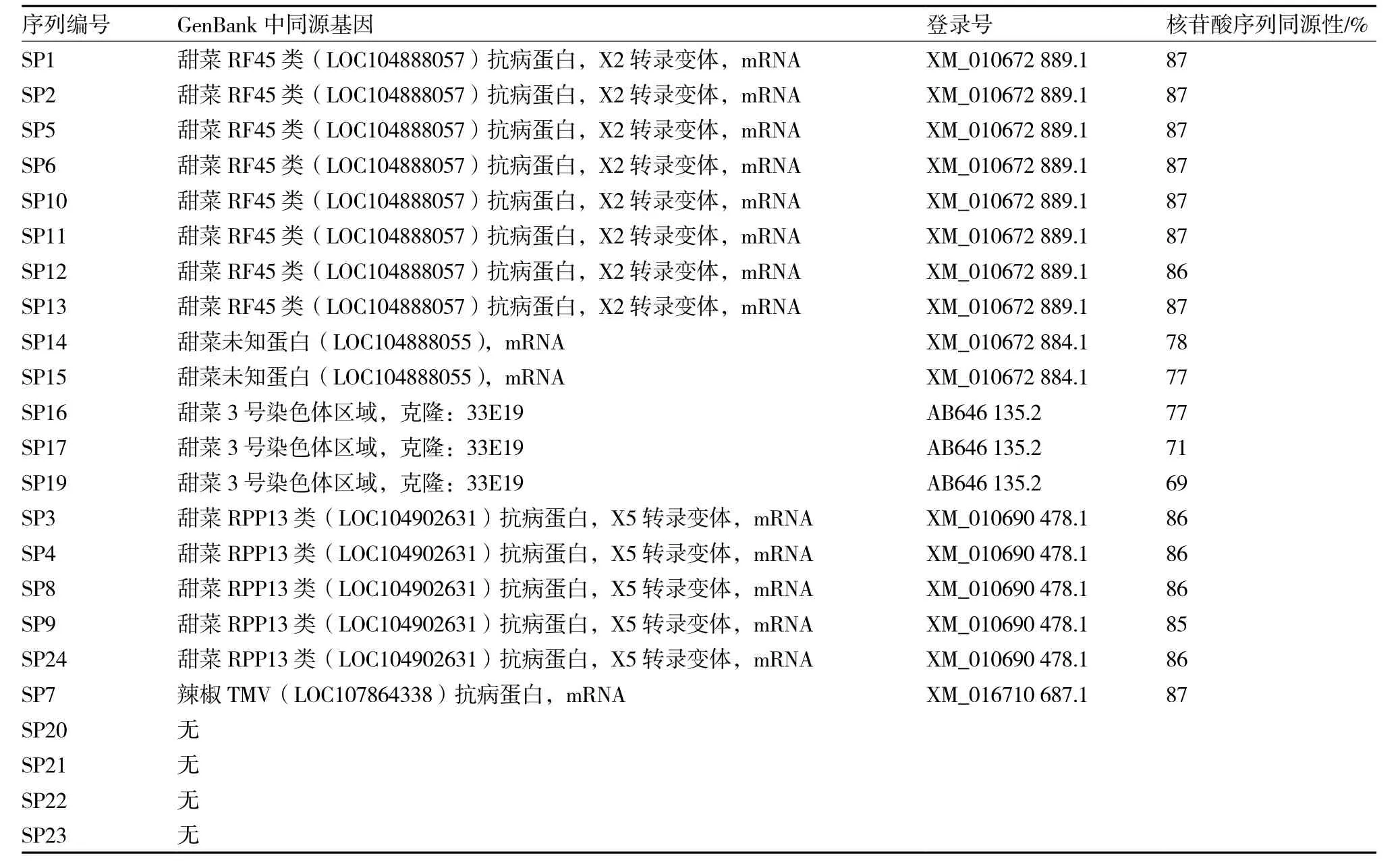
表3 GenBank中与菠菜RGAs具有高同源性的基因序列
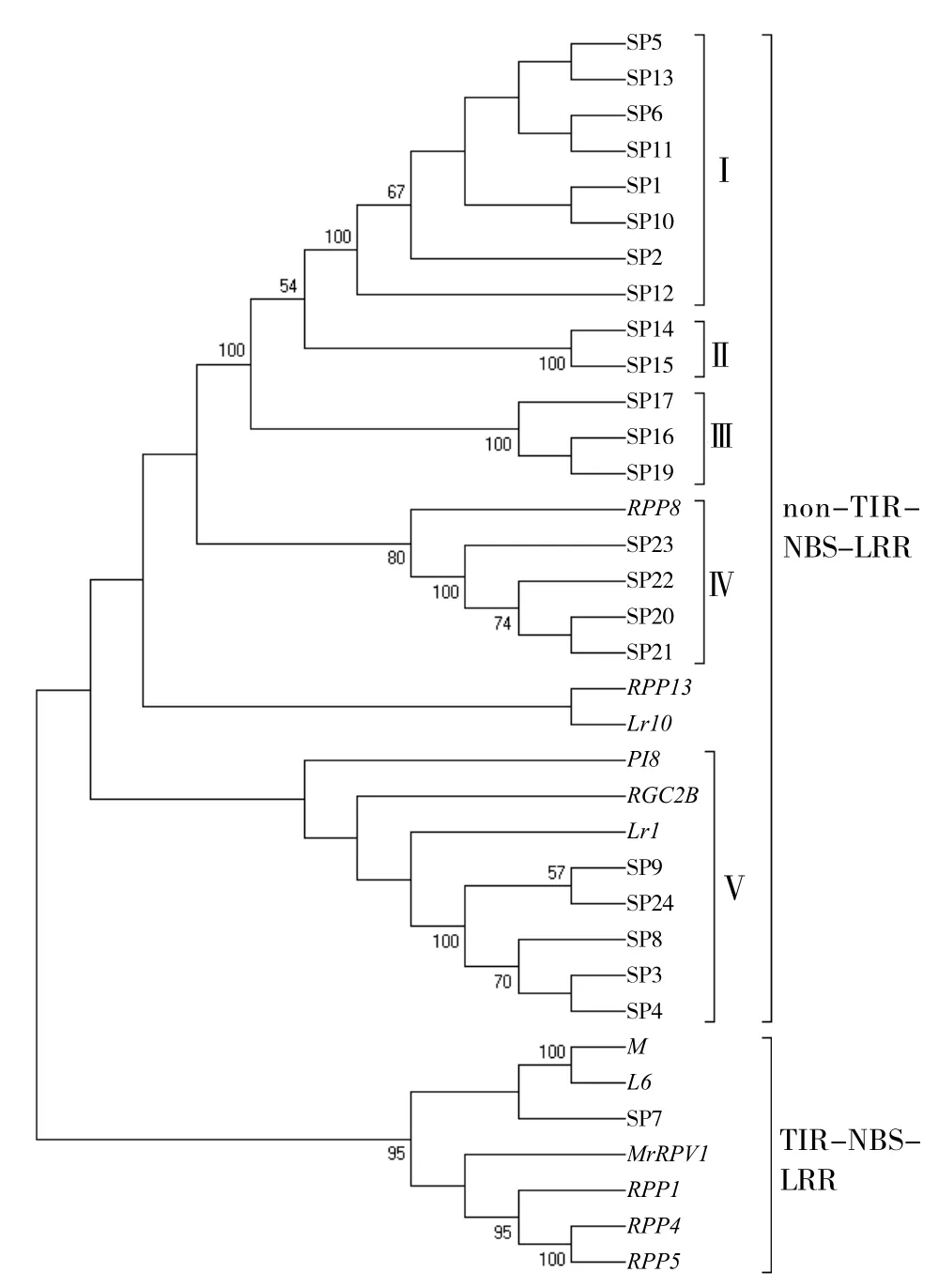
图3 菠菜RGAs氨基酸序列聚类分析结果
3 结论与讨论
目前关于菠菜抗病基因克隆的报道仅有一例(Feng et al.,2015),本试验依据NBS-LRR类抗病基因的保守区域设计2对简并引物Ploop-1/ GLPL-1和Ploop-2/GLPL-2,经过PCR扩增、克隆和测序等步骤,从菠菜中分离出23条抗病基因同源序列(RGAs),除SP20没有P-loop保守结构域外,其余RGAs序列均含有NBS-LRR类抗病基因的4个保守结构域(P-loop、Kinase-2、Kinase-3和GLPL)。聚类分析结果表明,除SP7外,其余RGAs可分为5类,每一类中的RGAs彼此之间氨基酸相似性较高,如第Ⅰ类RGAs间氨基酸相似性高达98%以上,第Ⅴ类RGAs间氨基酸相似性高达99%。产生这样的原因主要有:① 每一类菠菜RGAs在核苷酸水平上虽有差异,但是由于密码子的简并性,不同的核苷酸序列翻译成氨基酸后这种差异变的更小;② 可能是测序误差引起的,使得同一序列测序后获得不同的重组子。
应用Blastn工具在GenBank中搜索,发现这23条RGAs序列主要与甜菜中的一些抗病蛋白或其他相关蛋白有较高的同源性。进一步研究发现,SP3、SP4、SP8、SP9、SP24这5条RGAs氨基酸序列之间有着99%以上的相似性,它们与向日葵抗霜霉病基因PI8相应区域的氨基酸序列相似性为32.20%~33.52%,且在进化树上聚为一类,推测这5条RGAs序列位于菠菜中类PI8抗病基因的基因簇上;SP1、SP2、SP5、SP6、SP10、SP11、SP12和SP13氨基酸序列之间的相似性在98%以上,且这些RGAs的核苷酸序列与甜菜RF45类抗病蛋白基因有着86%以上的同源性,推测它们属于同一基因簇并在菠菜中编码RF45类抗病蛋白;此外,SP20、SP21、SP22和SP23序列在GenBank中没有找到与之相匹配的核苷酸序列,但在进化树上与拟南芥抗霜霉病基因RPP8聚在一起,推测这4条序列成簇的存在于菠菜基因组中且结构类似RPP8基因。这些研究结果为菠菜抗病基因的分离和克隆提供了条件,同时也说明利用简并引物对菠菜基因组DNA进行扩增获得RGAs是一种可行的途径。
NBS-LRR类抗病基因是植物R基因中最大的一类,这类基因根据N-端的结构特点可以细分为两类:一类是果蝇Toll蛋白和哺乳动物白细胞介素-1受体同源域(TIR-NBS-LRR或TNL蛋白),在双子叶植物中发现,如烟草的N基因、亚麻的M和L6基因等;另一类是N端卷曲螺旋域(CCNBS-LRR或CNL蛋白 ), 也 称non-TIR-NBSLRR,在单子叶、双子叶植物中均有发现,如单子叶植物小麦的Lr1、Lr10基因以及双子叶植物拟南芥的RPP13、RPP8基因等。此外,根据Kinase-2区域末端氨基酸也可以区分这两类抗病基因,当该氨基酸是色氨酸(W)时为CNL类抗病基因;是天冬氨酸(D)时为TNL类抗病基因(Meyers et al.,1999)。菠菜属于黎科菠菜属双子叶植物,通过对菠菜23条RGAs序列进行分析发现,SP7氨基酸序列的Kinase-2区域最后一个氨基酸为天冬氨酸(D),且在进化树上与TIR类的亚麻M、L6和葡萄MrRPV1基因聚在一起,判断其为TIR-NBSLRR类抗病基因;其余22条RGAs的Kinase-2区域末端氨基酸为色氨酸(W),且在进化树上与non-TIR类的抗病基因聚在一起,故判断其为non-TIR-NBS-LRR类抗病基因。两种分析结果是一致的。由于此前菠菜R基因的报道较少,且本试验所获RGAs片段多数没有与已知R基因聚为一类,且含有NBS结构的RGAs并不全部和抗病相关,因此,菠菜RGAs的类型、功能以及与抗病基因的关系等问题仍有待于进一步研究。
丁国华.2004.黄瓜抗病基因同源序列的克隆及其对霜霉病抗病基因标记的研究〔博士论文〕.哈尔滨:东北农业大学.
丁海,宛煜嵩,朱美霞,方宣钧.2003.大豆抗病基因同源序列的克隆与分析.分子植物育种,(2):217-223.
李金堂,默书霞,杨子亮,吴金鹏.2013.菠菜霜霉病的识别及防治.长江蔬菜,(9):46-47.
刘登全,王园秀,崔朝宇,秦双林,欧阳慧,蒋军喜.2016.晚熟温州蜜柑NBS-LRR类抗病基因同源序列的克隆及分析.江西农业大学学报,38(1):83-89.
钱伟,张合龙,刘伟,徐兆生.2014.菠菜遗传育种研究进展.中国蔬菜,(3):5-13.
谢欢,周颖,杨文香,张娜,刘大群.2016.TcLr24小麦NBS-LRR同源基因的克隆与分析.西北农业学报,25(4):518-522.
赵敬,万红建,杨悦俭,侯喜林.2012.辣椒NBS-LRR类型抗病基因同源序列的克隆及分析.江苏农业学报,28(3):611-616.
郑先武,翟文学,李晓兵,王文君,徐吉臣,刘国振,朱立煌.2001.水稻NBS-LRR类R基因同源序列.中国科学(C辑),31(1):43-51.
Azhar M T,Amin I,Bashir A,Mansoor S.2011.Characterization of resistance gene analogs from Gossypium arboreum and their evolutionary relationships with homologs from tetraploid cottons.Euphytica,178(3):351-362.
Dangl J L,Jones J D G.2001.Plant pathogens and integrated defence responses to infection.Nature,411(6839):826-833.
Dohm J C,Minoche A E,Holtgräwe D,Capella-Guti é rrez S,Zakrzewski F,Tafer H,Rupp O,Sörensen T R,Stracke R,Reinhardt R,Goesmann A.2014.The genome of the recently domesticated crop plant sugar beet(Beta vulgaris).Nature,505(7484):546-549.
Doyle J J.1987.A rapid DNA isolation procedure for small quantities of fresh leaf tissue.Phytochem Bull,19:11-15.
Feng C,Bluhm B H,Correll J C.2015.Construction of a spinach bacterial artificial chromosome(BAC)library as a resource for gene identification and marker development.Plant Molecular Biology Reporter,33(6):1996-2005.
Harris C J,Slootweg E J,Goverse A,Baulcombe D C.2013.Stepwise artificial evolution of a plant disease resistance gene.Proceedings of the National Academy of Sciences,110(52):21189-21194.
Huang S,Li R,Zhang Z,Li L,Gu X,Fan W,Lucas W J,Wang X,Xie B,Ni P,Ren Y.2009.The genome of the cucumber, Cucumis sativus L.Nature Genetics,41(12):1275-1281.
Hunger S,Gaspero G D,Mohring S,Bellin D,Schäfer-Pregl R,Borchardt D C,Charles-Eric D,Werber M,Weisshaar B,Salamini F,Schneider K.2003.Isolation and linkage analysis of expressed disease-resistance gene analogues of sugar beet(Beta vulgaris L.).Genome,46(1):70-82.
Meyers B C,Dickerman A W,Michelmore R W,Sivaramakrishnan S,Sobral B W,Young N D.1999.Plant disease resistance genes encode members of an ancient and diverse protein family within the nucleotide-binding superfamily.The Plant Journal,20(3):317-332.
Meyers B C,Kozik A,Griego A,Kuang H,Michelmore R W.2003.Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis.The Plant Cell,15(4):809-834.
Rose T M,Schultz E R,Henikoff J G,Pietrokovski S,McCallum C M,Henikoff S.1998.Consensus-degenerate hybrid oligonucleotide primers for amplification of distantly related sequences.Nucleic Acids Research,26(7):1628-1635.
Rose T M,Henikoff J G,Henikoff S.2003.CODEHOP(COnsensus-DEgenerate hybrid oligonucleotide primer)PCR primer design.Nucleic Acids Research,31(13):3763-3766.
Soosaar J L M,Burch-Smith T M,Dinesh-Kumar S P.2005.Mechanisms of plant resistance to viruses.Nature Reviews Microbiology,3(10):789-798.
Tian Y,Fan L,Thurau T,Jung C,Cai D.2004.The absence of TIR-type resistance gene analogues in the sugar beet(Beta vulgaris L.)genome.Journal of Molecular Evolution,58(1):40-53.
Williamson V M.1999.Plant nematode resistance genes.Current Opinion in Plant Biology,2(4):327-331.
Zhao H,Li G.2004.Cloning and expression analysis of downy mildew resistance-related cDNA sequences in melon.Agricultural Sciences in China,4(11):839-844.
Cloning and Analysis of NBS-LRR Type Disease-resistant Gene Analogs in Spinach oleracea L.
SHE Hong-bing1,FAN Gui-yan1,ZHANG He-long2,WANG Xiao-wu1,WU Jian1,QIAN Wei2*,XU Zhao-sheng2*
(1InstituteofVegetablesandFlowers,ChineseAcademyofAgriculturalSciences,Beijing100081,China;2China VegetableSeedTechnologyCo.,Ltd.,Beijing100081,China)
In order to understand better the resistant mechanism of spinach(Spinach oleraceaL.)downy mildew and lay foundation for further clone of spinach downy mildew resistance genes and marker-assistedselection of resistance breeding,2 pares of degenerate primers were designed from the conserved motifs of NBSLRR type plant resistance gene.There are 3 types of spinach material-commercial hybrid‘RZ51-147’,inbred line‘12S3’and‘12S4’.Two pares of primers were used for amplifying the disease-resistant gene analogs(RGAs)by polymerase chain reaction(PCR)from the genomic DNA of these 3 material.After cloning and sequencing the PCR products,23 resistance gene analogs with uninterrupted open reading frames(ORFs)and conserveddomains of NBS were obtained from spinach,and deposited in GenBank with the accession number of KX914865-KX914887.Homology research showed that most of these 23 RGAs shared high homology with some putative disease-resistant genes or the other relative protein genes in Beta vulqaris.For example,the nucleotides of SP1,SP2,SP5,SP6,SP10,SP11,SP12,SP13 were over 86% identical to disease resistance protein RF45 from sugar beet,and the nucleotides of SP3,SP4,SP8,SP9,SP24 were over 85% identical to sugar beet disease resistance RPP13 protein.In addition,these 5 RGAs shared 32.20%-33.52% amino acid sequence homology with sunflower downy mildew resistance genePI8,and clustered a group withPI8gene in phylogenetic tree,suggesting that these 5 RGAs might be related to downy mildew gene.Homologous evolution analysis demonstrated that all of RGAs were ranked into non-TIR-NBS-LRR typeRgene except SP7,which was consistent with the result based on multiple alignment of deduced amino acid sequences.
Spinacia oleraceaL.;Resistance gene analogs;Nucleotide binding site-leucine rich repeat(NBS-LRR);Spinach downy mildew
折红兵,男,硕士研究生,专业方向:蔬菜遗传育种,E-mail:13051127571@163.com
*通讯作者(Corresponding authors):徐兆生,研究员,硕士生导师,专业方向:蔬菜遗传育种,E-mail:xuzhaosheng@caas.cn;钱伟,副研究员,专业方向:蔬菜遗传育种,E-mail:qianwei@caas.cn
2017-01-07;接受日期:2017-04-10
国家“十二五”科技支撑计划项目(2012BAD02B04,2014BAD01B08),国家自然科学基金项目(31401872),中国农业科学院科技创新工程项目(CAAS-ASTIP-IVFCAAS),农业部园艺作物生物学与种质创制重点实验室项目

