染色体特异位点筛选方法在无创产前检测中的应用*
孔令印,王挺,贺权责,冒燕,申静静,宣黎明,祝轶君,薛永峰,孙丹凤,刘慧敏,梁波
(1.苏州贝康医疗器械有限公司,江苏苏州 215001;2.苏州市立医院遗传中心,江苏苏州 215002;3.上海交通大学生命科学与生物技术学院,上海 200240)
·临床检验技术研究·
染色体特异位点筛选方法在无创产前检测中的应用*
孔令印1,王挺2,贺权责2,冒燕1,申静静1,宣黎明1,祝轶君1,薛永峰1,孙丹凤1,刘慧敏1,梁波3
(1.苏州贝康医疗器械有限公司,江苏苏州 215001;2.苏州市立医院遗传中心,江苏苏州 215002;3.上海交通大学生命科学与生物技术学院,上海 200240)
目的 建立一种基于染色体特异位点测序进行无创产前检测的方法,以替代目前传统的基于全基因组测序的无创产前检测技术。方法 采集200例已知胎儿核型的孕妇血浆样本,通过数据库筛选挑选出13、18、21号染色体上特异性的位点,通过探针捕获的方式进行特异性片段的捕获,针对特异性片段进行测序分析,通过Z值计算来判断是否存在染色体非整倍体异常。结果 染色体特异位点筛选法检出7例21三体胎儿、3例18三体胎儿,1例13三体胎儿,结果与全基因组高通量测序法一致。结论 基于染色体特异位点测序法具有检测成本低、检测通量高的优势,有望替代全基因组高通量测序法进行无创产前筛查。
染色体特异位点测序;无创产前筛查;高通量测序
1997年,Lo等[1]在孕妇的外周血浆中发现了胎儿的游离DNA,使得从理论上可以通过孕妇血浆游离DNA的检测判断胎儿染色体是否异常。然而孕妇血浆中的胎儿游离DNA片段短,含量低,高背景的母体游离DNA给全面检测胎儿染色体带来了诸多困难。近年来研究发现,可用高通量测序来分析母体外周血中的胎儿游离DNA,并通过随后大量的临床试验验证其准确率可达99%[2-4]。然而,该技术需要对整个基因组DNA片段进行检测,测序量较大,导致检测价格较高。2012年,Ariosa公司发现,通过捕获21、18号染色体上的特异性片段进行测序分析,可显著降低测序所需的数据量[5]。因此,本研究设计捕获了13、18、21号染色体上的特异性片段,通过Z检验分析是否存在13、18、21号染色体非整倍体,旨在提高1张测序芯片的样本检测通量,降低检测成本,以期替代目前无创产前检测常规的高通量测序方法。
1 材料与方法
1.1 研究对象 收集2014年5月至2015年3月在苏州市立医院产前诊断中心就诊的孕妇外周血样本200例。孕龄为22~40周岁,平均孕龄34.5周岁,取样时孕周14~28周,平均孕周20.1周。纳入标准:高龄孕妇(≥35岁)、唐氏生化筛查高风险(唐氏风险≥1/270, 18三体风险≥1/350)及中等风险(唐氏风险1/270~1/1 000, 18三体风险1/350~1/1 000)孕妇。本研究经孕妇及家属知情同意,获该医院医学伦理学委员会批准。
1.2 主要仪器及试剂 2720 ABI PCR仪、Sorvall ST 16R冷冻离心机、Qubit 2.0荧光定量仪、Ion Proton高通量测序仪(美国Thermo Fisher公司),DH300恒温金属浴(杭州瑞诚仪器公司); AMPure XP 磁珠纯化试剂(美国Beckman公司),血浆核酸提取试剂盒(Circulating Nucleic Acid Kit,德国Qiagen公司),Pfusion 聚合酶(Fermentas公司),TdT末端原位转录酶、dNTPs(美国Enzymatics公司),Myone C1磁珠(Dynal,美国Thermo Fisher公司),生物素-16-dUTP(Roche公司)。
1.3 方法
1.3.1 常规无创产前检测 采集各孕妇外周血样本10 mL,4 ℃、1 600×g离心 10 min,取上清液,16 000×g离心10 min,去沉淀,收集血浆600 μL,送苏州市立医院进行常规无创产前高通量测序。高通量测序为高风险的孕妇送苏州市立医院遗传科进行羊水或脐血采集及染色体核型分析。
1.3.2 血浆DNA提取 取2 mL血浆样本,按照血浆核酸提取试剂盒说明书操作提取DNA,样本用30 μL无核酸酶的ddH2O洗脱,并用Qubit 2.0荧光定量仪进行浓度检测,取浓度>0.5 ng/μL DNA样本用于后续文库构建,样本置-20 ℃保存。
1.3.3 染色体位点筛选 根据美国国立生物技术信息中心(NCBI)的人类基因组序列(GRCH Build 37),对13、18、21号染色体特异位点进行初步筛选,每个染色体上筛选500~1 000个初筛位点序列,需满足以下条件:(1)序列长度范围50~70 bp;(2)GC含量在45%~55%之间;(3)序列中不包含字符“N”;(4)与NCBI单核苷酸多态性位点库(dbSNP)进行比较,序列中不存在任何单核苷酸多态性位点(SNP);(5)与NCBI基因组变异数据库(DGV)中拷贝数变异(CNV)信息进行比较,位点不能包含在任何已知的CNV中。染色体特异位点筛选流程见图1。
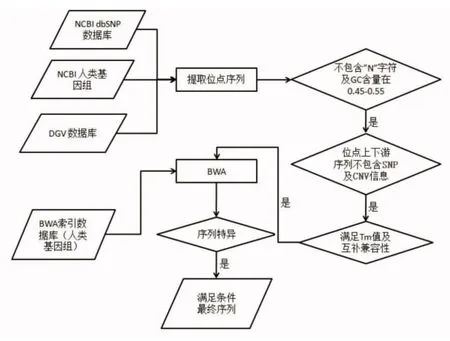
图1 染色体特异位点筛选流程图
1.3.4 探针合成 按照参考文献[5-6],分别从21、18、13号染色体上,随机选择500个特异位点序列,并在位点序列5′端、3′端以及序列中部各设计1段特异性位点结合寡核苷酸引物。所有的探针及引物均由上海生工公司设计并合成。
1.3.5 特异性位点片段分离 取20 μL血浆游离DNA,加入3 μL 150 pmoL的生物素-16-dUTP,0.5 μL TdT末端原位转录酶和5 μL 1×TdT末端原位转录酶缓冲液,ddH2O补足至30 μL。37 ℃温育1 h。用异丙醇沉降法除去多余的生物素,30 μL TE(pH 8.0,含10 mmol/L Tris-HCl, 0.1 mmol/L EDTA)缓冲液重悬DNA。反应体系为50 μL,含50 μg/μL Myone C1磁珠(Dynal) 2 μL, Tris-HCl(60 mmol/L,pH 8.0)1 μL, EDTA(6 mmol/L)1 μL, NaCl2(100 mmol/L)3 μL, 35%甲酰胺2 μL,土温-80 5 μL,3段探针各4 nmol/L(探针浓度为2 nmol/L, 各加入2 μL),ddH2O补足体积。25 ℃温育2 h,将连接有生物素的DNA固定至生物磁珠。反应后用磁力架吸附磁珠, 50 μL清洗缓冲液(10 mmol/L Tris-HCl pH 8.0, 1 mmol/L EDTA, 50 mmol/L NaCl2) 清洗磁珠,吸弃缓冲液后加入1 μL Taq 连接酶,37 ℃温育1 h。用清洗缓冲液洗涤2次, 30 μL TE重悬,95 ℃反应3 min,将连接好的探针从磁珠上洗脱下来,探针置-20 ℃保存。
1.3.6 模板链扩增 通用引物序列根据Ion Proton测序平台文库的接头序列设计,由上海生工公司合成。上游通用PCR引物序列:5′-CCATCTCATCCCTGCGTGTCTCCGACTCAGNNNNNNNNNNTACACCGGCGTTATGCGTCGAGAC-3′,下游通用PCR序列:5′-CCTCTCTATGGGCAGTCGGTGATTCAAGCAGAAGACGGCATACGAGAT-3′,其中10个N代表10个碱基的index序列。将1.3.5中的特异性位点片段进行PCR扩增,PCR反应体系为50 μL;包括 Pfu高保真聚合酶(1 U/μL)1 μL, Pfu聚合酶缓冲液5 μL, dNTPs(200 μmol/L)1 μL,ddH2O补足体积。用AMPure XP磁珠纯化试剂盒纯化样品,并经Qubit 2.0核酸蛋白定量仪进行浓度测定,取>0.5 ng/μL的样本进行上机测序,样本置-20 ℃保存。
1.3.7 高通量测序和数据分析 取上述PCR纯化产物,送贝康医疗公司进行高通量测序(Ion Proton测序平台),并将获得的测序结果与选择的片段数进行匹配分析,分别统计13、18、21号染色体上每个位点片段数量。根据测序得到的13、18、21号染色体的片段数量进行Z值计算,公式:
pj:21号染色体(或13、18号染色体)上位点的标准化后的平均片段数量除以13、18、21号染色体上位点总和的标准化后的平均片段数量。平均片段数量按照20%切尾均值方法计算,即去除掉10%的最低值以及10%的最高值。p0:1个测序泳道中上所有样品pj的中值。nj:13、18、21号染色体上位点平均片段数量的总和;Zj:样本j的21号(或13号、18号)染色体值。根据以上公式,计算所有样本13、18、21号染色体对应的Z值,当Z≥6时,判断为三体,当Z<6时,判断为单体。
2 结果
2.1 常规无创产前检测结果 200例血清学筛查高危孕妇中,经常规高通量测序检测,发现其中有7例孕有21三体患儿,3例孕有18三体患儿,1例孕有13三体患儿,且均通过核型分析得到确诊。
2.2 特异性位点捕获结果 统计21、18、13号染色体上捕获的片段数量,对200份血浆游离DNA的不同染色体片段捕获效率进行了统计,结果显示,13号染色体上的平均捕获效率为85.5%,18号染色体上的平均捕获效率为92.2%,21号染色体上的平均捕获效率为90.2%,均满足分析需求。
2.3 捕获片段测序结果 染色体特异位点测序法检测出21号染色体三体(T21)7例,18号染色体三体(T18)3例,13号染色体三体(T13)1例。染色体特异位点无创产前检测方法对21三体,18三体,13三体的检测结果和常规高通量测序方法检测结果一致。200例样本对应的Z值分布见图2。
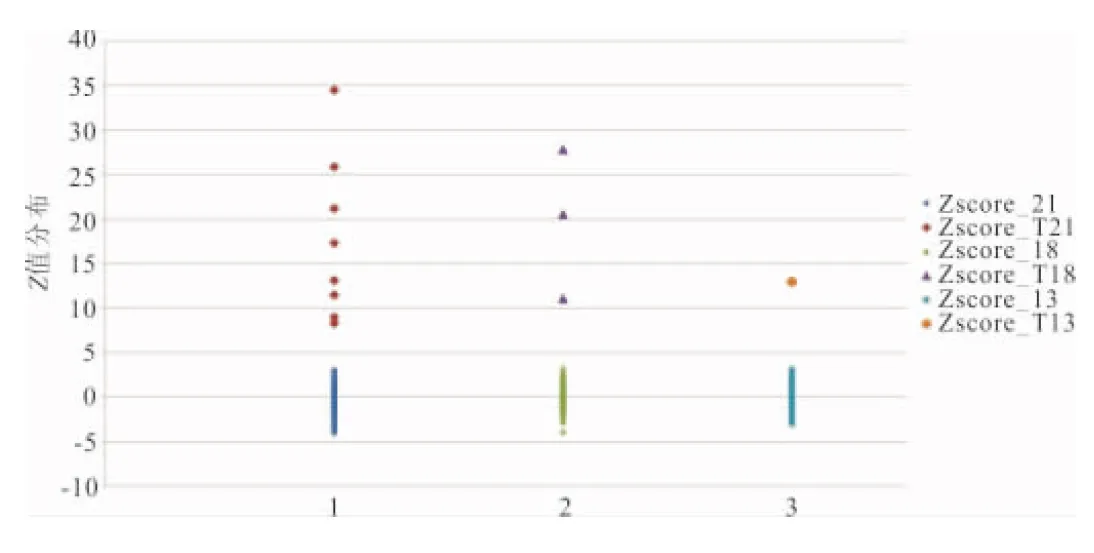
注:1~3分别为13、18、21号染色体。
图2 13、18、21号染色体的Z值分布
3 讨论
本研究建立了利用染色体特异位点进行无创产前筛查的检测体系。利用该体系对200例样本验证,发现其中189例阴性,7例T21,3例T18,1例T13,与传统高通量测序的无创产前检测一致性达100%。相比高通量测序法,本研究建立的染色体特异位点进行无创产前筛查的优点主要表现为克服了高通量测序技术针对整个基因组的所有染色体进行检测时需要测量大量无关数据的缺陷。全基因组高通量测序获得的待检染色体特异片段仅占20%,而染色体特异位点测序获得的待检染色体特异片段超过90%,需要的测序通量更低。此外,传统的高通量测序无创产前检测在Ion Proton平台上最多可以同时检测16个样本,而本研究建立的方法可以同时检测70个样本,显著降低无创产前检测成本。
基于染色体特异位点的高通量测序法相比于全基因组测序法,在操作流程上增加了捕获的步骤但省去了传统的文库构建步骤,在操作步骤和时间花费上并无太大差异,但值得一提的是全基因组测序是对全部的游离DNA进行检测,需要的血浆样本量较少,一般临床上采用600 μL的血浆提取DNA即可满足检测需求,而基于染色体特异位点的高通量测序法需要捕获目标染色体上的DNA片段进行检测,需要的样本量较大,本研究中采用了2 mL的血浆样本提取DNA,但仍是临床上合理的取样范围。
本研究虽然对部分三体阳性样本进行了准确检测,但值得注意的是,本次研究中所用的样本孕周均超过14周,胎儿游离DNA的含量较高,缺乏对于更早孕周样本的检测数据,且使用的均是既往样本,缺乏双盲实验的数据,后续将加大样本量开展临床试验,进一步确认该技术的准确性。此外,本研究仅对21,18,13三体进行了非整倍体的检测,后续研究可将该技术扩展到对其他染色体上的检测以及对于染色体微缺失微重复的检测,扩大检测范围。
[1]Lo YM, Corbetta N, Chamberlain PF,etal. Presence of fetal DNA in maternal plasma and serum[J]. Lancet, 1997, 350(9076):485-487.
[2]林颖,胡平,季修庆,等.22q 11.2微重复伴先天性心脏缺损的产前分子遗传学诊断[J].临床检验杂志,2011,29(2):103-105.
[3]Wong AI, Lo YM. Noninvasive fetal genomic, methylomic, and transcriptomic analyses using maternal plasma and clinical implications[J]. Trends Mol Med, 2015, 21(2):98-108.
[4]Zhang H, Gao Y, Jiang F,etal. Non-invasive prenatal testing for trisomies 21, 18 and 13: clinical experience from 146,958 pregnancies[J]. Ultrasound Obstet Gynecol, 2015,45(5):530-538.
[5]Sparks AB, Wang ET, Struble CA,etal. Selective analysis of cell-free DNA in maternal blood for evaluation of fetal trisomy[J]. Prenat Diagn, 2012, 32(1):3-9.
[6]Sparks AB, Struble CA, Wang ET,etal. Noninvasive prenatal detection and selective analysis of cell-free DNA obtained from maternal blood: evaluation for trisomy 21 and trisomy 18[J]. Am J Obstet Gynecol, 2012, 206(4):319 e1-e19.
(本文编辑:许晓蒙
The application of chromosome specific site selection method in noninvasive prenatal testing
KONGLing-yin1,WANGTing2,HEQaun-ze2,MAOYan1,SHENJing-jing1,XUANLi-ming1,ZHUYi-jun1,XUEYong-feng1,SUNDan-feng1,LIUHui-min1,LIANGBo3
(1.SuzhouBasecareMedicalDeviceCo.,Ltd.Suzhou215001,Jiangsu; 2.CenterforReproductionandGenetics,SuzhouHospitalaffiliatedtoNanjingMedicalUniversity,Suzhou215002,Jiangsu;3.SchoolofLifeSciencesandBiotechnology,ShanghaiJiaoTongUniversity,Shanghai200240,China)
Objective To establish a noninvasive prenatal testing(NIPT) method based on the chromosome specific site sequencing instead of the conventional whole genome sequencing. Methods Blood plasma samples from 200 pregnant women with known fetus karyotypes were collected. First, the specific loci on chromosome 13, 18 and 21 were selected by the database filter. Then, these specific loci were captured by probes, and were performed sequencing. Finally, chromosome aneuploidy was identified by the Z-score. Results Seven fetuses with 21-trisomy syndrome, 3 with 18-trisomy syndrome and 1 with 13-trisomy syndrome were detected by the chromosome specific site sequencing, and the results were accordant with those of the whole genome sequencing method. Conclusion The NIPT method based on the chromosome specific site sequencing has the advantages of low cost and high throughput, which may replace the NIPT method based on the whole genome sequencing.
chromosome specific site sequencing; noninvasive prenatal testing; high-throughput sequencing
10.13602/j.cnki.jcls.2017.03.02
江苏省科技支撑计划社会发展项目(BE2013654)。
孔令印,1980年生,男,硕士,从事生物化学与分子生物学研究。
梁波,博士,E-mail:liang01@basecare.cn。
R715.5
A
2016-11-25)

