辣椒类Pto蛋白激酶(PLPK)基因家族全基因组分析和进化研究
Jelli Venkatesh Molly Jahn Byoung-Cheorl Kang *
(1. 植物科学与植物基因组育种研究所, 蔬菜育种研究中心, 首尔大学 首尔, 151–921韩国; 2. 威斯康星大学, 麦迪逊市 威斯康星州,WI53706美国)
1 前言
植物应对病原物产生了多种防御反应。抗病性通常是由植物抗病基因(R)编码的蛋白和病原物无毒基因(Avr)编码的相应效应蛋白的基因对基因方式互作产生的。这种互作激活了寄主中抑制病原物入侵的防御相关级联反应[1-2]。一些植物的R基因已经被克隆,对寄主抗性机制的研究也取得了很大进展[3-8]。
植物的R基因根据保守结构域分为8类[7,9]。醋栗番茄(Solanum pimpinellifoliumL.)的Pto基因是R基因的最好例子[10]。Pto编码的丝氨酸/苏氨酸激酶(STK)与抗丁香假单胞菌(P. syringae)的效应蛋白AvrPto和AvrPtoB直接互作,从而引发寄主植物的抗性[12,14-15]。AvrPto和AvrPtoB在其原本不存在的细菌菌株中表达时显示毒性会增加[16-18]。AvrPtoB的致病机理是通过抑制细胞程序性坏死来扰乱寄主的防御反应[18]。有趣的是,AvrPto也能够抑制本氏烟(Nicotiana benthamiana)和番茄中非寄主病原体导致的细胞程序性坏死[19]。在抗Pst系统中已经确定了三类下游效应器。Pti1是增强体内超敏反应的蛋白激酶;Pti4、Pti5和Pti6是防御相关的类乙烯应答转录因子(EREBP)[16,20-21],Prf是一个结合核苷酸的富亮氨酸重复(NB-LRR)的蛋白质。通过Prf的识别,AvrPto效应子使Pto激酶磷酸化,从而产生抗性[22-25]。
尽管对植物特别是番茄中的Pto通路基因有了广泛的研究,但除了茄属植物,其他植物都没有鉴定到识别AvrPto的Pto功能同系物。在一些植物物种中寻找类Pto序列一直都在尝试[26-27]。在这些研究中,有人认为识别AvrPto的分子可能类似于番茄中的Pto激酶。然而,突变分析显示AvrPto蛋白在番茄和烟草中被差异性识别[17],这表明烟草中的AvrPto识别蛋白可能与番茄Pto序列无关[17]。已有报道在大豆中鉴定出了AvrPto[28],这表明AvrPto的识别和下游信号级联可能在物种间保守[28]。植物中对AvrPto的识别也引起了关于AvrPto识别蛋白在Pst非寄主物种中的作用问题。
最近,已经在一些茄属植物[27]以及菜豆[26]、葡萄[29]、黄瓜[30]、香蕉[31]、草莓[32]和柑橘[33]等植物中鉴定出类Pto基因。但是,两种不同的番茄(S.lycopersicum)基因型中不存在Pto同源物[34-35]。而由特异性AvrPto和Pto介导的抗性可能是从醋栗番茄(Solanum pimpinellifoliumL.)中进入到一些栽培番茄品种中[10,36]。尽管对Pto基因在分子遗传学方面研究有所进展,但对Pto基因的分子进化研究仍然知之甚少。了解Pto基因的进化关系对于解开其功能差异以及了解其在非寄主抗病性中的作用非常重要。
辣椒是一种重要的香料作物,一直被认为是Pst的非寄主。有证据表明辣椒可能具有识别AvrPto蛋白并诱导抗性的能力[19]。然而,Pto激酶并未被确定为识别组分。本研究中,我们使用PVX系统检测辣椒能否识别AvrPto。此外,我们对辣椒中类Pto基因家族(PLPKs)进行了全基因组分析,共确定了25个PLPK基因,同时基于序列相似性和系统发育关系将其分为8个亚族(PLPKⅠ-PLPKⅧ)。为了解植物类Pto基因的进化多样性,使用其他茄科植物如番茄、马铃薯、本氏烟(Nicotiana benthamiana)、拟南芥和水稻的Pto家族基因进行系统发育比较和分子进化分析。根据计算机工具研究辣椒PLPK的结构特征。最后,我们使用RNA-seq数据对各种辣椒基因型的PLPK基因表达谱进行分析。
2 材料与方法
2.1 植物材料
本研究使用Robert Stall提供的C.annuum‘NuMex Rnaky’(RNaky),Early CalWonder 30(ECW),‘Criollo de Morelos 334’(CM334),‘Perennial’,C. chinense‘PI159234’(234)和‘Habanero’等不同基因型的辣椒品种。试验品种包括生产中重要的易感病品种(ECW和RNaky),以及抗病品种(Perennial,CM334,Habanero和PI159234)。使用本氏烟(Nicotiana benthamiana)进行PVX载体扩增。
2.2 使用PVX衍生载体在辣椒中瞬时表达AvrPto
将G.Martin和X.Tang提供的AvrPto和AvrPtoI96T突变体克隆到由D.Baulcombe提供的pPVX201载体中,来构建PVX衍生载体的瞬时表达系统[37]。选 择 5'-ATATCGATGGGAAATATATGTGTC-3'和5'-GAGGTCGACATTATGACGCC-3'作 为 引 物,利用PCR从质粒pPtE6[28]和pPtE6:I96T[17]中扩增每个AvrPto基因的528 bp编码区。引物中的ClaI和Sall位点用下划线标注。扩增片段克隆到pGEMT载体中,并通过测序检测。用限制酶ClaI和Sall消化基因并亚克隆到相应载体位点。所得到的pPVX201衍生物称为pPVX201::AvrPto和pPVX201::AvrPtoI96T。携带GFP cDNA的pPVX201衍生物pPVX204当作对照[37]。利用15%膨润土悬浮液和43 mM磷酸钠缓冲液(pH 7.0),将≥50 μg的质粒摩擦接种到4~6周大小的本氏烟草(Nicotiana benthamiana)中[37],产生接种物。使用pPVX201、pPVX204或pPVX载体,系统侵染本氏烟草(Nicotiana benthamiana),在接种后14 d其出现了系统花叶病症状,而pPVX::AvrPto导致植株白化和出现死斑,然后出现系统坏死症状[38]。没有发生坏死症状的本氏烟(Nicotiana benthamiana)的烟叶在接种后10~14 d用50 mM磷酸钠缓冲液(pH 7.0)进行匀浆,然后摩擦接种到6~8周大小的辣椒植株中。模拟接种的辣椒作为常规对照。通过杂交分析和抗PVX抗体的酶联免疫吸附测定(ELISA)试剂盒(Agdia, Elkhart, IN)确定感染[39]。接种14 d后从植物的未接种上部叶提取总RNA,将其印迹到尼龙膜N+上,并与放射性标记的PVX衍生序列和AvrPto序列杂交。
2.3 Pto通路同源物基因定位和全基因组分析
利用已发表的辣椒遗传连锁图谱[40]来确定Pto通路基因的位置。Pto、Fen和Prf克隆的cDNA由S.D.Tansksley(康奈尔大学,伊萨卡,纽约)提供。Pti1、Pti4、Pti5和Pti6的克隆基因由G.Martin(康奈尔大学,伊萨卡,纽约)提供。为研究PI159234和NuMex Rnaky品种的限制性片断长度多态性(RFLPs),用EcoRⅠ、EcoRⅤ、DraⅠ、BclⅠ、BstNⅠ、HindⅢ和XbaⅠ限制酶消化亲代DNA,并与Pto通路基因杂交。根据Livingstone等[40]描述的75个植物F2代的定位来收集RFLP标记物的分离。通过MAPMAKER软件对照原始框架标记数据集,来确定PCR克隆片段的图谱位置。
以番茄(S.pimpinellipolium)中具有Pto基因同源序列的LpimPth2(AAF76305),LpimPth3(AAF76304),LpimPth4(AAF76303),Fen_kinase(AAF76307) 和Pto_kinase(AAF76306) 作 为引用数据,使用BLAST程序在CM334 20140109(v1.5)PROTEINS数 据 库(http://cab.pepper.snu.ac.kr/)中鉴定出辣椒(C.annuum)基因组的Pto基因家族成员。从茄科基因组学资源数据库(http://solanaceae.plantbiology.msu.edu/)中获得预测的马铃薯Pto蛋白同源序列。在拟南芥信息资源数据库(TAIR)和水稻基因组注释计划(http://rice.plantbiology.msu.edu/index.shtml)中以番茄Pto同源序列作为引用数据,利用BLAST程序获得拟南芥和水稻的类Pto蛋白激酶序列。利用番茄Pto序列作为引用数据在茄科基因组学数据库(https://solgenomics.net/)中进行BLASTP比对得到番茄和烟草的类Pto蛋白序列。我们也使用番茄Prf1(AAF76308)、Pti1(U28007)、Pti4(U89255)、Pti5(U89256)和Pti6(U89257)序列作为引用数据,通过BLAST程序鉴定出番茄Prf同系物和辣椒Pti同系物。
2.4 辣椒类Pto基因和其他辣椒Pto通路相关基因的染色体定位
通过辣椒基因组数据库(http://cab.pepper.snu.ac.kr/)获得辣椒PLPK基因和其他Pto通路基因的染色体定位。使用MapChart程序将PLPK定位到辣椒染色体上[41]。从辣椒基因组数据库(http://cab.pepper.snu.ac.kr/)获得辣椒基因组的DNA和CDS序列。在GSDS网站(http://gsds.cbi.pku.edu.cn/) 中通过比较基因组DNA和其对应的CDS序列,做出PLPK基因和其他Pto通路基因外显子-内含子结构图。
2.5 确定进化分析与保守模体
进化树中包括辣椒、番茄、马铃薯、烟草、拟南芥和水稻等植物的蛋白序列以确定进化关系。为确定辣椒和其他物种PLPKs的系统发育关系,利用所有可得到的植物物种PLPK序列,例如水稻、拟南芥、番茄、烟草和辣椒预测的PLPK序列,使用 Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/)程序(参数默认)进行多重序列比对。各种类型的拟南芥蛋白激酶[42]也包括在系统发育分析中,用来区分Pto和PLPK成员。使用软件JalView 2.8[43]编辑由高差异区域或缺失造成不确定比对的序列,并将其排除在进一步分析之外。使用MEGA 6.0软件(邻接法)对全长PLPK序列进行系统发育分析[44],Bootstrap参数为1 000。类似地,对辣椒的PLPK蛋白进行了单独的系统进化分析。在进化树根是辣椒类受体激酶(PLK)蛋白。由其他植物的PLPK成员的进化关系对辣椒的PLPK蛋白成员进行分类。通过EuLoc网站对辣椒PLPK进行亚细胞定位预测[45]。利用SMART网站(http://smart.embl-heidelberg.de/)来分析PLPK蛋白的结构和功能域。
2.6 选择压力检测
利用Datamonekey在线服务器(http://www.datamonkey.org/)来估计核酸密码子的非同义(dN)和同义(dS)替换速率,以及dN与dS的比值(ω),以此确定Pto和PLPK蛋白残基的丝氨酸/苏氨酸激酶(STK)结构域选择压力的性质。为避免假阳性,我们进行了重组检测的遗传算法(GARD)[46]。在Datamonekey网站上[47]我们使用几种基于密码子的最大似然法计算特定位点的选择压力:固定效应似然法(FEL),内部固定效应似然法(IFEL)和单一似然祖先计算(SLAC)。使用REV核酸替换模型来研究核酸位点。为确定多样化选择的单独密码子,我们进行了进化的混合效应模型(MEME)分析。ω值< 1表示负(纯化)选择,ω值> 1表示正(多样化)选择,ω=1表示位点不受选择压力的影响。
2.7 RNA-seq数据分析
利用前人研究的RNA-seq数据对各种辣椒基因型中PLPK基因的表达模式进行分析[48]。使用软件CLC Genomics Workbench v8.0(CLC Bio,Aarhus, Denmark)(参数默认)将测序片段比对到辣椒转录组数据库中(http://cab.pepper.snu.ac.kr)。所得数据用每千个碱基转录每百万映射读取的读长(RPKM)标准化。RPKM值进行log2转换,并使用R中ggplot包的heatmap.2函数生成热图。
3 结果
3.1 辣椒中AvrPto特异性诱导超敏反应和PVX抗性

图1 接种PVX载体的辣椒幼苗用于表达GFP,AvrPto和AvrPtoI96T
为了确定辣椒中是否发生了AvrPto的特异性识别,我们使用PVX衍生载体[49]表达AvrPto和AvrPtoI96T,体外和体内实验中96位点的取代突变阻止AvrPto和Pto的相互作用,使其对毒力没有影响[17]。在预实验中,我们对包括Perennial、234、Habanero、Rnaky、ECW和CM334等6种不同的辣椒基因型进行PVX的易感性筛选(附图1)。单独接种载体时,234、Habanero、ECW和Rnaky基因型的整个植株出现感病症状(附图1)。随后生长的234和ECW基因型植株被单独接种PVX载体或者GFP、AvrPto和AvrPtoI96T来表达构建体(图1)。病原和PVX同源序列在接种部位 (数据未显示)和上部未接种组织(图1)都存在,并且接种GFP构建体的植株中,其未接种的叶片在紫外线下产生荧光(数据未显示),这些都证明单独接种载体(附图1) 和接种包含GFP的PVX构建体 (图1A中Ⅰ,Ⅱ)都导致了系统症状的感染。相比之下,当植物接种pPVX::AvrPto时,症状被限制在接种叶片下端的叶上,并表现出局部坏死症状(图1A中Ⅲ,Ⅳ)。另外,仅在接种组织中检测到PVX抗原、PVX-和AvrPto-同源序列(数据未显示),而在未接种组织没有检测到(图1B)。在感病的辣椒植株中,PVX从感染部位散布到整个植株。同样,PVX::AvrPto也被认为是全身性移动。然而,PVX::AvrPto的散布在辣椒中受到限制,只能在感染的下部叶片中检测到。这可能是由于AvrPto是通过Pto同系物定位在接种叶上,从而抑制PVX::AvrPto的全身移动。因此,在未接种的上部叶片中PVX和AvrPto都没有检测到(图1B,道4)。
根据局部病变特征性坏死的出现,所有对PVX敏感的辣椒基因型似乎都识别AvrPto,因此不适用需要不同寄主反应的常规遗传方法。与Pto不互作的突变激发子AvrPtoI96T的获得,让我们有了一种替代方法。如果辣椒Pto同源序列在体内编码可以识别AvrPto的功能基因产物,且该识别对定位PVX感染是重要的,那么表达AvrPtoI96T的PVX载体应该是系统性扩散的。当辣椒幼苗接种pPVX::AvrPtoI96T后,在不亲和互作中观测到在接种的叶片上出现褪绿斑点,局部性坏死发展为相同尺寸的坏死性晕圈。这些症状随后遍布整个植株(图1A中Ⅴ, Ⅵ)。由于PVX抗原的存在(数据未显示)和上部未接种叶片的总RNA与PVX-和AvrPto-同源探针的杂交证实系统感染(图1B)。
总之,只有两种辣椒品种表达AvrPto的PVX载体限于接种组织。其他所有PVX载体全身移动,这与辣椒中Pto同系物可以在体内识别AvrPto假设一致。
3.2 Pto和PLPK的鉴定、分类和进化关系
为鉴定AvrPto可能的互作因子,我们对栽培种CM334进行全基因组分析,寻找辣椒中的番茄Pto同系物。我们用许多植物的Pto同源物和拟南芥的PLK构建系统进化树[42],将Pto同系物与含STK结构域的其他类型的蛋白区分开。只有在系统进化树中包括STK结构域亚域Ⅰ和Ⅺ之间的区域,包括PLPK在内的Pto同系物清晰地形成了拟南芥PLK和其他蛋白激酶的独立簇(附图S2)。根据序列相似性和系统发育分析,我们在辣椒基因组中鉴定出25个Pto类蛋白激酶基因(表1)。
为了确定Pto和PLPK同源物之间的进化关系,并对辣椒PLPK进行分类,我们用预测的辣椒PLPK序列和许多植物(比如番茄、烟草、马铃薯、拟南芥和水稻)的全长Pto和PLPK同源物构建进化树(图2)。系统进化分析显示在Pto同系物之间,Pto和PLPK存在两个不同的簇(图2)。茄科植物的Pto进化枝不同,并且在这个进化枝中番茄Pto同源物聚在一起。然而,在辣椒中没有发现Pto进化枝成员,这表明Pto的起源发生在茄科亚系的早期进化阶段。辣椒PLPK进一步分成八个亚族(PLPKⅠ至PLPKⅧ)。在PLPKⅠ和PLPKⅣ亚族中,许多双子叶植物的PLPK聚集在一起,而PLPKⅡ、PLPK Ⅲ、PLPKⅤ、PLPKⅥ 和PLPKⅦ亚族中,双子叶植物和单子叶植物PLPK成员都存在。然而,在PLPKⅡ和PLPKⅢ亚族中,单子叶和双子叶植物形成了不同的进化枝。除了在茄科中比较特别的PLPKⅧ亚族,其他所有的PLPK亚族中都观测到了至少一个拟南芥的PLPK成员。总之,系统进化分析表明辣椒PLPK基因簇模式与其他双子叶植物相似。此外,在进化树中的双子叶植物成员中间发现来自水稻的PLPK成员,这意味着PLPK蛋白家族是在单子叶和双子叶植物的共同祖先中进行进化。
3.3 辣椒PLPK蛋白家族系统进化分析
我们单独构建了辣椒PLPK蛋白系统进化树,来确定辣椒PLPK蛋白之间的系统进化关系。从这个进化分析来看,辣椒PLPK蛋白可以分为 PLPKⅠ和 PLPK Ⅷ 8个亚族(图3)。亚族PLPKⅠ和PLPKⅥ包括2个成员,PLPKⅢ和PLPKⅤ包括3个成员,PLPKⅡ、PLPKⅣ、PLPKⅦ和PLPKⅧ分别包括1、3、6、5个成员。
3.4 辣椒PLPKs和番茄Pto与PLPK的比较系统进化分析
为了确定辣椒和番茄Pto同源物的进化关系,我们生成了一个系统进化树(图F3),这表明辣椒中不存在类Pto成员。除了PLPKⅥ族在番茄中没有存在外,其他所有的PLPK同源物在辣椒和番茄中都显示出相同的聚类模式。辣椒和番茄的PLPKⅠ、 PLPKⅡ和PLPKⅣ族成员数量是一样的,分别是1、2和3个。番茄和辣椒的PLPKⅦ族中分别鉴定到5和6个成员。在PLPKⅧ族中,在辣椒中发现了5个成员,而在番茄中只有1个成员。这些观测结果表明,茄科植物中存在Pto和PLPK亚系扩大和分化。

表1 辣椒中类Pto基因和其他与Pto途径相关基因
3.5 辣椒PLPK和Pto通路基因的染色体分布和结构
我们根据基因在染色体上的顺序将辣椒PLPK基因命名为PLPK1到PLPK25。表1中列出了辣椒基因名称和其ID的详细信息。25个PLPK基因中有7个未被定位到染色体上。其余的18个PLPK基因定位到6条染色体上。Chr1、Chr4、Chr5、Chr8和Chr12上的染色体数差别很大,Chr2上最多有7个,Chr3和Chr6各有3个PLPK基因。Chr9和Chr10分别观测到了2个PLPK基因,然而Chr7只有1个基因(附图S4A)。基于连锁图谱分析,在未分配到任何染色体的7个PLPK基因中,至少有一个PLPK基因能分配到Chr12,并且2个PLPK基因(HPto-a和HPto-d)与Prf同系物Prf1.2(HPrf-c)和Prf1.3(HPrf-b)可以定位到Chr11(附图S4B)。HPto-a和HPrf-b(Prf1.3)被定位在番茄Pto进化枝的对应位置,这表明HPto-a是番茄Pto的直系同源物。然而,与嵌入Pto簇的番茄Prf相比,HPrf-b与HPto-a距离约2 cM。

图2 番茄Pto旁系同源基因与PLPK基因的系统进化分析
在 4个 Prf同 源 物 中,Prf1.1位 于 Chr4,Prf1.2和Prf1.3位于Chr11,Prf1.4不与任何染色体相关。在4个Pti1同源基因中,Pti1.1、Pti1.2(HPti1-a)和Pti1.3分别位于Chr3、Chr5和Chr12,基于连锁图,Pti1.4(Hpti1-b)可能位于Chr5(附图S4B)。在辣椒中检测到的2个Pti1同系物HPti1-a和Hpti1-b与番茄Chr5具有共线性,且Pti4、Pti5和Pti6.1分别位于Chr5、Chr2和Chr6。Pti6.2不位于任何染色体。
外显子-内含子结构分析表明,辣椒大多数PLPK基因的内含子较少,少数例外;PLPK3、PLPK5、PLPK6、PLPK7和PLPK18显示有 1个内含子,PLPK4显示有2个内含子(图3B)。相反,辣椒Prf基因显示有3到6个不同长度和位置的内含子(附图S5)。同样,Pti基因也被发现是高度分散的,并且显示具有0到7个内含子的复杂结构 (附图 S5)。
3.6 辣椒PLPK基因的保守基序分析
使用Clustal Omega程序对25个辣椒PLPK蛋白的预测氨基酸序列和番茄Pto的相应区域进行多序列比对。辣椒PLPK序列预测长度从683到898个氨基酸不等。辣椒的PLPK蛋白的预测氨基酸序列分析显示,其与番茄Pto有54%~71%的序列同源性。在辣椒PLPK进化组中,氨基酸序列的同源性变化范围从35%~96%。所有辣椒PLPK蛋白都显示出约275个氨基酸的高度保守的STK结构域(图4)。8个亚族都含有在大多数植物的STK结构域亚域Ⅰ至Ⅺ中发现的保守氨基酸残基。氨基酸序列比对表明高度保守的Pto同源物PLPK的几个显著特征,如STK结构域,位于亚域Ⅶ和Ⅷ之间的激活域,以及负责与AvrPto效应器特异性结合的内部P+1环结合位点[50]。STK结构域也包含几个分散的可变氨基酸位点。另外,Pto激活域的4个自磷酸化位点(Ser或Thr)中的3个保留在所有辣椒的相应区域中[51]。据报道,高度保守的Val55和His94的突变破坏了酵母中的Pto-AvrPto的相互作用,并抑制Pto介导的抗性[52]。

图3 辣椒PLPK蛋白系统进化分析
我们用SMART程序来确认PLPK的结构域,发现大部分PLPK蛋白包含4个功能域:信号肽区、麦角蛋白样区、跨膜区和STK结构域(图3C)。相比之下,番茄Pto只包含STK结构域。PLPKⅤ成员PLPK18在其C端具有额外的激酶结构域。PLPKⅧ成员PLPK7在其N端有2个抗病性的stress-antifung结构域。另外,几种辣椒PLPK蛋白区域显示出低复杂度区域(low complexity regions)。蛋白质亚细胞定位预测工具EuLoc[45]表明,所有辣椒的PLPK蛋白质都预测定位到质膜上。所有的Pti1蛋白也都预测定位在质膜上,而Pti4、Pti5和Pti6都预测定位于细胞核,Prfs被预测定位于细胞质。
为鉴定辣椒的PLPK激活域中保守的Pto自磷酸化位点和其他关键残基,我们用辣椒和番茄的Pto序列进行了多重序列比对分析(图4)。 5个Pto自磷酸化位点中的3个,包括Thr195、Ser198和Thr199[51]在辣椒PLPKs中高度保守,有少数例外。Thr195被PLPK24和PLPK25中的Gly取代,Ser198被 PLPK10和 PLPK13中 的 Val、PLPK24和PLPK25序列中的Thr取代。 Ser198是AvrPto-Pto介导的超敏反应所必需的,并且在辣椒PLPKs中高度保守。Pto自磷酸化位点Thr190在大多数辣椒PLPK中被Pro取代。在一些PLPK中,例如PLPK3、PLPK4、PLPK5 和 PLPK7,Ser被 Thr取代,产生了另一个磷酸化位点。在PLPK22中,Thr190被Leu取代。另外两个Pto磷酸化残基Thr204和Tyr207对于Pto功能至关重要,但不是对体外自体磷酸化[14,53]。这两个Pto磷酸化残基高度保守;然而,在所有辣椒PLPK中Thr204被Ser取代。
3.7 选择压力分析
我们对选择压力的变化进行了分析,检验类Pto和PLPK基因在基因倍增之后是否经历了适应性进化。通过使用Datamonkey网站中的各种模型多序列比对,我们基于密码子来检测选择压力[47]。我们还使用GARD分析比对以排除重组断裂点,分析显示没有任何重组事件的证据。
为鉴定单个密码子过去的选择,我们使用基于密码子的似然方法(FEL、IFEL和SLAC)计算每个密码子位点上的非同义(dN)和同义(dS)替换率以及dN/dS(ω)值。有趣的是,这些方法并没有显示在任何位点正向选择的证据(表2和附图S6)。大部分位点都显示为负(净化)选择,而位点13和22 (取决于使用哪种模式)是以中性速率进化。有趣的是,进化混合效应模型(MEME)确定了16个氨基酸位点(附图S6)是处于偶发性正选择(0.1显著水平)。其中,位于STK结构域的N末端有两个氨基酸位点,Ⅱ、Ⅲ、Ⅵ-b和Ⅶ亚结构域各有一个氨基酸位点,Ⅷ、Ⅸ和Ⅹ亚基有两个位点,在Ⅺ亚基有3个位点。这些结果表明,除纯化作用之外,正向选择在植物Pto同源物的进化过程中发挥了重要作用,并在适应性抗病性的进化中也发挥了重要作用。
3.8 Pto通路基因表达图谱
Pto通路基因的表达谱是用来自5种不同辣椒种质的RNA-seq数据生成的[48]。为分析不同材料间辣椒PLPK基因的表达图谱,我们根据log2转化的RPKM值生成热图和层次聚类(图5)。根据表达模式,我们将辣椒PLPK基因分为3组:C1、C2和C3(图5)。在C1组中,相对高表达的PLPK1,PLPK12-PLPK13和PLPK16-PLPK20聚集在一起。有趣的是,除了Ⅷ亚族以外,在C1中观测到每个辣椒至少一个PLPK亚族的基因。所有的亚族Ⅷ成员(PLPK3-PLPK7)和分别属于亚族Ⅴ和Ⅶ的PLPK22、PLPK23都聚类到C2组,并且都是温和水平表达。辣椒PLPK基因PLPK2、PLPK8 -PLPK11和PLPK14 - PLPK15都聚类到C3组,并以低水平表达。有趣的是,除了PLPKⅡ和PLPKⅧ亚族,每个亚族中至少有一个基因在C3组中。包括Pti和Prf的许多Pto通路基因也表现出中等到低的表达水平。一般来说,植物R基因在感染或未感染的植物中表现出低水平的组成型表达,这与其在病原体中识别一般规律一致[54-55]。总之,5种辣 椒 种 质(C.annuum'PI201234','YCM334',C.chinense'Aji Dulce','PI152225'和C.chacoense'PI260429')基因型中Pto通路基因的表达谱非常相似(图5),这表明辣椒物种中Pto信号基因调控机制是高度保守的。
4 讨论
在番茄中,Pto位点由5个存在于5号染色体上的Pto同源物组成,这些Pto同源物可能是通过连续的基因复制和缺失进化的[10,34]。Pto位点及其在AvrPto识别特异性中的基因对基因模式在茄科物种是保守的[19,54]。然而,Pto的识别在许多茄科物种(包括辣椒和马铃薯)中是否是具有特异性还是未知的。尽管许多植物物种中的AvrPto已经被鉴定了,但其识别是否由类Pto激酶介导尚不清楚。另外,除了番茄和烟草之外,在其他植物物种中没有鉴定出识别AvrPto的功能性类Pto蛋白。辣椒PVX载体系统的结果为AvrPto的体内识别提供了证据。当然,我们不能排除辣椒中识别AvrPto的是类Pto基因或者其他PLKs基因的可能性,除非使用反向遗传方法来验证该结果。Pto通过与NBLRR和Prf相互作用介导番茄中AvrPto/AvrPtoB的识别[56]。然而,AvrPto/AvrPtoB的靶点是各种RLK[57-60]的激酶结构域,而不是Pto的STK结构域。Pto被Rrf当作分子诱饵来与病原效应蛋白相互作用[61-62]。AvrPto/AvrPtoB效应物与几种植物分子互作,如拟南芥GTPase RabE[63-64]和几种PLK激酶结构域,如 CERK1,BAK1,EFR1 和 FLS2[57-60,65]。

图4 STK结构域的多重序列比对
为了鉴定辣椒中可能与AvrPto相互作用的番茄Pto同系物,我们进行了全基因组分析,共鉴定了25个全长的类Pto(PLPK)基因。大多数R基因是多基因家庭的成员,并且在植物基因组中以簇的形式出现。基因重复和多样化被认为是R基因簇进化扩张的原因[5]。一些植物物种的Pto同系物的进化分析鉴定出了Pto同源物的几个亚族[27,30,32]。在趣的是,辣椒中没有发现Pto进化枝成员。我们利用3个不同的辣椒基因组数据库“CM334”、“Chiltepin”和“Zunla-1”(http://cab.pepper.snu.ac.kr/) 和NCBI中的辣椒表达序列标签(ESTs)证实了辣椒基因组中没有番茄Pto同系物(Pto 进化枝)。Wan等人[66]在使用针对Pto基因保守区域设计的引物时,未在辣椒中扩增出真正的Pto同系物。此外,在番茄中进行R基因定位实验没有检测出番茄类Pto的同源物[67]。除Pto进化枝外,辣椒和其他植物的茄科PLPK蛋白在进化树枝干中均匀分布,这表明PLPK蛋白是先于单子叶和双子叶分化由共同的祖先基因产生。辣椒中番茄Pto同源物的缺失进一步表明茄科的分支特异性扩大了Pto进化枝。这一观察结果也表明,Pto基因在病原识别和抗病中的作用可能是茄科早期进化过程中产生的[27,35],或者是从辣椒中丧失的。

表2 多物种类Pto基因STK结构域正选择分析
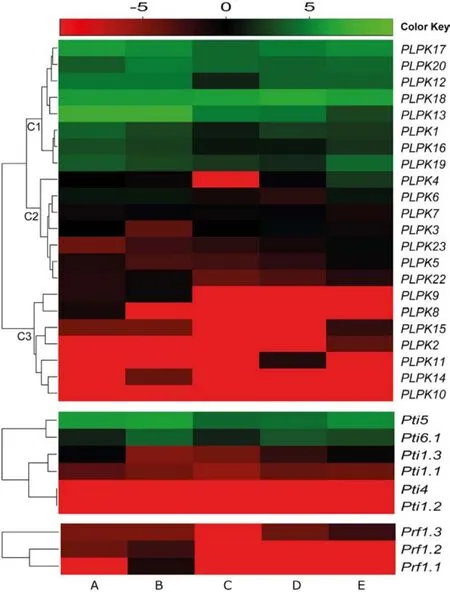
图5 5个不同辣椒品种的PLPK基因表达谱
可能导致辣椒和一些番茄基因型中Pto进化枝缺失的潜在机制尚不清楚[34-35]。一些栽培番茄(S.lycopersicum)品种表现出对AvrPto特异的,从番茄(S.pimpinellifolium)引入的Pto介导的Pst抗性[10,36]。有2个可能的原因来解释Pto特异识别在一些番茄品种中保留,并在一些亲缘关系很近的物种中丧失的现象。第一,在野生番茄(S.lycopersicum)的驯化过程中,可能发生了强烈的遗传瓶颈效应,并导致缺乏Pto基因的番茄品系的选择[35]。第二,在缺乏具有AvrPto和Pst的基因的情况下,与Pto位点相关的适合度代价可能导致对含有Pto基因的番茄品系的选择[35]。虽然Pto基因位点的有毒效应可能难以通过实验确定[35],但是没有证据显示在含有Pto位点和不含有Pto位点的番茄的近等基因与Pst抗性有相关的适合度代价[35,68]。
尽管对番茄进行了广泛的研究,但其他茄科植物中Pto同系物的功能特异性还不太清楚。Pto同源物与番茄Pto有78%~91%的核苷酸相似性,并且其具有功能性蛋白激酶活性,但都不能与AvrPto和AvrPtoB效应子互作[12,69]。Pto基因家族的基因复制以及随后的多样化可能导致了可变的特异识别性[70]。Pto和Pto同系物仅在几个氨基酸残基上变化,这可能引起蛋白质构象变化,导致与其他蛋白质物理性相互作用能力的改变[36]。Fen基因是5个Pto旁系同源物之一,与Pto基因有87%的相似度,并参与共同的导致超敏反应的信号事件。但是,它是由一个不同的信号激活的[70-71]。相反,LhirPto显示出与Pto基因97%的相似度,只有17个氨基酸的不同使其与Pto区分开来,但是LhirPto不能取代Pto的抗病性[35]。这些观察结果进一步表明在Pto-AvrPto的识别特异性上有选择压力[72]。
多样化选择和适应性选择被假定在R基因的进化中起关键作用。适应性选择被认为是推动寄主-病原体识别功能基因区进化的重要力量[5,73]。有趣的是,纯化选择似乎是对含有STK结构域以保持其祖先状态的基因的主要选择性压力[32]。然而前人的研究认为是偶发性正选择,这表明在进化过程中选择压力可能发生了变化。有报道称,适应性进化常常涉及在某个基因的少数几个位点上偶发的突变,并可能只影响系统进化的一部分基因[74]。与此一致的是,MEME分析显示一些密码子属于偶发性正选择。Pto的激活结构域(氨基酸182和211之间的区域),在AvrPto识别中起重要作用[27]。有趣的是,我们结果显示Ser198和Ile208这两个位置都经历了片段正选择,这意味着它们在适应性进化中发挥了重要作用。最近的文献显示,已被用作Pto/AvrPto反应的阴性对照的本氏烟草(N.benthamiana)也可以用基因对基因的方式识别AvrPto,并且这种识别依赖于Prf[19]。AvrPto的识别使植物产生抗性也在远距离的分类群中观察到,例如在大豆中[28]。这些观察结果进一步表明,除了进化和平衡选择外[72],适应性选择也可能影响Pto位点的进化。
总之,在不同辣椒基因型中观察到来自于Pst的AvrPto效应子的识别相关的非寄主应答。AvrPto的识别可能是由Pto同源物或其他RLK蛋白介导的。我们通过全基因组分析鉴定了25个辣椒PLPK基因,发现它们分为8个系统进化亚族。Pto进化枝代表茄科植物最近起源的基因家族,但PLPK基因代表了古代共同起源的分歧基因。在辣椒基因组中没有番茄Pto旁系同源物,这表明Pto基因家族经历了对Pst抗性的不同适应性进化过程。辣椒PLPK基因的特点是存在高度保守的STK结构域。PLPK基因中与碳水化合物结合的麦芽糖结合蛋白类似结构域的存在暗示糖代谢与疾病抗性信号传导通路可能具有某种联系[75]。在全部5种的辣椒基因型中观察到Pto通路基因的类似表达谱,这些观察结果表明具有高度保守功能域的PLPK基因与辣椒基因型中的Pto信号传导通路有共同的高度保守的基因调控机制。对于Pto和PLPK,以及它们与AvrPto或其他效应物的相互作用的进一步研究将对植物-病原体共同进化过程提供更多思路,对于辣椒PLPK基因的全基因组分析为进一步功能研究提供了重要的基因资源,也为揭示辣椒非寄主抗病性提供了分子基础。
[1] Chisholm S T, Coaker G, Day B, Staskawicz B J. Hostmicrobe interactions: shaping the evolution of the plant immune response[J]. Cell, 2006, 124(4):803–814
[2] Cui H, Tsuda K, Parker J E. Effector-triggered immunity:from pathogen perception to robust defense[J]. Annu Rev Plant Biol., 2015, 66:487– 511
[3] Martin G B. Functional analysis of plant disease resistance genes and their downstream effectors[J]. Curr Opin Plant Biol., 1999, 2(4):273–279
[4] Pan Q, Wendel J, Fluhr R. Divergent evolution of plant NBS-LRR resistance gene homologues in dicot and cereal genomes[J]. J. Mol. Evol., 2000, 50(3):203–213
[5] Richter T E, Ronald P C. The evolution of disease resistance genes[J]. Plant Mol Biol, 2000, 42:195–204
[6] White F F, Yang B, Johnson L B. Prospects for understanding avirulence gene function[J]. Curr Opin Plant Biol., 2000, 3(4):291–298
[7] Gururani M A, Venkatesh J, Upadhyaya C P, Nookaraju A, Pandey S K, Park S W. Plant disease resistance genes:current status and future directions[J]. Physiological and Molecular Plant Pathology, 2012, 78:51–65
[8] Belkhadir Y, Subramaniam R, Dangl J L. Plant disease resistance protein signaling: NBS–LRR proteins and their partners[J]. Curr Opin Plant Biol., 2004, 7(4):391–399
[9] Hammond-Kosack K E, Parker J E. Deciphering plant–pathogen communication: fresh perspectives for molecular resistance breeding[J]. Curr Opin Biotechnol, 2003,14(2):177–193
[10] Martin G B, Brommonschenkel S, Chunwongse J, Frary A, Ganal M, Spivey R, Spivey R, Wu T, Earle E D,Tanksley S D. Map-based cloning of a protein kinase gene conferring disease resistance in tomato[J]. Science, 1993,262: 1432–1436
[11] Collmer A, Badel J L, Charkowski A O, Deng W L, Fouts D E, Ramos A R, et al.Pseudomonas syringaeHrp type Ⅲsecretion system and effector proteins[J]. Proc. Natl. Acad.Sci. USA, 2000, 97(16):8770–8777
[12] Kim Y J, Lin N C, Martin G B. Two distinctPseudomonaseffector proteins interact with the Pto kinase and activate plant immunity[J]. Cell, 2002, 109(5):589–598
[13] Lin N C, Martin G B. Pto-and Prf-mediated recognition of AvrPto and AvrPtoB restricts the ability of diversePseudomonas syringaepathovars to infect tomato[J]. Mol Plant Microbe Interact, 2007, 20(7):806–815
[14] Pedley K F, Martin G B. Molecular basis of Pto-mediated resistance to bacterial speck disease in tomato[J]. Annu Rev Phytopathol, 2003, 41(1):215–243
[15] Mathieu J, Schwizer S, Martin G B. Pto kinase binds two domains of AvrPtoB and its proximity to the effector E3 ligase determines if it evades degradation and activates plant immunity[J]. PLoS Pathog, 2014, 10(7):e1004227
[16] Chang J H, Tobias C M, Staskawicz B J, Michelmore R W. Functional studies of the bacterial avirulence protein AvrPto by mutational analysis[J]. Mol Plant Microbe Interact, 2001, 14(4):451–459
[17] Shan L, Thara V K, Martin G B, Zhou J M, Tang X. ThePseudomonasAvrPto protein is differentially recognized by tomato and tobacco and is localized to the plant plasma membrane[J]. Plant Cell, 2000, 12 (12):2323–2337
[18] Abramovitch R B, Kim Y J, Chen S, Dickman M B, Martin G B.Pseudomonastype III effector AvrPtoB induces plant disease susceptibility by inhibition of host programmed cell death[J]. EMBO J., 2003, 22(1):60–69
[19] Kang L, Tang X, Mysore K S. Pseudomonas typeⅢ effector AvrPto suppresses the programmed cell death induced by two nonhost pathogens inNicotiana benthamianaand tomato[J]. Mol Plant Microbe Interact,2004, 17(12):1328–1336
[20] Gu Y Q, Wildermuth M C, Chakravarthy S, Loh Y T, Yang C, He X, Han Y, Martin G B. Tomato transcription factorsPti4, Pti5, andPti6activate defense responses when expressed inArabidopsis[J]. Plant Cell, 2002, 14:817–831
[21] He P, Warren R F, Zhao T, Shan L, Zhu L, Tang X, Zhou J M. Overexpression ofPti5in tomato potentiates pathogeninduced defense gene expression and enhances disease resistance toPseudomonas syringaepv. tomato[J]. Mol Plant Microbe Interact, 2001, 14(12):1453–1460
[22] van der Hoorn R A, Kamoun S. From guard to decoy: a new model for perception of plant pathogen effectors[J].Plant Cell, 2008; 20(8):2009–2017
[23] Cui H, Xiang T, Zhou J M. Plant immunity: a lesson from pathogenic bacterial effector proteins[J]. Cell Microbiol,2009, 11(10):1453–1461
[24] Balmuth A, Rathjen J P. Genetic and molecular requirements for function of the Pto/Prf effector recognition complex in tomato andNicotiana benthamiana[J]. Plant J.,2007, 51(6):978–990
[25] Oh C S, Martin G B. Effector-triggered immunity mediated by the Pto kinase[J]. Trends Plant Sci., 2011, 16(3):132–140
[26] Vallad G, Rivkin M, Vallejos C, McClean P. Cloning and homology modelling of a Pto-like protein kinase family of common bean (Phaseolus vulgarisL.) [J]. Theor Appl Genet., 2001, 103(6–7):1046–1058
[27] Vleeshouwers V G, Martens A, van Dooijeweert W, Colon L T, Govers F, Kamoun S. Ancient diversification of the Pto kinase family preceded speciation inSolanum[J]. Mol Plant Microbe Interact, 2001, 14(8):996–1005
[28] Ronald P C, Salmeron J M, Carland F M, Staskawicz B J. The cloned avirulence gene avrPto induces disease resistance in tomato cultivars containing thePtoresistance gene[J]. J. Bacteriol, 1992, 174(5):1604–1611
[29] Di Gaspero G, Cipriani G. Nucleotide binding site/leucinerich repeats, Pto-like and receptor-like kinases related to disease resistance in grapevine[J]. Mol Genet Genomics,2003, 269 (5):612–623
[30] Wan H, Qian C, Malik A A, Zhao Z, Chen J. Isolation,phylogeny and evolutionary analysis ofPto-typedisease resistance gene analogues from aCucumis hystrixintrogression line of cucumber (C. sativus) [J]. Functional Plant Biology. 2010; 37(6):513–523.
[31] Peraza-Echeverria S, James-Kay A, Canto-Canché B,Castillo-Castro E. Structural and phylogenetic analysis of Pto-type disease resistance gene candidates in banana[J].Mol Genet Genomics, 2007, 278(4):443–453
[32] Zamora M M, Castagnaro A P, Ricci J D. Genetic diversity of Pto-like serine/threonine kinase disease resistance genes in cultivated and wild strawberries[J]. J. Mol. Evol., 2008,67(2):211– 221
[33] Deng Z, Gmitter F Jr. Cloning and characterization of receptor kinase class disease resistance gene candidates inCitrus[J]. Theor Appl Genet, 2003, 108(1):53–61
[34] Michelmore R W, Meyers B C. Clusters of resistance genes in plants evolve by divergent selection and a birth-anddeath process[J]. Genome Res., 1998, 8(11):1113–1130
[35] Riely B K, Martin G B. Ancient origin of pathogen recognition specificity conferred by the tomato disease resistance genePto[J]. PNAS, 2001, 98(4):2059–2064
[36] Jia Y, Loh Y T, Zhou J, Martin G B. Alleles ofPtoandFenoccur in bacterial speck-susceptible and fenthioninsensitive tomato cultivars and encode active protein kinases[J]. Plant Cell, 1997, 9(1):61–73
[37] Baulcombe D C, Chapman S, Cruz S. Jellyfish green fluorescent protein as a reporter for virus infections[J].Plant J., 1995, 7(6):1045–1053
[38] Tobias C M, Oldroyd G E, Chang J H, Staskawicz B J.Plants expressing thePtodisease resistance gene confer resistance to recombinant PVX containing the avirulence geneAvrPto[J]. Plant J., 1999, 17(1):41–50
[39] Grube R C, Zhang Y, Murphy J F, Loaiza-Figueroa F,Lackney V K, Provvidenti R, Jahn M K. New source of resistance toCucumber mosaic virusinCapsicum frutescens[J]. Plant Dis., 2000, 84(8):885–891
[40] Livingstone K D, Lackney V K, Blauth J R, Van Wijk R, Jahn M K. Genome mapping inCapsicum and the evolution of genome structure in theSolanaceae[J].Genetics, 1999, 152(3):1183–1202
[41] Voorrips R E. MapChart: software for the graphical presentation of linkage maps and QTLs[J]. J. Hered., 2002,93(1):77–78
[42] Hardie D G. Plant protein serine/threonine kinases:classification and functions[J]. Annu Rev Plant Physiol Plant Mol Biol, 1999, 50(1):97–131
[43] Waterhouse A M, Procter J B, Martin D M, Clamp M,Barton G J. Jalview Version 2-a multiple sequence alignment editor and analysis workbench[J].Bioinformatics, 2009, 25(9):1189–1191
[44] Tamura K, Peterson D, Peterson N, Stecher G, Nei M,Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance,and maximum parsimony methods[J]. Mol Biol Evol.,2011, 28(10):2731–2739
[45] Chang T H, Wu L C, Lee T Y, Chen S P, Huang H D,Horng J T. EuLoc: a web-server for accurately predict protein subcellular localization in eukaryotes by incorporating various features of sequence segments into the general form of Chou’s PseAAC[J]. J. Comput Aided Mol Des., 2013, 27(1):91–103
[46] Pond S L K, Posada D, Gravenor M B, Woelk C H, Frost S D. Automated phylogenetic detection of recombination using a genetic algorithm[J]. Mol Biol Evol., 2006,23(10):1891–1901
[47] Delport W, Poon A F, Frost S D, Pond S L K.Datamonkey 2010: a suite of phylogenetic analysis tools for evolutionary biology[J]. Bioinformatics, 2010,26(19):2455–2457
[48] Kang J H, Yang H B, Jeong H S, Choe P, Kwon J K, Kang B C. Single nucleotide polymorphism marker discovery from transcriptome sequencing for marker-assisted backcrossing inCapsicum[J]. Korean J Hortic Sci Technol,2014, 32(4):535–543
[49] Gilardi P, Garcia-Luque I, Serra M T. Pepper mild mottle virus coat protein alone can elicit theCapsicumspp. L3gene-mediated resistance[J]. Mol Plant-Microbe Interact,1998, 11(12):1253–1257
[50] Frederick R D, Thilmony R L, Sessa G, Martin G B.Recognition specificity for the bacterial avirulence protein AvrPto is determined by Thr-204 in the activation loop of the tomato Pto kinase[J]. Mol. Cell, 1998; 2(2):241–245
[51] Sessa G, D'Ascenzo M, Martin G B. Thr38 and Ser198 are Pto autophosphorylation sites required for the AvrPto–Pto‐mediated hypersensitive response[J]. EMBO J., 2000,19(10):2257–2269
[52] Scofield S R, Tobias C M, Rathjen J P, Chang J H. Molecular basis of gene-for-gene specificity in bacterial speck disease of tomato[J]. Science, 1996,274(5295):2063–2065
[53] Rathjen J P, Chang J H, Staskawicz B J, Michelmore R W. Constitutively active Pto induces a Prf‐dependent hypersensitive response in the absence of avrPto[J].EMBO J., 1999, 18(12):3232–3240
[54] Hulbert S H, Webb C A, Smith S M, Sun Q. Resistance gene complexes: evolution and utilization[J]. Annual review of phytopathology. 2001; 39(1):285–312
[55] Yuan M, Chu Z, Li X, Xu C, Wang S. Pathogen-induced expressional loss of function is the key factor in race-specific bacterial resistance conferred by a recessiveRgene xa13 in rice[J]. Plant Cell Physiol, 2009, 50(5):947–955
[56] Gutierrez J R, Balmuth A L, Ntoukakis V, Mucyn T S,Gimenez‐Ibanez S, Jones A M, Rathjen J P. Prf immune complexes of tomato are oligomeric and contain multiple Pto-like kinases that diversify effector recognition[J]. Plant J., 2010, 61(3):507–518
[57] Gimenez-Ibanez S, Hann D R, Ntoukakis V, Petutschnig E, Lipka V, Rathjen J P. AvrPtoB targets the LysM receptor kinase CERK1 to promote bacterial virulence on plants[J].Curr Biol., 2009, 19 (5):423–429
[58] Göhre V, Robatzek S. Breaking the barriers: microbial effector molecules subvert plant immunity[J]. Annu Rev Phytopathol, 2008, 46:189–215
[59] Shan L, He P, Li J, Heese A, Peck S C, Nürnberger T, Martin G B, Sheen J. Bacterial effectors target the common signaling partner BAK1 to disrupt multiple MAMP receptor-signaling complexes and impede plant immunity[J]. Cell Host Microbe, 2008, 4(1):17–27
[60] Xiang T, Zong N, Zou Y, Wu Y, Zhang J, Xing W, Li Y,Tang X, Zhu L, Chai J, Zhou J M.Pseudomonas syringaeeffector AvrPto blocks innate immunity by targeting receptor kinases[J]. Curr Biol, 2008; 18(1):74–80
[61] Collier S M, Moffett P. NB-LRRs work a “bait and switch”on pathogens[J]. Trends Plant Sci., 2009, 14(10):521–529
[62] Saur I M-L, Conlan B F, Rathjen J P. The N-terminal domain of the tomato immune protein Prf contains multiple homotypic and Pto kinase interaction sites[J]. J Biol Chem., 2015, 290 (18):11258–11267
[63] Bogdanove A J, Martin G B. AvrPto-dependent Ptointeracting proteins and AvrPto-interacting proteins in tomato[J]. Proc Natl. Acad. Sci. USA, 2000, 97(16):8836–8840
[64] Speth E B, Imboden L, Hauck P, He S Y. Subcellular localization and functional analysis of theArabidopsisGTPase RabE[J]. Plant Physiol, 2009, 149(4):1824–1837
[65] Yeam I, Nguyen H P, Martin G B. Phosphorylation of thePseudomonas syringaeeffector AvrPto is required for FLS2/BAK1‐independent virulence activity and recognition by tobacco[J]. Plant J., 2010, 61(1):16–24
[66] Wan H, Yuan W, Ruan M, Ye Q, Wang R, Li Z, Zhou G,Yao Z, Yang Y. Identification, phylogeny, and expression analysis ofPto-like genes in pepper[J]. Plant Mol Biol
Rep., 2013; 31(4):901–916
[67] Pflieger S, Lefebvre V, Caranta C, Blattes A, Goffinet B,Palloix A. Disease resistance gene analogs as candidates for QTLs involved in pepper-pathogen interactions[J].Genome, 1999, 42(6):1100–1110
[68] Tanksley S, Bernacchi D, Fulton T, Beck-Bunn T, Emmatty D, Eshed Y, et al. Comparing the effects of linkage drag in a set of processing tomato lines nearly isogenic for theMigene for resistance to root knot nematodes[J]. Rep Tomato Genet Coop, 1997, 47:35–36
[69] Chang J H, Tai Y S, Bernal A J, Lavelle D T, Staskawicz B J,Michelmore R W. Functional analyses of the Pto resistance gene family in tomato and the identification of a minor resistance determinant in a susceptible haplotype[J]. Mol Plant Microbe Interact, 2002, 15(3):281–291
[70] Martin G B, Frary A, Wu T, Brommonschenkel S,Chunwongse J, Earle E D, Tanksley S D. A member of the tomato Pto gene family confers sensitivity to fenthion resulting in rapid cell death[J]. Plant Cell, 1994, 6(11):1543–1552
[71] Loh Y T, Martin G B. ThePtobacterial resistance gene and theFeninsecticide sensitivity gene encode functional protein kinases with serine/threonine specificity[J]. Plant Physiol, 1995, 108(4):1735–1739
[72] Rose L E, Michelmore R W, Langley C H. Natural variation in thePtodisease resistance gene within species of wild tomato (Lycopersicon). Ⅱ. Population genetics of Pto[J]. Genetics, 2007, 175(3):1307–1319
[73] Chen Q, Han Z, Jiang H, Tian D, Yang S. Strong positive selection drives rapid diversification of R-genes in Arabidopsis relatives[J]. J. Mol Evol., 2010, 70(2):137–148
[74] Pond S L K, Murrell B, Fourment M, Frost S D, Delport W, Scheffler K. A random effects branch-site model for detecting episodic diversifying selection[J]. Mol Biol Evol.2011, 28(11):3033–3043
[75] Hok S, Danchin E G, Allasia V, Panabieres F, Attard A,Keller H. AnArabidopsis(malectin-like) leucine-rich repeat receptor-like kinase contributes to downy mildew disease[J]. Plant, Cell & Environment, 2011, 34(11):1944–1957
(文献来源:PLoS ONE 11(8): e0161545)华中农业大学园艺林学学院 胡震竺 刘越 孔秋生 译

