胡萝卜类甜蛋白家族鉴定与生物信息学分析
刘 潮 韩利红 王海波 宋培兵 唐利洲
(曲靖师范学院云南高原生物资源保护与利用研究中心,生物资源与食品工程学院,云南省高校云贵高原动植物多样性及生态适应性进化重点实验室,云南曲靖,655011)
胡萝卜类甜蛋白家族鉴定与生物信息学分析
刘 潮 韩利红 王海波 宋培兵 唐利洲*
(曲靖师范学院云南高原生物资源保护与利用研究中心,生物资源与食品工程学院,云南省高校云贵高原动植物多样性及生态适应性进化重点实验室,云南曲靖,655011)
为了全面了解胡萝卜基因组中类甜蛋白家族基因结构和蛋白功能,利用胡萝卜基因组数据库,通过生物信息学方法对筛选的32个胡萝卜类甜蛋白家族成员进行鉴定、聚类及结构功能分析。结果表明:胡萝卜类甜蛋白家族基因分布在8条染色体上,该家族蛋白保守性较强。系统发育分析显示,胡萝卜类甜蛋白家族属于10个进化组,其中进化组5中的基因主要来自1号染色体,该组成员具有抑菌潜力,值得深入研究。
胡萝卜;类甜蛋白;系统进化;生物信息学
类甜蛋白(thaumatin-like protein,TLP)又称为PR5蛋白,广泛分布于多种植物、动物及微生物中(Abad et al.,1993;Shatters et al.,2006),具有广谱的抗真菌活性、β-1,3葡聚糖酶活性、酶抑制活性、过敏原活性以及抗冻活性等(Liu et al.,2010),当植物遭受胁迫时被诱导表达,在植物的防御反应中发挥作用(van Loon et al.,2006;Beatrice et al.,2016;Rout et al.,2016),过表达TLP基因植物显著延缓了病症(Kalpana et al.,2006;Maruthasalam et al.,2007;Rout et al.,2016)。TLP蛋白除了参与植物的胁迫反应之外,也参与了生长发育的多项进程,有些植物(如樱桃、苹果、香蕉)果实成熟时很多TLPs高表达(Fils-Lycaon et al.,1996;Kim et al.,2003;Ho et al.,2007)。目前在拟南芥、水稻和杨树基因组中已鉴定到多个TLPs(Liu et al.,2010;Zhao & Su,2010;Petre et al.,2011),对这些物种的TLP蛋白系统发育分析发现,水稻和拟南芥TLP分布于多个支系,并存在染色体内和染色体间的复制(Shatterset al.,2006)。研究植物基因组中TLP家族基因结构、分布与系统进化,对全面了解该家族蛋白在植物生命进程中的作用具有重要意义。
胡萝卜(Daucus carota)原产于西亚,属于伞形科胡萝卜属,根作蔬菜食用,含有多种维生素及胡萝卜素,在世界各国普遍栽培。胡萝卜苗期、成株期均有虫害和白粉病、细菌性软腐病等病害发生,往往造成胡萝卜生产上毁灭性的损失。尽管对TLP家族的研究有了重大进展,但这些研究主要限于拟南芥和水稻等模式植物。为了全面了解胡萝卜基因组中TLP家族基因结构和蛋白功能,本试验通过生物信息学方法利用胡萝卜基因组数据库,从全基因组水平上分析TLP家族基因在胡萝卜中的数目、基因结构、染色体定位以及系统进化,为进一步研究该家族成员的生物学功能奠定基础。
1 材料与方法
1.1胡萝卜TLP家族数据获取与鉴定
胡萝卜(D. carota)全基因组数据下载于GenBank数据库,其基因组大小为473 Mb,分布于9条染色体,包括了32 113个基因座位,产生44 740个编码蛋白转录本(Iorizzo et al.,2016)。以典型TLP蛋白1Z3Q(Musa acuminate)序列为探针,搜索胡萝卜蛋白数据库,在Pfam数据库(http:// pfam.sanger.ac.uk/)和SMART数据库(http://smart. embl-heidelberg.de/)中对候选蛋白序列功能域进行确认。通过Expasy(http://www.expasy.org/tools/)对蛋白生理生化特征进行预测。
1.2胡萝卜TLP基因和蛋白结构及染色体定位分析
从GenBank数据库中获得胡萝卜TLP蛋白对应的基因序列和CDS序列。使用基因结构显示系统(http://gsds.cbi.pku.edu.cn/index.php)绘制基因结构示意图。通过MEME SUITE在线工具(Bailey et al.,2009)(http://meme-suite.org/tools/meme)预测胡萝卜TLP家族蛋白序列的保守Motif,Motif搜索数目为5,最大宽度20个氨基酸。通过 MapInspect(http://mapinspect.software.informer. com/)进行染色体定位作图。利用Swiss-Model构建蛋白同源三级结构模型。使用VMD 1.8.5(http:// www.ks.uiuc.edu/Research/vmd/vmd-1.8.5/)进行3D结构的可视化。
1.3胡萝卜TLP蛋白序列比对与系统进化分析
所有 蛋 白使 用ClustalX(Thompson et al.,1997)进行序列比对,应用MEGA 5.0(Tamura et al.,2011)软件,采用NJ(Neighbor-Joining)法构建系统进化树。
2 结果与分析
2.1胡萝卜TLP家族成员的鉴定
采用生物信息学方法,鉴定到32个具有典型THN结构域的胡萝卜TLP家族成员(表1)。32条TLPs序列中,有25条与1Z3Q序列一致性高于40%,所有序列E值均小于1E-30。蛋白理化性质分析显示,最长的蛋白序列XP_017252308.1包含650个氨基酸,最短的XP_017229268.1也包含171个氨基酸,30个成员的蛋白氨基酸数目介于220~330之间,蛋白分子量介于18.64~68.39 kD,蛋白等电点介于4.17~8.99,69%的蛋白属于酸性蛋白。
利用ClustalX进行序列比对,发现所有胡萝卜TLP均含有16个半胱氨酸残基,多数均具有索玛甜家族标签G-X-[GF]-X-C-X-T-[GA]-D-C-X-(1,2)-G-X-(2,3)-C和5个保守的REDDD氨基酸残基,后者参与蛋白维持适当的拓扑结构和酸裂周围的表面静电势,对TLPs抗真菌活性必不可少(Liu et al.,2012)。
2.2胡萝卜TLP家族基因和蛋白结构分析
通过对胡萝卜TLP家族基因结构、内含子位置和相位进行分析,发现5种基因结构类型,其中不含内含子和含有1、2、3(及以上)个内含子的基因数分别为6和17、6、3;相同数量内含子的基因类型中,内含子的相位又有多种类型,其基因结构类型较丰富。根据剪接中位置的不同,内含子分为3种相位类型,0型内含子位于2个密码子之间,1型内含子位于密码子的第1和第2碱基之间,2型内含子位于密码子的第2和第3碱基之间(Sharp,1981)。相位的改变会导致后续阅读框发生改变,因此内含子的相位通常是比较保守的。胡萝卜TLP家族基因17个含有1个内含子的TLP基因中,7个均为1型相位。含有同样内含子相位的基因可能来源于相同的祖先,可能是新近由基因扩张复制而来,而含有不同内含子相位的基因可能发生了独立的内含子获取或丢失,可能属于较古老的基因。XP_017237350.1含有1个内含子,属于数量最多的基因结构类型;XP_017233153.1和XP_017216393.1分别为不含内含子和含2个内含子的基因,属于数量次之的基因结构类型(图1)。
使用MEME SUITE在线软件对胡萝卜TLP家族蛋白序列保守氨基酸Motif进行分析,搜索参数选择,最大宽度20个氨基酸。发现5类Motif的保守性较强,它们的正则表达式如图2所示。30条序列均含有全部5种Motif,仅有蛋白XP_017229268.1缺少Motif 2和Motif 4,蛋白XP_017254223.1缺少Motif 5,说明该家族蛋白保守性较强。XP_017233153.1蛋白三维结构模型显示,蛋白具有典型的3个功能域:DomainⅠ为N端核心功能域,由2个反向平行的β片层(分别包含5个和6个β折叠)组成;DomainⅡ由半胱氨酸二硫键形成的3个较短的α螺旋构成;Domain Ⅲ由2个β折叠和1个大环构成(Leone et al.,2006)。Motif 1位于蛋白“V”字形裂缝的内部,Motif 2位于功能域Ⅰ和功能域Ⅱ的结合部位,Motif 3位于功能域Ⅱ和功能域Ⅲ的结合部位,Motif 4位于蛋白“V”字形裂缝的外缘,属于功能域Ⅱ,Motif 5位于蛋白序列的碳端,属于功能域Ⅰ(图3)。Motif 1、Motif 2、Motif 3和Motif 4构成了“V”字形裂缝,该部分保守的蛋白氨基酸残基保证了蛋白催化功能的实现。
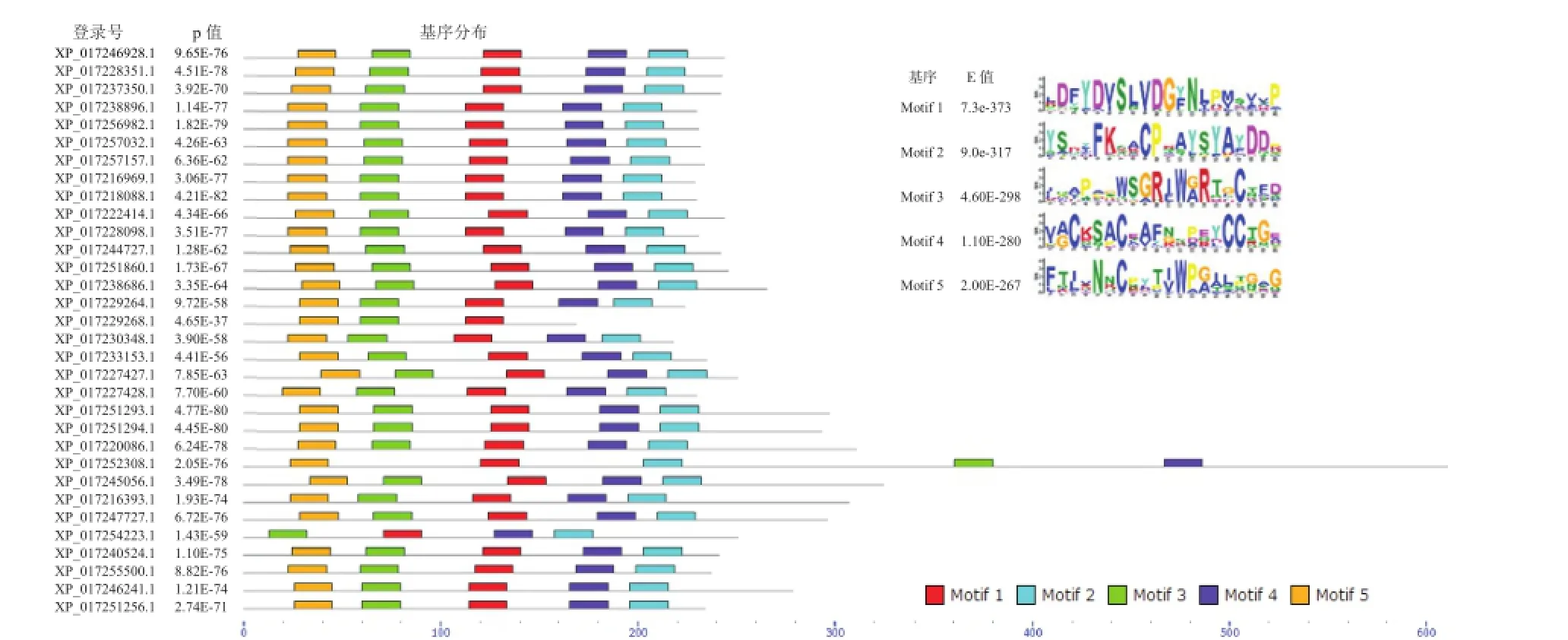
图2 胡萝卜TLP蛋白保守氨基酸基序分布

图3 蛋白XP_017233153.1三维结构预测
2.3胡萝卜TLP家族系统进化分析
为全面了解胡萝卜TLP与其他物种同源基因的进化关系,选取了10个拟南芥(Arabidopsis thaliana)TLPs、10个 水 稻(Oryza sativa)TLPs和10个毛果杨(Populus trichocarpa)TLPs与32个胡萝卜TLPs一起构建NJ系统进化树(图4),该进化树以稻瘟病菌(Magnaporthe grisea)TLP(NCBI登录号EAQ71431)作为外群。发现拟南芥、水稻和毛果杨的10个TLPs分别属于10个进化组(Shatters et al.,2006;Jami et al.,2007),胡萝卜TLPs在10个进化组中均有成员分布。其中进化组2成员最多,包含9个胡萝卜TLP成员,其次为进化组5中含有6个DcTLPs,该组的其他物种成员能对病原或环境胁迫作出响应,并具有抑菌活性(Leone et al.,2006;Liu et al.,2012),进化组5中的胡萝卜TLPs可能也具有抑菌活性。
2.4胡萝卜TLP家族基因染色体定位分析
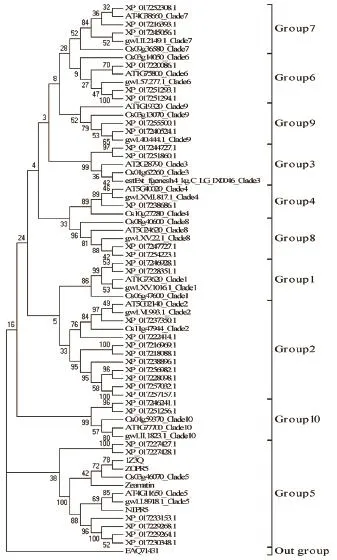
图4 胡萝卜TLP家族蛋白系统发育树
采用MapInspect软件绘制基因染色体定位图,发现胡萝卜TLP基因分布在9条染色体中的8条上(图5)。其中1号、4号和6号染色体上分布最多,均有5个TLP基因;其次为3号和5号染色体,有4个TLP基因;8号和9号染色体最少,只有2个TLP基因。分析发现,胡萝卜TLP基因在染色体上存在聚集现象,如在1号染色体的50 Mb、6号染色体的32 Mb、3号染色体的36 Mb、5号染色体的5 Mb、7号染色体的5 Mb、9号染色体的10 Mb位置都存在2个或2个以上的基因集中分布的现象。
3 结论与讨论
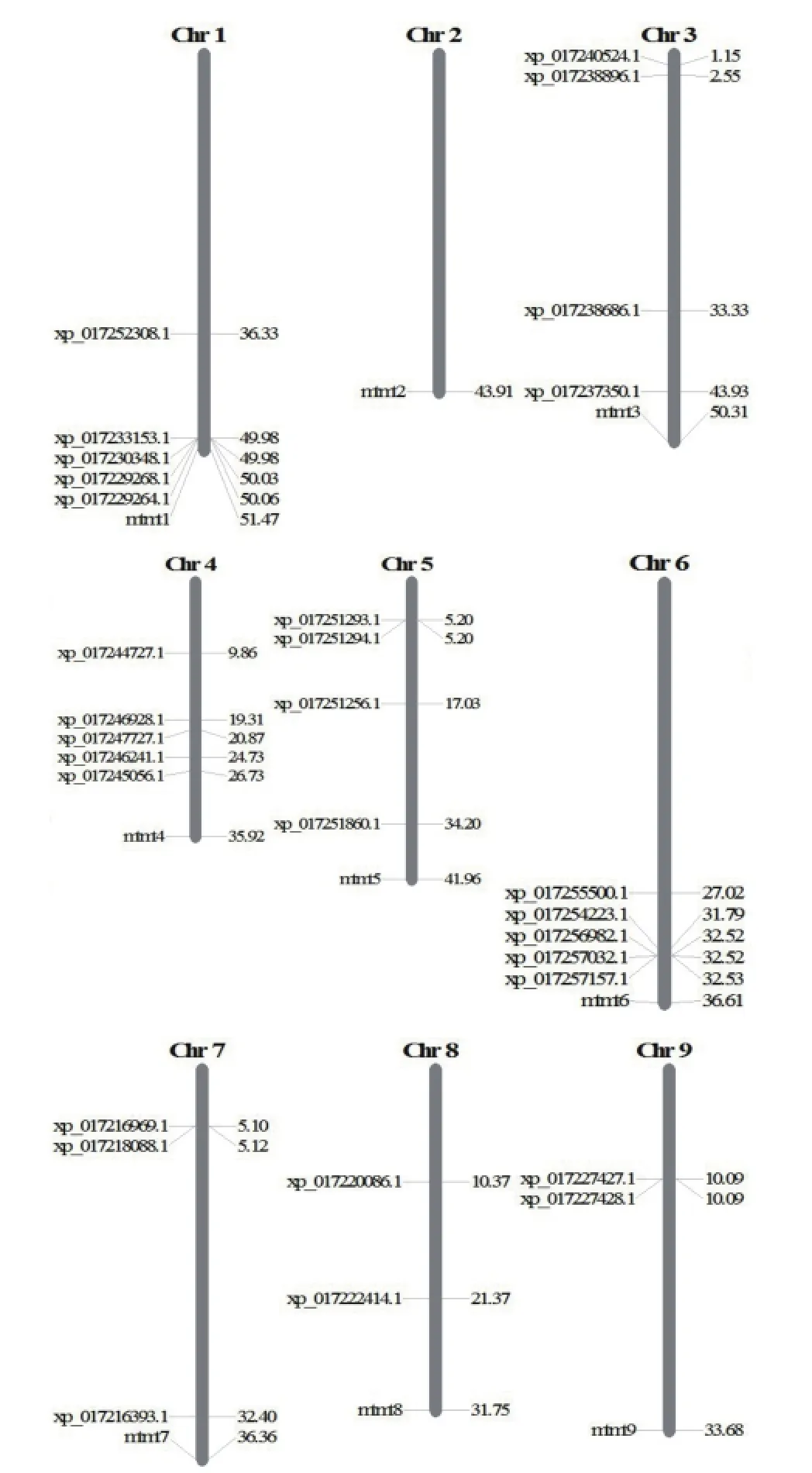
图5 胡萝卜TLP家族基因染色体定位
TLP转基因作物显著提高了其对病原真菌的抗性和对多种非生物胁迫的耐受性(Kalpana et al.,2006;Maruthasalam et al.,2007;Rout et al.,2016),因此,对植物TLP家族的研究受到越来越多的关注。本试验通过生物信息学方法共获得32条胡萝卜TLPs序列,均具有THN功能域(PF00314)。通过搜索Superfamily 1.75 database(http://supfam.org/SUPERFAMILY/hmm.html)发现,植物基因组编码TLP蛋白数量差异很大,如番木瓜(Carica papaya)编码TLP蛋白数量为18,黄瓜(Cucumis sativus)29,木薯(Manihot esculenta)33, 番 茄(Solanum lycopersicum)34,桃(Prunus persica)38,小果野蕉(Musa acuminata)41,粳稻(Oryza sativa ssp. japonica)46, 谷 子(Setaria italica)46, 杨 树(Populus trichocarpa)54, 柳 枝 稷(Panicum virgatum)65,玉米(Zea mays)67,面包小麦(Triticumaestivum)84,火炬松(Pinus taeda)87。蔬菜中编码的TLP蛋白数量相对较少,粮食作物编码数量较多,乔木火炬松中最多,这可能与植物所生存的胁迫环境有关,蔬菜作物一般种植面积相对较小,多为多样性种植,所面临的病理及环境胁迫压力较小;而粮食作物一般为大面积单一化种植,病原微生物种类复杂多样,很容易造成病害的大面积蔓延,面临的环境胁迫压力大,所以植物通过基因扩张产生更多的抗病相关基因以应对不良环境。虽然胡萝卜TLP家族基因数量不是很多,但其基因结构类型仍较丰富,说明该家族基因容易发生内含子获取或丢失,这与该家族基因功能的多样性是一致的。
比较基因组通过对已知的基因和基因组结构进行比较,来了解基因的功能、表达机理和物种进化,通过对不同种生物的基因组数据及其垂直进化、水平演化过程进行比较研究,可以了解基因的结构及其调控作用。系统发育分析显示,胡萝卜TLPs家族基因数目和系统发育与拟南芥、水稻和杨树类似(Liu et al.,2010;Zhao & Su,2010),主要分为10个进化组,说明单子叶植物和双子叶植物进化上发生分离前TLP家族已经形成了10个进化祖先(Shatters et al.,2006),后续的进化过程中该家族基因较保守。染色体定位分析发现,胡萝卜TLP家族多个基因存在集中分布现象,说明这些基因可能是通过基因复制进化而来的。各个进化组中TLP数量不一致,其中进化组2成员最多,成员数量次之的进化组5中含有6个TLPs,进化组5中的基因主要来自1号染色体,说明这些基因可能是染色体内复制的结果。Shatters等(2006)发现动物TLP蛋白可能是以单一祖先序列的形式来自于植物,而水稻和拟南芥TLP蛋白分布于多个支系,并存在染色体内和染色体间的复制,单子叶植物和双子叶植物进化上发生分离后,TLP基因在10个进化枝上发生了不对称的增加。本试验结果与Shatters等(2006)的研究结果一致,即胡萝卜的TLP蛋白也分布于多个支系,且可能存在染色体内和染色体间的复制现象。研究发现,进化组5中的其他物种TLPs能对病原或环境胁迫作出响应,如AT4G11650又称为ATOSM34,编码了拟南芥渗调蛋白,参与了拟南芥响应病原微生物和盐害胁迫的信号途径(Narasimhan et al.,2009),预示该组中胡萝卜TLPs很可能也具有类似功能,具体活性值得深入研究。
为了更好的了解胡萝卜TLP家族基因和蛋白的结构和功能,本试验通过生物信息学方法对胡萝卜TLP家族基因结构类型、基因染色体定位和系统进化进行详细分析。共获得32个胡萝卜TLP家族基因,其基因结构类型多样,且分布在多条染色体上,多个基因存在聚集现象,第5进化组中的成员具有抑菌潜力,值得深入研究。
Abad L R,D’Urzo M P,Liu D,Narasimhan M L,Reuveni M,Zhu J K,Niu X,Singh N K,Hasegawa P M,Bressan R A.1996.Antifungal activity of tobacco osmotin has specificity and involves plasma membrane permeabilization.Plant Science,118(1):11-23.
Bailey T L,Boden M,Buske F A,Frith M,Grant C E,Clementi L,Ren J,Li W W,Noble W S.2009.MEME SUITE:tools for motif discovery and searching.Nucleic Acids Research,w202-w208.
Beatrice C,Linthorst J M H,Cinzia F,Luca R.2016.Enhancement of PR1 and PR5 gene expressions by chitosan treatment in kiwifruit plants inoculated with Pseudomonas syringae pv.actinidiae.European Journal of Plant Pathology,doi:10.1007/s10658-016-1080-x.
Fils-Lycaon B R,Wiersma P A,Eastwell K C,Sautiere P.1996.A cherry protein and its gene,abundantly expressed in ripening fruit,have been identified as thaumatin-like.Plant Physiology,111:269-273.
Ho V S,Wong J H,Ng T B.2007.A thaumatin-like antifungal protein from the emperor banana.Peptides,28:760-766.
Iorizzo M,Ellison S,Senalik D,Zeng P,Satapoomin P,Huang J,Bowman M,Iovene M,Sanseverino W,Cavagnaro P,Yildiz M,Macko-Podgorni A,Moranska E,Grzebelus E,Grzebelus D,Ashrafi H,Zheng Z,Cheng S,Spooner D,van Deynze A,Simon P.2016.A high-quality carrot genome assembly provides new insights into carotenoid accumulation and asterid genome evolution.Nature Genetics.48(6):657-666.
Jami S K,Anuradha T S,Guruprasad L,Kirti P B.2007.Molecular,biochemical and structural characterization of osmotinlike protein from black nightshade(Solanum nigrum).Journal of Plant Physiology,164:238-252.
Kalpana K,Maruthasalam S,Rajesh T,Poovannan K,Kumar K K,Kokiladevi E,Raja J A J,Sudhakar D,Velazhahan R,Samiyappan R,Balasubramanian P.2006.Engineering sheath blight resistance in elite indica rice cultivars using genes encodingdefense proteins.Plant Science,170(2):203-215.
Kim S H,Lee J R,Kim S R.2003.Molecular characterization of a fruit-preferential thaumatin-like gene from apple(Malus domestica cv.Fuji).Journal of Plant Biology,46:52-58.
Leone P,Menu-Bouaouiche L,Peumans W J,Payan F,Barre A,Roussel A,van Damme E J M,Rougé P.2006.Resolution of the structure of the allergenic and antifungal banana fruit thaumatin-like protein at 1.7-A.Biochimie,88(1):45-52.
Liu D,He X,Li W,Chen C,Ge F.2012.Molecular cloning of a thaumatin-like protein gene from Pyrus pyrifolia and overexpression of this gene in tobacco increased resistance to pathogenic fungi.Plant Cell,Tissue and Organ Culture,111:29-39.
Liu J J,Sturrock R,Ekramoddoullah A K M.2010.The superfamily of thaumatin-like proteins:its origin,evolution,and expression towards biological function.Plant Cell Reports,29:419-436.
Maruthasalam S,Kalpana K,Kumar K K,Loganathan M,Poovannan K,Raja J A J,Kokiladevi E,Samiyappan R,Sudhakar D,Balasubramanian P.2007.Pyramiding transgenic resistance in elite indica rice cultivars against the sheath blight and bacterial blight.Plant Cell Reports,26(6):791-804.
Narasimhan M L,Bressan R A,D’Urzo M P,Jenks M A,Mengiste T.2009.Unexpected turns and twists in structure/function of PR-proteins that connect energy metabolism and immunity.Advances in Botanical Research,51:439-489.
Petre B,Major I,Rouhier N,Duplessis S.2011.Genome-wide analysis of eukaryote thaumatin-like proteins(TLPs)with an emphasis on poplar.Plant Biology,11:33.
Rout E,Nanda S,Joshi R K.2016.Molecular characterization and heterologous expression of a pathogen induced PR5 gene from garlic(Allium sativum L.)conferring enhanced resistance to necrotrophic fungi.European Journal of Plant Pathology,144(2):345-360.
Sharp P A.1981.Speculations on RNA splicing(minireview).Cell,23(643):621.
Shatters Jr R G,Boykin L M,Lapointe S L,Hunter W B,Weathersbee A A.2006.Phylogenetic and structural relationships of the PR5 gene family reveal an ancient multigene family conserved in plants and select animal taxa.Journal of Molecular Evolution,63(1):12-29.
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S.2011.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Molecular Biology and Evolution,28(10):2731-2739.
Thompson J D,Gibson T J,Plewniak F,Jeanmougin F,Higgins D G.1997.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools.Nucleic Acids Research,25(24):4876-4882.
van Loon L C,Rep M,Pieterse C M J.2006.Significance of inducible defense-related proteins in infected plants.Annual Review of Phytopathology,44:135-162.
Zhao J P,Su X H.2010.Patterns of molecular evolution and predicted function in thaumatin-like proteins of Populus trichocarpa.Planta,232(4):949-962.
Identification and Bioinformatics Analysis of Thaumatin-like Protein Family in Daucus carota
LIU Chao,HAN Li-hong,WANG Hai-bo,SONG Pei-bing,TANG Li-zhou*
(Center for Yunnan Plateau Biological Resources Protection and Utilization,College of Biological Resource and Food Engineering,Key Laboratory of Yunnan Province Universities of the Diversity and Ecological Adaptive Evolution for Animals and Plants on YunGui Plateau,Qujing Normal University,Qujing 655011,Yunnan,China)
This study analyzed and identified gene structure and phylogenesis of 32 carrot thaumatinlike proteins through genome database and bioinformatics method. The results showed that the carrot TLP genes distributed on eight out of nine chromosomes,the protein sequences were more conserved. The phylogeny analysis showed that carrot TLPs were classified into 10 evolutionary groups,most members of group fifth derived from chromosome one,which have potential antifungal activity.
Daucus carota;Thaumatin-like protein;Molecular evolution;Bioinformatics
刘潮,男,博士研究生,讲师,主要从事分子植物病理研究,E-mail:liuchao@mail.qjnu.edu.cn
*通讯作者(Corresponding author):唐利洲,博士,教授,主要从事动物遗传多样性研究,E-mail:tanglizhou@163.com
2016-11-17;接受日期:2017-01-12
国家自然科学基金项目(31460179),云南省高校云南特境内生菌资源的开发与利用科技创新团队项目

