一株海南野生灵芝的rDNA—ITS鉴定与系统发育分析
范平杰++姜芳燕++李珍++马红梅++陈永敢

摘 要:灵芝是一类重要的木腐真菌,在我国海南岛有广泛分布。研究扩增了1株分离自海南五指山的灵芝菌株rDNA-ITS序列,构建系统发育树,并比较了其与部分亲缘关系较近灵芝的rDNA-ITS序列差异。结果表明,分离自海南五指山的菌株Wzs1与G.resinaceum类群亲缘关系较近,支持聚类的自展值为26%,表明灵芝Wzs1与G.resinaceum类群较相近。
关键词:海南;灵芝;rDNA-ITS;系统发育
中图分类号 Q933 文献标识码 A 文章编号 1007-7731(2016)02-14-03
灵芝(Ganoderma spp.)是一种重要的腐生型真菌,隶属于真菌界(Kingdom Fungi),担子菌门(Basidiomycota),担子菌纲(Basidiomycetes),灵芝目(Ganodermatales),灵芝科(Ganodermataceae)[1]。野生灵芝的形态特征常常由于受到外部环境的影响产生很大的差异,有伞形、马蹄形等,菌盖有圆形、半圆形或肾形等,菌盖表面一般有光泽或环形纹理。海南岛的热带和亚热带雨林气候为野生灵芝生长发育提供了适宜的温度和湿度,丰富的原始森林为灵芝生长提供充足的营养成分。至今已发现野生灵芝科有103个种,分为4个属,3个亚属,其中海南有72个种,约占总数的70%,表明海南岛是我国野生灵芝分布最丰富的地区之一[2]。本研究利用rDNA-ITS引物,扩增1株灵芝菌株的序列,并构建系统发育树,研究菌株的分类学地位及亲缘关系,以便今后进一步的资源探索和菌株进化研究提供参考借鉴。
1 材料与方法
1.1 供试菌株分离、培养及DNA的提取 供试菌株Wzs1为灵芝子实体经酒精-次氯酸钠-酒精常规表面消毒后,置放于PDA培养基表面,于25℃恒温培养至长出菌落,纯化后4℃冷藏保存。菌种斜面上挑取菌丝接种至30mLGY液体培养基中,25℃摇床培养14d,收集菌丝用于DNA提取[3]。
1.2 PCR扩增及产物的克隆和测序 扩增片段为ITS1、5.8S和ITS2区域,引物为ITS1 (5-TCCGTAGGTGAACCTGCGG-3)和ITS4 (5-TCCTCCGCTTATTGATATGC-3) [3]。目的片段的扩增及克隆参照Chen等方法[4],将含有目的基因片段的菌液委托北京华大基因科技股份有限公司测序。
1.3 系统发育学分析及DNA序列差异性比较 根据菌株的rDNA-ITS测序结果,在GenBank数据库中进行Blast比对,搜索同源序列。选取不同种的代表性菌株作进一步的分析。采用Clustalx 1.83[5]将序列对齐,利用MEGA 5.0[6]软件进行系统发育分析,并以自展法(bootstrap)进行检测,循环1 000次,采用邻接法构建系统发育树,其中虎皮香菇(Lentinula tigrinus)作为外群。利用Clustalx 1.83[5]软件将实验菌株序列及部分亲缘关系较近的代表性菌株序列进行比对,找出不同序列间的碱基差异。
2 结果与分析
2.1 分离菌株的形态特征 在PDA培养基上25℃培养,从表面消毒的子实体边缘生长出白色菌落,具有典型的腐生真菌形态特征。菌落正面白色,棉质,质地紧密,中央隆起或稍有皱褶,边缘疏松,背面泛黄色或淡褐色。
2.2 DNA序列组成分析 灵芝菌株Wzs1进行了rDNA-ITS的扩增和测序。扩增产物包括ITS1-5.8S-ITS2区段以及部分18S、25S区段,其中菌株Wzs1扩增长度为674bp,ITS1、5.8S、ITS2序列长度分别为206bp、158bp、215bp,同时将测得的序列提交到GenBank(HQ689698)。
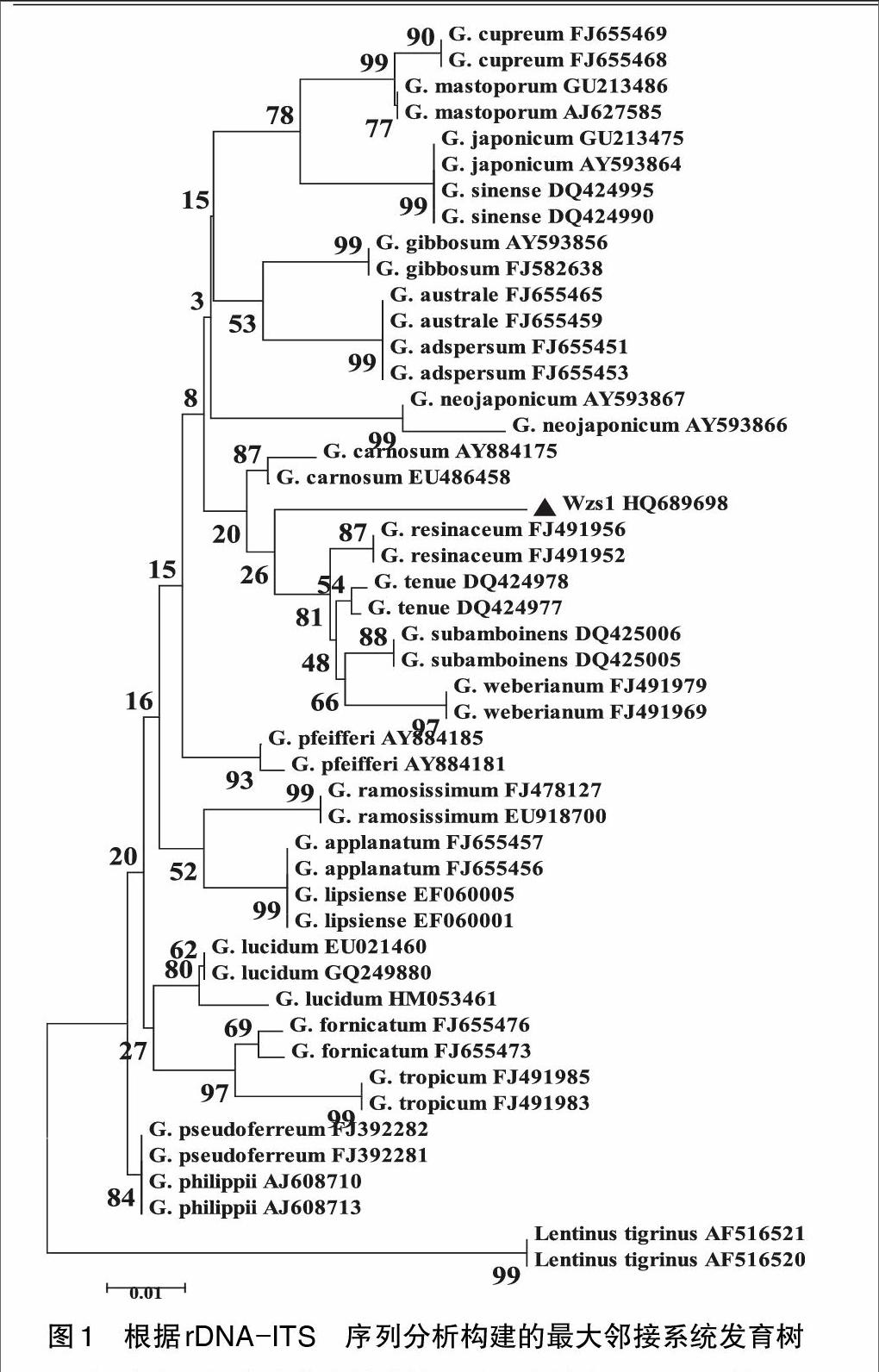
2.3 系统发育分析 利用MEGA5.0软件进行系统发育分析,构建NJ树branch length=0.391。系统发育树结构显示:灵芝菌株Wzs1与G.resinaceum类群聚为一支,自展值为26%;这一支包含了G.resinaceum、G.tenue、G.subamboinens和G.weberianum共4个种(图1)。此外,rDNA-ITS系统发育树显示灵芝不同种间亲缘关系的远近,同时G.japonicum与G.sinense这2个种的4株菌株聚为一支,与此相似的还有G.australe与G.adspersum,G.applanatum与G.lipsiense,G.pseudoferreum与G.philippii 6个种,其它各个种菌株各自聚为一支,这些结果反映了rDNA-ITS能够较好地对不同种菌株进行区分(图1)。
2.4 DNA序列差异分析 将属于G.resinaceum类群的9个菌株进行rDNA-ITS序列差异性比较,以序列的一端作为起始点,菌株Wzs1与其它8个菌株的差异主要分布在3个不同区域,分别位于90~140bp,160~200bp,420~460bp,这反映了菌株Wzs1与其它G.resinaceum类群菌株产生差异可能由这3个差异显著的片段决定(图2)。
3 讨论与结论
随着分子生物学技术的发展,灵芝真菌的形态分类容易受到外界环境的影响被越来越多的研究证明,形态数据难以准确区分不同种的菌株[3]。采用rDNA-ITS序列研究发现,中国栽培灵芝菌株中树舌亚属、紫芝组的菌株各自聚成一组,赤芝组的菌株分成3组,85.7%赤芝组菌株均聚于同一组,且树舌亚属、紫芝组和赤芝组间的遗传差异较大,赤芝组内存在一定的遗传差异,但多样性并不丰富[7-8]。菌株Wzs1与G.resinaceum类群聚为一支,说明这些灵芝遗传关系较为紧密。此外,Wzs1灵芝子实体也具有类似G.resinaceum类群4种灵芝的形态特征。因此,系统发育分析结果表明,菌株Wzs1可能与G.resinaceum类群较相近,但与G.resinaceum这个种有较明显的区别,并且序列差异比较分析也表明Wzs1与G.resinaceum类群其它菌株存在3个不同区域的差异,这也反映了菌株Wzs1仅与这一类群相近但有别于G.resinaceum的菌株。这些结果为进一步研究海南产灵芝菌株的系统发育学信息提供了参考借鉴。
参考文献
[1]Chang S T,Miles P G.Mushrooms cultivation,nutritional value,medicinal effect,and environmental impact[M].CRC Press,2004:357-372.
[2]吴兴亮,戴玉成.中国灵芝图鉴[M].北京:科学出版社,2005:1-14.
[3]陈永敢,陈光宙,袁学军,等.中国产Ganoderma lucidum的系统发育学分析及栽培技术研究[J].北方园艺,2014,08(1):92-95,96.
[4]Chen Y G,Ji Y L,Yu H S,et al.A new Neotyphodium species from Festuca parvigluma Steud.grown in China [J].Mycologia,2009,101(5):681-685.
[5]Thompson J D,Higgins D G,Gibson T J.Clustal W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice [J].Nucleic Acids Res,1994,22:4673-4680.
[6]Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28:2731-2739.
[7]苏春丽,唐传红,张劲松,等.基于rDNA-ITS序列探讨中国栽培灵芝菌株的亲缘关系[J].微生物学报,2007,47(1):11-16.
[8]Zheng L Y,Jia D H,Fei X F,et al.An assessment of the genetic diversity within Ganoderma strains with AFLP and ITS PCR-RFLP [J].Mycol Res.,2009,164:312-321.
(责编:张宏民)

