4株海洋红球菌产类胡萝卜素分析及其全基因组测序
张 帝,张 宁,陈吉刚,杨季芳*
(1.上海海洋大学 水产与生命学院, 上海 201306;2. 浙江万里学院 生物与环境学院, 浙江 宁波 315100)
4株海洋红球菌产类胡萝卜素分析及其全基因组测序
张 帝1,2,张 宁2,陈吉刚2,杨季芳*2
(1.上海海洋大学 水产与生命学院, 上海 201306;2. 浙江万里学院 生物与环境学院, 浙江 宁波 315100)
以源于东太平洋海水的4株红球菌分离菌Rhodococcussp. EPR-134、Rhodococcussp. EPR-147、Rhodococcussp. EPR-157和Rhodococcussp. EPR-279为研究对象,开展了菌株的色素提取和色素全波长扫描,并基于全基因组测序分析了4株细菌类胡萝卜素代谢通路中的相关基因。色素全波长扫描结果显示,菌株EPR-134不具备类胡萝卜素产生能力,而其它3株红球菌能够产生类胡萝卜素,且所产类胡萝卜素组分不同。基因组分析表明,4个细菌基因组中存在较为完整的促使类胡萝卜素形成的基因簇。对菌株参与番茄红素形成的3个关键基因crtE、crtB和crtI的氨基酸序列同源性两两比对分析表明,菌株EPR-134 3个基因氨基酸序列与其它3个菌株相应基因氨基酸序列同源性最低,这可能是导致该菌株不产类胡萝卜素的关键原因。该研究结果为产类胡萝卜素红球菌的遗传改造,以及为产类胡萝卜素工程菌的构建奠定了前期基础。
海洋红球菌;类胡萝卜素;基因组;代谢通路
0 引言
类胡萝卜素是一类在自然界中普遍存在的有机化学色素,属于四萜烯有机分子。类胡萝卜素具有体内清除自由基、抗氧化、抗癌等多重生理作用,在已有的600多种类胡萝卜素中有10多种已被多数国家批准使用于各个领域[1-3]。随着类胡萝卜素药用价值的不断发现,人类对类胡萝卜素的种类和产量需求将越来越大。然而,类胡萝卜素很难用化学方法合成,迄今为止,只有少数几种类胡萝卜素,如β胡萝卜素、虾青素、角黄素等能通过化学合成商业生产;通过天然微生物发酵途径,也能生产β胡萝卜素、虾青素,但由于其产量较低所占市场份额较低[4]。因此寻找胡萝卜素产量较高且其结构新颖的微生物,并通过分子生物学手段进一步提高菌株的类胡萝卜素产量尤为必要。
红球菌在海洋中广泛分布,其中大部分类群通过产生色素来抵抗海洋中的不良环境,其中广泛存在能够生产结构新颖的类胡萝卜的微生物类群。张加蓉 等[3]在北极海水中分离获得一株产胡萝卜素的红球菌Rhodococcossp. B7740。其后,ZHANG et al[5]对该菌株进行了全基因组测序,在其基因组中发现了参与类胡萝卜素合成的相关基因,这与此前赵文恩 等[6]报道类胡萝卜素合成过程中基因特性表现一致。继而,陈亚淑 等[7]采用液相色谱-质谱串联鉴定了B7740类胡萝卜素的结构特征,表明该菌株产生的类胡萝卜素类型为甲基萘醌类胡萝卜素。
近年来,依托多个航次的大洋科学考察,从深远海中分离获得了一些红球菌,通过菌落培养特征结合菌株的16S rDNA比较分析,筛选获得了亲缘关系较远,菌落颜色明显不同的4株红球菌(Rhodococcussp. EPR-134、Rhodococcussp. EPR-147、Rhodococcussp. EPR-157和Rhodococcussp. EPR-279)。本实验通过对4株红球菌色素的全波长扫描以及菌株的全基因组测序,分析4株细菌的产类胡萝卜素能力以及参与类胡萝卜的相关基因。研究结果将为后续产类胡萝卜素红球菌的遗传改造,以及为产类胡萝卜素工程菌的构建奠定前期基础。
1 材料与方法
1.1 材料
1.1.1 菌种信息
菌株Rhodococcussp. EPR-134、Rhodococcussp. EPR-147、Rhodococcussp. EPR-157和Rhodococcussp. EPR-279分离自中国大洋科学考察22航次采集的CTD海水样品,样品采集站位分别为-2 855 m(1°37.074′N,102°15.415′W)、-1 500 m(1° 44.951′N,102°17.552′W)、-2 855 m(1° 37.074′N,102° 15.415′W)和表层(5° 56.573′S,105° 41.102′W)。菌株保藏于宁波市微生物与环境工程重点实验室。
1.1.2 培养基
红球菌生长培养基配方为:牛肉膏7 g,酵母提取物5 g,葡萄糖5 g,乳糖3 g,加原位陈海水定容到1 L。固体培养基的琼脂含量为1.5%。
1.1.3 主要试剂
溶菌酶、RNase A和蛋白酶K购自北京全式金生物公司;DNA回收试剂盒、小剂量质粒提取试剂盒购自上海生工生物工程股份有限公司;DL2000,λ/HindIII DNA Marker购自Sigma公司;生化鉴定试剂购自青岛海博生物公司;主要生化试剂为国药或者进口分析纯。
1.2 实验方法
1.2.1 红球菌色素的提取及全波长扫描
红球菌色素的提取按照MAKUS et al[8]报道的方法进行。既在菌体中加入适量甲醇与丙酮(7∶3,V/V),避光水浴1~2 h并每间隔20 min轻轻震荡,离心收集上清液,超微量分光光度计Thermo Scientific NanoDrop 2000进行色素的全波长扫描。
1.2.2 基因组DNA的提取及测序
细菌基因组的提取采用CTAB法[9],即挑取红球菌单菌落接种于液体生长培养基,培养5~7 d至菌液适当混浊后离心收集菌体,菌体经溶菌酶消化和蛋白酶K与SDS裂解后加入CTAB/NaCl预热液,待蛋白充分变性后采用酚、氯仿混合液抽提细菌基因组DNA。提取的基因组DNA加入RNAse A去除RNA。基因组测序测定委托杭州迈英图生物科技有限公司完成,测序平台为Illumina Hiseq 2000。
1.2.3 基因组分析软件
通过RNAmmer 1.2软件[10]预测rRNA;通过tRNAscan-SE 1.23软件[11]预测tRNA区域;使用CRISPRsII软件[12]分析规律成簇间隔短回文重复序列;采用IslandViewer软件[13]预测基因组岛;采用RAST(http://rast.nmpdr.org/)在线数据库[14]对基因组进行注释;采用Clustal X2[15]、MEGA 5.0[16]进行基因比对分析。
2 结果与分析
2.1 色素全波长扫描分析
菌株EPR-134、EPR-147、EPR-157和EPR-279菌落颜色存在明显差异,其菌落颜色分别为无色、深黄色、浅黄色和深红色。为了分析4株菌株是否产生类胡萝卜素,以及分析它们产生的类胡萝卜素的组成是否存在差异,本实验通过全波长扫描对4株菌株色素提取物的吸收光谱进行了初步分析,结果如图1~图4所示。类胡萝卜素代谢过程中形成的多种中间产物具有特征性的吸收光谱,如八氢番茄红素、番茄红素、β-胡萝卜素和γ-胡萝卜素的最大吸收峰通常位于270~300 nm,445~500 nm、425~480 nm和435~490 nm之间,此外,β-胡萝卜素(无羟基)、β-隐黄质(单羟基)和玉米黄素(双羟基)实际上具有相同λmax(在乙醇中分别为428 nm、450 nm和476~478 nm)[17]。
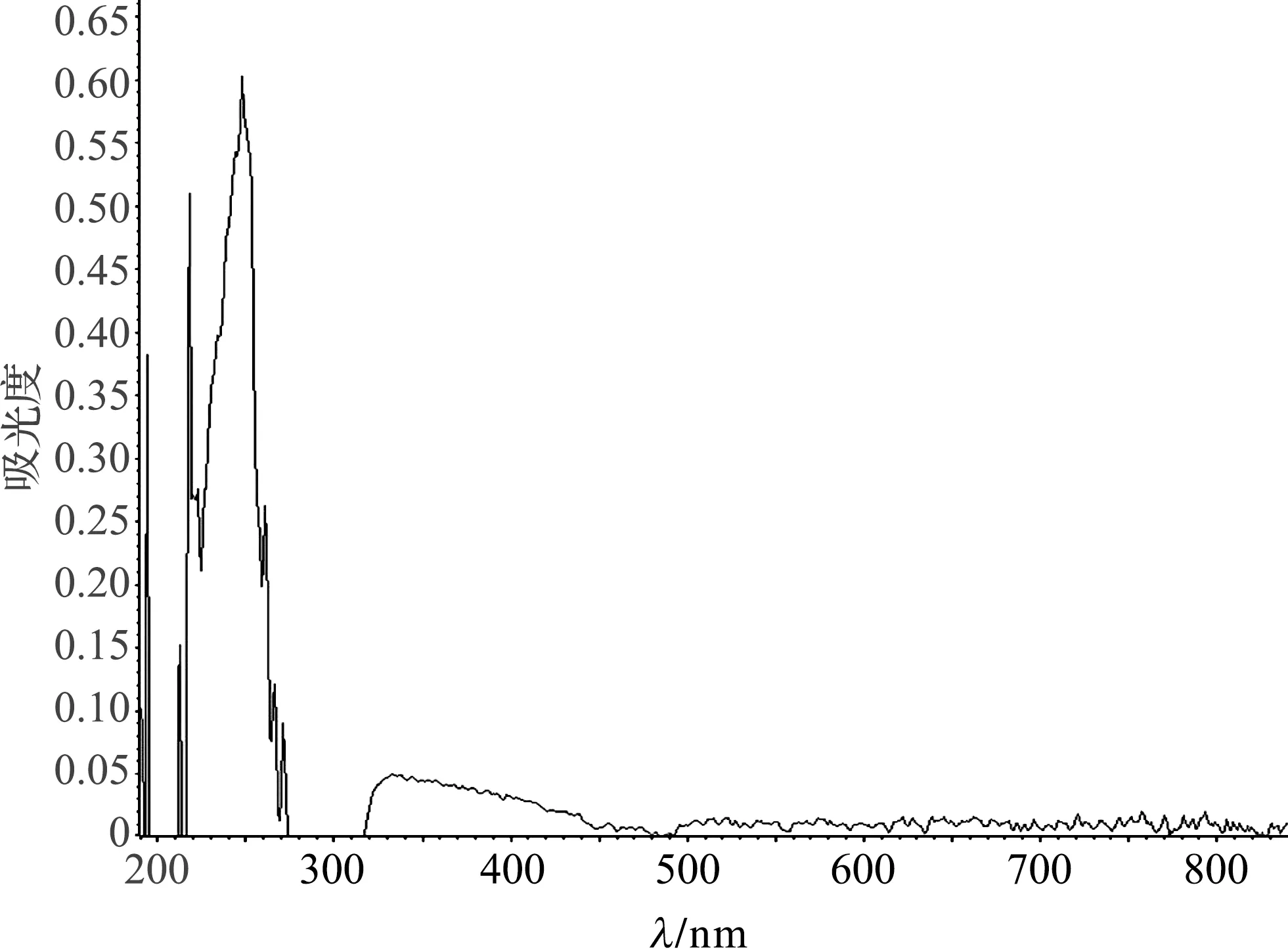
图1 红球菌Rhodococcus sp. EPR-134色素全波长扫描结果Fig.1 Full wavelength scanning results of pigments from Rhodococcus sp. EPR-134
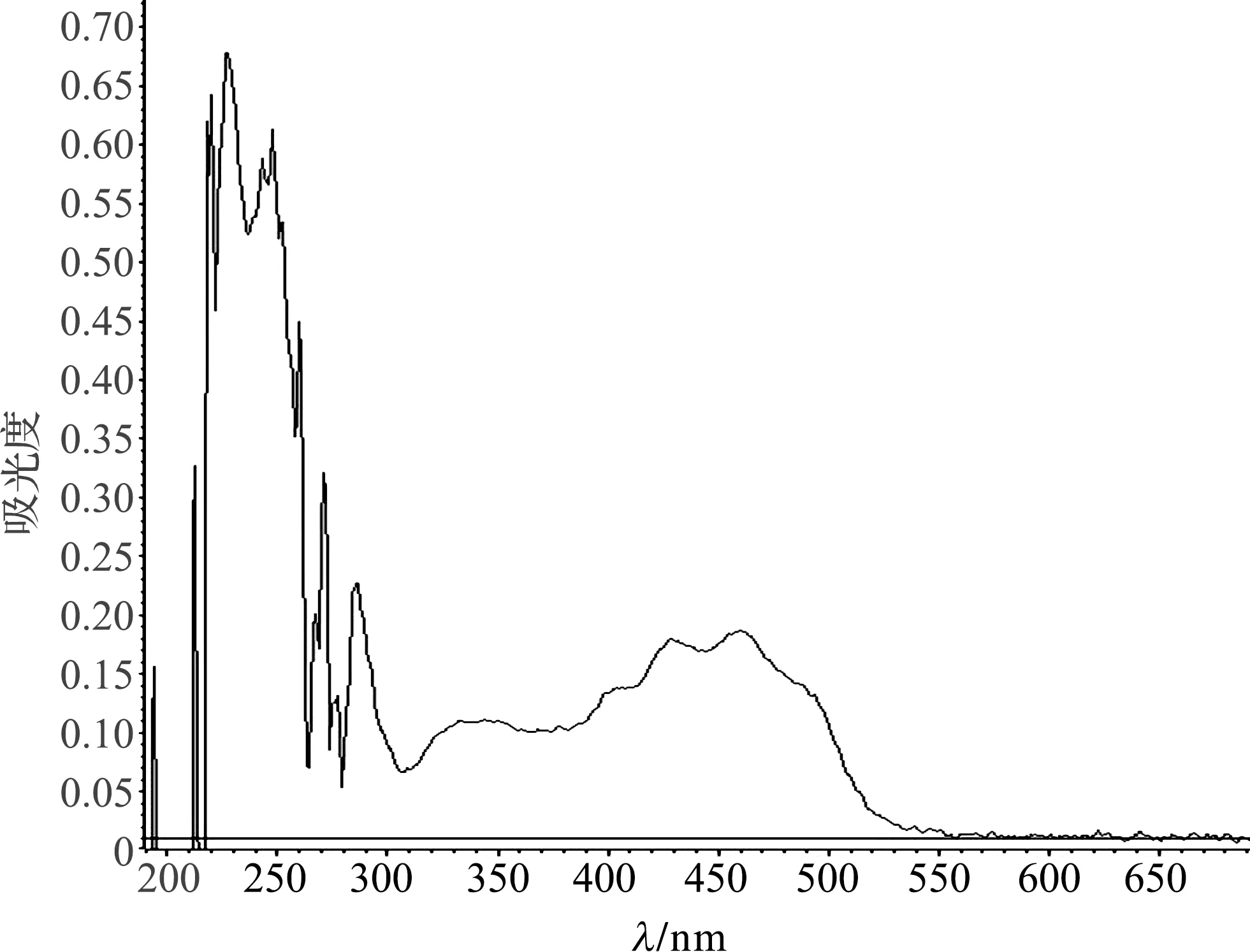
图2 红球菌Rhodococcus sp. EPR-147色素全波长扫描结果Fig.2 Full wavelength scanning results of pigments from Rhodococcus sp. EPR-147
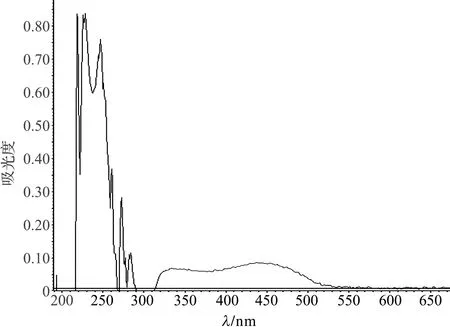
图3 红球菌Rhodococcus sp. EPR-157色素全波长扫描结果Fig.3 Full wavelength scanning results of pigments from Rhodococcus sp. EPR-157
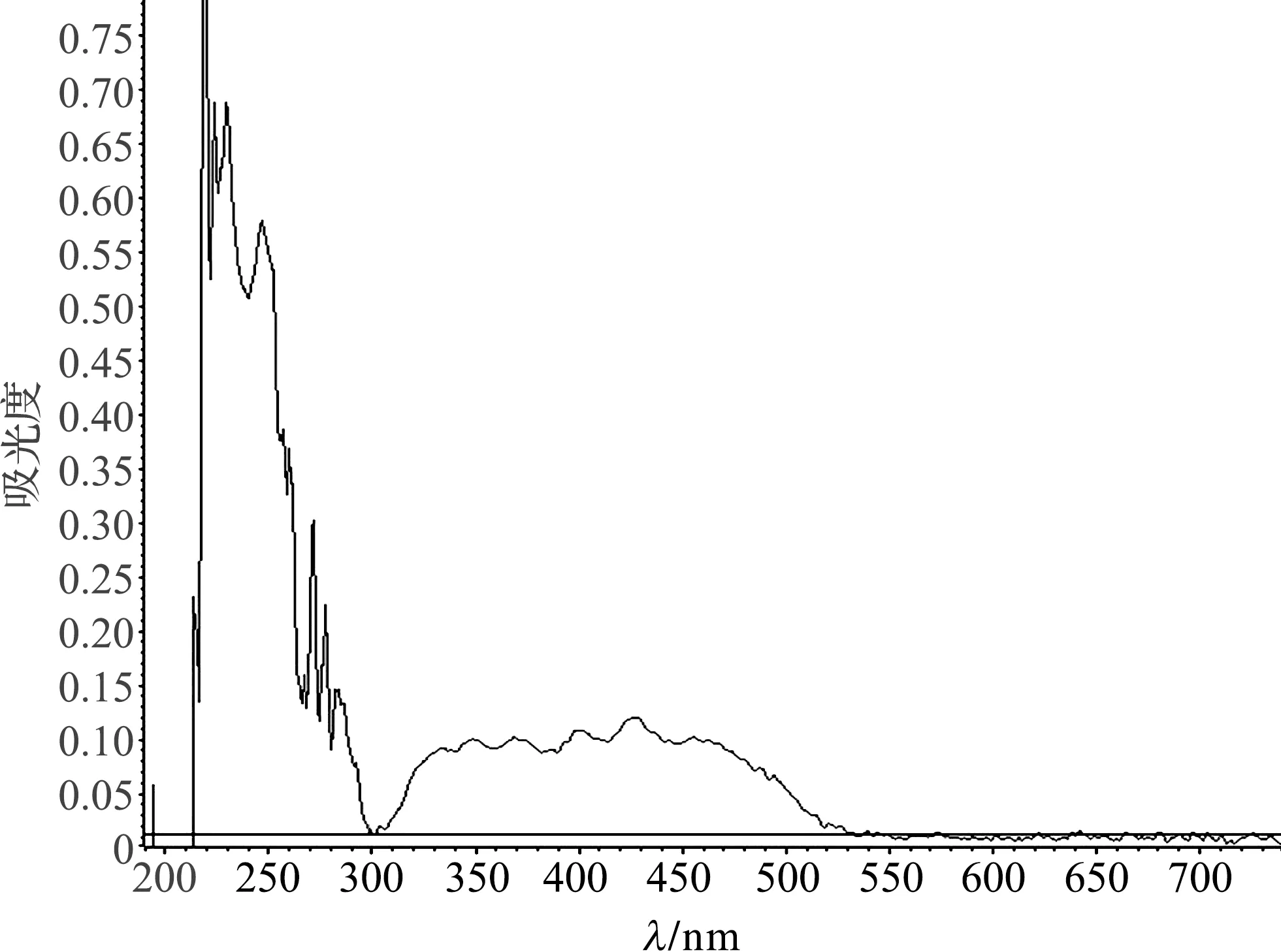
图4 红球菌Rhodococcus sp. EPR-279色素全波长扫描结果Fig.4 Full wavelength scanning results of pigments from Rhodococcus sp. EPR-279
对4株红球菌色素的全波长扫描结果分析发现,除了菌株EPR-134色素在250~300 nm范围内不存在明显吸收峰外,其余3株菌色素在该波长范围内均存在比较明显的吸收峰。此外,除了菌株EPR-134色素在450~500 nm波长范围左右未检测出较大的吸光度外,其余3株菌色素均检测到明显的吸收度高值。在450~500 nm范围左右,EPR-147、EPR-157和EPR-279 3个菌株在不同波长范围的吸光度存在明显差异,其吸光度较大的波长主要停留在430和470 nm、450 nm以及400、430和460 nm。
2.2 基因组基本特征分析
EPR-134、EPR-147、EPR-157和EPR-279 4株细菌的基因组草图的DDBJ/ENA/GenBank数据库收录号分别为LVCT00000000、LVCU00000000、LVCV00000000和LVCW00000000。对4株菌的基因组草图基本特征分析表明(表1),4株细菌的基因组草图含有的contigs数量为626~894个,含有的scaffolds数量为56~94个;GC含量为62.39%~64.83%;4个基因组编码蛋白的基因数量为5 464~7 251个,其中5 132~6 782个编码的蛋白具有已知功能;4株细菌中,菌株EPR-134基因组含有的编码蛋白及已知功能的蛋白数量最多,其次为菌株EPR-157;4个基因组含有tRNA、5s rRNA、16s rRNA、23s rRNA和sRNA数量基本相同。
2.3 参与胡萝卜素通路的基因分析
目前对原核微生物的类胡萝卜素的代谢通路研究得已比较透彻。原核生物的类胡萝卜素代谢通路起始于前体物质IPP,继而通过中间产物的异构化、缩合和脱氢以及系列酶的催化,最终形成结构较为复杂的类胡萝卜素[4]。本实验通过对4株红球菌的基因组进行分析注释及类胡萝卜素代谢通路分析,发现了参与产类胡萝卜素形成的相关基因,这些基因包括八氢番茄红素合成酶(phytoene synthase,crtB)、八氢番茄红素脱氢酶(phytoene dehydrogenase,crtI)、八氢番茄红素脱氢酶相关蛋白(phytoene dehydrogenase and related protein)、牻牛儿基牻牛儿基焦磷酸合成酶(geranylgeranyl pyrophosphate synthetase,crtE)、β-胡萝卜素环化酶(Beta-carotene ketolase)、番茄红素β环化酶(lycopene beta cyclase)和Phi-类胡萝卜素合成酶(phi-carotenoid synthase),其中Phi-类胡萝卜素合成酶在菌株EPR-134基因组中缺失,而番茄红素β环化酶在菌株EPR-157基因组中缺失。
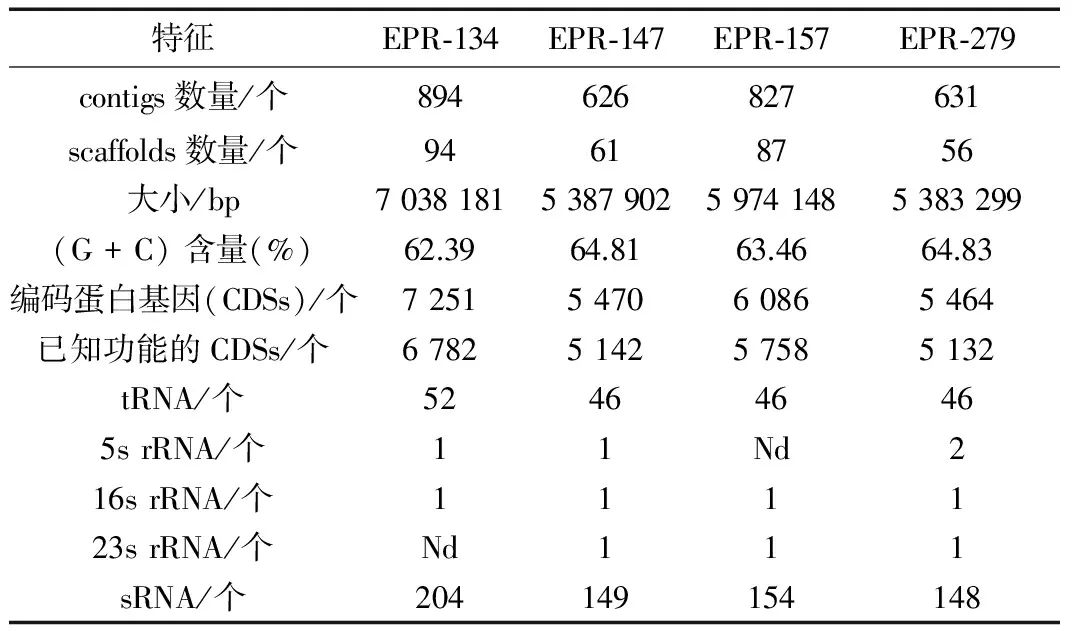
表1 Rhodococcus sp. EPR-134,Rhodococcus sp. EPR-147,Rhodococcus sp. EPR-157和Rhodococcus sp. EPR-279细菌基因组基本特征
注:Nd为未发现,可能由于草图测序过程中gap所造成
2.4 类胡萝卜素代谢通路关键基因crt E,crt B和crt I比较分析
原核微生物类胡萝卜素代谢通路存在3个关键基因:八氢番茄红素合成酶(phytoene synthase,crtB),八氢番茄红素脱氢酶(phytoene dehydrogenase,crtI),牻牛儿基牻牛儿基焦磷酸合成酶(geranylgeranyl pyrophosphate synthetase,crtE),这3个基因属于类异戊二烯类物质合成酶蛋白家族。crtE主要催化GGPP的合成,crtB主要催化两个GGPP缩合形成八氢番茄红素,crtI主要催化八氢番茄红素进一步脱氢形成番茄红素。
对菌株间的crtE、crtB和crtI3个基因的氨基酸序列两两比较发现(表2),菌株EPR-147 3个基因与EPR-279 3个相应基因氨基酸同源性均最高(100%),而 EPR-134 3个基因与另3个菌株的相应基因同源性均较低,其中EPR-134crtB、crtI和crtE分别与菌株EPR-157、EPR-147 和EPR-279 相应基因氨基酸同源性最低,其同源性分别为49.86%、24.34%和59.37%。

表2 Rhodococcus sp. EPR-134,Rhodococcus sp. EPR-147,Rhodococcus sp. EPR-157和Rhodococcus sp. EPR-279 4 株细菌crt E、crt B和crt I 3个基因氨基酸序列比对
3 讨论
本研究涉及的4株红球菌EPR-134、EPR-147、EPR-157和EPR-279分离自东太平洋海水,其中菌株EPR-279分离自表层海水,而后3株均分离自深层海水。4株菌株菌落颜色存在明显差异,其颜色与菌株来源的海水深度具有一定的联系,即样品来源越接近表层其菌落的颜色就越深,这可能因为位于海水表层的红球菌受到环境因子,尤其是紫外线的胁迫较深层严重,生长在表层海水中的红球菌通过产生色素来抵抗不利环境。通过颜色的差异,初步推断这4株菌株在产类胡萝卜素方面也可能存在差异。对4株菌株的全波长扫描结果初步证实了这一假设。菌落无色的菌株EPR-134不具有八氢番茄红素特征性吸收峰。相比另外3株菌,菌株EPR-134的色素中也未检测到番茄红素、β-胡萝卜素和γ-胡萝卜素等高等类胡萝卜素所具有的典型峰值。实验结果说明菌株EPR-134不具备产生类胡萝卜素的能力。全波长扫描结果发现菌株EPR-147、EPR-157和EPR-279虽然在450~500 nm范围左右均有吸光度高值的出现,但是3个菌株无论在吸光度的范围还是吸光度值的大小上均存在差异,这说明这3株细菌产生的类胡萝卜素的组成存在差异。
基于产类胡萝卜素菌株一定存在参与类胡萝卜素形成的相关基因的这一事实,我们对4株细菌进行了全基因组测定,寻找参与类胡萝卜相关基因的差异。测序结果表明,除了菌株EPR-134缺失Phi-类胡萝卜素合成酶以及EPR-157缺失番茄红素β环化酶(这可能是由于基因组草图的gap导致),其余参与类胡萝卜素形成的相关基因均能在4个菌株基因组中找到。尤其值得一提的是,参与番茄红素形成的3个基因crtE、crtB和crtI在不产类胡萝卜素的菌株EPR-134中也存在。进一步的序列比对发现,EPR-134中的crtE、crtB和crtI3个基因的氨基酸序列与其它菌株相应基因氨基酸序列的同源性较低,且该同源序列存在于相应基因氨基酸序列的高度保守区,这可能导致了该菌株的3个酶功能丧失,从而使得该菌株不产八氢番茄红素和番茄红素。EPR-134crtE、crtB、crtI及其他类胡萝卜素形成相关基因的存在是否说明该菌曾经能够产类胡萝卜素,而在其进化过程中由于生存环境的改变导致了类胡萝卜素产生能力的丧失,目前尚不得而知。
本研究基于全波长扫描确定了3株海洋红球菌能够产类胡萝卜素,通过全基因组测序找到了类胡萝卜素代谢通路中的关键基因,但有关这些菌株产生的类胡萝卜素的组成及结构还需依赖更加精确的纯化分析,开展类胡萝卜素结构特征与构效研究,在此基础上,进一步改造或调控红球菌的类胡萝卜素代谢途径,构建工程菌,以期提高目标类胡萝卜素的产量。
[1] GAMMONE M A, RICCIONI G, D’ORAZIO N. Marine Carotenoids against oxidative stress: Effects on human health[J]. Mar Drugs,2015,13(10):6 226-6 246.
[2] MIYASHITA K. Function of marine carotenoids[J]. Forum Nutrition,2009(61):136-146.
[3] ZHANG Jia-rong, YANG Ji-fang, CHEN Fu-sheng. Optimization of growing conditions and a method for the extraction of crude carotenoids produced byRhodococcussp.(strain B7740) isolated from the Arctic sea[J]. Chinese Journal of Polar Research,2012,24(2):168-177. 张加蓉,杨季芳,陈福生.一株分离自北极海水红球菌(Rhodococcossp.)B7740的生长条件优化及其粗类胡萝卜素提取方法的优化[J].极地研究,2012,24(2):168-177.
[4] TAO Jun, ZHANG Shang-long, XU Chang-jie, et al. Gene and gene engineering of carotenoid biosynthesis[J]. Chinese Journal of Biotechnology,2002,18(3):276-281. 陶俊,张上隆,徐昌杰,等.类胡萝卜素合成的相关基因及其基因工程[J].生物工程学报,2002,18(3):276-281.
[5] ZHANG Di, LI Li, ZHU Si-dong, et al. Complete genome sequence ofRhodococcussp. B7740, a Carotenoid producing bacterium isolated from the Arctic Sea[J]. Genome Announc,2015,3(2):.DOI:10.1128/genomeA.00333-15.
[6] ZHAO Wen-en, LI Yan-jie, CUI Yan-hong, et al. Carotenoid biosynthetic pathway and its control, and genetic manipulation[J]. Acta Bot Boreal-Occident Sin,2004,24(5):930-942. 赵文恩,李艳杰,崔艳红,等.类胡萝卜素生物合成途径及其控制与遗传操作[J].西北植物学报,2004,24(5):930-942.
[7] CHEN Ya-shu, WANG Rong, XIE Bi-jun, et al. Optimization of extraction of carotenoids and identification of menaquinone carotenoids fromRhodococcussp. B7740[J]. Food Science,2016,37(2):25-30. 陈亚淑,汪荣,谢笔钧,等.北极海洋红球菌(Rhodococcussp.)B7740产类胡萝卜素的提取条件优化及甲基萘醌类类胡萝卜素鉴定[J].食品科学,2016,37(2):25-30.
[8] MARKUS D, GREENWOOD M C, FOREMAN C M. Carotenoid pigmentation in Antarctic heterotrophic bacteria as a strategy to withstand environmental stresses[J]. Arctic, Antarctic and Alpine Research,2010,42(4):396-405.
[9] PANG Jian, LIU Zhan-Ying, HAO Min, et al. Comparison and optimization of methods for genomic DNA extraction from Gram positive bacteria[J]. Microbiology China,2015,42(12):2 482-2 486. 庞建,刘占英,郝敏,等.革兰氏阳性细菌基因组DNA提取方法的比较及优化[J].微生物学通报,2015,42(12):2 482-2 486.
[10] LAGESEN K, HALLIN P, RODLAND E A, et al. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Res,2007,35(9):3 100-3 108.
[11] GARDNER P P, FASOLD M, BURGE S W, et al. Conservation and losses of non-coding RNAs in avian genomes[J]. PLOS One, 2015,10(3):e0121797. DOI: 10.1371/journal.pone.0121797.
[12] RISSA I, VERGNAUD G, POURCEL C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007, 8:172.DOI:10.1186/1471-2105-8-172.
[13] LANGILLE M G, BRINKMAN F S. IslandViewer: an integrated interface for computational identification and visualization of genomic islands[J]. Bioinformatics,2009,25(5):664-665.
[14] MCCALL C A. Metagenomic comparison of antibiotic resistance genes associated with liquid and dewatered biosolids[J]. J Environ Qual,2016,45(2):463-470.DOI:10.2134/jeq2015.05.0255.
[15] CHENG Y, GUINDON S, RODRIGO A, et al. Cumulative viral evolutionary changes in chronic hepatitis B virus infection precedes hepatitis B e antigen seroconversion[J]. GUT,2013,62(9):1 347-1 355.DOI:10.1136/gutjnl-2012-302408.
[16] ZHAO Y, ZHANG H, ZHAO J, et al. Evolution of infectious bronchitis virus in China over the past two decades[J]. J Gen Virol, 2016,97(7):1 566-1 574.DOI:10.1099/jgv.0.000464.Epub 2016 Mar 23.
[17] WANG Hai-bin. UV-VIS characteristics of carotenodis and its utilization in structural identification[J].Journal of Wuhan Polytechnic University,2004,23(4):10-13. 王海滨.类胡萝卜素的紫外可见光谱特性及其应用[J].武汉工业学院学报,2004,23(4):10-13.
Analysis on caroteniod producing of four marine Rhodococcus and its genome sequence determining
ZHANG Di1,2, ZHANG Ning2, CHEN Ji-gang2, YANG Ji-fang*2
(1.CollegeofFisheriesandLifeSciences,ShanghaiOceanUniversity,Shanghai201306,China;2.FacultyofBiological&EnvironmentalScience,ZhejiangWanliUniversity,Ningbo315100,China)
Rhodococcussp. EPR-134,Rhodococcussp. EPR-147,Rhodococcussp. EPR-157 andRhodococcussp. EPR-279 were isolated from water samples of East Pacific Ocean, extraction of pigment and pigment full wavelength scan were carried out in this study. Based on the whole genome sequencing, the related genes in carotenoid metabolic pathways were analyzed. Pigment full wavelength scan results show that strain EPR-134 does not have a kind of caroteniod production ability, while other 3 strains ofRhodococcuscan produce carotenoids, and the carotenoid composition was different. Genome analysis show that there are more complete gene clusters in the 4 bacterial genomes.crtEgene、crtBgene andcrtIgene are very important in the metabolic pathway of caroteniod producing, and by analyzing amino acid sequence homology of these genes in different strains, it is find that amino acid sequences of these genes in the strain EPR-134 share the lowest sequence homology compared with other 3 strains. This may be the key reason why strain EPR-134 can’t produce carotenoid. The results of this study may lay a preliminary foundation for genetic modification of the carotenoid producingRhodococcusand construction of carotenoids producing engineering bacteria.
marineRhodococcus; carotenoid; genome; metabolic pathway
10.3969/j.issn.1001-909X.2016.04.010.
2016-04-25
2016-05-19
国家海洋公益性行业科研专项项目资助(201405015-5);宁波市科技公关重大项目资助(2012C10038);浙江省重中之重学科学生创新计划项目资助(CX2015007)
张帝(1991-),男,湖北仙桃市人,主要从事海洋资源微生物方面的研究。E-mail:396358077@qq.com
*通讯作者:杨季芳(1963-),男,研究员,主要从事海洋微生物生态过程、海洋微生物资源与环境研究。E-mail:jfkwlq@163.com
Q93;Q78
A
1001-909X(2016)04-0078-06
10.3969/j.issn.1001-909X.2016.04.010
张帝,张宁,陈吉刚,等.4株海洋红球菌产类胡萝卜素分析及其全基因组测序[J].海洋学研究,2016,34(4):78-83,
ZHANG Di, ZHANG Ning, CHEN Ji-gang, et al. Analysis on caroteniod producing of four marineRhodococcusand its genome sequence determining[J]. Journal of Marine Sciences, 2016,34(4):78-83, doi:10.3969/j.issn.1001-909X.2016.04.010.

