基于质谱技术应用CLINPROT系统建立乳腺癌诊断模型*
杨 帆,陈英剑,胡成进△
(1.锦州医科大学研究生院,辽宁锦州 121001;2.济南军区总医院实验诊断科,济南 250031)
·论 著·
基于质谱技术应用CLINPROT系统建立乳腺癌诊断模型*
杨 帆1,陈英剑2,胡成进2△
(1.锦州医科大学研究生院,辽宁锦州 121001;2.济南军区总医院实验诊断科,济南 250031)
目的 应用液体蛋白芯片-飞行时间质谱技术(CLINPROT)系统筛选乳腺肿瘤患者血清中潜在的肿瘤标志物,并基于差异蛋白建立乳腺癌诊断预测模型。方法 收集病理确诊的乳腺癌患者血清标本43例及健康女性血清标本24例,并随机分为建模组和验证组。应用ClinProt Tools系统对建模组中的乳腺癌患者和健康女性血清进行差异蛋白的筛选,建立差异蛋白质图谱,应用ClinProTools v3.0 软件建立乳腺癌诊断预测模型,而后利用该诊断模型对验证组血清标本进行分组验证,初步评估该模型的诊断效能。结果 通过分析病例组和对照组质谱峰信息,得到24个具有明显差异的蛋白峰(P<0.05)。根据遗传算法选取表达差异最显著的3个蛋白质建立乳腺癌诊断预测模型,该模型对建模组乳腺癌的识别能力为91.67%。后利用验证组标本对该模型的诊断效能进行评估,结果显示其对验证组乳腺癌识别准确率为85.7%。 结论 利用ClinProt Tools v3.0软件成功建立并评估了乳腺癌诊断预测模型,为乳腺癌诊断提供了新的途径。
液体蛋白芯片-飞行时间质谱技术; 乳腺癌; 诊断模型
液体蛋白芯片-飞行时间质谱技术(CLINPROT)系统是由德国布鲁克道尔顿公司基于质谱开发的蛋白质组学分析和鉴定系统。该系统主要由4个子系统组成:磁珠富集低丰度蛋白质/多肽、质谱采集、CLINPROT软件和体液样品自动处理系统。基本过程:将患者或健康对照的临床样品通过磁珠进行分离,去除样品中的高丰度蛋白和杂质,将分离后的蛋白质/多肽样本与基质混合后,直接点在抛光靶上,通过MALDI TOF/TOF MS进行分析,得到两组蛋白的总质谱图,通过CLINPROT系统比较病例组与对照组表达差异的蛋白,生成平均质谱图、胶状图等,用于临床未知样品的预测[1]。目前,多项研究均利用了CLINPROT系统对肿瘤标志物进行了前瞻性研究,包括宫颈鳞状细胞癌、食管鳞状细胞癌、胃癌、口腔鳞状细胞癌等[2-5]。CLINPROT系统作为蛋白质组学研究的新技术,为发现新型肿瘤标志物提供了新的途径。本研究应用该技术对乳腺癌患者血清蛋白进行了初步探讨。
1 资料与方法
1.1 一般资料 收集济南军区总医院甲状腺乳腺外科2015 年 1 月至2016 年 1 月收治的乳腺癌女性患者血清标本43例作为病例组,平均年龄(45.0±3.4) 岁。所有标本均经术后病理学确诊,术前均未进行临床治疗。另选济南军区总医院同期健康体检的女性血清24例作为对照组,纳入对照女性既往无乳腺癌及相关疾病,体检未见其他恶性疾病,平均年龄(43.0±5.2)岁。病例组与对照组年龄等一般资料差异无统计学意义(P>0.05)。
1.2 试剂与仪器 弱阳离子交换磁珠分析试剂盒(MB-WCX)、疏水磁珠分析试剂盒(MB-HIC C8)、金属螯合磁珠分析试剂盒(MB-IMAC-Cu)和标准蛋白/多肽购自德国 Bruker公司。三氟乙酸(TFA)、乙腈、丙酮购自Sigma-Aldrich公司。血清蛋白质谱图的采集采用德国Bruker公司基质辅助激光解析串联飞行时间/飞行时间质谱仪,并使用仪器原装进口配套的试剂和定标品。
1.3 方法
1.3.1 标本采集和预处理 受检对象均进行清晨空腹采血,采集静脉血5 mL,3 000 r/min离心5 min分离血清,置于-80 ℃冰箱保存。
1.3.2 3种磁珠分析试剂盒富集血清低丰度蛋白 分别采用不同分离原理的磁珠试剂盒MB-WCX、MB-HIC C8、MB-IMAC-Cu提纯4例乳腺癌患者和4例健康对照女性血清中的低丰度蛋白,根据平均出峰量、信噪比、峰强度等系数比较3种分离试剂盒的提纯效果。
1.3.3 CLINPROT系统重复性检测 将已富集的4例乳腺癌患者和4例健康对照女性血清中的低丰度蛋白经MALDI-TOF/TOF MS连续检测5次,分别获得不同批间质谱图。再应用ClinProt Tools v3.0软件分析采集质谱图,计算每批质谱图的变异系数(CV),通过比较批间变异系数变化评价本方法的重复性。
1.3.4 MB-WCX富集血清低丰度蛋白 根据磁珠分选结果,采用弱阳离子交换磁珠分析试剂盒处理43例乳腺癌和24例健康女性血清标本。(1) 磁珠吸附标本。将10 μL MB-WCX磁珠、10 μL结合缓冲液(BB)和5 μL 待测血清加入样品管,颠倒混匀;室温静置 5 min;利用磁性分离器分离磁珠1 min,使磁珠与上清液彻底分离;用加样枪吸出上清液,应避免枪头接触和吸走磁珠;(2)洗涤磁珠。加入100 μL 洗涤缓冲液(WB);在磁性分离器相邻位置反复移动,彻底分离上清液;(3)洗脱磁珠。将分离后的上清液中加入5 μL洗脱缓冲液(EB)混匀,将样品管置于磁珠分离器中静置2 min,分离上清液,最后加入5 μL稳定缓冲液(SB),充分混匀后,24 h内利用MALDI TOF/TOF MS进行分析。
1.3.5 样品上样分析 稀释1 μL多肽样品溶液于10 μL混合基质溶液中,充分混匀;加1 μL混合液于MTP 384 target plate polished steel BC靶板的相应位置上,室温风干后进行质谱分析,采集每例血清标本低丰度蛋白表达质谱图。
1.3.6 生物信息学及数据统计学分析 (1)FlexAnalysis软件数据分析。在FlexAnalysis软件中打开待分析数据,编辑找峰参数,通过平滑、基线处理、校正等方法对数据进行处理后,进行标峰,并检查已采集图谱质量。(2)ClinProt Tools v3.0软件建立乳腺癌诊断模型。打开ClinProt Tools v3.0软件,设置一般参数并对数据进行平滑、对齐、归一化处理,然后将模型组(36例乳腺癌患者和17例健康女性标本)质谱图调入软件,对所有峰进行统计分析,同时生成峰统计列表和准胶图,在对每张谱图中的峰高和峰面积进行标注。最后利用对峰参数的统计结果,根据遗传算法建立乳腺癌诊断预测模型。(3)乳腺癌诊断模型的验证。利用ClinProt v3.0软件用建立的乳腺癌诊断模型对验证组标本(7例乳腺癌和7例健康对照)进行双盲验证,检测诊断模型的诊断价值。
2 结 果
2.1 磁珠分选结果 本研究首先对3种磁珠分离蛋白质/多肽方法MB-WCX、MB-HIC C8、MB-IMAC-Cu进行了比较,以选择最佳的富集方法进行后续的大样本试验。利用3种磁珠富集4例乳腺癌患者和4例健康对照女性血清中的低丰度蛋白,然后通过MALDI TOF/TOF MS的FlexControl软件进行质谱图的采集,分别生成3种方法的峰统计列表,经比较后发现 MB-WCX在平均出峰量、平均峰面积、平均峰强度等方面均优于MB-HIC C8和MB-IMAC-Cu。因此选择MB-WCX作为后续大样本血清富集方法。
2.2 CLINPROT系统重复性检测 反复测定8例标本(4例乳腺癌血清、4例健康女性血清)富集的低丰度蛋白,经MALDI-TOF/TOF MS和ClinProt Tools v3.0软件分析,得到5次测定质谱图信息和平均CV,结果显示5次测定质谱图基本一致,病例组平均CV为24.5%,对照组平均CV为19.7%,当CV<30%,即表明该方法重复性良好。
2.3 血清差异蛋白的定量分析 将乳腺癌患者和健康对照血清经MB-WCX分析试剂盒富集得到的蛋白质/多肽通过MALDI TOF/TOF MS分别采集两组质谱图,应用FlexAnalysis软件将质谱图进行均一化处理后,导入ClinProt Tools v3.0软件进行统计学分析,生成平均质谱图、峰统计列表、胶状图等,发现了24个具有明显差异的蛋白峰(P<0.05),其中差异最明显的分别是Mass 3 348.9、Mass 4 787.5、Mass 5 750.1和Mass 8 141.6(表1)。
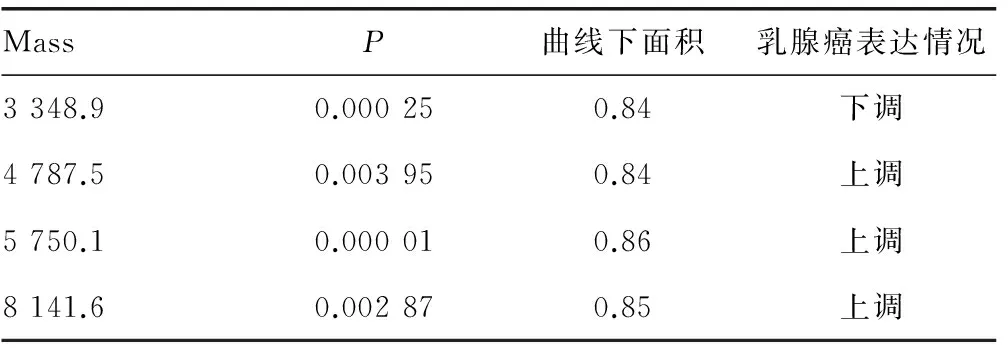
表1 乳腺癌和健康对照相比具有明显差异的蛋白质
2.4 乳腺癌诊断预测模型的建立 将模型组质谱图导入ClinProt Tools v3.0软件,根据遗传算法选取表达差异最显著的3个蛋白质建立乳腺癌诊断预测模型,对乳腺癌和健康对照的识别能力分别为91.6%和100.0%,建立模型的3个蛋白质曲线下面积分别为0.84、0.86和0.85。后为了验证该模型的诊断效能,利用验证组标本对该诊断模型进行验证,结果显示其对乳腺癌和健康对照的识别准确率均为85.7%(6/7)。诊断乳腺癌的灵敏度为85.7%,特异度为85.7%,Youden指数为0.714。根据ClinProt Tools选取的两个差异较为明显的蛋白质Mass 3 348.9和Mass 5 750.1进行聚类分析,可见两组交叉面积较小,且趋势明显,说明该模型鉴别能力较好。
3 讨 论
肿瘤性疾病的早期发现、诊断和治疗是降低病死率和改善患者生活质量的重要途径。乳腺癌早期临床症状不明显,患者一般至预后较差的中后期就诊,大大降低了生存率[6-7]。目前乳腺癌诊断还是依靠影像学检查和病理诊断为依据[8-10],而有研究表明在肿瘤性疾病发病早期,血液中肿瘤标志物的升高要先于影像学检查[11]。在肿瘤发生、发展的过程中,会同时伴随着多种生物标志物不同程度的升高或降低。目前,癌胚抗原(CEA)和癌抗原CA15-3等已作为乳腺癌血清标志物开始应用于临床乳腺癌患者的辅助诊断和预后判断,但多项研究表明两者灵敏度以及对乳腺癌的早筛准确率都不够理想[12-13]。因此,需要更多有关于乳腺血清肿瘤标志物的研究来满足临床乳腺癌诊断的需求。
液体芯片-MALDI TOF/TOF MS-CLINPROT系统是近年来蛋白质组学进行高通量分析的一种新技术,其主要优点是操作简单,节省标本,且能够直接对临床标本进行检测。该技术已经在前列腺癌、卵巢癌和膀胱癌等早期诊断方面进行了一系列的前瞻性研究[14-16],建立了多种预测诊断模型,同时发现数个有潜力的肿瘤标志物,并通过Mascot搜库成功鉴定,为肿瘤性疾病致病机制的进一步探索奠定了基础。
本研究应用液体芯片-MALDI-TOF/TOF MS-CLINPROT系统分析了36例乳腺癌患者和17例健康体检女性血清蛋白质谱,通过比较发现了24个差异具有统计学意义(P<0.05)的蛋白质/多肽。其中Mass 5 750.1和8 141.6在乳腺癌血清中均呈现表达下调,诊断乳腺癌的ROC曲线下面积分别为0.86和0.85,证明两个差异蛋白有潜力成为有价值的乳腺肿瘤新型标志物。ClinProt Tools v3.0软件根据遗传算法选择3个差异最显著的蛋白Mass 3 348.9、Mass 5 750.1和Mass 8 141.6建立了乳腺癌预测诊断模型,该模型能有效地区分乳腺癌和健康人。后通过7例乳腺癌患者和7例健康对照血清对该模型进行了验证,结果显示其对乳腺癌诊断的准确率达到85.7%,灵敏度为85.7%,特异度为85.7%,Youden 指数为0.714。该模型的诊断效能明显高于目前临床血清标志物的联合诊断,具有重要的临床应用前景。
本研究建立了诊断效能较高的乳腺癌预测诊断模型,同时发现了两个有价值的血清肿瘤标志物,为后续的鉴定和体内生物学效应的探究奠定了基础。由于本研究时间有限,病例数少,建立的诊断模型有其局限性,今后有待于扩大样本量进行深入研究。
[1]Leung SM,Pitts RL.A novel approach using MALDI-TOF/TOF mass spectrometry and prestructured sample supports (AnchorChip Technology) for proteomic profiling and protein identification[J].Methods Mol Biol,2008,441(1):57-70.
[2]Liu Y,Wei F,Wang F,et al.Serum peptidome profiling analysis for the identification of potential biomarkers in cervical intraepithelial neoplasia patients[J].Biochem Biophys Res Commun,2015,465(3):476-480.
[3]Jia K,Li W,Wang F,et al.Novel circulating peptide biomarkers for esophageal squamous cell carcinoma revealed by a magnetic bead-based MALDI-TOFMS assay[J].Oncotarget,2016,7(17):23569-23580.
[4]Ebert MP,Niemeyer D,Deininger SO,et al.Identification and confirmation of increased fibrinopeptide a serum protein levels in gastric cancer sera by magnet bead assisted MALDI-TOF mass spectrometry[J].J Proteome Res,2006,5(9):2152-2158.
[5]Jiang WP,Wang Z,Xu LX,et al.Diagnostic model of saliva peptide finger print analysis of oral squamous cell carcinoma patients using weak cation exchange magnetic beads[J].Biosci Rep,2015,35(3):203-210.
[6]张勇.应用ClinProt系统分析结肠腺癌患者血清蛋白表达谱[J].中国现代药物应用,2013,7(18):23-25.
[7]唐金海,姚宇锋.早期乳腺癌的诊断和外科治疗进展[J].中国普外基础与临床杂志,2011,18(9):916.
[8]刘为敏,王晓红,孔庆聪,等.乳腺肿瘤性病变的钼靶X线征象分析[J].临床放射学杂志,2010,29(11):1469-1472.
[9]李素荣,朱会玲.乳腺钼靶摄影对早期乳腺癌的诊断价值[J].中国辐射卫生,2010,19(4):500-501.
[10]龚西瑜,丁华野.乳腺肿瘤病理学[M].北京:人民卫生出版社,2009:294-443.
[11]郑航,罗荣城.TPS、CA153和CEA联合检测对乳腺癌的诊断价值[J].第一军医大学学报,2005,25(10):1293-1294.
[12]赵晶,刘红,张爱敏,等.乳腺癌患者血清可溶性肿瘤标志物的临床价值研究[J].中国肿瘤临床,2008,35(24):1411-1414.
[13]叶强,陈峻.多种肿瘤标志物在检验乳腺癌的临床应用[J].肿瘤基础与临床,2009,22(4):350-351.
[14]董德鑫,李汉忠,严维刚,等.应用磁珠联合 MALDI-TOF MS 技术建立前列腺癌血清差异蛋白诊断模型[J].协和医学杂志,2012,3(4):437-440.
[15]Petricoin EF,Ardekani AM,Hitt BA,et al.Use of proteomic patterns in serum to identify ovarian cancer[J].Lancet,2002,359(9306):572-577.
[16]Villanueva J,Shaffer DR,Philip J,et al.Differential exoprotease activities confer tumor-specific serum peptidome patterns[J].J Clin Invest,2006,116(1):271-284.
Establishment of diagnostic model of breast cancer by using CLINPROT system based on MALDI-TOF/TOF MS*
YANGFan1,CHENYingjian2,HUChengjin2△
(1.PostgraduatesSchool,JinzhouMedicalUniversity,Jinzhou,Liaoning121001,China;2.DepartmentofLaboratoryDiagnosis,GeneralHospitalofJinanMilitaryRegion,Jinan,Shandong250031,China)
Objective To use the liquid protein chip-MALDI-TOF/TOF MS CLINPROT system for screening the serum potential tumor markers in the patients with breast tumor and to establish the breast cancer diagnostic prediction model basic on differential protein.Methods Forty-three serum samples from the patients with pathologically diagnosed breast cancer and 24 serum samples from healthy female controls were collected and randomly divided into the construction model group and verification group.The differential protein screening in the breast cancer patients of the constructing model group and healthy female serum was performed by using the CLINPROT system.The differential protein atlas was established.The breast cancer diagnotic prediction model was established by using the Clin Prot Tools v3.0 software.Then the diagnostic model was applied to perform the grouping verification in the serum samples of verification group.The diagnostic efficiency of this model was preliminarily evaluated.Results Twenty-four protein peaks with significant difference were obtained by analyzing the mass spectrum peak information of the patients group and control group(P<0.05).Three proteins expressing the difference most significantly were selected to build a breast cancer diagnostic prediction model according to genetic algorithm,and the recognition ability of this model for breast cancer in the construction model group was 91.67%.Then the diagnostic efficiency of this model was evaluated by using the samples of verification group,and the results showed that its recognition accuracy rate to breast cancer in the verification group was 85.7%.Conclusion Using the ClinProt Tools v3.0 software successfully constructs and evaluates the diagnostic prediction model of breast cancer,which provides a new pathway for the diagnosis of breast cancer.
CLINPROT system; breast cancer; diagnostic model
国家自然科学基金资助项目(81472497)。
杨帆,女,技师,主要从事临床检验研究。△
,E-mail:hcj6289@163.com。
10.3969/j.issn.1673-4130.2016.24.002
A
1673-4130(2016)24-3388-03
2016-09-07
2016-10-26)

