谷子CO家族基因的全基因组鉴定与分析
穆彩琴,屈聪玲,张瑞娟,李燕,杨致荣
(1.山西农业大学文理学院,山西太谷030801;2.山西农业大学研究生院,山西太谷030801;3.山西农业大学生命科学学院,山西太谷030801)
谷子CO家族基因的全基因组鉴定与分析
穆彩琴1,2,屈聪玲2,3,张瑞娟2,3,李燕1,2,杨致荣1
(1.山西农业大学文理学院,山西太谷030801;2.山西农业大学研究生院,山西太谷030801;3.山西农业大学生命科学学院,山西太谷030801)
抽穗开花是高等植物从营养生长向生殖生长转变的过程,该过程受到众多基因的调控。CONSTANS(CO)是光周期开花途径中的一个关键基因,是生物节律钟基因和开花基因之间的关键枢纽。利用生物信息学的方法,从谷子基因组中鉴定了34个CO基因;与拟南芥CO家族的17个成员进行同源比对,发现Seita.7G007800与Seita.9G020100相似性最高;在此基础上,分析了谷子CO基因的启动子,发现启动子有光响应元件Sp1、脱落酸响应元件ABRE以及赤霉素响应元件P-box等;RNA-Seq转录组测序发现,这些CO基因在叶片中表达量存在显著差异,其中,Seita.4G192300基因表达量最高,而Seita.1G304900,Seita.3G080700和Seita.4G001600这3个基因在叶片中不表达。这些结果不仅为深入研究CO基因与抽穗开花的关系提供依据,而且为后续谷子抽穗开花途径的研究奠定理论基础。
谷子;全基因组;CONSTANS;光周期途径;抽穗;开花
谷子(Setaria italica)是禾本科狗尾草属植物,具有抗旱性强、耐贫瘠等优点,广泛种植于干旱和半干旱地区。2012年,由美国国家能源部所属的联合基因组研究所和中国华大基因分别进行的豫谷1号和张谷的全基因组测序工作相继完成,从此谷子功能基因组学研究进入了一个新的时代[1-2]。随着我国和世界其他国家研究人员对谷子的高度重视,其逐渐发展为禾谷类重要的旱生C4模式作物[3]。
抽穗开花是植物生长发育过程中的重要阶段,受到光周期、温度、激素和自身发育等众多内外因素的综合调控,且不同植物间的调控机制也不尽相同。在众多外界环境因素中,植物对光周期信号尤其敏感。CO基因在响应光周期调节中起着重要作用[4-6],是调控植物开花的重要基因之一。Putterill等[7]最早报道了拟南芥中的CO基因。CO是一类锌指蛋白转录因子,含有B-box和CCT结构域两类保守结构域[8-9]。该基因是植物开花的正调控因子,其表达受到mRNA和蛋白水平的双重调控。在短日照条件下,CO基因在黑夜会出现高表达,而CO蛋白在黑暗条件下会被迅速降解,因此,不能促进开花;而在长日照条件下,CO基因在傍晚和黑夜都会出现高表达,并且CO蛋白在光照下可以稳定存在,从而促进开花[5]。CO受到众多基因的调控,包括CYCLING DOF FACTOR1(CDF1),FLAVIN-BINDING KELCH REPEAT F-BOX 1(FKF1),GIGANTEA(GI),CONSTITUTIVEPHOTOMORPHOGENESIS1(COP1)和EFL3等。在蛋白水平上,PHYTOCHROME B(PHYB)在上午能促进CO蛋白泛素化,导致其降解,而PHYA,CRYPTOCHROME 1(CRY1)以及CRY2等能在傍晚稳定CO蛋白[10]。最终,CO基因通过调控FLOWERING LOCUS T(FT)基因的表达进而调控植物开花[5]。
目前关于拟南芥、水稻等模式作物的开花途径研究的比较清楚,但关于谷子的开花研究目前较少,尤其对谷子一些重要的开花基因研究鲜有报道。本研究对谷子进行了全基因组分析,从中间发现了34个CO基因,进一步对这些基因进行了生物信息学分析,并利用RNA-Seq高通量测序技术检测了其在营养生长时期的表达情况,旨在为深入揭示谷子抽穗开花的调控机制提供依据。
1 材料和方法
1.1 供试材料
晋谷21号由山西农业大学生物工程研究所提供。
1.2 试验方法
1.2.1 谷子CO基因的生物信息学分析利用Phytozome v11.0中豫谷1号基因组数据库(https:// phytozome.jgi.doe.gov/pz/portal.html)进行关键词搜索,进一步结合NCBI,GenBank等数据库获取谷子CO蛋白的信息,并下载整理。使用Phytozome v 11.0中JBrowse功能下载豫谷1号CO基因在染色体上的位置信息。使用MEGA 7.0软件中的Neibor-joining算法对下载的谷子CO氨基酸序列构建系统发育树。通过PlantCARE网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在线分析启动子元件。
1.2.2 谷子CO基因的表达情况分析将晋谷21号播种在人工气候箱培养,光照时间为16 h光照/8 h黑暗,光照强度为50 000 lx,温度为25℃(光照)/ 22℃(黑暗)。播种28 d,提取总RNA用于RNASeq测序。RNA提取采用植物总RNA提取试剂盒(天根生化科技(北京)有限公司,货号DP432),操作方法按照试剂盒使用说明进行。利用Nanodrop和Agilent 2100检测RNA的纯度(OD260/OD280)、浓度和完整性,检测合格的样品用于测序文库的构建和RNA-Seq测序。文库构建和RNA-Seq测序参照王兴春等[11]的方法进行。
2 结果与分析
2.1 谷子CO基因的基本信息
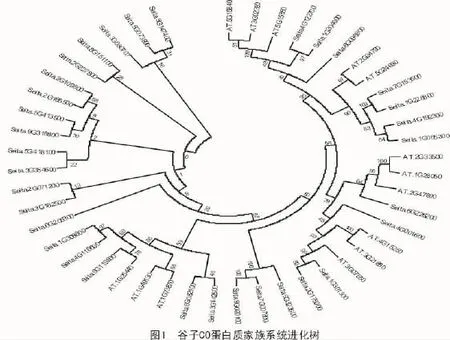
利用拟南芥已知17个CO蛋白序列在豫谷1号基因组中进行Blast分析,筛选到34个谷子CO基因(图1),比拟南芥CO基因家族多出17个基因,但是否存在未被筛选出的CO-like基因依旧不清楚。比较它们的蛋白序列发现,部分基因之间的相似性非常高,其中,Seita.7G007800与Seita.9G020100相似性最高,达到85.09%,Seita.1G301300与Seita. 2G175000,Seita.7G153500与Seita.1G228800以及Seita.4G192300与Seita.1G065300的相似性也较高。
2.2 谷子CO基因的理化性质
从豫谷1号的CO基因的cDNA序列预测得到的氨基酸序列,比较发现CO蛋白的氨基酸序列长度在174~1 033 aa,分子量处于10.65~113.86 kDa,理论等电点在4.74~11.8(表1)。
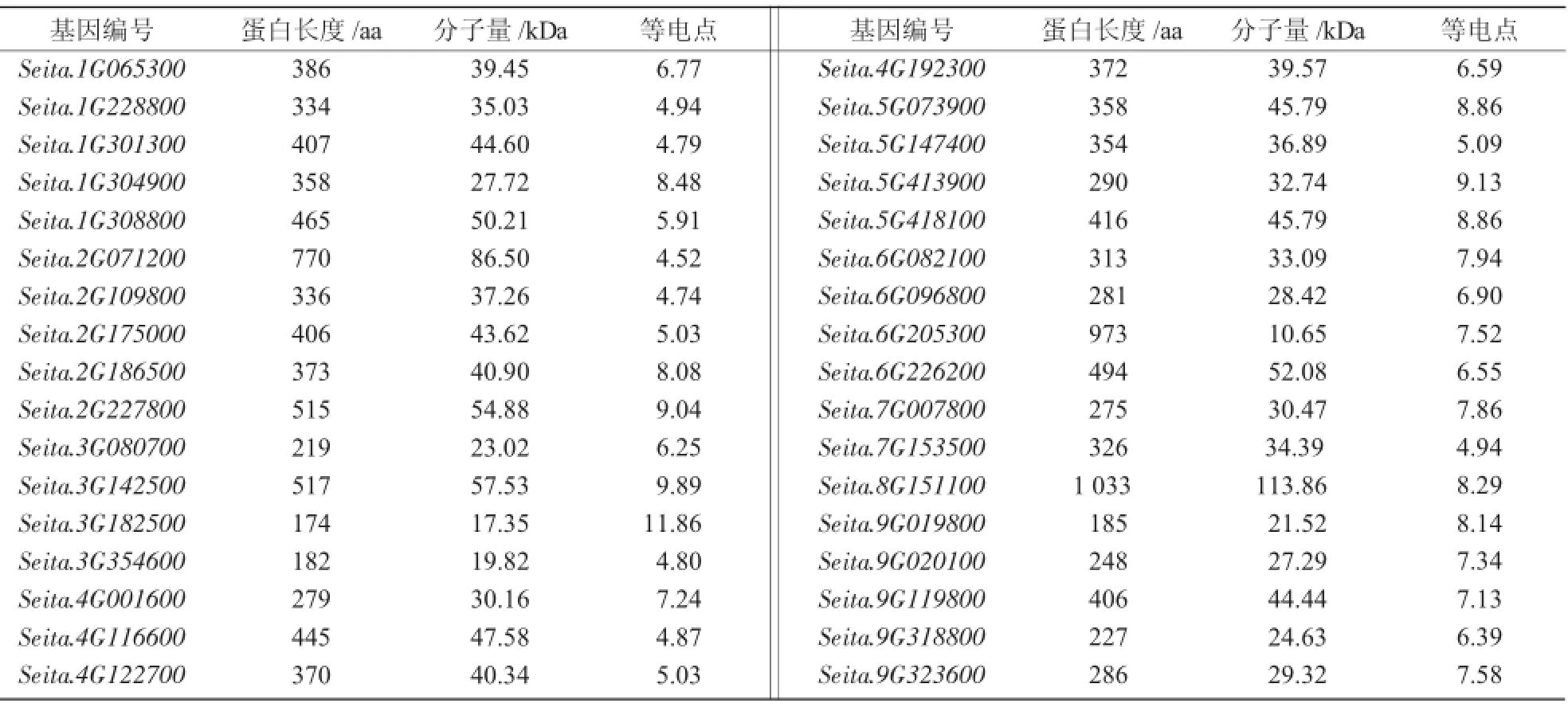
表1 谷子CO蛋白家族理化性质
2.3 谷子CO基因家族启动子元件
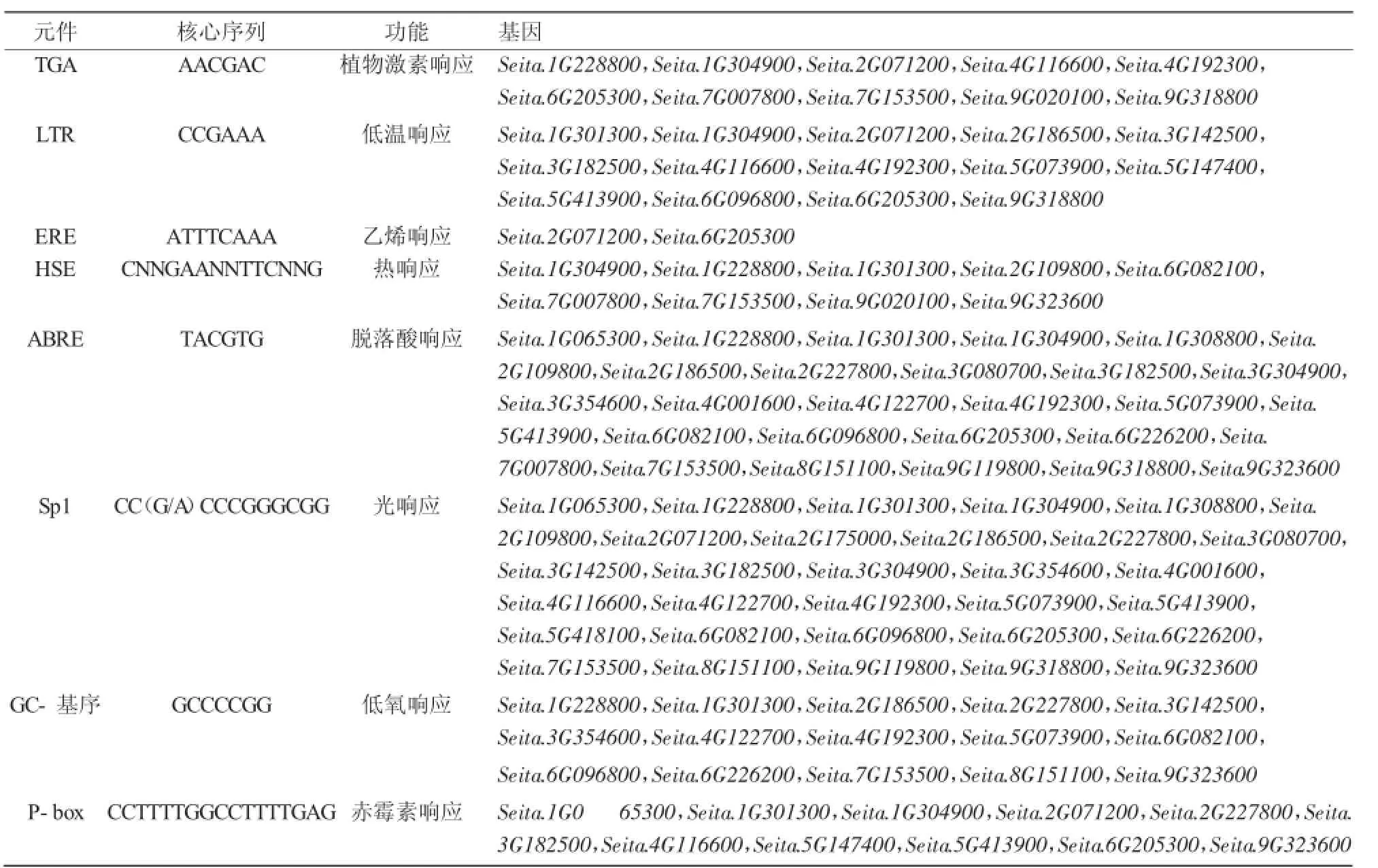
表2 谷子CO基因家族启动子顺式元件预测
根据豫谷1号全基因组序列,获取谷子扩展蛋白基因CDS区上游1 500 bp的核苷酸序列,用PlantCARE进行启动子在线分析,发现除了基本启动子元件TATA-box和CAAT-box外,谷子CO基因还包含多种与胁迫响应相关的顺式作用元件,包括热胁迫响应元件HSE、低温胁迫响应元件LTR、植物激素响应元件TGA-element、赤霉素响应元件P-box以及光响应元件Sp1等(表2)。
将豫谷1号CO家族基因的基本启动子元件进行统计,发现90%以上的基因含有光响应元件Sp1,80%以上的基因含有脱落酸响应元件ABRE,说明谷子中的CO基因参与对光照和胁迫的响应(表2)。
2.4 CO基因在营养生长期的表达分析
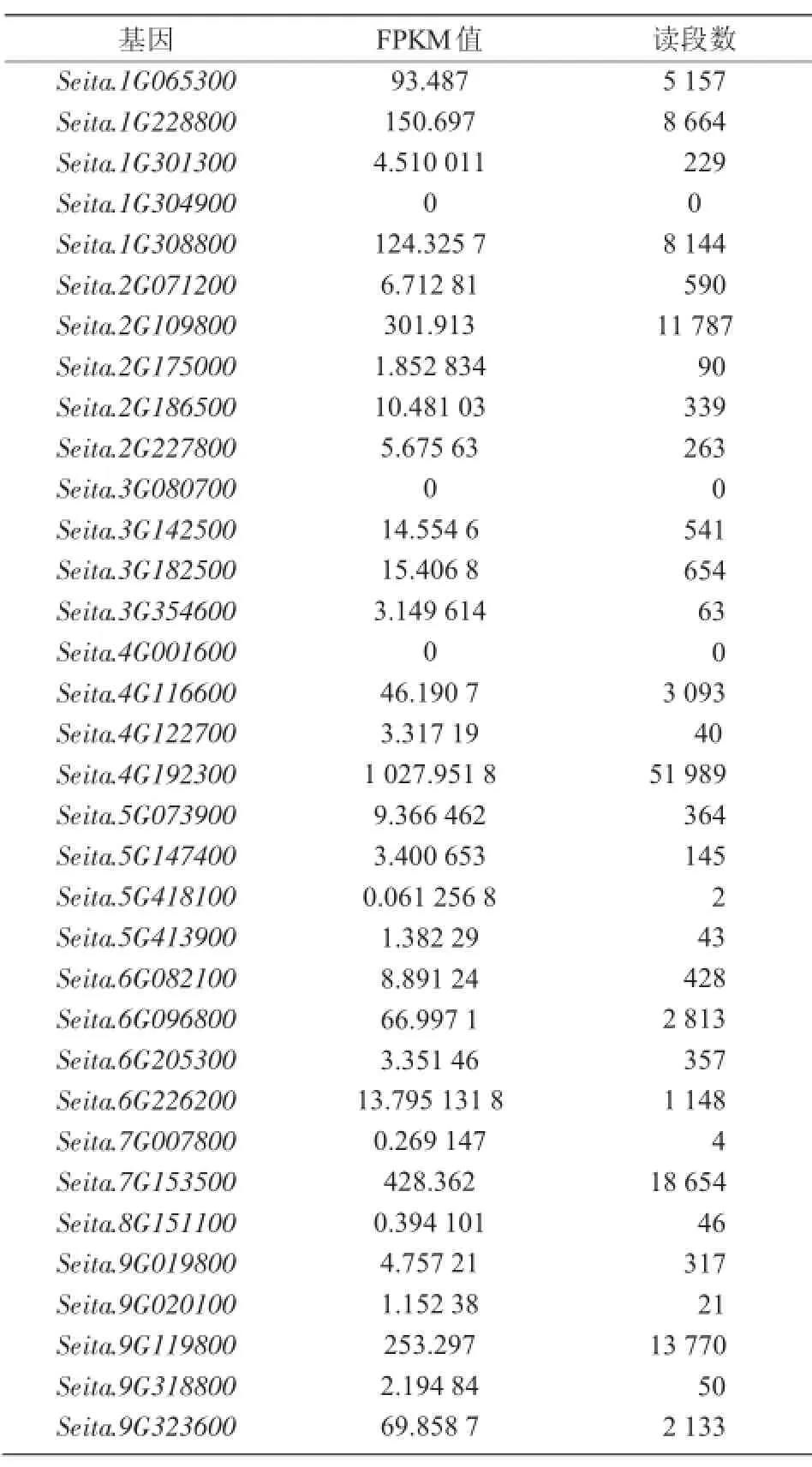
表3 叶片中CO基因的表达量
将播种28 d的晋谷21号提取RNA进行转录组测序分析,对CO基因的表达进行统计,发现表达量较低(≤10)的基因为20个,占58.82%;高表达(>1 000)的基因为1个,占2.94%(表3)。其中,Seita.4G192300的表达量最高(表3)。
3 讨论与结论
CO是光周期途径的一个重要基因,在植物的生物钟与开花之间起重要的枢纽作用。通过对谷子CO基因组的分析,有利于了解CO基因特征及功能,对后续谷子开花途径的研究具有重要意义。
目前已经对拟南芥和水稻的开花途径研究比较清楚,在拟南芥中发现CO家族有17个成员[12],水稻中有16个成员[13],然而在谷子中发现有34个成员。在同源性高的谷子家族成员中含有B-box和CCT两类保守结构域,其功能可能与拟南芥的CO基因类似。
谷子抽穗开花会受到内外因素的综合调控,通过对谷子CO基因上游约1 500 bp的启动子序列分析发现,启动子中含有多个与逆境相关的顺式作用元件,包括热胁迫响应元件HSE、低温胁迫响应元件LTR、植物激素响应元件TGA-element、赤霉素响应元件P-box、以及光响应元件Sp1,其中,90%以上的基因含有光响应元件Sp1,CO基因家族可能通过以上顺式作用元件参与谷子对光照的响应。
本研究通过对谷子CO基因进行生物信息学分析,并对晋谷21号转录组测序结果统计,发现CO家族成员的表达水平存在差异。初步了解了谷子CO基因与谷子开花途径的关系,为进一步理解CO基因在谷子抽穗开花过程中的作用提供了依据,同时为后续深入研究谷子开花途径奠定了理论基础。
[1]Quan ZW,ChengSF,Xu X,et al.Genome sequence offoxtail millet(Setaria italica)provides insights into grass evolution and biofuel potential[J].Nat Biotechnol,2012,30(6):549-554.
[2]Bennetzen J L,Schmutz J,Wang H,et al.Reference genome sequence ofthe model plant Setaria[J].Nature biotechnology,2012,30(6):555-561.
[3]Doust A N,Kellogg E A,Devos K M,et al.Foxtail millet:a sequence-driven grass model system[J].Plant Physiol,2009,149(1):137-141.
[4]Samach A,Onouchi H,Gold S E,et al.Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis[J].Science,2000,288:1613-1616.
[5]An H,Roussot C,Suarez-Lopez P,et al.CONSTANS acts in thephloem to regulate a systemic signal that induces photoperiodic flowering ofArabidopsis[J].Development,2004,131(15):3615-3626.
[6]Suarez-Lopez P,WheatleyK,Robson F,et al.CONSTANS mediates between the circadian clock and the control of flowering in Arabidopsis[J].Nature,2001,410:1116-1120.
[7]Putterill J,Robson F,Lee K,et al.The CONSTANS gene of Arabidopsis promotes floweringand encodes a protein showingsimilarities to zinc finger transcription factors[J].Cell,1995,80(6):847-857.
[8]Strayer C,Oyama T,Schultz T F,et al.Cloning of the Arabidopsis clock gene TOC1,an autoregulatory response regulator homolog[J]. Science,2000,289:768-771.
[9]Borden K L.RING fingers and B-boxes:zinc-binding protein-protein interaction domains[J].Biochem Cell Biol,1998,76(2/3):351-358.
[10]Endo M,Tanigawa Y,Murakami T,et al.hytochrome-dependent Late-flowering accelerates flowering through physical interactions with phytochrome B and CONSTANS[J].Proc Natl Acad Sci USA,2013,110(44):18017-18022.
[11]王兴春,谭河林,陈钊,等.基于RNA-Seq技术的连翘转录组组装与分析及SSR分子标记的开发[J].中国科学:生命科学,2015,45(3):301-310.
[12]Robson F,Costa MM,Hepworth S R,et al.Functional importance of conserved domains in the flowering-time gene CONSTANS demonstrated byanalysis of mutant alleles and transgenic plants[J]. Plant J,2001,28(6):619-631.
[13]Griffiths S,Dunford R P,Coupland G,et al.The evolution of CONSTANS-like gene families in barley,rice,and Arabidopsis[J].Plant Physiol,2003,131(4):1855-1867.
Genome-wide Identification and Analysis of CO Family Genes in Foxtail Millet
MUCaiqin1,2,QUCongling2,3,ZHANGRuijuan2,3,LI Yan1,2,YANGZhirong1
(1.College ofArts and Sciences,Shanxi Agricultural University,Taigu 030801,China;2.College ofGraduate,Shanxi Agricultural University,Taigu 030801,China;3.College ofLife Sciences,Shanxi Agricultural University,Taigu 030801,China)
Heading and flowering is the transition stage from vegetative to reproductive growth in higher plants,which is regulated bymultiple genes.CONSTANS(CO)is a keygene in photoperiodic floweringpathwayand is a keyjunction between the circadian clock and flowering gene.In this study,34 CO genes were identified using bioinformatics in foxtail millet.The paper analyzed the relationship between these CO genes and the 17 CO genes in Arabidopsis thaliana,and founded that Seita.7G007800 and Seita.9G020100 had the highest similarity,further analyzed their promoter sequence,and identified light responsive element Sp1,ABA response element ABRE, gibberellin-responsive element P-box.RNA-Seq transcriptom sequencing revealed that the expression level of these CO genes was significantly different in leaves.Seita.4G192300 had the highest expression,while Seita.1G304900,Seita.3G080700 and Seita. 4G001600 were not expressed in leaves.These results not only provide a foundation for further study on the relationship between CO genes and flowering,but alsocontribute tofurther research on headingand floweringpathways in foxtail millet.
foxtail millet;genome-wide;CONSTANS;photoperiod pathway;heading;flowering
S515
A
1002-2481(2016)09-1243-05
10.3969/j.issn.1002-2481.2016.09.02
2016-06-26
山西省回国留学人员科研资助项目(2015-067);山西省留学回国人员科技活动择优资助项目(2014-11)
穆彩琴(1990-),女,山西柳林人,在读硕士,研究方向:谷子抽穗开花的调控机制。屈聪玲同为第一作者。杨致荣为通信作者。

