枯草芽胞杆菌谷氨酰胺转氨酶在谷氨酸棒杆菌中的分泌表达及诱导条件的优化
黄 琳,刘逸寒,李明杰,李 瑞,郭 伟,桂 爽,路福平
(工业发酵微生物教育部重点实验室,天津市工业微生物重点实验室,工业酶国家工程实验室,天津科技大学生物工程学院,天津 300457)
枯草芽胞杆菌谷氨酰胺转氨酶在谷氨酸棒杆菌中的分泌表达及诱导条件的优化
黄 琳,刘逸寒,李明杰,李 瑞,郭 伟,桂 爽,路福平
(工业发酵微生物教育部重点实验室,天津市工业微生物重点实验室,工业酶国家工程实验室,天津科技大学生物工程学院,天津 300457)
本文通过重叠PCR技术构建获得枯草芽胞杆菌(Bacillus subtilis)来源的谷氨酰胺转氨酶(transglutaminase,TG)(BTG)重组基因Sprodbtg,该基因依次包含SD序列、谷氨酸棒杆菌信号肽ΔS0949序列、proD-BTG基因,并将该目的基因克隆入大肠杆菌-谷氨酸棒杆菌穿梭表达载体pXMJ19中构建重组质粒pXMJ19-Sprodbtg.随后,重组质粒电转入谷氨酸棒杆菌(Corynebacterium glutamicum)ATCC13032中进行诱导表达,并对重组菌株的诱导条件进行优化.结果表明:重组菌株C. glutamicum ATCC13032/pXMJ19-Sprodbtg表达重组酶原蛋白proD-BTG经活化后,BTG的活力达到(41.23±2.01)U/L;在重组菌培养12 h后诱导、IPTG终浓度0.8 mmol/L、诱导40 h的最优诱导条件下,BTG的活力达到最高,为(55.62±2.34)U/L,较优化前提高了约 34.90%,.
谷氨酰胺转氨酶;谷氨酸棒杆菌;枯草芽胞杆菌;分泌表达;条件优化
谷氨酰胺转氨酶(transglutaminase,TG)是一种催化酰基转移反应的转移酶,催化蛋白质分子内和分子间发生交联、蛋白质和氨基酸之间的连接以及蛋白质分子内谷氨酰胺基的水解反应[1].由于其安全、特有的交联性质,可用于改善蛋白质的结构和功能性质,在植物蛋白制品、焙烤制品、鱼、肉、乳类制品、固定化酶、可食性包装等行业已有广泛应用[2-6].
目前,仅茂源链霉菌(Streptomyces mobaraense)谷氨酰胺转氨酶(MTG)实现了商业化生产,但茂源链霉菌生长慢、发酵周期长、酶活力收率低[7],造成MTG价格较高;另外,随着食品加工业种类逐渐增多,MTG的特性无法满足一些食品加工底物和过程的需求,因此,亟需开发不同特性的TG.枯草芽胞杆菌(Bacillus subtilis)谷氨酰胺转氨酶(BTG)是在1996年被Kobayashi等[8]首次发现,且经验证BTG的分子结构没有信号肽和前肽;Suzuki等[9]发现BTG最适作用pH为8.2,最适作用温度为60 ℃,可催化酪素液及BSA蛋白分子的凝胶化和交联;并且,经分析BTG和MTG在氨基酸序列和空间结构上有很大区别,这为开发新型TG奠定了基础.
本研究室在前期研究中从枯草芽胞杆菌(B. subtilis)168ATCC23857基因组克隆了BTG基因,该基因仅编码成熟肽,对异源宿主细胞有毒性[10],影响其正常生长,难以实现BTG高效表达.研究发现,来自于生暗灰链霉菌(S.caniferus)的TG前肽proD对BTG活性的抑制率最高,达到70%[11].然而,大肠杆菌表达系统难以实现proD-BTG酶原的高效表达.谷氨酸棒状杆菌蛋白分泌机制健全,生长迅速,培养简便,使用安全,无致病性,是表达外源基因的良好受体菌,本研究室已利用谷氨酸棒杆菌表达系统实现了BTG的异源表达[12].因此,基于上述研究,本文以谷氨酸棒杆菌(Corynebacterium glutamicum)ATCC13032为宿主菌株,构建重组菌株C. glutamicum ATCC13032/pXMJ19-Sprodbtg以实现proD-BTG的高效分泌表达,并对其诱导条件进行优化,旨在为BTG的研究奠定基础.
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
大肠杆菌(Escherichia coli)JM109、谷氨酸棒状杆菌(C.glutamicum)ATCC13032、大肠杆菌-谷氨酸棒杆菌穿梭表达质粒pXMJ19和克隆有prodbtg基因的质粒pET28a-prodbtg均为本实验室保藏.
1.1.2 酶与主要试剂
限制性内切酶HindⅢ和BamHⅠ、T4 DNA连接酶、Probest高保真酶,TaKaRa公司;溶菌酶、RNase,上海生工生物工程有限公司;小量琼脂糖凝胶DNA试剂盒、PCR产物纯化回收试剂盒、质粒小量提取试剂盒,北京庄盟国际生物基因科技有限公司;BCA蛋白浓度试剂盒,碧云天生物技术研究所;蛋白胨和酵母浸粉,Oxoid公司;Factor Xa,Sigma公司;其他试剂均为国产分析纯.
1.1.3 培养基
LB培养基(g/L):胰蛋白胨10,酵母浸出粉5,NaCl 5,pH 7.0.
LA培养基:LB+2%,琼脂粉,pH 7.0~7.2.
谷氨酸棒杆菌复苏培养基:LB+0.5%,葡萄糖,pH 7.0~7.2.
谷氨酸棒杆菌产MTG培养基(MMTG)(g/L):葡萄糖60,MgSO41,(NH4)2SO430,KH2PO41.5,FeSO4·7H2O 0.01,MnSO4·4H2O 0.01,盐酸硫胺素4.5×10-4,生物素4.5×10-4,甲硫氨酸0.15,CaCO350,pH 7.5.
1.1.4 引物设计
为了使得外源蛋白在谷氨酸棒状杆菌中实现高效分泌表达,设计含SD序列及C.glutamicum R来源的有高效分泌能力信号肽ΔS0949序列[13]的重组proD-BTG基因序列.依据引物设计原则[12]设计引物. 上游引物P1:5′-TATCGGCGCTGCCAGCATGT TTATGCCAAAGGCCAACGCCCAAGGAGCACTCG AGGCCAGCGGCGGC-3′;P2:5′-CCCAAGCTT AAA GGAGGACACGCATGCAAATAAACCGCCGAGGCT TCTTAAAAGCCACCGCAGGACTTGCCACTATCG GCGCTGCCAGCATG-3′;下游引物R1:5′-CGCGG ATCC TTAGCGGACGATGCGG-3′.上游引物P1 5′端不含有酶切位点;上游引物P2 5′端含有HindⅢ酶切位点(AAGCTT);下游引物R1 5′端含有BamHⅠ酶切位点(GGATCC).
1.2 方法
1.2.1 Sprodbtg基因的扩增
通过重叠PCR将SD序列和信号肽ΔS0949序列克隆于proD-BTG基因的N端,获得目的基因.第1轮PCR以质粒pET28a-prodbtg为模板,以P1、R1为引物扩增目的基因;第2轮PCR以第1轮扩增产物为模板,以P2、R1为引物扩增出最终目的基因含SD序列及ΔS0949 全长序列的proD-BTG基因.PCR反应条件:95 ℃预变性5 min;95 ℃变性30s,55 ℃退火30 s,72 ℃延伸1 min,30 个循环;72 ℃10 min.
1.2.2 重组质粒pXMJ19-Sprodbtg的构建
经切胶、回收、纯化后的PCR产物(Sprodbtg基因)及pXMJ19质粒均用HindⅢ和BamHⅠ进行双酶切,分别回收酶切产物,经纯化后,用T4 DNA连接酶于16,℃连接过夜并转化至E.coli JM109,然后涂布于含50 µg/mL氯霉素的平板,挑取转化子,提取质粒,酶切验证.将验证正确的重组质粒命名为pXMJ19-Sprodbtg.
1.2.3 重组菌株C.glutamicum ATCC13032/pXMJ 19-Sprodbtg的构建
取6 µL 构建好的质粒pXMJ19-Sprodbtg电转化至C.glutamicum ATCC13032感受态细胞,电击条件同挑取转化子酶切验证,将验证正确的重组菌株命名为C.glutamicum ATCC13032/pXMJ19-Sprodbtg.质粒pXMJ19转化子C.glutamicum ATCC13032/ pXMJ19作为对照菌株.
1.2.4 重组菌C. glutamicumATCC13032/pXMJ19-Sprodbtg及C. glutamicumATCC13032/pXMJ 19的诱导表达
分别挑取1环重组菌C.glutamicum ATCC 13032/pXMJ19-Sprodbtg或C.glutamicum ATCC 13032/pXMJ19单菌落,分别接种于装有50,mL LB培养基的250,mL三角瓶中,30 ℃、200,r/min振荡培养过夜,以1%,的接种量分别转接于装有100 mL MMTG的500 mL三角瓶中,30 ℃、200 r/min振荡培养12,h,加入终浓度为1,mmol/L的IPTG诱导酶原蛋白proD-BTG的表达,继续培养48 h.分别取1 mL重组菌发酵液于12,000,r/min离心1,min,获取上清液,向其中分别加入130 µL 100%, TCA,冰上放置30 min,12,000,r/min离心5,min,弃去上清液,加1,mL丙酮洗涤2次.待丙酮挥发完全后,沉淀分别溶解于20,µL无菌蒸馏水中,各加入5,µL 5×SDS-PAGE电泳上样缓冲液,沸水浴5,min,12,000,r/min离心10,min,分别取上清液进行SDS-PAGE分析.
1.2.5 酶原蛋白proD-BTG的酶活力测定[15]
含有酶原蛋白proD-BTG的发酵液通过4,000,r/min离心15,min回收上清液,一定量的Factor Xa加入到表达proD-BTG的重组谷氨酸棒杆菌上清液中切割重组蛋白.随后通过荧光定量分析法分析酶原蛋白proD-BTG的酶活力.
酶活力检测反应体系:90,µL(5,mg/mL)的重组酶和1.98,mL 50,mmol/L Tris-HCl(pH,7.5)包括0.2%, N,N-二甲基酪蛋白,12.5,µmol/L 丹磺尸胺(MDC),4.5,mmol/L DTT.反应体系于37,℃反应30,min,加入42,mmol/L(NH4)2SO4终止反应,于激发波长350,nm下检测发射波长500,nm下的荧光强度.
酶活力定义:每分钟催化1 nmol/L的MDC插入到N,N-二甲基酪蛋白所需的酶量定义为1个活力单位(U).
1.2.6 重组菌C.glutamicumATCC13032/pXMJ19-Sprodbtg诱导条件的优化
重组菌生长曲线的绘制:将培养过夜的菌液按1%,的接种量接入50,mL含有10,µg/mL氯霉素的MMTG培养中,30 ℃、200 r/min振荡培养,间隔一定时间取样,以MMTG培养基为空白对照,检测培养液600,nm下的吸光度.以该菌种培养时间为横坐标,以吸光度为纵坐标,绘制生长曲线.
最佳诱导时机的确定:分别在接种4、8、12、16、20,h后加入终浓度为1,mmol/L的IPTG开始诱导,30,℃诱导48,h,检测分析诱导时机对proD-BTG表达量的影响.
最佳诱导时间的确定:在最佳诱导时机加入终浓度为1 mmol/L的IPTG开始诱导,30,℃诱导20、30、40、50、60 h,检测分析诱导时机对proD-BTG表达量的变化.
IPTG最佳诱导浓度的确定:在最佳诱导时机加入终浓度分别为0.4、0.6、0.8、1.0、1.2,mmol/L的IPTG开始诱导,30,℃诱导至最佳时间,分析IPTG浓度对proD-BTG表达的影响.
2 结果与分析
2.1 Sprodbtg基因的扩增
本实验室前期构建的质粒pET28a-prodbtg中目的基因未发生突变,以该质粒作为模板,分别用引物P1、R1和P2、R1扩增基因Sprodbtg.PCR产物进行琼脂糖凝胶电泳,获得约为1,100,bp的目的片段(图1),其大小与Sprodbtg预计大小相符,表明Sprodbtg扩增成功.该基因上游外源加入了一段SD序列及C.glutamicum高效分泌蛋白的信号肽序列ΔS0949,使得prodbtg具有了在C.glutamicum中分泌表达的潜力.
2.2 重组质粒pXMJ19-Sprodbtg的构建
将PCR产物切胶回收后用限制性内切酶HindⅢ和BamHⅠ进行双酶切,后与同样经过HindⅢ和BamHⅠ双酶切的质粒 pXMJ19连接,然后转化至E.coli JM109,涂布含氯霉素质量浓度为50,µg/mL的平板.经筛选挑取阳性菌落活化后提取质粒,用HindⅢ和BamHⅠ双酶切验证,同时将pXMJ19 空质粒用 HindⅢ进行单酶切作为对照,结果如图2所示.空质粒经HindⅢ单酶切获得约6,600,bp的片段,重组质粒经HindⅢ和BamHⅠ双酶切获得约6,600,bp和1,100,bp的两条带,其中大片段与空质粒pXMJ19大小(6,600,bp)相符,小片段与目的基因Sprodbtg大小相符,表明Sprodbtg基因已成功连接到表达载体上.
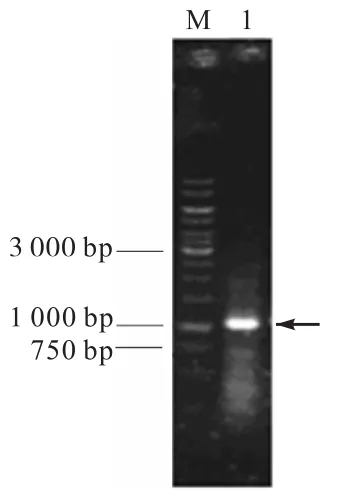
图1 Sprodbtg PCR扩增产物电泳图Fig. 1 Identification of Sprodbtg
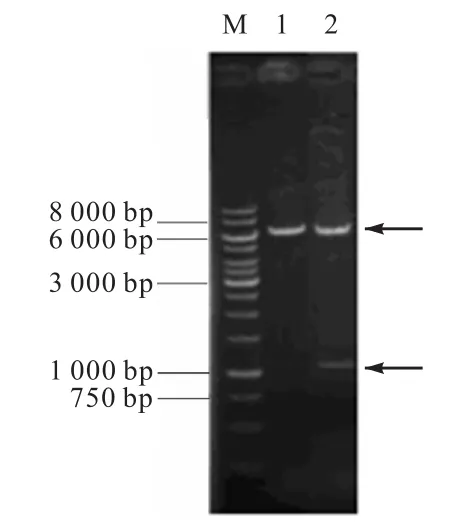
图2 质粒pXMJ19-Sprodbtg酶切验证Fig. 2 Recombined plasmids pXMJ19-Sprodbtg digested by restricted endonuclease
2.3 谷氨酸棒杆菌的电转化及鉴定
提取重组质粒pXMJ19-Sprodbtg并电击转化谷氨酸棒杆菌感受态细胞,以空质粒pXMJ19作为对照菌株.涂布于含氯霉素10,µg/mL的LA抗性平板,挑取转化子酶切验证.
2.4 重组菌的诱导表达
按照1.2.4方法对转化子进行诱导表达,将诱导48,h后的重组菌株C.glutamicum ATCC13032/ pXMJ19-Sprodbtg和对照菌株C.glutamicum ATCC13032/pXMJ19 发酵液经TCA沉淀浓缩后进行SDS-PAGE分析,结果如图3所示.

图3重组谷氨酸棒杆菌发酵上清液 SDS-PAGE 分析Fig. 3SDS-PAGE analysis of supernatant of C. glutamicum of the recombined strains
泳道1阳性转化子C. glutamicum ATCC 13032/pXMJ19-Sprodbtg在大约3.8×104处出现1条特异性蛋白条带,其大小与预测的proD-BTG的相对分子质量一致.泳道2中对照菌C. glutamicum ATCC13032/pXMJ19未见相应目的蛋白条带,从而初步说明proD-BTG在谷氨酸棒杆菌中被成功分泌表达.
2.5 酶原蛋白proD-BTG的酶活力测定
重组菌C. glutamicum ATCC13032/pXMJ19-Sprodbtg表达酶原蛋白 proD-BTG 经Factor Xa切割后,荧光法测定酶活力,结果显示BTG活力为(41.23±2.01)U/L.
2.6 重组菌C. glutamicum ATCC13032/pXMJ19-Sprodbtg诱导条件优化
2.6.1 生长曲线的绘制
重组菌在氯霉素浓度为10 µg/mL的MMTG培养基中的生长曲线如图4所示.由图4可知:重组菌株4,h后进入对数生长期,14,h后生长减缓,18 h后进入稳定期.
2.6.2 诱导时机对目的蛋白proD-BTG表达的影响
取不同诱导时机的发酵液添加Factor Xa并测定重组菌BTG的活力,结果如图5所示.在培养4 h后进行诱导,目的蛋白在重组菌中表达量非常低;然后随着菌体密度的增大而进行诱导,目的蛋白的表达量也随之增加.在培养12 h(A600约为2.2)时进行诱导达到最高酶活力(47.43±2.34)U/L,此时为重组菌对数生长期的中后期,目的蛋白的表达量基本达到最大,之后表达量开始下降,这可能是后期培养基中的营养被大量消耗,缺乏充足的营养用于目的蛋白的合成,因此当培养12,h后开始诱导,为最适的诱导时机.
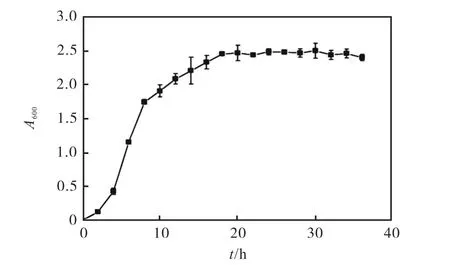
图4 C. glutamicum ATCC13032/pXMJ19-Sprodbtg生长曲线Fig. 4 Growth curve of C. glutamicum ATCC13032/ pXMJ19-Sprodbtg

图5 不同诱导时机对proD-BTG蛋白表达的影响Fig. 5 Effect of different inducing period on protein proD-BTG expression
2.6.3 诱导时间对目的蛋白proD-BTG表达的影响
如图6所示,当诱导时间为10~40 h时,目的蛋白的表达量随着诱导时间的增加而增加,最高达到(54.69±3.35)U/L;而当诱导时间超过40 h后,表达量有所下降,推测可能由于菌体开始进入衰亡期,裂解的菌体释放出的蛋白酶降解了部分重组蛋白,导致重组蛋白表达量的下降.所以最佳诱导时间定为40 h.
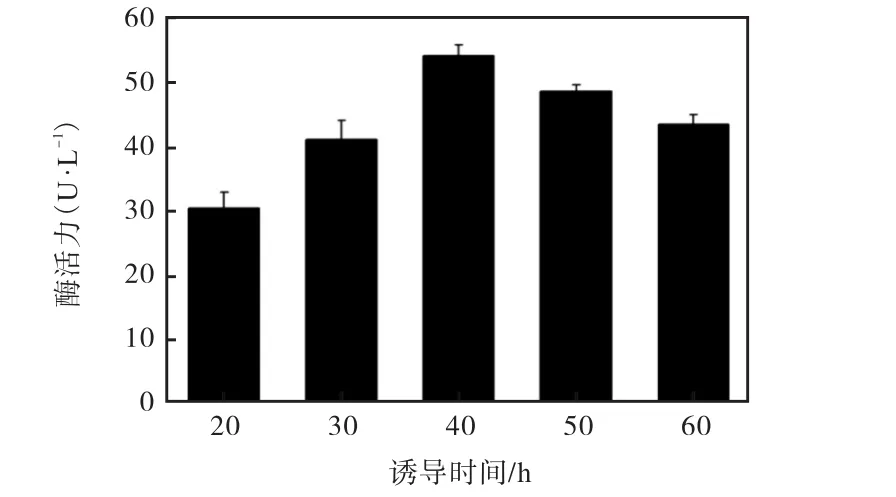
图6 不同诱导时间对proD-BTG蛋白表达的影响Fig. 6 Effect of induction time on protein proD-BTG expression
2.6.4 IPTG浓度对目标蛋白proD-BTG表达的影响
IPTG对细胞有一定的毒性[16],所以加入合适浓度的IPTG,才能使目的蛋白得到高效表达.IPTG浓度在0.01~5.0,mmol/L的范围内可能对表达产生影响[17].IPTG浓度对目标蛋白proD-BTG表达的影响如图7所示.
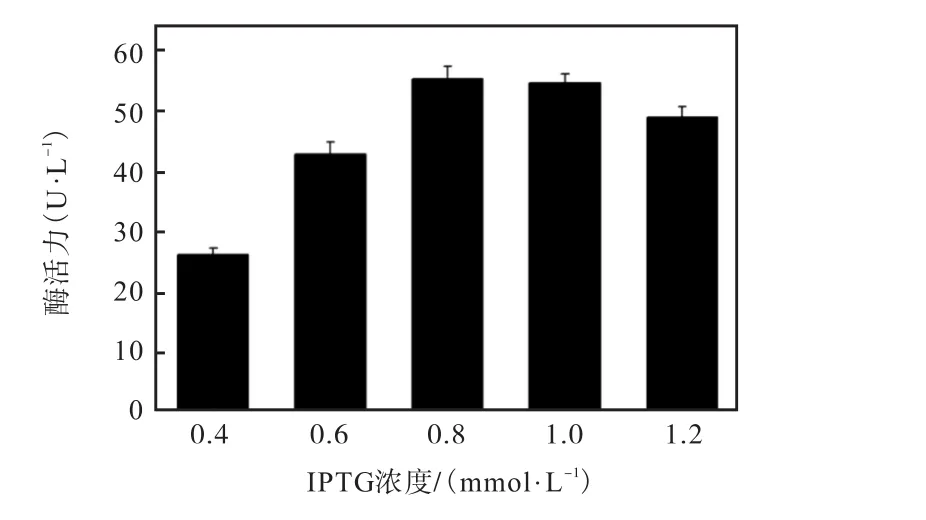
图7 不同IPTG浓度对proD-BTG蛋白表达的影响Fig. 7Effect of different concentration of IPTG on protein proD-BTG expression
当IPTG浓度为0.4~0.8 mmol/L时,目的蛋白的表达量随着IPTG浓度的增加而增加,最高达到(55.62±2.34)U/L;当IPTG浓度增大到1.0 mmol/L时,目的蛋白的表达量也能达到最高,但考虑到成本的节约,将0.8 mmol/L作为IPTG的最优诱导浓度.
3 结 语
本文在前期研究的基础上,对谷氨酸棒杆菌重组菌株C. glutamicum ATCC13032/pXMJ19-Sprodbtg的发酵条件进行优化.重组菌培养12 h后A600约为2.2时,添加IPTG至终浓度为0.8 mmol/L,诱导40 h.优化后的发酵上清液酶活力由(41.23±2.01)U/L提高到(55.62±2.34)U/L,比优化前提高了约34.90%,,为BTG进一步的高效制备奠定了基础.
[1] Folk J E. Transglutaminase[J]. Annual Review Biochemistry,1980,49:517-531.
[2] Yeoh S Y,Alkarkhi A F,RamLi S B,et al. Effect of cooking on physical and sensory properties of fresh yellow alkaline noodles prepared by partial substitution of wheat flour with soy protein isolate and treated with cross-linking agents[J]. International Journal of Food Science and Nutrition,2011,62(4):410-417.
[3] Benjakul S,Phatcharat S,Tammatinna A,et al. Improvement of gelling properties of lizardfish mince as in-fluenced by microbial transglutaminase and fish freshness[J]. Journal of Food Science,2008,73(6):239-246.
[4] Agyare K K,Damodaran S. pH-stability and thermal properties of microbial transglutaminase-treated whey protein isolate[J]. Journal of Agricultural and Food Chemistry,2010,58(3):1946-1953.
[5] Zhu Y,Rinzema A,Tramper J. Microbial transglutaminase:A review of its production and application in food processing[J]. Applied Microbiology Biotechnology,1995,44(3):277-282.
[6] Seguro K,Nio N,Motoki M. Some characteristics of a microbial protein cross-linking enzyme:Transglutaminase[J]. ACS Symposium Series,1996,650:271-280.
[7] 周楠迪,田亚平,华子安,等. 微生物转谷氨酰胺酶的纯化方法和酶学性质研究[J]. 工业微生物,2004,34(3):20-23.
[8] Kobayashi K,Kumazawa Y,Miwa K,et al. ε -(γ -Glutamyl)lysine cross-links of spore coat proteins and transglutaminase activity in Bacillus subtilis[J]. FEMS Microbiology Letters,1996,144(2/3):157-160.
[9] Suzuki S,Izawa Y,Kobayashi K,et al. Purification and characterization of novel ansglutaminase from Bacillus subtilis spores[J]. Bioscience Biotechnology and Biochemistry,2000,64(11):2344-2351.
[10] Bernard B K,Tsubuku S,Shioya S. Acute toxicity and genotoxicity studies of a microbial transglutaminase[J]. International Journal of Toxicology,1998,17(6):703-721.
[11] Liu Y,Lin S,Zhang X,et al. A novel approach for improving the yield of Bacillus subtilis transglutaminase in heterologous strains[J]. Journal of Industrial Microbiology & Biotechnology,2014,41(8):1227-1235.
[12] 刘凯,刘逸寒,张艳,等. 枯草芽胞杆菌谷氨酰胺转氨酶的异源表达[J]. 天津科技大学学报,2012,27(3):1-5.
[13] Watanabe K,Tsuchida Y,Okibe N,et al. Scanning the Corynebacterium glutamicum R genome for highefficiency secretion signal sequences[J]. Microbiology,2009,155:741-750.
[14] Van der Rest M E,Lange C,Molenaar D. A heat shock following electroporation induces highly efficient transformation of Corynebacterium glutamicum with xenogeneic plasmid DNA[J]. Applied Microbiology Biotechnology,52(4):541-545.
[15] Yasueda H,Kumazawa Y,Motoki M. Purification and characterization of a tissue-type transglutaminase from red sea bream[J]. Bioscience Biotechnology & Biochemistry,1994,58(11):2041-2045.
[16] 萨姆布鲁克J,拉塞尔D W. 分子克隆实验指南[M]. 3版. 北京:科学出版社,2002:1230.
[17] 张毅,屈贤铭,杨胜利. 乳糖作为诱导剂对重组目的蛋白表达的影响[J]. 生物工程学报,2000,16(4):464-468.
责任编辑:郎婧
Expression of the Transglutaminase from Bacillus subtilis and Optimization of Fermentation Conditions for Corynebacterium glutamicum
HUANG Lin,LIU Yihan,LI Mingjie,LI Rui,GUO Wei,GUI Shuang,LU Fuping
(Key Laboratory of Industrial Fermentation Microbiology,Ministry of Education,Tianjin Key Laboratory of Industrial Microbiology,National Engineering Laboratory for Industrial Enzymes,College of Biotechnology,Tianjin University of Science & Technology,Tianjin 300457,China)
Overlap PCR was used to produce Sprodbtg encode with transglutaminase(BTG)from Bacillus subtilis.The recombined gene Sprodbtg contains signal peptide ΔS0949 from Corynebacterium glutamicum,SD sequence and the proDBTG gene.The sprodbtg was cloned into pXMJ19 to construct secretion expression vector pXMJ19-Sprodbtg.After that,the recombined plasmid was transformed into C.glutamicum ATCC13032 to express proD-BTG.The optimized fermentation conditions were also studied.The results showed that the recombined strain C.glutamicum ATCC13032/pXMJ19-Sprodbtg accumulated proD-BTG up to a level of (41.23±2.01)U/L.The optimized conditions for the induction of expression of the recombined protein proD-BTG in strain 13032/pXMJ19-Sprodbtg were as follows:growing in MMTG medium for 12 h,adding IPTG with a final concentration of 0.8 mmol/L and the subsequent incubation for 40 h.The activity of BTG reached (55.62±2.34)U/L,a 34.90%, increase compared with the non-optimized control.
transglutaminase;Corynebacterium glutamicum;Bacillus subtilis;secretion expression;condition optimization
Q814.4
A
1672-6510(2016)06-0011-06
10.13364/j.issn.1672-6510.20160044
2016-02-18;
2016-05-16
天津市应用基础与前沿技术研究计划资助项目(14JCYBJC23800);天津市科技特派员资助项目(15JCTPJC56500)
黄 琳(1984—),女,天津人,实验师;
路福平,教授,lfp@tust.edu.cn.
数字出版日期:2016−07−11;数字出版网址:http://www.cnki.net/kcms/detail/12.1355.N.20160711.1615.010.html.

