狼尾草属牧草遗传多样性的RAPD分析
刘伟民 乔良普 (山东省临沂市河东区畜牧兽医局 276000)
狼尾草属牧草遗传多样性的RAPD分析
刘伟民 乔良普 (山东省临沂市河东区畜牧兽医局 276000)
采用RAPD分子标记技术对狼尾草属10个品种(品系)间的遗传关系进行了分析。从100条RAPD引物中筛选出8条RAPD引物用于RAPD分析。8条RAPD引物共检测到70个位点,其中多态位点占88.57%,Nei’s基因多样度为0.3080,Shannon信息指数为0.5334; RAPD标记显示狼尾草属牧草具有丰富的遗传多态性。基于RAPD扩增构建相应的Nei’s(1979)遗传距离矩阵以及UPGMA树图。狼尾草属牧草品种(系)间遗传相似系数(GS)值为0.5102~0.9350。粗茎象草与美洲狼尾草的遗传相似系数最小,为0.5102,表明其亲缘关系最远;粗茎象草与未知品种的遗传相似系数最大,为0.9350,说明其亲缘关系最近。
狼尾草属牧草 RAPD 遗传多样性
狼尾草属(Pennisetum Rich.)牧草为一年生或多年生禾本科牧草,主要分布在热带和亚热带,全世界约有80种,多数原产于非洲[1]。目前我国人工栽培利用的种主要有多年生的象草(P. purpureum Schumach)和一年生的美洲狼尾草(P. americanum Rich.)及其种间杂交种。在发展草食动物生产中发挥了越来越重要的作用[2]。
RAPD(Random Amplified Polymorphic DNA,随机扩增多态性DNA)技术是1990年由Welsh and Mcclelland[3]和Williams[4]同时提出的一种DNA检测技术,是继RFLP技术之后发展起来的一种以PCR为基础的分子生物学技术。RAPD技术存在的局限性是:因其使用随机引物扩增,分子基础未知,致使扩增片段的基因来源不明了;反应的影响因素太多,稳定性差,各实验室的结果很难重复,因此必须严格控制条件[5,6]。目前,RAPD分子标记已经应用到了棉属[7]、石蒜属[8]、杜鹃属[9]、松毛虫属[10]、卷柏属[11]等植物。
1 材料与方法
1.1 试验材料
在福建省福州市和建阳市采集了10个品系共计45份的牧草样本,根据狼尾草属的分布状况选取具有代表性的样本。要求每个样本的株间距至少在50m以上,每株采摘的鲜嫩叶片都做好标记,塑料袋低温保存,立即拿到实验室,进行基因组总DNA的提取。植株的品系名、采摘地点、电泳泳道见表1。
1.2 试验方法
1.2.1 基因组DNA的提取及检测 本试验DNA的提取采用天根植物基因型DNA提取试剂盒,通过1%琼脂糖凝胶电泳和上海尤尼柯公司UV-2102紫外分光光度计检测DNA浓度和纯度。DNA浓度均调至50~100ng/μl,于-20℃冰箱内保存备用。
1.2.2 RAPD引物的筛选 RAPD引物购自上海生工生物工程公司。
1.2.3 RAPD反应条件 总体积为20μl,反应体系中含有1.5μl Mg2+(25mM)、0.4μl dNTPs(10mM)、1.2μl primer(10uM)、0.4μl Taq DNA Polymerase(5U/μl)、2.0μl 10×PCR Buffer、2.0μl DNA,加ddH2O补足到20μl;扩增反应在美国Bio-RAD公司Mycycler上进行,RAPD反应程序为:95℃ 预变性5min
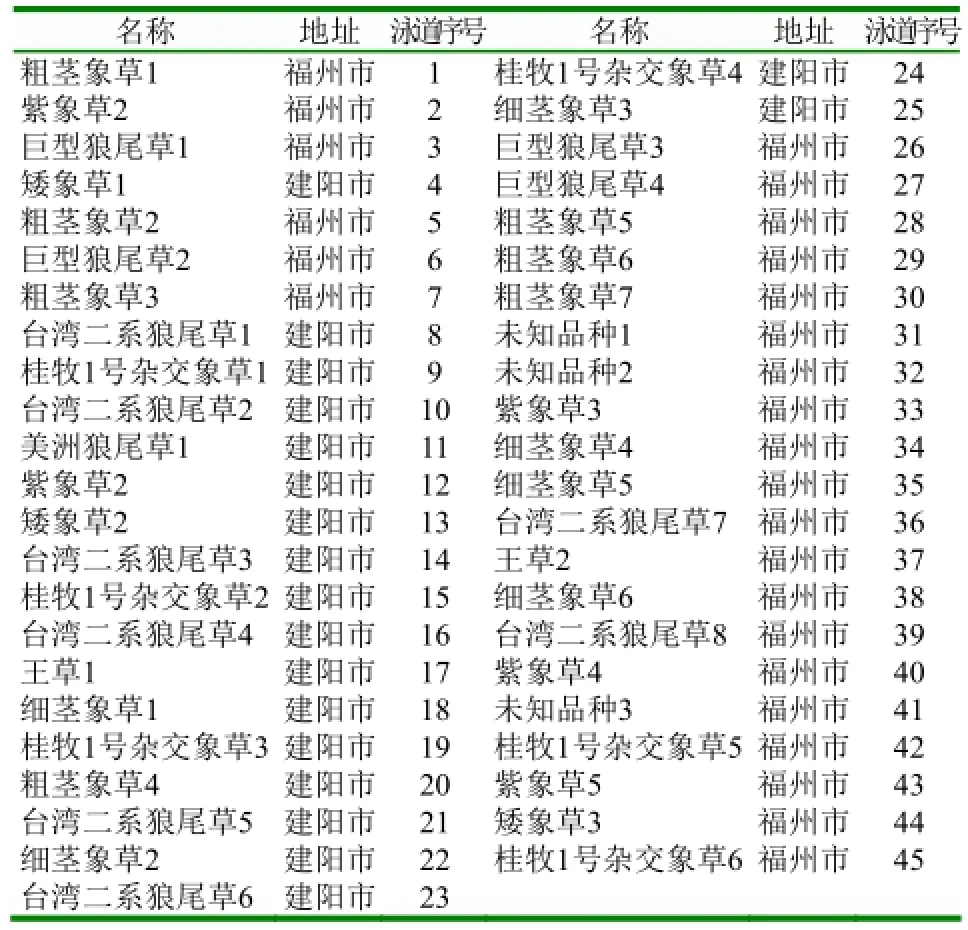
表1 狼尾草属样本的采集地点及数目

1.2.4 RAPD引物筛选 以台湾二系狼尾草2和矮象草1为材料筛选引物,共对上海生物工程有限公司生产的100条随机引物在20μL反应体系,10pM引物浓度下进行筛选,共筛选出8条引物能扩增出条带清晰稳定、明亮的条带。用筛选出的8条RAPD引物对45份材料进行PCR扩增,并对引物进行了统计分析。所使用引物的序号、序列及退火温度如表2。
1.2.5 PCR产物电泳检测 PCR产物经含EB1.4%琼脂糖凝胶电泳100V电压大约1h后,在凝胶成像系统上拍照。
1.2.6 数据处理和聚类分析 采用Popgen32,根据10个品系间的遗传相似度构建系统聚类图谱。采用DPSv6.85分析软件,通过类平均法UPGMA构建45个样本的系统聚类树状图。

表2 引物及测试的退火温度 (℃)
2 结果与分析
2.1 RAPD多态性分析
RAPD反应扩增的条带分子量一般在100~2000bp范围内。8个RAPD引物共扩增出70条带,平均每个引物扩增出8.75条带,具体如表3。

表3 狼尾草属RAPD标记的多态性分析
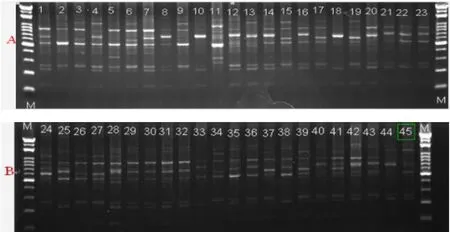
图1 (A-B)引物176在狼尾草属牧草材料中的RAPD图谱
2.2 狼尾草属牧草RAPD分子标记遗传差异的聚类分析

表4 10个品种遗传多样性水平(RAPD)

表5 10种狼尾草属牧草的遗传距离(对角线下)和遗传相似性(对角线上)
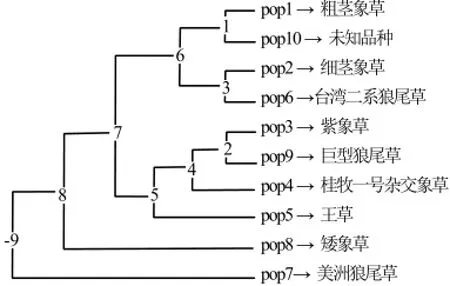
图2 狼尾草属牧草10个品种间RAPD分析的聚类图
2.3 狼尾草属种间的遗传变异
(1)利用POPGENE 32软件分析RAPD扩增结果,得到狼尾草属品系间遗传一致度与遗传距离矩阵与亲缘关系聚类图2,遗传距离(对角线下)和遗传相似性(对角线上)表5。结果显示:狼尾草属牧草品系间遗传距离在0.0672~0.6730范围之内,其中粗茎象草与美洲狼尾草的遗传距离最大为0.6730,粗茎象草与未知品系间的遗传距离最小,只有0.0672。10种狼尾草属植物可明显地分为两类:一类包括粗茎象草、细茎象草、紫象草、桂牧一号杂交象草、王草、台湾二系狼尾草、矮象草、巨型狼尾草、未知品种;另一类只包括美洲狼尾草。对第一类又可细分为三类:矮象草单独聚为一类;粗茎象草、细茎象草、未知品种、台湾二系狼尾草聚为第二小类;紫象草、桂牧一号杂交象草、王草、巨型狼尾草聚为第三小类。(2)依据RAPD扩增结果,狼尾草属牧草品系间遗传相似系数(GS)值为0.5102~0.9350。粗茎象草与美洲狼尾草的遗传相似系数最小,为0.5102,表明它们之间的亲缘关系最远;粗茎象草与未知品种的遗传相似系数最大,为0.9350,说明其亲缘关系最近。
3 讨论与小结
狼尾草作为一种重要的牧草资源,在世界各地广泛种植,并选育出许多品种,这使得狼尾草的遗传分化越来越多样,这种情况下单靠传统的分类法已经很难识别。DNA 指纹技术由于能鉴定出新品种的一致性和特异性,在植物新品种保护中发挥了重要作用[12]。基于RAPD 分子标记技术建立的指纹图谱也能够对特异性DNA 片段及种族特异性差异进行鉴定。
3.1 狼尾草属DNA提取质量
DNA的提取是PCR反应的第一步,DNA提取的质量直接影响到后续反应的进行。本试验提取的DNA纯度高,A260nm/A280nm值在1.8左右,没有RNA及蛋白质污染。ISSR-PCR和RAPD-PCR扩增出来的条带清晰明亮,辨别率高,完全满足RAPD和ISSR反应的需要。
3.2 RAPD标记揭示的遗传参数
由RAPD扩增结果计算出的Nei’s基因多样性指数和Shannon信息指数较高,多态性条带百分率为88.57%。RAPD分析方法可检测到较多的遗传多态性信息。Shannon多样性指数是使用某一扩增产物的存在频率作为该位点的表型频率来计算遗传多样性[13]。本研究由RAPD标记计算出的狼尾草属牧草的Shannon多样性指数分别为0.4541,Nei’s遗传多样性指数分别为0.3086,Nei’s遗传多样性指数的变化趋势与Shannon多样性指数估测的基本相同,只是Shannon多样性指数估计值有些偏大,这主要是统计方法不同造成的。因此,采用这种指数估计遗传多样性都是可行的。

表6 狼尾草属10个品系间的遗传变异分析
由表6可以得知:RAPD标记分析10个狼尾草属品系间基因分化系数分别为59.17%,而品系间基因分化系数表示品系间遗传变异占总遗传变异的百分比,因此可以判定,狼尾草属牧草的遗传变异主要存在于品系间。还有应用RAPD分子标记方法计算出的基因流分别为0.3450,小于1,表明10个品系间的基因交流还是有限的。
[1] 林洁荣, 刘建昌. 福建南亚热带狼尾草属牧草品比试验[J]. 福建农林大学学报(自然科学版), 2003, 32(1): 110-112.
[2] 卓坤水, 苏水金, 杜仲清等. 闽引象草(Pennisetum purpureum schum MIN-YIN)的选育[J]. 热带作物学报, 2009, 30(8): 1196-1200.
[3] Welsh J, Mclelland M.Fingerprinting genomes using PCR with arbitrary primers[J]. Nucleic Acids Research, 1990(18): 7213-7218.
[4] Williams J. G.K,Kubelik A.R., Livak K.L, et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research, 1990(18): 6531-6535.
[5] 汪小全, 邹喻苹, 张大明. RAPD应用于遗传多样性和系统学研究中的问题[J]. 植物学报, 1996, 38(12): 954-962.
[6] 汪永庆, 徐来祥, 张知彬. RAPD技术的标准化问题[J]. 动物学杂志, 2000, 35(4): 57-60.
[7] 吴玉香. 棉属遗传多样性与栽培棉种间杂交研究[D]. 浙江大学, 2007.
[8] 袁菊红. 中国石蒜属(LycorisHerb.)种间亲缘关系与居群分子标记研究[D]. 南京农业大学, 2007.
[9] 周兰英. 杜鹃属植物亲缘关系及遗传多样性研究[D]. 四川农业大学, 2008.
[10] 南宫自艳. 松毛虫属部分种类遗传多样性及影响因素研究[D].河北农业大学, 2008.
[11] 万定荣. 湖北省卷柏属药用植物的鉴定和品质研究[D]. 湖北中医学院, 2006.
[12] 李晓辉, 李新海, 张世煌. 植物新品种保护与DUS测试技术[J].中国农业科学, 2003, 36(11): 1419-1422.
[13] Wang Q Y, Ren X Q,Jiang J. Genetic diversity for the provenance of Piceakor analysis by RAPD markers[J]. Journal of Northeast Forestry University, 2004, 32(6): 1-3.
S816.5+1
A
1007-1733(2016)05-0005-03
2016–01–29)

