水稻粒形性状QTL定位分析
彭 强,张大双,吴健强,张玉珊,王际凤,黄培英,朱速松*
(1.贵州省水稻研究所,贵州 贵阳 550006;2.贵州省生物技术研究所,贵州 贵阳 550006)
水稻粒形性状QTL定位分析
彭 强1,张大双1,吴健强1,张玉珊2,王际凤1,黄培英1,朱速松1*
(1.贵州省水稻研究所,贵州 贵阳 550006;2.贵州省生物技术研究所,贵州 贵阳 550006)
为稻米品质分子育种改良提供新的辅助选择标记和理论依据,以籼爪交(籼稻×爪哇稻)组合V20B/CPSLO17衍生的150份重组自交家系(RIL)为作图群体,结合RIL群体的粒形性状表型数据,运用QTL IciMapping 4.0软件进行粒形性状QTL检测及其遗传效应分析。结果表明,在7条染色体(1,2,3,4,5,7,10)上共检测出5个粒长性状QTLs(qGL-1,qGL-2,qGL-3,qGL-7,qGL-10)、3个粒宽性状QTLs (qGW-2,qGW-4,qGW-5)和4个长宽比性状QTLs(qLWR-1,qLWR-3,qLWR-5,qLWR-7)。第3染色体的Marker910209-Marker823024标记区间对粒长和长宽比具有较大遗传效应,表型变异贡献率分别为21.37 %和28.35 %;第5染色体的Marker1642127-Marker1514505标记区间对粒宽和长宽比具有较大遗传效应,表型变异贡献率分别为29.71 %和17.42 %。
籼稻; 外观品质; 分子标记; 连锁图谱; 数量性状座位
随着生活水平的提高,稻米品质越来越受到人们的重视,稻米品质改良已成为水稻育种领域的重点和热点。稻米品质通常包括外观品质、碾磨品质、蒸煮与食味品质和营养品质4个方面。其中,外观品质直接体现着稻米商品属性的优劣,而粒形(包括粒长、粒宽、粒厚和长宽比)是衡量稻米外观品质优劣的重要指标[1]。因此,开展稻米粒形性状QTL研究对稻米品质分子改良具有重要的理论和应用价值。
近年来学者们运用新兴的分子生物技术已获得200多个粒形(粒长、粒宽和长宽比)性状QTLs,每条染色体都分布着多个粒形QTLs[2],但目前已克隆的粒形性状主效控制基因不多[3-10]。GL7[3-4]基因位于第7染色体上,是一个控制粒长和粒宽的主效QTL,也影响长宽比;GW2[6]和GW5[7]基因是粒宽主效控制基因;PGL1[8]和PGL2[9]基因位于同一位点,都是粒长正调节子,且两者存在功能冗余。上述QTL定位研究多以籼籼交(籼稻×籼稻)、籼粳交(籼稻×粳稻)组合衍生的遗传群体为研究材料,鲜有利用籼爪交(籼稻×爪哇稻)组合构建重组自交系进行稻谷粒形性状QTL定位的报道。为此,笔者以广亲和性的爪哇稻品种CPSLO17和配合力强的籼稻品种V20B作为亲本材料,构建遗传作图群体,进行稻谷粒形性状(粒长、粒宽和长宽比)QTL定位分析,旨在为稻米品质分子育种改良提供新的辅助选择标记和理论依据。
1 材料与方法
1.1 试验材料
研究所用的RIL群体是由V20B/CPSLO17杂交F1代通过单粒传法得到的F10代。2014年夏季在贵州省水稻研究所(贵阳)试验田种植150份RIL群体,每份材料种植2行,每行10株,水稻成熟期随机选取10株混合收种。
1.2 稻谷粒形性状考查
稻谷晾干贮藏 3 个月后(含水量13 %左右),进行稻谷粒形性状统计分析。
稻谷粒长(Grain length,GL)测定:随机挑取10粒饱满的种子首尾相接,排成一行,不重叠,不留隙,用电子游标卡尺测量10粒种子总长度,重复3次,取平均值。
粒长(mm)=10粒种子总长度平均值/10
稻谷粒宽(Grain width,GW)、粒厚(Grain thickness,GT)测定:随机挑选10粒饱满种子,以宽度最宽处或厚度最厚处摆成一排,不重叠,不留隙,分别测量10粒种子总宽度和总厚度,重复3次,取平均值。
粒宽(mm)=10粒种子总宽度平均值/10
粒厚(mm)=10粒种子总厚度平均值/10
根据粒长和粒宽计算稻谷的长宽比(length-width ratio,LWR)。
1.3 QTL分析
研究所用的遗传连锁图谱及其相关分子数据(未发表)是由北京百迈客生物科技有限公司利用SLAF-seq(Specific-Locus Amplified Fragment Sequencing)技术[11]和HighMap 软件[12]开发获得。该图谱覆盖水稻全基因组2508.65 cM,共有8602个多态性SLAF标签较均匀地分布在12条染色体上,标记间平均距离为0.292 cM。结合RIL群体粒形表型数据,利用软件IciMapping 4.0的ICIM-ADD方法进行QTL分析,扫描步长设定为0.1 cM,LOD值设定为3.0,并计算每个 QTL的贡献率和加性效应,QTL的命名原则遵循McCouch等[13]提出的方法。加性效应为正值表示增效等位基因来源于亲本V20B,负值表示来源于亲本CPSLO17。
2 结果与分析
2.1 群体中谷粒的粒形性状
2.1.1 粒形表型值 从150份RIL群体的粒形性状(粒长、粒宽、粒厚和长宽比)表型数据分析结果(表1)可知,RIL群体稻谷粒长的均值为(9.1455±0.864)mm,变异幅度为7.22~11.56 mm;稻谷粒宽均值为(2.9063±0.2444)mm,变异幅度为2.2667~3.4833 mm;稻谷粒厚均值为(2.0005±0.2873)mm,变异范围为1.4133~4.02 mm;群体稻谷长宽比的均值为3.172±0.4279,变异范围为2.2833~4.7967 mm。
2.1.2 粒形指标分布 4个稻米品质性状在RIL群体中的分布频率如图1所示,RIL群体的粒长、粒宽、粒厚和长宽比在各自的变异范围内均呈连续分布,都表现出受多基因控制的数量性状遗传特征。
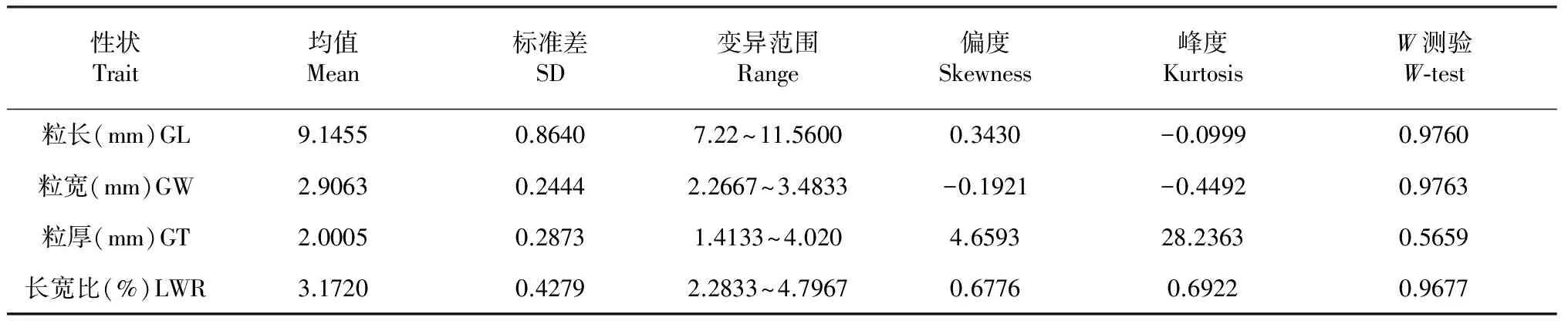
表1 RIL 群体中谷粒性状的表现

表2 RIL 群体粒形性状的相关系数
注:GL: 粒长,GW: 粒宽,GT: 粒厚,LWR: 长宽比.; **表示极显著水平(P<0.01), *表示显著水平(P<0.05)。

图1 粒形性状在RIL群体中的分布Fig.1 The distribution of grain shape trait in RIL population
2.2 群体粒形性状间的相关性
4个粒形性状间的相关性分析结果(表2)表明,粒长与长宽比,粒长与粒厚、粒厚与长宽比呈正相关,粒长与粒宽、粒宽与长宽比、粒宽与粒厚呈负相关。t检验结果表明,4个性状间6组相关性分析的P值都小于0.01,达极显著水平。
2.3 粒形性状的QTL定位
利用软件IciMapping 4.0的ICIM-ADD方法进行品质性状QTL分析,共检测到12个外观品质性状相关QTLs,LOD 值在3.28~16.60,贡献率( percentage of variance explained,PVE)变幅为4.39 %~29.71 %(表3)。5个与粒长性状相关的QTLs(分别命名为qGL-1、qGL-2、qGL-3、qGL-7和qGL-10),分别位于第1、2、3、7和10染色体上,其表型贡献率变幅为4.39 %~21.37 %,其中qGL-3(LOD=14.59)的贡献率最高(21.37 %)。3个与粒宽性状相关的QTLs(分别命名为qGW-2、qGW-4和qGW-5),分别位于第2、4和5染色体上,其表型贡献率变幅为6.51 %~29.71 %,其中,qGW-5(LOD=12.61)对表型贡献率最高(29.71 %)。4个与长宽比相关的QTLs(分别命名为qLWR-1、qLWR-3、qLWR-5和qLWR-7),分别位于第1、3、5和7染色体上,其表型贡献率变幅为6.73 %~28.35 %,其中,qLWR-3(LOD=16.60)对表型贡献率最高(28.35 %)。

表3 稻米粒形QTL定位及其遗传效应

图2 粒形性状QTL在遗传图谱上的位置分布Fig.2 The QTL position of grain shape trait in genetic mapping
研究检测到的12个QTLs分布在7条染色体上(图2),第4和10染色体各有1个QTLs,而第1、2、3、5和7染色体上各有2个不同性状的QTLs,其中qLWR-3和qGL-3 QTLs在遗传图谱上的位置很近,均位于Marker910209~Marker823024标记之间;qGW-5和qLWR-5 QTLs在遗传图谱上的位置很近,均位于Marker1642127~Marker1514505标记之间。
3 结论与讨论
大量研究表明,数量性状具有复杂的遗传背景和广泛的遗传多样性,不同遗传背景的作图群体、不同种植环境得到的QTL分析结果不同[14-16]。本研究利用籼爪交(籼稻×爪哇稻)组合衍生的RIL群体进行粒形QTL定位研究,共检测到10个影响粒形性状的染色体区域,其中,第3染色体短臂Marker910209~Marker823024标记区间和第5染色体短臂Marker1642127~Marker1514505标记区间是粒形主效QTL的重点区域,这与前人研究结果相一致[17-19]。第3染色体Marker910209~Marker823024区间内检测出控制粒长和长宽比的主效QTL(qGL-3和qLWR-3),贡献率分别为21.37 %和28.35 %,其增效等位基因均来自于亲本V20B;第5染色体Marker910209~Marker823024区间检测出控制粒宽和长宽比的主效QTL(qGW-5和qLWR-5),贡献率分别为29.71 %和17.42 %,其增效等位基因分别来自于亲本V20B和CPSLO17。
研究结果表明,粒形性状间广泛存在着一因多效,且性状间(如粒长与长宽比,粒宽与长宽比之间)存在遗传相关性,这与目前大多数研究结论一致[2]。此外,根据NCBI(http://www.ncbi.nlm.nih.gov/)和RiceVarMap(http://ricevarmap.ncpgr.cn/)网站相关信息,选择MSU/TIGR Rice Psedomolecule Release 7.0进行blast比对分析发现,Marker1642127~Marker1514505区间覆盖了粒宽基因GW5[7];而Marker910209~Marker823024区间与粒形基因GS3[10]无关,两者所对应的物理位置(MSU/TIGR 7.0)相距约37 kb,表明该区间的粒形QTL极可能是一个新的粒形主效控制基因,且具有一因多效功能,这需要后续工作进一步探索验证。本研究结果不仅为克隆粒形控制基因和开发相关分子标记提供参考,也展现了不同稻种资源在水稻性状基因及其育种改良研究中的潜在价值。
[1]Juliano B O. Rice chemistry and technology[M]. American: Association of Cereal Chemists, 1985.
[2]康美花, 王 丰,贺浩华. 稻米外观品质性状的遗传与标记定位研究进展[J]. 广东农业科学, 2010(5):168-170.
[3]Wang S, Li S, Liu Q, et al. The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]. Nat Genet, 2015, 47(8):949-954.
[4]Wang Y, Xiong G, Hu J, et al. Copy number variation at the GL7 locus contributes to grain size diversity in rice[J]. Nat Genet, 2015, 47(8):944-948.
[5]Shomura A, Izawa T, Ebana K, et al. Deletion in a gene associated with grain size increased yields during rice domestication[J]. Nat Genet, 2008, 40(8):1023-1028.
[6]Song X J, Huang W, Shi M, et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nat Genet, 2007, 39(5):623-630.
[7]Weng J F, Gu S H, Wan X Y, et al. Isolation and initial characterization of GW5, a major QTL associated with rice grain width and weight[J]. Cell Research, 2008, 18(12):1199-1209.
[8]Heang D ,Sassa H. An atypical bHLH protein encoded by POSITIVE REGULATOR OF GRAIN LENGTH 2 is involved in controlling grain length and weight of rice through interaction with a typical bHLH protein APG[J]. Breed Sci, 2012, 62(2):133-141.
[9]Heang D ,Sassa H. Antagonistic actions of HLH/bHLH proteins are involved in grain length and weight in rice[J]. PLoS One, 2012, 7(2):e31325.
[10]Fan C, Xing Y, Mao H, et al.GS3, a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]. Theor Appl Genet, 2006, 112(6):1164-1171.
[11]Sun X, Liu D, Zhang X, et al. SLAF-seq: an efficient method of large-scale de novo SNP discovery and genotyping using high-throughput sequencing[J]. PLoS One, 2013, 8(3):e58700.
[12]Liu D, Ma C, Hong W, et al. Construction and analysis of high-density linkage map using high-throughput sequencing data[J]. PLoS One, 2014, 9(6):e98855.
[13]MaCouch S R, Cho Y G, Yang M, et al. Report on QTL nomenclature[J]. Rice Genet Newslett, 1997, 14:11-13.
[14]杨 莉, 蒋开锋, 涛 张, 等. 水稻再生力与产量性状的 QTL 分析[J]. 西南农业学报, 2013, 26(6):2179-2184.
[15]强 彭, 张大双, 吴健强, 等. 水稻苗期耐冷性的QTL定位分析[J]. 贵州农业科学, 2015, 43(5):11-13.
[16]俊 严, 杨荣志, 薛文韬, 等. 不同生态环境下四倍体小麦千粒重的QTL分析[J]. 贵州农业科学, 2011, 39(11):6-10.
[17]梅德勇, 朱玉君,樊叶杨. 籼稻稻米碾磨与外观品质性状的 QTL 定位[J]. 遗传, 2012, 34(12):1591-1598.
[18]Lou J, Chen L, Yue G H, et al. QTL mapping of grain quality traits in rice[J]. Journal Of Cereal Science, 2009, 50(2):145-151.
[19]Zheng T Q, Xu J L, Li Z K, et al. Genomic regions associated with milling quality and grain shape identified in a set of random introgression lines of rice (OryzasativaL.)[J]. Plant Breeding, 2007, 126(2):158-163.
(责任编辑 姜 萍)
QTL Analysis of Grain Shape Trait of Rice
PENG Qiang1, ZHANG Da-shuang1, WU Jian-qiang1, ZHANG Yu-shan2, WANG Ji-feng1, HUANG Pei-ying1, ZHU Su-song1*
(1.Guizhou Rice Research Institute, Guiyang Guizhou 550006, China; 2.Guizhou Institute of Bio-technology, Guiyang Guizhou 550006, China)
A mapping population of 150 lines (recombination inbred lines, RIL), derived from a cross between rice varieties V20B and CPSLO17, and their grain shape phenotype data were used to analyze QTLs location and evaluate their genetic effects by using ICIM-ADD method of IciMapping 4.0 software. The results showed that a total of 12 QTLs for grain shape trait were detested, of whcih 5 for grain length (namedqGL-1,qGL-2,qGL-3,qGL-7 andqGL-10, respectively), 3 for grain width (namedqGW-2,qGW-4 andqGW-5, respectively), 4 for length/width ratio (namedqLWR-1,qLWR-3,qLWR-5 andqLWR-7, respectively). These QTLs distributed on chromosomes 1, 2, 3, 4, 5, 7 and 10. A QTLs cluster with major effects on grain length and length/width ratio was located in the Marker910209-Marker823024 interval on chromosomes 3. The phenotypic variances were explained by this QTLs were 21.37 % and 28.35 %, respectively. QTLs control grain width and length/width ratio were clustered in the Marker1642127-Marker1514505 region of chromosomes 5, which contributed 29.71 % and 17.42 % to the phenotypic variances, respectively.
Indicarice; Appearance quality; Molecular marker; Linkage map; Quantitative trait locus
1001-4829(2016)08-1766-05
10.16213/j.cnki.scjas.2016.08.002
2015-10-10
贵州省联合基金项目“水稻高密度遗传图谱构建和垩白QTL定位分析”[黔科合LH字(2014)7689]
彭 强(1986-),男,助理研究员,硕士,从事水稻分子育种研究,E-mail:450058876@163.com,*为通讯作者。
S511
A

