基于COⅠ基因分析辽东湾海蜇群体遗传多样性
李玉龙,王 彬,王文波,董 婧
( 辽宁省海洋水产科学研究院,辽宁省海洋生物资源与生态学重点实验室,辽宁 大连 116023 )
基于COⅠ基因分析辽东湾海蜇群体遗传多样性
李玉龙,王 彬,王文波,董 婧
( 辽宁省海洋水产科学研究院,辽宁省海洋生物资源与生态学重点实验室,辽宁 大连 116023 )
为研究海蜇群体的遗传多样性状况,采用PCR扩增获得20个海蜇野生群体样品的线粒体COⅠ基因部分序列,并结合GenBank上其他15个海蜇样品的同源序列,对其序列变异和遗传分化进行分析。结果显示,35条序列的A、T、C、G碱基含量分别为26.7%、36.4%、18.8%、18.1%;在长度624 bp的线粒体COⅠ基因片段中共发现21个多态位点,定义了17种单倍型,单倍型多态性为0.91±0.03,核苷酸多态性为0.0056±0.0032。同其他几种大型水母相比,海蜇种群的遗传多样性处于较高或中等水平。研究结果表明,不同海蜇种群尤其是相距较远的群体在其分布范围内可能存在遗传分化现象。
海蜇;线粒体DNA;COⅠ基因;遗传多样性
海蜇(Rhopilemaesculentum)是经济价值最高的一种大型食用水母且兼有药用功效,属于海产八珍之一,是我国重要的海洋渔业资源。海蜇在分类学上隶属刺胞动物门、钵水母纲、根口水母目、根口水母科、海蜇属。该物种为暖水性河口种类,适应的水温和盐度范围较广,曾广泛分布于我国北至鸭绿江口,南至北部湾近岸浅海水域,且在日本西部、朝鲜半岛南部和俄罗斯远东地区也有分布[1-4]。在我国从南到北沿海一带,曾有多个地理种群,如粤东、闽南、闽东、浙南、杭州湾、海州湾、莱州湾、渤海湾和辽东湾等群体。近十年来,由于人为及自然原因,加上灾害水母频发,海蜇自然资源量呈显著下降趋势,海蜇的多个地理种群可能已不复存在,如黄海北部的海蜇群体自1986年后已经逐渐消失,渤海湾、莱州湾、浙江近海、福建近海等海域曾经丰富的海蜇资源也已经逐渐衰退[1,5]。
目前,对海蜇的研究主要集中在形态学分类、生活史、生态及增殖放流等[1,5-14]方面,对海蜇的生态习性、种群特征,资源管理、苗种培育、池塘养殖、沿岸增殖等领域进行了较为系统全面的研究。但迄今为止,有关海蜇自然群体的遗传变异及群体间遗传分化等方面的研究尚不多,所用的分子标记主要是核基因分子标记如扩增片段长度多态性和内转录间隔区序列。高祥刚等[15]采用扩增片段长度多态性技术研究了辽宁沿海海蜇和沙蜇(Nemopilemanomurai)野生群体和养殖群体的遗传多样性,发现辽宁沿海的海蜇和沙蜇遗传多样性仍处于较高水平。乔洪金等[16]同样采用扩增片段长度多态性技术对蓬莱、江苏2个海蜇捕获群体以及江苏2个海蜇养殖群体共56个个体的遗传多样性进行了分析,结果同样表明海蜇遗传多样性水平较高,但并未发现海蜇不同群体间存在遗传分化现象。孙国华等[17]利用内转录间隔区序列对烟台莱州湾、江苏海州湾海蜇捕获群体和威海养殖群体共24个个体进行的序列分析亦表明海蜇群体间无明显的遗传分化,但遗传多样性水平较低。虽然上述研究为海蜇群体的遗传变异提供了一定的参考,但所用的分子标记或者样品数量均相对较少,而要全面了解这一物种的遗传多样性背景,还需利用更多的遗传标记方法对更多的海蜇样品进行研究。
线粒体DNA因具有结构简单、无重组、进化速率较快且多数母性遗传等特点,已成为进行群体遗传多样性分析的理想研究手段,被广泛应用于海洋动物群体遗传学研究中。线粒体CO Ⅰ基因和核糖体内转录间隔区基因是研究水母类遗传多样性及其遗传结构常用的分子标记,利用两种分子标记对几种钵水母类如肺状根口水母(Rhizostomapulmo)和夜光游水母(Pelagianoctiluca)的研究结果表明,线粒体CO Ⅰ基因片段比内转录间隔区序列具有更多的变异位点,更适合作为检测钵水母类群体遗传多样性及遗传分化的分子标记[18-19]。目前,尚未见利用线粒体DNA作为分子标记对海蜇进行遗传多样性分析的相关报道。本研究以辽东湾野生海蜇群体为研究对象,对其CO Ⅰ基因片段进行比较分析,并与GenBank数据库中不同海域的海蜇同源序列进行比较,了解不同海域海蜇群体的遗传背景及遗传分化等情况,以期为海蜇遗传育种及种质资源保护提供科学依据。
1 材料与方法
1.1 样品采集
因野生海蜇样品数量较少难以采集,本研究仅对20个海蜇样品进行分析,其中19个取自2012年6—8月进行的辽东湾海蜇跟踪监测调查中所获得的样品(取样站位见图1黑色圆点所示),每个站位随机取样(1~5个样品不等),伞径40~440 mm,剩余1个样品为2013年取自韩国江华岛附近海域。样品采集后取伞部及肩板组织于95%酒精中保存备用。

图1 辽东湾海蜇样品取样站位
1.2 DNA提取、扩增及测序
采用CTAB法提取基因组DNA,PCR扩增在Eppendorf AG型PCR仪上进行,扩增引物序列均为:FFDL 5′-TTTCAACTAACCAYAAAGAYATWGG-3′ 和FRDL2 5′-TANACTTCWGGRTGNCCRAAGAATCA-3′[20]。25 μL反应体积,包括:0.2 mmol/L每种dNTPs,0.2 μmol/L每种引物,1 μLDNA模板,1 UTaq,2.0 mmol/L MgCl2,2.5 μL 10×缓冲液,灭菌超纯水补足剩余体系。反应程序:95 ℃预变性3 min后,95 ℃变性30 s,51 ℃退火30 s,72 ℃延伸45 s,运行35个循环,最后72 ℃延伸5 min。扩增产物纯化后进行双向测序(上海英潍捷基有限公司)。
1.3 数据分析
将测定的COⅠ序列进行拼接和人工校对,并通过将COⅠ基因序列翻译成氨基酸序列来证实其准确性和排除假基因干扰。从GenBank数据库中下载了15条海蜇样品的同源序列与本研究所获得的序列进行分析,根据GenBank数据库中信息按样品的地理来源将海蜇划分为3个群体,采自日本海的3个样品HF930514-15、KF840718归于日本海群体(JP),辽东湾19个海蜇样品及2个采自渤海的样品EU373722-23属于渤海群体(BH),采自韩国的1个样品与采自黄海的6个样品EU3737224、JX845347、HF536571-74归为黄海群体(HH),另有4条序列HF930546、HF930550、HF937282、HF937287为采自于中国的海蜇样品,未标明采集地,最后分子变异分析时将其归为中国组群进行计算(表1)。利用DnaSP v5[21]软件确定单倍型。单倍型数目、多态位点、单倍型多态度、核苷酸多态度等分子多样性指数由Arlequin 3.01[22]软件计算获得。通过MEGA 3.0[23]计算碱基含量及单倍型间的遗传距离。使用Arlequin 3.01软件中的分子变异分析[24]来评估群体间遗传变异,其显著性通过1000次重复抽样来检验,群体间的遗传距离采用Kimura2-parameter模型计算。
利用Mega 3.0软件[23]构建海蜇群体COⅠ单倍型的邻接系统树,系统树的可靠性采用1000次重复抽样进行评估。此外,为探讨海蜇单倍型的谱系结构,采用中介网络法[25]构建单倍型网络关系图。
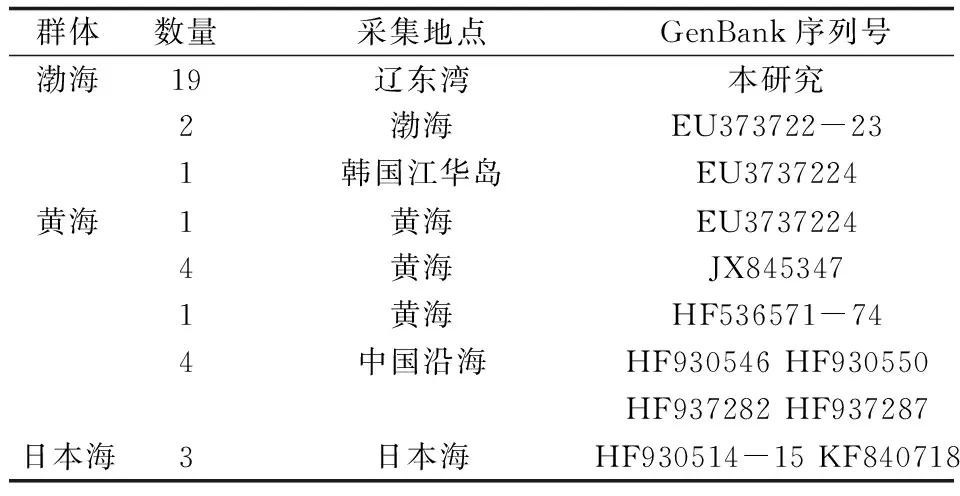
表1 试验用海蜇样品的采集数量、地点及引用序列登录号
2 结果与分析
2.1 碱基组成及序列变异
经比对去掉两端引物本研究分析的海蜇CO Ⅰ基因同源序列长度为624 bp,其A、T、G、C 4种碱基的平均含量分别为26.7%、36.4%、18.8%、18.1%,A+T(63.1%)含量明显高于G+C(36.9%)含量。624个位点中共检测到21个多态位点,包括9个简约信息位点和12个单态核苷酸变异位点。大部分突变(86%)发生在密码子第3位上,有3个突变发生在密码子第1位,所有突变位点均未导致氨基酸的改变。21个变异位点共定义了17种单倍型,定义为单倍型1~17,单倍型间遗传距离为0.2%~1.5%,平均遗传距离0.7%。单倍型6、9、10、11、14是海蜇群体的共有单倍型,其中单倍型6、9、10为主体单倍型,其所占频率分别为17%、23%、14%。除此之外,其他单倍型均为各群体特有单倍型。单倍型频率及其在3个群体中的分布见表2。
计算了海蜇不同群体及总群体的单倍型多样性指数和核苷酸多样性指数,其中因日本海海蜇样品数量太少(n=3),未计算该群体的多样性指数,同时根据已有资料比较了海蜇与其他几种大型水母的相关遗传多样性参数(表3),结果表明,海蜇群体的COⅠ变异水平较高,除夜光游水母外,与沙蜇或肺状根口水母相比,海蜇的遗传多样性水平最高。

表2 海蜇变异位点及单倍型在各群体中的分布

表3 海蜇与其他3种大型水母COⅠ基因遗传多样性参数比较
2.2 群体间遗传分化
基于COⅠ基因对海蜇3个群体的分子变异分析表明,80.71%的差异属于群体内差异,群体间差异为19.29%,遗传分化指数为0.1929且差异显著(P=0.005),说明海蜇群体间有较明显的遗传分化(表4)。进一步将海蜇分为两组,中国沿海海蜇样品为一组,日本海海蜇样品为另一组,计算结果表明遗传分化指数为0.418且差异极显著(P=0)。

表4 海蜇群体的AMOVA分析
2.3 分子系统分析
利用Mega 3.0软件构建了海蜇的单倍型邻接关系树(图2)。由图2可见,邻接关系树分成2个明显的进化枝,其中来自日本海的2个单倍型首先聚为一支,再与来自渤海的7个单倍型以及韩国的1个单倍型聚为类型Ⅰ;而来自黄海的3个单倍型和渤海剩余的单倍型则聚为类型Ⅱ。

图2 海蜇单倍型邻接关系树
采用中介网络法构建了海蜇单倍型的网络关系图(图3)。与单倍型邻接关系树的拓扑结构一致,单倍型网络关系图显示出2个明显的单倍型谱系分支,主体单倍型6和9、10分别位于2个分支Ⅰ和Ⅱ的中心,其他单倍型通过一步或多步突变分别与主体单倍型相连。

图3 海蜇单倍型的中介网络圆圈面积表示单倍型的频率,数字表示突变位点
3 讨 论
3.1 海蜇群体COⅠ基因遗传多样性分析
海蜇曾是我国近海重要的渔业资源,全国的海蜇渔业最高产量曾高达581 930 t[5],但由于过度捕捞及海洋生态环境破坏等不利因素影响,近年来海蜇资源量呈显著下降趋势。近几年,辽宁、山东、江苏等省份均在其传统的海蜇渔场开展了海蜇生产性放流工作,取得一些效果,增加了海蜇产量。但是,有研究表明,人工增殖放流在增加资源量的同时会污染原物种自然群体的遗传基因,破坏遗传结构,造成种群遗传多样性降低。本文基于线粒体CO Ⅰ基因部分片段的研究结果显示,与其他几种大型水母相比较,海蜇群体的遗传多样性处于中等或较高水平,这与用扩增片段长度多态性方法[15-16]对海蜇群体进行研究得出的研究结果相一致,而与孙国华等[17]利用内转录间隔区序列对烟台莱州湾、江苏海州湾群体和威海养殖群体得出的结果不同。基于内转录间隔区序列对海蜇群体进行的分析表明海蜇群体的遗传多样性水平较低,这一方面可能是由于内转录间隔区序列变异较少所致,另一方面其试验样品取自放流海蜇的捕捞群体,也会导致所研究群体的遗传多样性水平较低。这种现象在本研究中也有体现,海蜇渤海群体的遗传多样性参数明显高于黄海群体,其中渤海的海蜇样品大多为采自辽东湾的野生海蜇群体,而黄海的海蜇样品大多为捕捞的放流海蜇样品,因此遗传多样性水平较低。
3.2 海蜇群体的遗传分化
分子变异分析的结果表明,在本研究中渤海海蜇群体和GenBank数据库中样品来自黄海的海蜇群体和日本海的海蜇群体间存在群体遗传结构,其遗传分化指数为0.1929且差异检验的结果显著(P=0.005),进一步将海蜇群体分成中国组群和日本族群两组后计算的遗传分化指数为0.418,且差异极显著(P=0),这表明相距较远的海蜇地理种群间具有明显的遗传分化,这与利用扩增片段长度多态性技术[16]和利用内转录间隔区序列[17]分析黄、渤海海蜇群体发现群体间无明显遗传分化的结果不一致。系统树与单倍型最小跨度树的结果亦表明,来自渤海的海蜇群体和黄海、日本海的海蜇群体间具有2个明显的单倍型谱系分支,其中日本海的单倍型全部属于类型Ⅰ,黄海的单倍型大部分属于类型Ⅱ,这说明海蜇不同地理群体间尤其是相距较远的群体间可能存在遗传分化。海蜇是河口性近海种类,具有水螅体世代、水母体世代交替出现的世代交替生活史,海蜇水母体世代生命周期一般为3—5个月,为季节性的浮游生物,而其水螅体世代生命周期至少为7—9个月,如果条件适宜甚至可以存活几年时间,占据其生活史大部分的阶段[1],水螅体阶段主要营固着生活,这种独特的生活习性决定了其移动能力十分有限,主要是随海流的作用随波逐流。因此,相距较远的地理群体间由于长期的隔离分化会产生明显的群体遗传结构,这种现象也存在于马赛克水母(Catostylusmosaicus)及海月水母(Aureliaaurita)的不同地理群体间[26]。
此外,本文的研究结果与利用扩增片段长度多态性技术和内转录间隔区序列对海蜇群体进行研究结果不同的原因,一方面可能是由于COⅠ属于线粒体基因标记,而扩增片段长度多态性和内转录间隔区序列属于核基因标记,两者间的遗传方式、进化速率等不同导致研究结果出现差异;另一方面扩增片段长度多态性[16-17]所分析的海蜇样品主要为黄海采集的海蜇放流捕捞群体及养殖群体,样品不同也可能是导致研究结果不同的重要原因[19]。
综上所述,本文利用COⅠ基因部分片段对辽东湾野生海蜇群体进行的分析表明辽东湾海蜇自然群体的遗传多样性尚处于中等或较高水平,分布于不同海域的海蜇群体间存在显著的遗传分化,这与利用扩增片段长度多态性技术和内转录间隔区序列对海蜇群体进行研究的结果不一致,样品不同是导致研究结果不一致的重要原因,因此还需要利用不同的分子标记对更多的海蜇样品进行分析,以期对海蜇这一重要经济水母的种群遗传结构及分子系统进化关系有更加全面准确的认识。
[1] 董婧,姜连新,孙明,等.渤海与黄海北部大型水母生物学研究[M].北京:海洋出版社,2013.
[2] 内田亨.日本动物分类:钵水母纲[M].东京:三省堂,1936.
[3] Kramp P L. Synopsis of the medusae of the world[J]. J Mar Biol Ass UK, 1961, 40(1):467-469 .
[4] 洪惠馨,张士美,王景池.海蜇[M].北京:科学出版社,1978.
[5] 姜连新,叶昌臣,谭克非,等.海蜇的研究[M].北京:海洋出版社,2007.
[6] 丁耕芜,陈介康.海蜇的生活史[J].水产学报,1981,5(2):93-102.
[7] 陈介康,丁耕芜.温度对海蜇横裂生殖的影响[J].动物学报,1983,29(3):195-206.
[8] 陈介康,丁耕芜,刘春洋.营养条件对海蜇横裂生殖的影响[J].水产学报,1985,9(4):321-329.
[9] 陈介康.海蜇的培育与利用[M].北京:海洋出版社,1985.
[10] 郭平.营养条件对海蜇螅状体形成足囊及足囊萌发的影响[J].水产学报, 1990, 14(3):206-211.
[11] 赵斌,张秀梅,陈四清,等.环境因子对海蜇早期幼体发育影响的生态学研究进展[J].海洋水产研究,2006, 27(1):87-92.
[12] 梁维波, 姜连新,于深礼.辽宁近海渔场海蜇增殖放流的回顾与发展[J].水产科学,2007,26(7):423-424.
[13] 游奎,马彩华,王绍军,等.短期低温条件对海蜇横裂生殖的影响[J].水生生物学报,2010,34(6):1223-1227.
[14] 王永顺,黄呜夏.海蜇增养殖技术[M].北京:金盾出版社,2004.
[15] 高祥刚,曹洁,董婧,等.辽宁沿海海蜇与沙海蜇遗传多样性的AFLP分析[J].水产科学,2009, 28(2):75-78.
[16] Qiao H J, Liu X Q, Zhang X J, et al. Genetic diversity of populations and clones ofRhopilemaesculentumin China based on AFLP analysis[J]. Chinese Journal of Oceanology and Limnology, 2013, 31(2):391-397.
[17] 孙国华,刘相全,杨建敏,等.海蜇养殖群体及自然捕获群体ITS序列遗传分析[J].海洋科学,2010, 34(10):90-95.
[18] Stopar K, Ramsak A, Trontelj P, et al. Lack of genetic structure in the jellyfishPelagianoctiluca(Cnidaria:Scyphozoa:Semaeostomeae) across European seas [J]. Mol Phylogenet Evol, 2010, 57(1):417-428.
[19] Andreja R, Katja S, Alenka M. Comparative phylogeography of meroplanktonic species,Aureliaspp. andRhizostomapulmo(Cnidaria:Scyphozoa) in European seas[J]. Hydrobiologia,2012,690(1):69-80.
[20] Armani A, Tinacci L, Giusti A, et al. What is inside the jar forensically informative nucleotide sequencing (FINS) of a short mitochondrial COⅠ gene fragment reveals a high percentage of mislabeling in jellyfish food products[J]. Food Research International, 2013, 54(2):1383-1393.
[21] Librado P, Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11):1451-1452.
[22] Excoffier L, Laval G, Schneider S. Arlequin ver 3.01: an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2005(1):47-50.
[23] Kumar S, Tamura K, Nei M. MEGA 3: integrated software for molecular evolutionary genetics analysis and sequence alignment [J]. Briefings in Bioinformatics, 2004, 5(2):150-163.
[24] Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data [J]. Genetics,1992,131(2):406-425.
[25] Bandelt H, Forster P, Rohl A. Median joining networks for inferring intraspecific phylogenics [J]. Molecular Biology Evolution, 1999, 16(1):37-48.
[26] Dawson M N, Gupta A S, England M H. Coupled biophysical global ocean model and molecular genetic analyses identify multiple introductions of cryptogenic species[J]. Proceedings of the National Academy of Sciences USA, 2005, 102(34):11968-11973.
GeneticDiversityAnalysisofJellyfishRhopilemaesculentuminLiaodongBayBasedontheCOⅠSequence
LI Yulong, WANG Bin, WANG Wenbo, DONG Jing
( Key Laboratory of Marine Biological Resources and Ecology, Liaoning Province, Liaoning Ocean and Fisheries Science Research Institute, Dalian 116023,China )
The 624 bp fragments of mitochondrial cytochrome oxidase Ⅰ(COⅠ) gene were sequenced and analyzed in 20 individuals jellyfishRhopilemaesculentumcollected from Liaodong Bay to investigate the genetic variation and diversity. In addition, the homologous sequences were analyzed in other 15 individuals sampled from the Bohai and Yellow Seas and Sea of Japan. The average contents were found to be 26.7% in A, 36.4% in T, 18.8% in C and 18.1% in G for COⅠ sequence. A total of 21 polymorphic nucleotide sites were detected, which defined 17 haplotypes. Compared with several other giant jellyfish species, the variation level of the jellyfish was moderate to high withHof 0.91 ± 0.03 and π of 0.0056±0.0032. With the aid of the homologous sequences retrieved from GenBank, both phylogenetic and molecular variance analysis supported significant differentiation among jellyfish populations in Sea of Japan and the Bohai Sea and Yellow Sea populations.
Rhopilemaesculentum; mtDNA; COⅠ gene; genetic diversity
S917
A
1003-1111(2016)04-0404-06
10.16378/j.cnki.1003-1111.2016.04.016
2015-5-12;
2016-03-01.
国家自然科学基金资助项目(31400406);国家海洋公益性行业科研专项项目(201405010).
李玉龙(1981—),男,助理研究员;研究方向:海洋生物分子生物学及渔业资源增殖放流.E-mail: liyudragon@126.com. 通讯作者:董婧(1966—),女,研究员;研究方向:水母生态学及渔业资源.E-mail: 1024470248 @qq.com.

