贵南沙蜥染色体核型与聚类分析
李嘉晟,童浩杰,张凯龙,杨坤,罗媛媛,金园庭
(中国计量大学,杭州310018)
贵南沙蜥染色体核型与聚类分析
李嘉晟,童浩杰,张凯龙,杨坤,罗媛媛,金园庭*
(中国计量大学,杭州310018)
贵南沙蜥Phrynocephalusguinanensis为近年沙蜥属新命名的物种,形态上区别于贵德沙蜥P.putjatia与青海沙蜥P.vlangalii,而最近沙蜥分子系统发育研究的结果不支持贵南沙蜥为一有效种,它在分子树拓扑结构上与贵德沙蜥一亚分支样本聚在一起。本文观察贵南沙蜥核型为2n=24I+24m,具有12对大染色体和12对小染色体,其中大染色体均为端部类型,且无性别相关的异型染色体。青海11个沙蜥种群染色体特征的聚类分析表明贵南沙蜥与贵德沙蜥聚于同一分支,该分支构成青海沙蜥分支的姐妹支,证实贵南沙蜥与贵德沙蜥具有较近的核型演化关系。研究结果不支持贵南沙蜥为有效物种。
贵南沙蜥;核型;聚类分析;系统分化
染色体的大小、形态和结构具有多样性。不同分类群核型特征不同,它不仅表现在属间、种间,也表现在种内不同种群间,这为探讨属、种系统进化关系以及生物的系统发育提供了重要的依据(Mayr,1969;Stebbins,1971)。染色体核型作为研究物种分类、演化的重要手段,已成为细胞遗传学研究的基本方法,并常被用来解决形态分类难以解决的问题,是现代综合分类的有效手段之一(吴甘霖,2006)。
沙蜥属Phrynocephalus属有鳞目Squamata爬行纲Reptilia鬣蜥科Agamidae,约40个物种,按繁殖方式可分为卵生沙蜥和胎生沙蜥(赵尔宓等,1998;赵肯堂,1999)。我国的沙蜥属核型均来自共同的核型祖先,可分为3个核型类群:(1)2n=48(包括12对大染色体和12对小染色体),所有胎生沙蜥属于这一类;(2)2n=46(包括11对大染色体和12对小染色体),所有卵生沙蜥(大耳沙蜥P.mystaceus除外)属于这一类;(3)2n=48(包括11对大染色体和13对小染色体),卵生的大耳沙蜥属于这一类(曾晓茂等,1997;邱清波等,2007)。
青藏高原上主要分布着6种胎生沙蜥:青海沙蜥P.vlangalii、西藏沙蜥P.theobaldi、红尾沙蜥P.erythrurus、南疆沙蜥P.forsythii、贵德沙蜥P.putjatia和近年命名的贵南沙蜥P.guinanensis(Jietal.,2009)。贵南沙蜥仅分布在青海省海南藏族自治州贵南县的一片连续沙丘(长宽各20~40 km),分布区与贵德沙蜥(主要分布在青海湖盆地及其东南部贵德、贵南)邻近但互不重叠(Jinetal.,2014)。两者个体大小与体色特征差异显著:贵南沙蜥体色具两性异形,两性成体在背部、尾与前肢侧面以及腹部周缘的颜色都存在差异(Jietal.,2009),而贵德沙蜥没有明显的体色两性异形。
贵德沙蜥异型性染色体的缺失证实它与具有异型性染色体的青海沙蜥存在遗传差异(王跃招等,2002)。近期研究证实贵德沙蜥为一有效的系统分类物种(Guo & Wang,2007;Jinetal.,2008;Jin & Brown,2013)。不同于贵南沙蜥仅仅分布于贵南县的一片连续沙丘,贵德沙蜥广泛分布于青海湖盆地及其东部与南部的荒漠和半荒漠地带(Jinetal.,2014)。应用线粒体基因和核基因研究证实贵南沙蜥与贵德沙蜥遗传分化不明显,贵南沙蜥系统分化上应归属于贵德沙蜥(Jinetal.,2014)。
本文采用骨髓细胞染色体制备的实验方法,旨在研究贵南沙蜥的核型特征,并分析贵南沙蜥与其他近缘沙蜥物种的染色体特征的聚类关系。以期揭示贵南沙蜥与青海沙蜥、贵德沙蜥及其他胎生沙蜥核型特征的演化关系,为胎生沙蜥系统分类与多样性研究提供新证据。
1 材料与方法
1.1 材料
贵南沙蜥成体(5♂♂,5♀♀)于2014年8月采自青海省海南藏族自治州贵南县过马营镇(35°47′N, 101°02′E)。样本处理前饲养于中国计量学院生命科学学院实验室。
1.2 骨髓染色体制备
分别在贵南沙蜥皮下和腹腔各注射2次一定量(按25 μg·g-1体质量比例)的0.5%秋水仙素溶液,间隔16 h。第二次注射2 h后将蜥蜴处死,取出四肢骨,在含有1 mL KCl低渗液(0.075 M)的培养皿中用止血钳将四肢骨粉碎,使骨髓细胞充分渗出。取渗出液于离心管中,37 ℃水浴20~30 min。1 200 r·min-1离心10 min,弃上清,加入固定液(甲醇∶冰醋酸=3∶1)1 mL,轻轻混匀后固定5 min。离心弃上清后加入固定液进行2次固定,固定时间15~20 min。1200 r·min-1离心10 min,弃上清,留下一小部分(约0.1 mL),用滴管轻轻地将沉淀物打匀。用滴管吸取悬浮液3~4滴于冰水浸泡过的载玻片上,在酒精灯上微烤,自然晾干。Giemsa染色8 min,用清水洗去多余染液,自然晾干。上述方法参照曾晓茂等(1997)的制片方法。
1.3 数据分析
选取雌、雄共44个较好的中期分裂相,用显微镜(E100,Nikon)对其放大照相。用Adobe Photoshop处理染色体核型图,Image J测量染色体并进行统计,染色体相对长度=待测的单个染色体长度/整套染色体长度×100%。染色体分类依据Levan等(1964)的标准,按臂比分为4 类:(1)中部着丝粒染色体(m),臂比为1.0~1.7;(2)亚中部着丝粒染色体(sm),臂比为1.7~3.0;(3)亚端部着丝粒染色体(st),臂比为3.0~7.0;(4)端部着丝粒染色体(t),臂比为7.0~∞。染色体臂数(NF)统计及类型缩写符号参照Gorman(1973)的标准:中部着丝粒染色体为V,亚中部着丝粒染色体为sV,端部着丝粒染色体为I,亚端部着丝粒染色体为sI,微小染色体为m。染色体特征加权采用王跃招等(2002)的方法进行:即Wi=Ai×(ΣMi)/n,其中Wi为加权值,Ai为编码值,有异型性染色体的Ai=1,无异型性染色体的Ai=0,Mi为第i对大染色体的相对长度,n为大染色体对数。染色体特征的聚类分析以居群为单位进行,先用Matlab计算居群间的欧式距离,构建欧式距离矩阵,然后用MEGA 6.0使用非加权组平均法(UPGMA)建树分析。
2 结果
2.1 贵南沙蜥染色体核型
对贵南沙蜥个体骨髓细胞的中期分裂相进行观察统计,结果显示所有分裂相染色体组均由12对大染色体和12对小染色体组成,且均为端部着丝粒染色体,未见与性别相关的异型性染色体(图1)。其中,最长的染色体的相对长度为12.953±0.034,最短的为4.937±0.016,其臂比均为∞(表1,统计结果表述为Mean±SE)。因此,贵南沙蜥核型模式为2n=24I+24m,臂数NF=48。
2.2 染色体特征聚类分析
应用已发表的贵德沙蜥与青海沙蜥染色体核型数据(王跃招等,2002),包含本研究取得的贵南沙
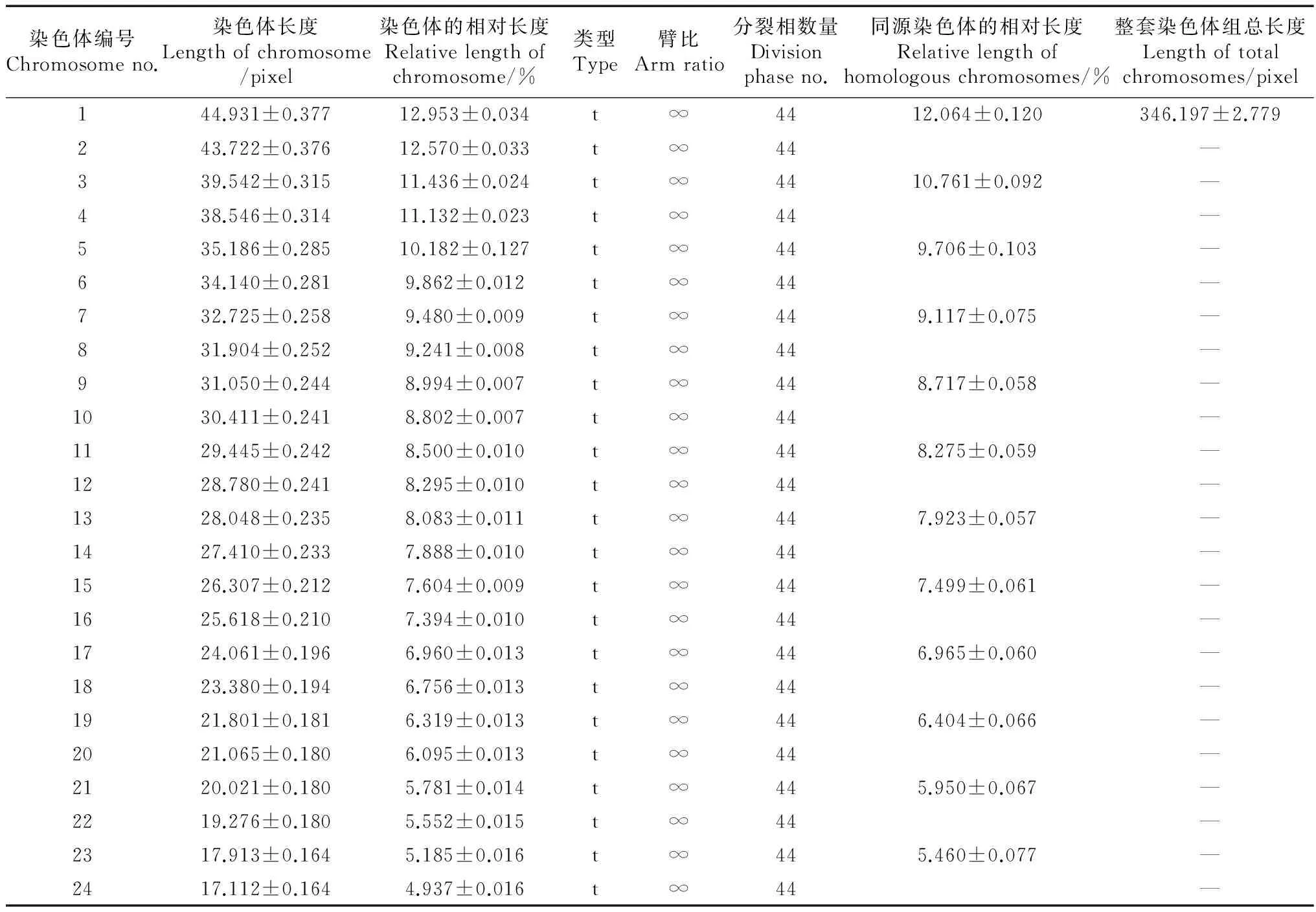
表1 贵南沙蜥染色体测量值

图1 贵南沙蜥核型图(100×)
A为雄性个体,B为雌性个体。
A represents male, B represents female.
蜥核型特征数据在内,我们对来自青海各地共计11个沙蜥地理种群(表2)进行了染色体特征的系统聚类分析。由于其中8个青海沙蜥种群雌性个体中存在异型性染色体(表2),我们分别进行了异型性染色体特征加权分析和异型性染色体特征非加权分析,然后分别用MEGA构建非加权组平均法(UPGMA)系统树(图2)。从系统树可见,11个种群分为2个分支,其中贵南沙蜥种群贵南(Guinan)与贵德沙蜥2种群贵德(Guide)和天祝(Tianzhu)聚为一个分支,剩余的8个青海沙蜥种群聚为另一分支。
3 讨论
研究结果显示贵南沙蜥染色体核型模式为2n=24I+24m。曾晓茂等(1997)将我国沙蜥核型分为3类:11对大染色体类群(2n=46=22I+24m)、12对大染色体类群(2n=48=24I+24m)和大耳沙蜥类型(2n=48=2V+8sV+12sI+26m)。贵南沙蜥为胎生蜥蜴,与贵德沙蜥、青海沙蜥、南疆沙蜥、泽当沙蜥P.zetangensis和红尾沙蜥等我国特有的其他胎生沙蜥(曾晓茂等,1997;王跃招等,2002)同归属具有12对大染色体类群的胎生沙蜥。
贵南沙蜥大染色体均为端部类型,未见与性别相关的异型染色体,这与贵德沙蜥相同;但不同于其他胎生沙蜥物种,如青海沙蜥(王跃招等,2002)。因此,从核型看贵南沙蜥与贵德沙蜥更相近。比较
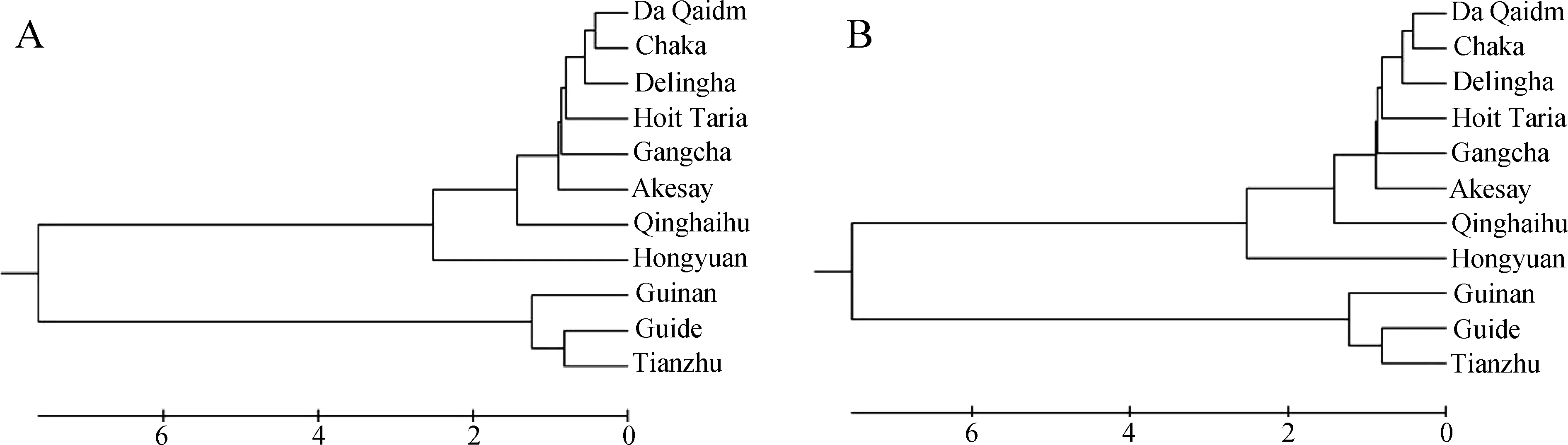
图2 青海11个沙蜥种群基于大染色体相对长度的系统聚类分析
Fig.2 The cluster relationship based on macrochromosomal relative length of elevenPhrynocephaluspopulations in Qinghai
A.ZW/ZZ为加权, B.ZW/ZZ为不加权。
A.ZW/ZZ represents the weighted, B.ZW/ZZ represents the unweighted.
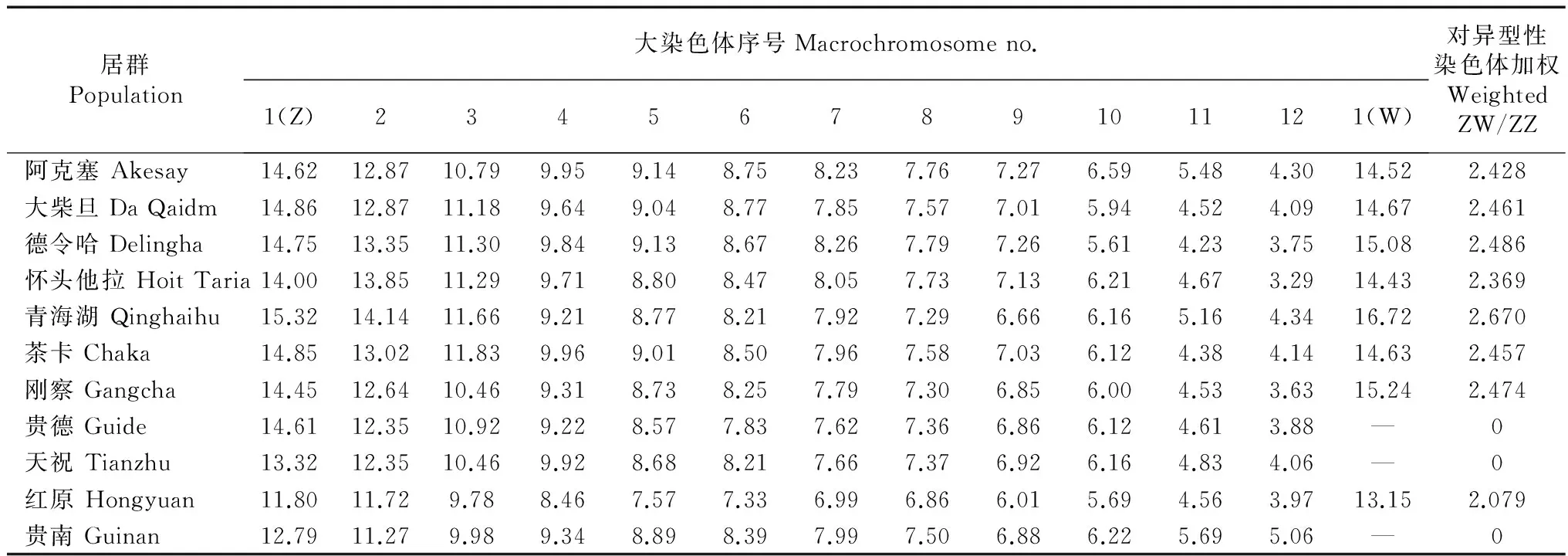
表2 沙蜥各居群大染色体相对长度
分析贵南沙蜥与青海沙蜥和贵德沙蜥样本染色体特征的聚类关系发现,贵南沙蜥与贵德沙蜥种群样本处于同一分支,而青海沙蜥不同种群样本聚于另一不同分支,两分支呈姐妹分歧关系;因此,染色体特征聚类分析结果依然支持贵南沙蜥与贵德沙蜥为同一物种,这与近期基于线粒体基因与核基因的分子系统分类的研究结果一致(Jinetal.,2014)。
然而由于贵南沙蜥和贵德沙蜥体色表型与生态地理分布差异显著(Jietal.,2009;Jinetal.,2014),且具有很近的起源祖先,在分子系统分化上,贵南沙蜥所有样本都聚在分布于青海湖盆地外的贵德沙蜥主分支的一亚分支内,表明贵南沙蜥和贵德沙蜥具有最近的起源祖先,而显著的表型分化提示贵南沙蜥与贵德沙蜥是一个有意思的生态进化类群,有待进一步开展表型特征与遗传分化研究。
致谢:感谢付昕蕾同学的实验工作、数据整理分析,以及李素芳、柳立军老师给予的染色体具体实验指导。
邱清波, 杜宇, 林炽贤. 2007. 沙蜥属13种蜥蜴基于16S rRNA序列的系统发生关系[J]. 海南师范大学学报(自然科学版), 20(4): 350-354.
王跃招, 曾晓茂, 方自力, 等. 2002. 沙蜥属一有效种贵德沙蜥及红原沙蜥的分类研究[J]. 动物分类学报, 27(2): 372-383.
吴甘霖. 2006. 核型分析在细胞分类学中的应用[J]. 生物学杂志, 23(1): 39-41.
曾晓茂, 王跃招, 刘志君, 等. 1997. 九种沙蜥的核型——论中国沙蜥属核型演化[J]. 动物学报, 43(4): 399-410.
赵尔宓, 江跃明, 黄庆云, 等. 1998. 拉汉英两栖爬行动物名称[M]. 北京: 科学出版社.
赵肯堂. 1999. 沙蜥属[M]// 赵尔宓, 赵肯堂, 周开亚, 等.中国动物志 有鳞目 蜥蜴亚目. 北京: 科学出版社: 151-193.
Gorman GC. 1973. The chromosomes of the Reptilia, a cytotaxonomic interpretation[M]// Chiarelli AB, Capanna E. Cytotaxonomy and vertebrate evolution. New York: Academic Press: 349-424.
Guo X, Wang Y. 2007. Partitioned Bayesian analyses, dispersal-vicariance analysis, and the biogeography of Chinese toad-headed lizards (Agamidae:Phrynocephalus): a re-evaluation[J]. Molecular Phylogenetics and Evolution, 45: 643-662.
Ji X, Wang YZ, Wang Z. 2009. New species ofPhrynocephalus(Squamata, Agamidae) from Qinghai, northwest China[J]. Zootaxa, 1988: 61-68.
Jin YT, Brown RP, Liu NF. 2008. Cladogenesis and phylogeography of the lizardPhrynocephalusvlangalii(Agamidae) on the Tibetan plateau[J]. Molecular Ecology, 17: 1971-1982.
Jin YT, Brown RP. 2013. Species history and divergence times of viviparous and oviparous Chinese toad-headed sand lizards (Phrynocephalus) on the Qinghai-Tibetan Plateau[J]. Molecular Phylogenetics and Evolution, 68: 259-268.
Jin YT, Yang ZS, Brown RP,etal. 2014. Intraspecific lineages of the lizardPhrynocephalusputjatiafrom the Qinghai-Tibetan Plateau: Impact of physical events on divergence and discordance between morphology and molecular markers[J]. Molecular Phylogenetics and Evolution, 71: 288-297.
Levan A, Fredga K, Sandberg AA. 1964. Nomenclature for centromeric position on chromosomes[J]. Hereditas, 52(2): 201-220.
Mayr E. 1969. Principles of systemstic zoology[M]. McGraw: Hill Book Company.
Stebbins GL. 1971. Chromosome evolution in higherplants[M]. London: Edward Aronld: 88.
Chromosomal Karyotype Characteristics and Systematic Cluster Analysis ofPhrynocephalusguinanensis
LI Jiasheng, TONG Haojie, ZHANG Kailong, YANG Kun, LUO Yuanyuan, JIN Yuanting*
(China Jiliang University, Hangzhou 310018, China)
Phrynocephalusguinanensiswas a newly named species and phenotypically different fromP.putjatiaandP.vlangalii.Recent molecular systematic analysis onPhrynocephalussuggested thatP.guinanensiswas an invalid species and its population was clustered into a subclade ofP.putjatiaon the tree topology.As a basic method of cellular genetics research, chromosomal karyotype analysis has been an alternative effective way for studying the modern comprehensive classification.In this study, the medullary cell ofP.guinanensiswas used to prepare chromosome for karyotype analysis and the chromosomal karyotype characteristics were used to investigate the cluster relationship amongP.guinanensisand otherPhrynocephalusspecies.The results showed that chromosomal karyotype ofP.guinanensiscould be expressed as 2n=24I+24m, which had twelve pairs of macrochromosomes belonging to end type and twelve pairs of microchromosomes.No heteromorphic sex chromosomes were observed.Based on the result of cluster analysis of elevenPhrynocephaluspopulations distributed in Qinghai province,P.guinanensisand allP.putjatiaspecimens were clustered as a sister clade, and this also indicated thatP.guinanensiswas an invalid species with the most closely evolutionary relationship withP.putjatia.
Phrynocephalusguinanensis; karyotype; cluster analysis; phylogenetic divergence
2015-12-31 接受日期:2016-01-30 基金项目:国家自然科学基金项目(31372183)
李嘉晟(1997—), 男,本科生, 主要从事分子进化与系统学研究
*通信作者Corresponding author,E-mail:jinyuanting@126.com
10.11984/j.issn.1000-7083.20150417
Q959.6
A
1000-7083(2016)03-0391-04

