刺参组蛋白脱乙酰基转移酶基因克隆及其在夏眠期间表达特征的研究*
陈慕雁, 孙 芮, 洪泽州, 朱爱军
(中国海洋大学 1.水产学院; 2.海水养殖教育部重点实验室,山东 青岛 266003)
刺参组蛋白脱乙酰基转移酶基因克隆及其在夏眠期间表达特征的研究*
陈慕雁1,2, 孙 芮1,2, 洪泽州1,2, 朱爱军1,2
(中国海洋大学 1.水产学院; 2.海水养殖教育部重点实验室,山东 青岛 266003)
刺参(Apostichopusjaponicus)利用夏眠的习性来应对夏季的高温胁迫,长期的代谢减退和基因转录抑制是刺参夏眠调控过程中的关键策略。本研究克隆分析了刺参体内关键组蛋白脱乙酰基转移酶(HDACs)的基因全长及结构特征,测定了该家族成员(HDAC1,HDAC3和HDAC4)在夏眠期间的基因表达模式。研究表明:刺参HDAC3和HDAC4与紫海胆(Strongylocehtrotuspurpturatus)相关基因的同源性最高,分属HDACs家族的Ⅰ型和Ⅱ型。定量表达分析表明,刺参不同组织中同属Ⅰ型的HDAC1和HDAC3基因在刺参夏眠不同阶段的表达均无显著差异;而属于Ⅱ型的HDAC4基因在深度夏眠阶段表达量显著上调。研究结果表明,II型HDACs在刺参夏眠基因转录抑制中具有潜在的特殊调节作用。
刺参; 组蛋白脱乙酰基转移酶; 基因特征; 夏眠; 转录抑制
夏眠是动物应对高温、低氧、干旱或缺少食物等极端环境时所采取的生存策略,广泛存在于脊椎动物和无脊椎动物中[1]。动物夏眠期间,机体进入代谢减退状态以降低休眠动物降低体内贮存的有限能量的消耗,保证基础代谢的正常进行。研究证实不同组织中细胞能量的1%~10%用以基因转录[2], 因而基因的转录表达抑制将会显著降低有机体在代谢减退状态下的能量消耗。已有研究表明卤虫 (Artemiafranciscana) 和滨螺 (Littorinalittorea)在缺氧胁迫响应过程中[3],哺乳动物在冬眠过程中[4-6]以及刺参在夏眠过程中都会发生严重的代谢减退并伴随着基因表达的整体抑制[7]。
表观遗传修饰作为调控基因表达抑制的重要手段之一,在生命过程中发挥了关键的作用。其中组蛋白的乙酰化和去乙酰化修饰是基因转录调控的关键表观遗传调节机制之一,通常组蛋白的乙酰化会诱导转录激活,而去乙酰化则引起转录抑制。这一修饰系统主要受组蛋白乙酰基转移酶(Histone acetyltransferases,HATs)和组蛋白脱乙酰基转移酶(Histone deacetylases, HDACs)调控[8]。如组蛋白H3 N-末端容纳了多个能够被乙酰化的Lys残基,由组蛋白乙酰化引发的结构改变致使装配紧密的DNA结构变得松散,以便于DNA的转录和翻译[9-10],组蛋白脱乙酰基转移酶则可通过移除组蛋白尾部的乙酰基团促使染色质的装配更为紧密从而抑制转录。已有研究表明组蛋白乙酰化在动物冬眠和缺氧的代谢减退过程中发挥着重要的调节作用[11-12],而关于乙酰化修饰在无脊椎动物夏眠过程中的调控作用研究还鲜有报道。刺参(Apostichopusjaponicus)又名仿刺参,属棘皮动物门(Echinodermata)海参纲(Holothuroiclea)楯手目(Aspidochirotida),是我国北方海域重要的水产养殖品种。刺参具有夏眠的特殊生态习性,当夏季水温升高到一定的阈值时,刺参即进入长达4个月的夏眠周期,夏眠期间刺参停止摄食,肠道退化萎缩,代谢减退,并伴随着耗氧率和氨氮排泄的减少[13],极大地延长了养殖周期,增加了养殖成本。本研究利用RACE技术首次克隆获得刺参HDAC3和HDAC4基因全长cDNA序列,并进行了同源比对和系统进化树分析;利用qRT-PCR技术测定了刺参在非夏眠期、深度夏眠时期、夏眠解除时期HDAC1、HDAC3和HDAC4基因在肠道、呼吸树和肌肉组织中的表达变化模式,以期为探讨刺参夏眠期间乙酰化在基因表达中的调控作用机制研究奠定基础。
1 材料与方法
1.1 实验材料
实验所用刺参采自青岛市胶南灵山卫海区,共采集了3个阶段的刺参样品:非夏眠期作为对照组(2014 年5月初,海水水温15 ℃);深度夏眠期(2014年8月上旬,海水水温28 ℃);及夏眠解除期(2014年10月上旬,海水水温18 ℃)。每个阶段选取5头健康且体重在100~120 g之间的刺参,快速解剖并取肠道、呼吸树和肌肉组织置于冻存管中,于液氮中速冻,之后保存在-80 ℃超低温冰箱中备用。
1.2 总RNA提取和cDNA 全长克隆
采用RNeasy mini kit 试剂盒(Cat No. 74104, Qiagen, Germany)提取刺参肠道,呼吸树和肌肉组织样品总RNA,用琼脂糖电泳检查RNA的完整性,NanoDrop 1000分光光度计(Thermo Scientific)检测计算总 RNA 样品浓度。根据刺参转录组中的HDAC3和HDAC4基因片段序列设计特异性引物(见表1),利用SMART RACE cDNA末端扩增试剂盒(Cat No. 634858, Clontech, USA)进行HDAC3和HDAC4基因cDNA 全长克隆,具体按照试剂盒说明书进行操作。利用1%琼脂糖电泳分析检测结果,割胶后利用OMEGA公司的胶回收试剂盒进行目的片断的回收,将目的片断连接至载体pMD19-T 质粒上,并将连接产物转化至感受态细胞中(Cat No. D9052, Takara, Japan),复苏1h后涂在预先制备好的含氨苄的LB平板上过夜培养。挑取阳性克隆于SOC液体培养基中培养12 h。以菌液为模板进行PCR 检测并送至Invitrogen公司测序。

表1 刺参基因CDNA全长扩增和QRT-PCR引物列表
1.3 序列分析
利用DNAStar软件进行序列拼接得到目的基因cDNA全长序列,并预测基因开放阅读框(Open reading frame, ORF)和氨基酸序列。利用NCBI 网站上的BLAST 工具(http://blast.ncbi.nlm.nih.gov/)进行同源比对分析。利用SMART version 4.0(http://www.smart.embl-heidelberg.de/)和Interpro: protein sequence analysis & classification(http://www.ebi.ac.uk/interpro/)预测推断氨基酸序列的功能结构域,并用SWISS-MODEL(http://swissmodel.expasy.org/)预测蛋白质结构。利用Clustal W程序和MEGA 6.0程序进行基因的多序列比对和系统进化树构建。
1.4 qRT-PCR测定基因表达水平
以不同组织的总RNA为模板,利用PrimeScript®RT Master Mix Perfect Real Time (Takara, Japan)试剂盒反转录第一链cDNA(不混样),-20 ℃保存。利用在线软件Primer 3 (http://frodo.wi.mit.edu/)设计荧光定量引物(如表1所示),以β-tubulin(TUBB)作为内参基因[14],应用SYBR®Premix Ex TaqTM试剂盒(Takara, Japan),在Mastercycler®eprealplexReal-Time PCR仪(Eppendorf, Germany)上进行荧光定量反应,每个反应设定3次技术重复。反应条件为:95 ℃预变性2 min,95 ℃变性30 s,57 ℃退伙30 s,72 ℃延伸30 s,共40个循环;并添加熔解曲线,检验反应产物是否单一。计算PCR 反应的效率和相关系数R。采用2-△△Ct方法测定目的基因的表达量。表达差异分析采用SPSS 17.0单因素方差分析和Duncan检验。相对表达量表示为平均值±平均数的标准误(Mean ± SEM)。P<0.05时认为差异显著。
2.3 软件设计流程 在完成Bmob后端云服务器的搭建与配置基础上,设计并开发由线性界面布局、数据获取模块和数据显示模块组成的应用程序,实现服务器连接、数据库监听、数据获取、数据显示等一系列流程。昆虫生境移动监测软件数据获取模块会实时监听Bmob后端云数据库中的昆虫生境数据的更新状态,如果发生了数据的更新,就将新上传的那条数据获取下来。数据获取模块获取到数据后就把数据交给数据显示模块,数据显示模块拿到数据后使用SimpleAdapter适配器将昆虫生境数据显示在ListView视图上。软件设计流程见图3。
2 结果
2.1 刺参HDAC3基因全长cDNA克隆及序列分析
刺参HDAC3基因cDNA全长为1885bp,其中包含85 bp的5′ UTR区域和495 bp的3′ UTR 区域。该基因开放阅读框(ORF)长度为 1 305 bp,编码了434个氨基酸,蛋白质分子量为49.7 kD(见图1)。利用SMART version 4.0预测刺参HDAC3蛋白的功能结构域见图1和表2(Genebank序列号:KX066363),包含1个NL结构域、1个PHD结构结构域,其中NL参与细胞分化进程,PHD结构域结合蛋白质和锌离子,Hist_deacetyl域是组蛋白脱乙酰基转移酶的代表功能域。蛋白质三级结构预测如图2A所示。

(编码区用大写字母表示,3’和5’非编码区用小写字母表示;星号(*)代表终止密码子;底部为刺参HDAC3功能结构域示意图。ORF are indicated in uppercase, and 3’UTR and 5’UTR are shown in lowercase. The asterisk indicates the translational termination codon.At the bottom of the page is a schematic diagram of domainsand characteristic motifs of the protein.)
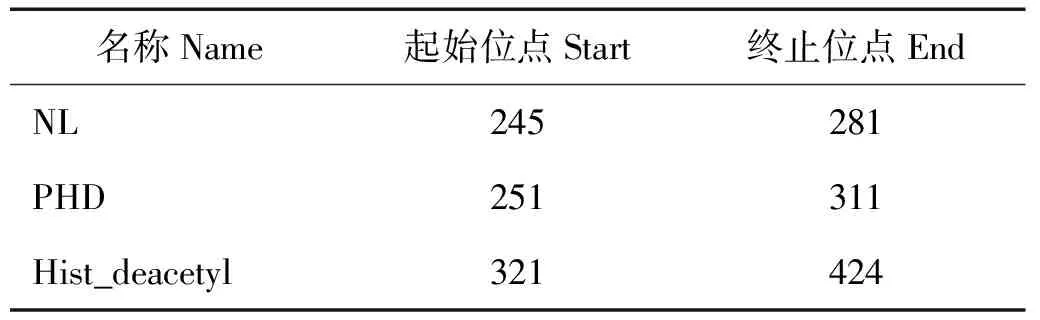
图1 刺参HDAC3基因结构序列及其编码的氨基酸序列
2.2 刺参 HDAC3序列同源性与系统进化树分析
序列同源性分析表明,刺参 HDAC3基因序列与紫海胆(Strongylocentrotuspurpuratus)(XP_794761.1)、密西西比鳄(Alligatormississippiensis)(XP_006265553.1)、矛尾鱼(Latimeriachalumnae) (XP_005995391.1)、美国鲎(Limuluspolyphemus)(XP_013784620.1)、舌囊虫(Saccoglouskowalevskii) (XP_002742058.1)的同源性分别为:84%、74%、75%、74%、80%(见图3)。其中与紫海胆同源性最高为84%,舌囊虫次之,与脊椎动物相似性相对较低。
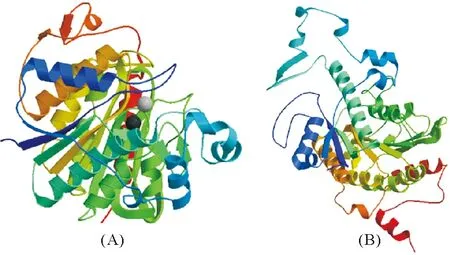
图2 HDAC3 (A)和HDAC4 (B) 蛋白的三级结构预测
系统进化树(见图4)分析表明:刺参HDAC3与同属棘皮动物的海胆(S.purpuratus)同源关系最为接近,并与鸭嘴海豆芽(Lingulaanatina)、舌囊虫(Saccoglossuskowalevskii)等无脊椎动物聚在一起,明显与脊椎动物分为2支。因此,刺参HDAC3的进化地位与刺参生物学分类地位一致。

(黑色代表具有相同的氨基酸,灰色代表具有相近的氨基酸。Identical and similar amino acid residues in all the proteins are indicated by black and grey highlights. Gaps are marked by hyphens. Percent identity of amino acid sequence to sea cucumber HDAC3 is shown at the end. AJ: 仿刺参Apostichopusjaponicus; AM:密西西比短吻鳄Alligatormississippiensis; LC:矛尾鱼Latimeriachalumnae; LP:美洲鲎Limuluspolyphemus; SK: 囊舌虫Saccoglossuskowalevskii; SP: 紫海胆S.purpuratus)
图3 刺参与其他物种HDAC3序列的同源性比对
Fig.3 Comparison of the amino acid sequence of sea cucumber HDAC3 with HDAC3s from other species
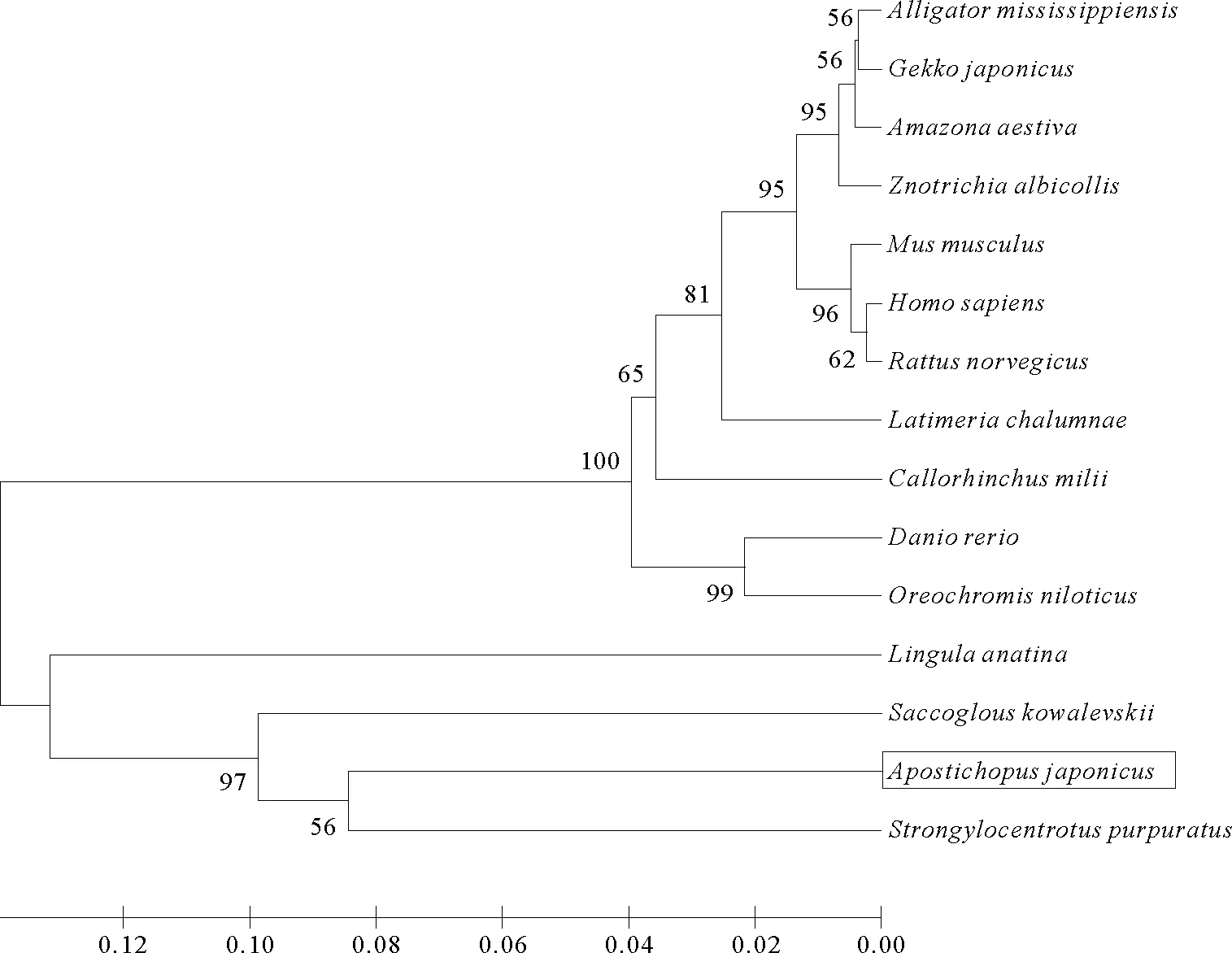
(系统发育树拓扑结构的可信度采用1000次重抽样来检测,树形图分支上的数字代表其置信值。The tree topology was evaluated by 1,000 bootstrap replications and numbers on each branch of the tree represent the bootstrap support value. Sequences were acquired from GenBank:密西西比短吻鳄(Alligatormississippiensis),XP_006265553.1;多疣壁虎(Gekkojaponicus),XP_015266654.1;青绿顶亚马逊鹦鹉(Amazonaaestiva),KQK74258.1;白喉带鹀(Zonotrichiaalbicollis);XP_005496373.2;家鼠(Musmusculus),AAF28798.1;智人(Homosapiens),AAC52038.1;褐家鼠(Rattusnorvegicus),NP_445900.1;矛尾鱼(Latimeriachalumnae),XP_005995391.1;叶吻银鲛(Callorhinchusmilii),XP_007906313.1;斑马鱼(Daniorerio),NP_957284.1;罗非鱼(Oreochromisniloticus),XP_003451863.1;鸭嘴海豆芽(Lingulaanatina),XP_013403048.1;囊舌虫(Saccoglossuskowalevski)i,XP_002742058.1;紫海胆(S.purpuratus),XP_794761.1.)
图4 不同物种的HDAC3系统进化树
Fig.4 Consensus neighbor-joining tree based on the sequences of HDAC3 from different species
2.3 刺参HDAC4基因全长 cDNA 克隆及序列分析
刺参HDAC4基因序列全长共3 248 bp,其中包括开放阅读框ORF3 012 bp,5’UTR区176 bp,3’UTR区60 bp,共编码1 003个氨基酸,预测蛋白分子量是268.52 kDa(见图5)。预测推断刺参HDAC4的功能结构域,包括2个HDAC4_Gln结构域,2个Hist_deacetyl结构域,1个NEBU结构域,1个PGRP结构域,1个锌指结构域,1个FTP结构域。其中,HDAC4_Gln结构域富含谷氨酰胺,形成许多螺旋体,与蛋白质的可逆性装配有关;Hist_deacetyl结构域是组蛋白脱乙酰基转移酶的代表性功能域,包括了3层二级结构(α螺旋-β折叠-α螺旋);ZnF_C2C2类结构域代表了在转录因子TFIIS中的锌指基序,NEBU为肌动蛋白结合域;PGRP为锌离子结合域,调节N—乙酰胞壁酰L—丙氨酸酰氨酶的活性(见图5和表3)(Genebank序列号: KX066364)。蛋白质三级结构预测如图2B所示。
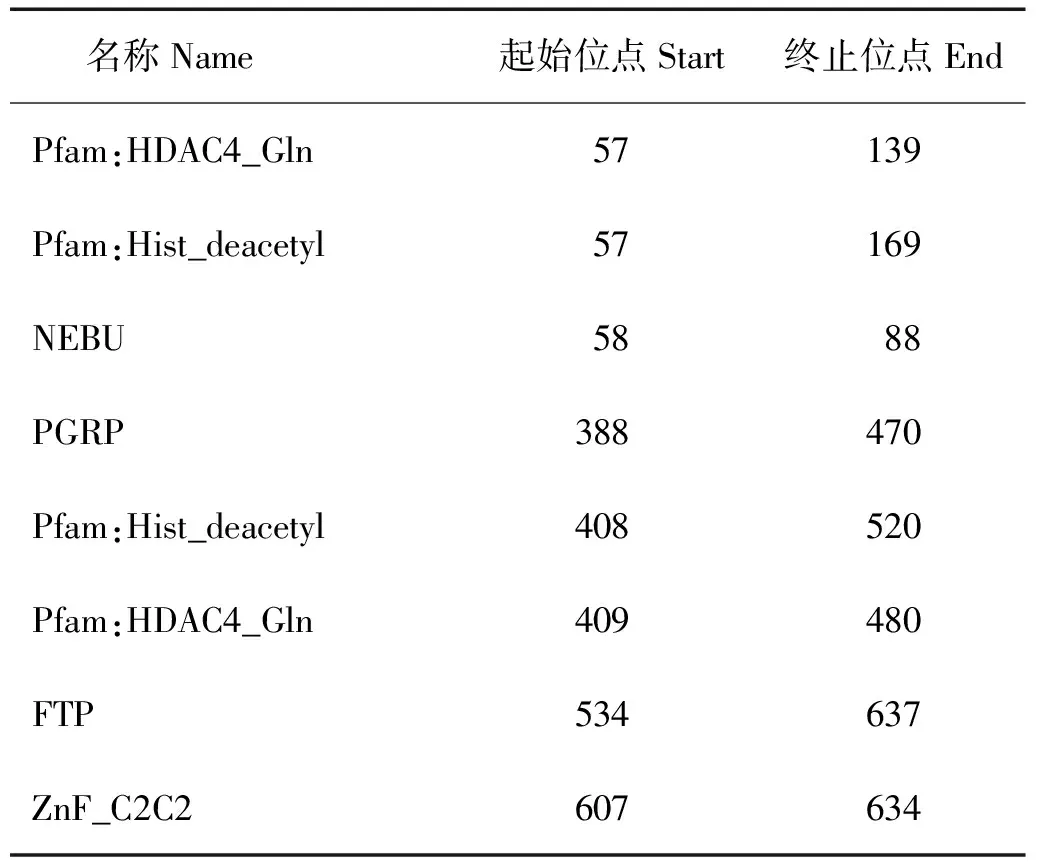
表3 刺参HDAC4保守结构域位置

(编码区用大写字母表示,3’和5’非编码区用小写字母表示;星号(*)代表终止密码子;底部为刺参HDAC4功能结构域示意图。ORF are indicated in uppercase, and 3’UTR and 5’UTR are shown in lowercase. The asterisk indicates the translational termination;At the bottom of the page is a schematic diagram of domains and characteristic motifs of the protein.)
图5 刺参HDAC4基因结构序列及其编码的氨基酸序列
Fig.5 Nucleotide sequence of sea cucumberHDAC4gene and deduced amino acid sequences
2.4 刺参 HDAC4序列同源性与系统进化树分析

(黑色代表具有相同的氨基酸,灰色代表具有相近的氨基酸。Identical and similar amino acid residues in all the proteins are indicated by black and grey highlights. Gaps are marked by hyphens. Percent identity of amino acid sequence to sea cucumber HDAC3 is shown at the end. AJ: 仿刺参(A.japonicus);CC: 胡椒鲷(C.cinctus); OA:O.abietinus; SP:紫海胆(S.purpuratus); VE:切叶蚁(V.emeryi).)
图6 刺参与其他物种HDAC4序列的同源性比对
Fig.6 Comparison of the amino acid sequence of sea cucumber HDAC4 with HDAC4s from other species
2.5 夏眠不同时期刺参各组织中HDAC1、HDAC3和HDAC4基因的表达模式
利用qRT-PCR 技术测定了夏眠不同阶段刺参肠道、呼吸树和肌肉组织中HDAC1、HDAC3和HDAC4基因的表达模式(如图8所示)。HDAC1和HDAC3在刺参夏眠的不同阶段不同组织内均无显著变化(见图8a、b)。HDAC4的表达量在深度夏眠期的肠道组织中显著增加,当夏眠解除时又恢复到正常水平;呼吸树组织中HDAC4的表达量在夏眠不同阶段没有显著变化;在肌肉组织中,HDAC4表达量在深度夏眠和夏眠解除时期均显著升高(见图8c)。
3 讨论
动物进入休眠状态伴随着耗能过程的整体抑制,比如转录和翻译过程的抑制[15]。我们前期的研究表明在刺参夏眠期间基因的转录和蛋白的翻译过程均受到整体的抑制[7, 16]。然而目前关于乙酰化修饰在动物夏眠期间基因表达抑制中调控作用的研究还鲜有报道。
DNA的结构以及在核中的易接近性是决定转录因子和RNA聚合酶等蛋白能否结合并起始转录的关键因素[17]。DNA片断紧紧包裹着组蛋白核心形成一个致密的结构,调节基因的表达。组蛋白尾部残基的乙酰化修饰导致紧密缠绕的染色质结构松散,从而促使参与转录调控的各种蛋白因子与DNA特异序列结合,进而发挥转录调控作用[18]。本研究重点关注组蛋白乙酰基转移酶 (HDACs)在刺参夏眠过程中的转录表达模式的变化,以期为乙酰化修饰在刺参夏眠过程中的基因表达的整体调控中的作用研究奠定基础。
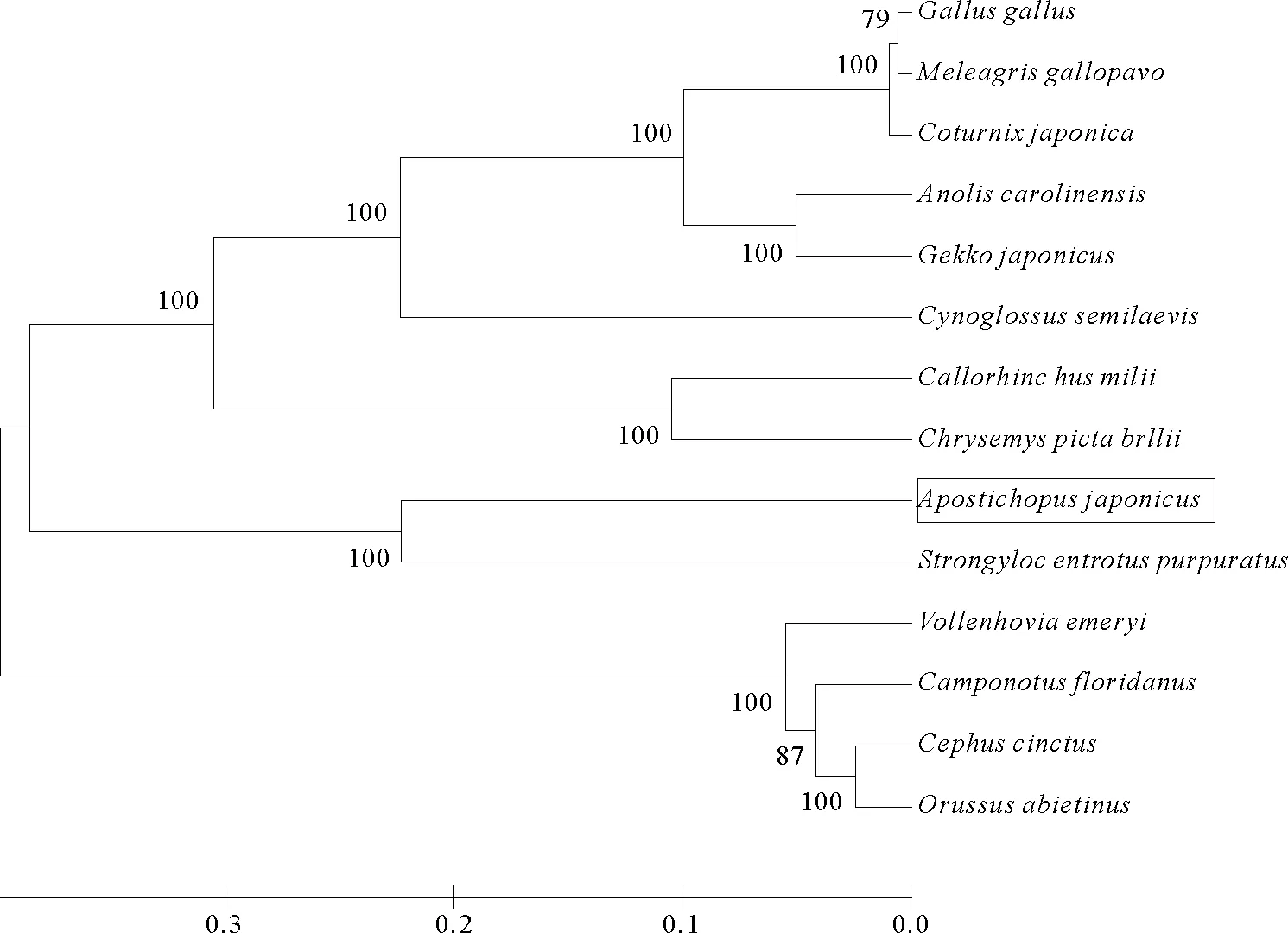
(系统发育树拓扑结构的可信度采用1000次重抽样来检测,树形图分支上的数字代表其置信。The tree topology was evaluated by 1,000 bootstrap replications and numbers on eachbranch of the tree represent the bootstrap support value. Sequences were acquired from GenBank:紫海胆(S.purpuratus), XP_011660540.1; 切叶蚁(Vollenhoviaemeryi), XP_011874060.1; 多疣壁虎(Gekkojaponicas), XP_015265702.1;叶吻银鲛 (Callorhinchusmilii), XP_007899619.1; 半滑舌鳎(Cynoglossussemilaevis), XP_008316615.1; 锦龟(Chrysemyspictabellii), XP_008160870.1; 锯蝇(Orussusabietinus), XP_012285216.1; 绿蜥蜴(Anoliscarolinensis), XP_008114639.1; 胡椒鲷(Cephuscinctus), XP_015593268.1; 日本鹌鹑(Coturnixjaponica), XP_015742338.1; 火鸡(Meleagrisgallopavo), XP_010718850.1; 原鸡(Gallusgallus), XP_015155729.1; 佛罗里达弓背蚁(Camponotusfloridanus), XP_011255713.1.)
图7 不同物种的HDAC4系统进化树
Fig.7 Consensus neighbor-joining tree based on the sequences of HDAC4 from different species
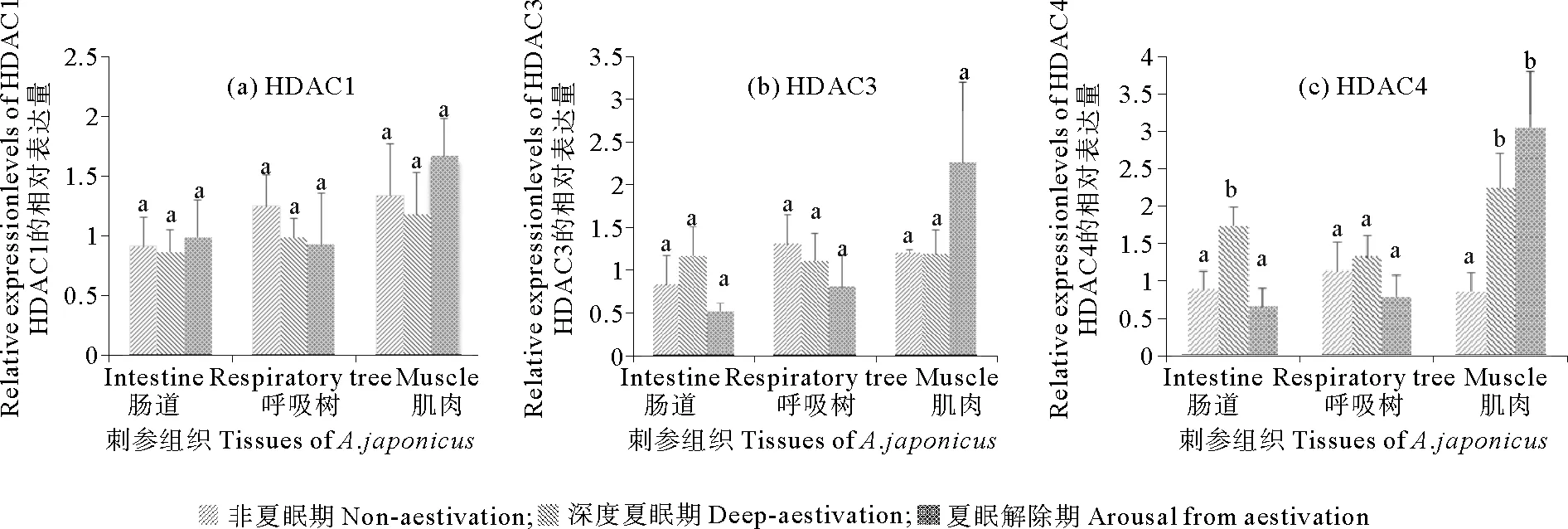
图8 夏眠不同时期刺参基因在肠道、呼吸树和肌肉组织中表达差异性分析
HDACs共分3个家族:与酵母 Rpd3 同源的I类HDACs,包括 HDAC1、HDAC2、HDAC3、 HDAC8;与酵母Hda1同源的II类HDACs,包括 HDAC4、HDAC5、HDAC6、HDAC7、HDAC9、HDAC10;与酵母Sir2同源的III类HDACs包括SIRT1~SIRT7。I 类 HDACs主要与Sin3, Mi2, NCoR /SMRT 等因子形成相关的转录抑制复合物,II类HDACs如HDAC4、HDAC5也参与形成NCoR/SMRT转录抑制复合物,目前在大多数细胞内都发现了Ⅰ类HDACs的表达,而类HDACs的表达更为局限,这暗示2类HDACs可能参与不同的细胞分化和发育进程[19]。
本研究发现同属I型的HDAC1和HDAC3在刺参夏眠的不同阶段不同组织内表达量均无显著变化,其中深度夏眠期间HDAC3表达量在肠道组织中略有所升高,这与王天明等[20]研究结果中长时间室内诱导夏眠(20天以上)刺参肠道组织中该基因表达量检测结果一致。HDAC4表达量在深度夏眠期肠道和肌肉组织中均保持在较高水平,这与之前在冬眠的十三条纹地松鼠(Ictidomystridecemlineatus)HDAC4的蛋白表达模式相吻合。Storey等[21]在十三条纹地松鼠冬眠期间的研究表明,在冬眠期间的褐色脂肪组织中,HDAC1和HDAC4的蛋白表达量分别上调了1.5和6倍。HDAC1和HDAC3属于Ⅰ型HDACs,而HDAC4则属于Ⅱ型a类。Ⅱ型a类HDACs 不同于其他HDACs,其氮末端具有和转录因子相结合以招募其靶标基因所需的功能域。通常Ⅱ型a类HDACs的表达具有组织特异性,已有研究表明此类HADCs在哺乳动物的骨骼、心脏、平滑肌、免疫系统、血管系统和脑组织中发挥了转录抑制的功能[22]。
HDAC4在动物休眠前后基因或蛋白表达量的显著改变提示Ⅱ型HDACs在动物休眠中的特殊作用。研究证实, HDAC4催化序列与Ⅰ类 HDACs相区别的特征是前者具有一段锌离子结合域,可容纳2种自分子中心分离的蛋白片段[23]。本研究中对HDAC3和HDAC4功能域的预测中也有同样的发现,我们推测HDAC4的特殊作用可能与这段锌离子结构有关,此假设还有待于今后深入探讨。
4 结论
本文分析了刺参体内关键组蛋白脱乙酰基酶的结构特征及其在刺参夏眠期间的基因表达模式,结果发现Ⅱ型的HDAC4基因在深度夏眠阶段表达量显著上升,暗示Ⅱ型HDACs在刺参夏眠基因转录抑制中具有潜在的特殊调节作用。本文为刺参夏眠期间乙酰化在基因表达中的调控作用机制研究奠定了基础。
[1] Zhao Y, Yang H, Storey K B, et al. Differential gene expression in the respiratory tree of the sea cucumberApostichopusjaponicusduring aestivation[J]. Mar Genomics, 2014, 18: 173-183.
[2] Rolfe D F, Brown G C. Cellular energy utilization and molecular origin of standard metabolic rate in mammals[J]. Physiol Rev, 1997, 77(3): 731-758.
[3] Ladare K, Storey K. A profile of the metabolic responses to anoxia in marine invertebrate[J]. Cell and Molecular Responses to Stress, 2002,3(2): 27-46.
[4] Van Breukelen F, Martin S. Reversible depression of transcription during hibernation[J]. J Comp Physiol, 2002, 172(5): 355-361.
[5] Osborne P G, Gao B, Hashimoto M. Determination in vivo of newly synthesized gene expression in hamsters during phases of the hibernation cycle[J]. Jpn J Physiol, 2004, 54(3): 295-305.
[6] Storey K B. Evidence for a reduced transcriptional state during hibernation in ground squirrels[J]. Cryobiology, 2006, 53(3): 310-318.
[7] Zhao Y, Yang H, Storey K B, et al. RNA-seq dependent transcriptional analysis unveils gene expression profile in the intestine of sea cucumberApostichopusjaponicusduring aestivation[J]. Comp Biochem Physiol D, 2014, 10: 30-43.
[8] 李保华, 张卫林. 组蛋白乙酰化/去乙酰化与基因表达调控研究进展[J]. 安徽农业科学, 2007, 35(18): 5364-5365.
Li B H, Zhang W L. Research Progress in the Histone Acetylation/ Deacetylation and Gene Expression and its Regulation [J]. J Anhui Agri Sci, 2007, 35(18): 5364-5365.
[9] Eberharter A, Becker P B. Histone acetylation: a switch between repressive and permissive chromatin[J]. EMBO Rep, 2002, 3(3): 224-229.
[10] Orphanides G, Reinberg D. RNA polymerase II elongation through chromatin[J]. Nature, 2000, 407(6803): 471-476.
[11] Storey K B. Evidence for a reduced transcriptional state during hibernation in ground squirrels[J]. Cryobiology, 2006, 53(3): 310-318.
[12] Krivoruchko A, Storey K B. Epigenetics in anoxia tolerance: a role for histone deacetylases[J]. Mol Cell Biochem, 2010, 342(1-2): 151-161.
[13] Yang H, Zhou Y, Zhang T, et al. Metabolic characteristics of sea cucumberApostichopusjaponicus(Selenka) during aestivation[J]. J Exp Mar Bio Ecol, 2006, 330(2): 505-510.
[14] Zhao Y, Chen M, Wang T, et al. Selection of reference genes for qRT-PCR analysis of gene expression in sea cucumberApostichopusjaponicusduring aestivation[J]. Chin J Oceanol Limnol, 2014, 32: 1248-1256.
[15] Storey K B, Storey J M. Metabolic rate depression in animals: transcriptional and translational controls[J]. Biol Rev, 2004, 79(1): 207-233.
[16] Chen M, Li X, Zhu A, et al. Understanding mechanism of sea cucumberApostichopusjaponicusaestivation: Insights from TMT-based proteomic study [J]. Comp Biochem Physiol D, 2016, 19: 78-89.
[17] Kornberg R D, Lorch Y. Twenty-five years of the nucleosome, fundamental particle of the eukaryote chromosome[J]. Cell, 1999, 98(3): 285-294.
[18] Cheung P, Tanner K G, Cheung W L, et al. Synergistic coupling of histone H3 phosphorylation and acetylation in response to epidermal growth factor stimulation[J]. Mol Cell, 2000, 5(6): 905-915.
[19] De Ruijter A J M, Van Gennip A H, Caron H N, et al. Histone deacetylases (HDACs): characterization of the classical HDAC family[J]. Biochem J, 2003, 370(3): 737-749.
[20] Wang T, Yang H, Zhao H, et al. Transcriptional changes in epigenetic modifiers associated with gene silencing in the intestine of the sea cucumber,Apostichopusjaponicus(Selenka), during aestivation[J]. Chin J Oceanol Limnol, 2011, 29: 1267-1274.
[21] Biggar Y, Storey K B. Global DNA modifications suppress transcription in brown adipose tissue during hibernation[J]. Cryobiology, 2014, 69(2): 333-338.
[22] Parra M. Class IIa HDACs-new insights into their functions in physiology and pathology[J]. FEBS J, 2015, 282(9): 1736-1744.
[23] Bottomley M J, Surdo P L, Di Giovine P, et al. Structural and functional analysis of the human HDAC4 catalytic domain reveals a regulatory structural zinc-binding domain[J]. J Biol Chem, 2008, 283(39): 26694-26704.
责任编辑 朱宝象
Molecular Cloning of Sea Cucumber Histone Deacetylases (HDACs) Genes and Their Expression Analysis During Aestivation
CHEN Mu-Yan1,2, SUN Rui1,2, HONG Ze-Zhou1,2, ZHU Ai-Jun1,2
(Ocean University of China 1.College of Fisheries; 2.The Key Laboratory of Mariculture, Ministry of Education, Qingdao 266003, China)
Sea cucumber,ApostichopusjaponicusSelenka, survives high summer temperature by entering into aestivation which is characterized by hypometabolism and global transcriptional depression. We investigated the hypothesis that aestivationis associated with acetylation-dependent epigenetic mechanisms by cloning, sequencing and quantifying the transcripts of important members of histone deacetylases (HDACs) family. The results showed that the deduced amino acid sequences and characteristic motifs of sea cucumber HDAC3 and HDAC4 were highly similar to those of their echinoderms. No significantly variation was observed inHDAC1 andHDAC3 belonging to class I in different tissues of sea cucumber during aestivation. However, the expression ofHDAC4 belonging to class II was significantly up-regulated in intestine and muscle during deep-aestivation.The findings in present study suggested that class II HDACs involved in transcriptional silencing during aestivation.
sea cucumber; histone deacetylase; gene characterization; aestivation; transcriptional depression
国家自然科学基金项目(31472257)资助
2016-07-18;
2016-09-05
陈慕雁(1979-),女,副教授。研究方向:无脊椎动物抗逆生理学。E-mail: chenmuyan@ouc.edu.cn
Q175
A
1672-5174(2016)11-052-10
10.16441/j.cnki.hdxb.20160260
陈慕雁, 孙芮, 洪泽州, 等. 刺参组蛋白脱乙酰基转移酶基因克隆及其在夏眠期间表达特征的研究[J]. 中国海洋大学学报(自然科学版), 2016, 46(11): 52-61.
CHEN Mu-Yan, SUN Rui, HONG Ze-Zhou, et al. Molecular cloning of sea cucumber histone deacetylases (HDACs) genes and their expression analysis during aestivation[J]. Periodical of Ocean University of China, 2016, 46(11): 52-61.
Supported by the National Natural Science Foundation of China (31472257)

