基于SRAP标记的花椒种质资源遗传多样性及群体结构分析
李立新,杨途熙,魏安智,冯世静,陈 旅,侯 娜
(西北农林科技大学 林学院,陕西 杨凌 712100)
基于SRAP标记的花椒种质资源遗传多样性及群体结构分析
李立新,杨途熙,魏安智,冯世静,陈 旅,侯 娜
(西北农林科技大学 林学院,陕西 杨凌 712100)
为探讨花椒种质资源的遗传多样性和群体结构特征,用SRAP分子标记技术对采自陕西、山西、云南、四川、甘肃5个种源地的269份花椒属样品的遗传多样性进行了研究。结果表明:从120对引物组合中筛选出了16对图谱带型清晰、丰富、重复性好的引物组合;269份花椒属样品用这些引物组合共扩增出169条清晰可重复的条带,其中多态性条带151条,多态性比率为93.8%;通过UPGMA分子系统聚类法,将269份花椒属种质划分为2个类群,即竹叶椒类群和花椒类群;在所研究的5个种源中,陕西花椒的遗传多样性水平最高,陕西和甘肃花椒的遗传距离最小,陕西和云南花椒的遗传距离最大;AMOVA 分子变异分析显示,在花椒总的遗传变异中,种源内占 74%,而种源间仅占 26%;Structure群体遗传结构和UPGMA聚类结果一致,且种源间存在明显的基因交流现象。结果为花椒资源的收集、分类和鉴定工作以及育种工作提供了理论依据。
花椒;SRAP分子标记;遗传多样性;遗传结构
花椒为芸香科花椒属植物,是我国特色辛香料和中药材树种,具有较高的经济价值,另外花椒抗旱、耐瘠薄,也是目前退耕还林中重要的生态型经济树种[1-2]。花椒目前在四川汉源、陕西韩城、凤县、山西芮城、甘肃武都、秦安、贵州、山东莱芜、河北涉县等地集中栽培[3-4],形成了一个巨大的特色产业。近年来,随着花椒科研和综合开发利用的深入发展,市场对花椒及其产品的需求量持续增加,花椒的发展前景十分广阔。
全世界花椒属植物约有250种,其中我国约有45种,较为常见、分布较广的主要有野花椒(Zanthoxyluymsimulans)、花椒(Z.bungeanumMaxim)、竹叶花椒(Z.armatum)、青花椒(Z.schinifolium)、川陕花椒(Z.piasezkiiMaxim)等[5-6]。在漫长的由野生种向栽培种驯化过程中,花椒也形成了诸多的品种,目前栽培较多的有大红袍、小红袍、枸椒、豆椒、白沙椒、秦安一号等[7]。由于各地分类依据不同使花椒的叫法颇多,花椒名称混淆、品种混杂等问题在科研与生产上都不利于我国花椒产业的发展,而且形态学、细胞学等常规的花椒分类方法容易受环境因素的影响[8-9]。因此,利用分子生物学方法,从基因水平开展相关研究能够更快速、准确、有效的鉴定花椒种质,这对花椒遗传改良、种质资源保存及开发利用研究等有重要的意义。
目前,在DNA分子水平对花椒的研究已有RAPD、ISSR及DNA测序等方面的报道。SRAP(相关序列扩增多态性)是一种基于PCR的新型分子标记技术,具有简单、高效及共显性高、重复性高等优点,现已在品种鉴定、遗传多样性检测、图谱构建、基因定位等方面得到应用[10]。目前,SRAP标记已广泛应用于我国的农作物[11-14]和蔬菜[15-17]等的遗传多样性分析、品种鉴定、遗传连锁图的构建、指纹图谱构建的研究,但在花椒中有关品种鉴定的应用目前相关报道很少。
鉴于此,本试验采用SRAP分子标记技术,对采自5省10个种源地的269份花椒属样品进行分子水平上的遗传多样性和群体遗传结构分析,以解析这些花椒种质之间的亲缘关系,分析遗传多样性及其变化规律,并进行花椒品种鉴定,希望能够为花椒丰产栽培、良种选育、良种的分子鉴别提供可靠的依据。
1 材料和方法
1.1 试验材料
从我国陕西、山西、云南、四川、甘肃5个种源地共收集到269份花椒属样品(表1),供试样株生长良好、无病虫害,能反映出群体的总体水平。根据花椒的栽培情况,按收集地点不同,每个地点随机取材,以幼嫩叶片作为试验材料。其中陕西109份,山西39份,云南20份,四川30份,甘肃71份。

表1 试验材料来源
1.2 花椒基因组DNA的提取
采集不同花椒种质的新鲜嫩叶,按不同种质密封包装,用冰袋保鲜,带回实验室置于-70 ℃冷冻保存备用;参照李晓、姜静等[18-19]CTAB方法对花椒嫩叶进行基因组DNA提取,用1.0%的凝胶电泳对DNA进行检测;将基因组DNA于-20 ℃保存备用。
1.3 SRAP-PCR引物组合的筛选
该反应在多功能PCR仪上进行。本试验PCR反应体系为:总体积20 μL,10 μL 2×TaqMasterMix(CWBIO北京生物技术有限公司(中国)有限公司),正反向引物(100 μmol/L)各1 μL,6.8 μL的双蒸水和1.2 μL的基因组DNA。PCR循环参照Feng 等[20]的程序。扩增产物经非变性聚丙烯酰胺凝胶电泳检测[21],凝胶浓度为8%,电泳缓冲液为0.5×TBE,稳压250 V,室温电泳2 h,用银染法显色。
用具有代表性的4 份花椒样品(凤县大红袍、秦安一号、青花椒、府谷花椒)的DNA作为模板对120对SRAP引物组合进行多态性引物的筛选,选用多态性好的引物用于后续的试验。
1.4 数据处理
选取可重复且清晰可辨的SRAP扩增条带,有SRAP谱带计为1,无SRAP谱带计为0,构建初始数据矩阵。计算多态性引物的总扩增条带和多态性条带及比率。用NTSYS-pc2.10软件[22]非加权平均法(UPGMA)计算遗传相似性系数(GS)并进行聚类分析构建聚类图;用Popgen32软件[23]计算群体的多态性位点百分率(PPB)、Nei′s 基因多样性指数(H)、观测等位基因数(Na)、Shannon′s信息指数(I)、有效等位基因数(Ne);用居群遗传分析软件GENALEX 6对群体进行分子方差分析(AMOVA)[24],计算种源内、种源间的变异方差;用Structure软件[25]对花椒群体结构进行基于模型的类群划分。先设定群体数目(K)为1~15,将 MCMC(Markov chain Monte Carlo)开始时的不作数迭代(Length of burn-in period)设为250 000次,再将不作迭代后的MCMC设为750 000次,每个K值重复10次,然后计算得到每个品种相应的ΔK值[25],选择合适的分组K值[26]。
2 结果与分析
2.1 SRAP-PCR引物的筛选与扩增
经 SRAP 引物筛选,其中16对引物组合能够扩
增出清晰且可重复的条带(表2),且16对引物组合共扩增出169条条带,DNA片段大小在 50~1 000 bp,平均每对引物的扩增条带数为10.56条,多态性条带151条,多态性比率为93.8%,表明花椒种质具有丰富的DNA序列多态性。

表2 本试验所用的SRAP引物
2.2 花椒种质的遗传多样性
2.2.1 Shannon′s多样性指数和Nei′s基因多样度分析 在假设等位基因频率符合Hardy-Weinberg 平衡的基础上,用Popgen32软件计算5个花椒种源内和整体的遗传多样性指数(表3)。
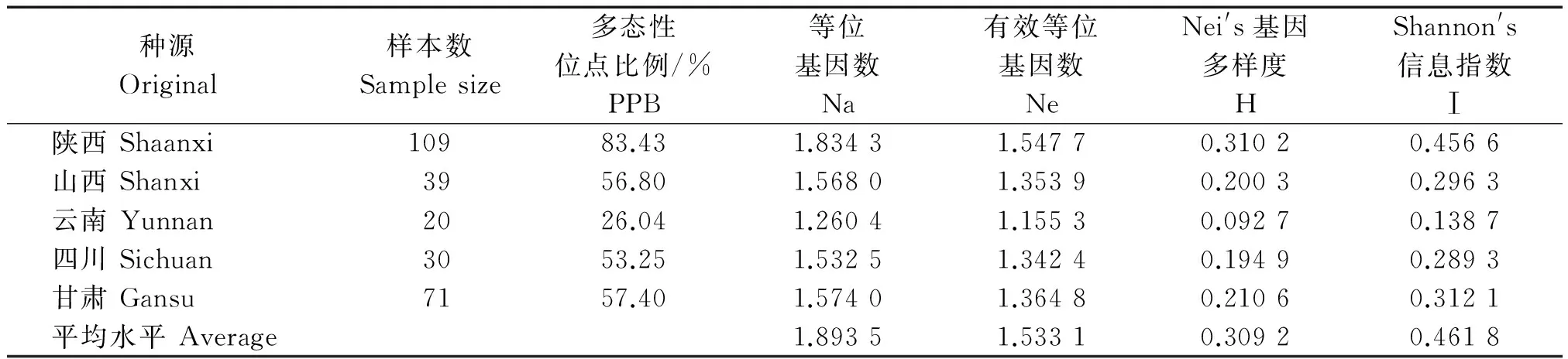
表3 SRAP标记估算花椒种源内的遗传多样性参数
表3分析结果表明,花椒各种源的多态位点百分率为26.04%~83.43%;5个花椒种源中,以陕西种源的遗传多样性最高(PPB=83.43%,Na=1.834 3,Ne=1.547 7,H=0.310 2,I=0.456 6),其次是甘肃、山西、四川、云南。花椒平均水平的遗传等位基因数是1.893 5,有效等位基因数为1.533 1,Nei′s基因多样度为0.309 2,Shannon′s信息指数为0.461 8。
2.2.2 遗传距离和遗传一致度分析 种源间的遗传一致度和遗传距离参数结果表明(表4):陕西和云南种源花椒的遗传距离最大(0.206 7),而遗传一致度最小(0.813 3);陕西和甘肃种源花椒的遗传距离最小(0.097 0),遗传一致度最大(0.907 5)。

表4 种源间遗传一致度(右上角)和遗传距离(左下角)

图1 基于SRAP标记的花椒聚类
Fig.1 Clustering map ofZanthoxylumbungeanum Maxim based on SRAP Markers
用Popgen32软件计算33个花椒群体的遗传距离与遗传一致度,结果表明,各花椒群体间遗传距离为0.000 0~0.648 1。其中陕西的竹叶椒与云南的野花椒遗传距离最大为0.648 1,其次是山西阳泉的黄金椒与四川的青花椒遗传距离为0.641 3;山西阳泉的花椒与山西忻州的黄金椒之间的遗传距离最小为0.000 0,其次为陕西韩城的狮子头与陕西韩城的大红袍遗传距离为0.021 5。遗传距离很好地体现了花椒种质之间的亲缘关系。
2.2.3 亲缘关系聚类分析 用NTSYS-pc2.10软件UPGMA法得聚类分析树状图(图1)。由图1可知,在相似系数0.59处所有花椒样品聚为2个类群,即竹叶椒类群和花椒类群。在相似系数为0.70处,所有的花椒样品可以聚为五类:山西阳泉的花椒、黄金椒和甘肃的白椒,枸椒、山西忻州的花椒、陕西韩城的臭椒、陕西榆林的府谷花椒以及山西的大红袍共8个品种为第Ⅰ类;陕西凤县的野花椒和甘肃的臭椒共2个品种为第Ⅱ类;陕西凤县大红袍山西的黄金椒、四川茂汶花椒、甘肃的大红袍,长柄大红袍,秦安一号、陕西凤县的秦安一号、云南的花椒,野花椒,四川的汉源花椒,大红袍共11个品种为第Ⅲ类;竹叶椒为第Ⅳ类,青花椒为第Ⅴ类。
2.2.4 AMOVA分子遗传变异分析 AMOVA分子变异分析结果表明(表5):在花椒种源总的遗传变异中,74% 的遗传变异发生在种源内,而只有 26% 的变异发生在种源间,即其遗传变异主要来源于种源内;种源遗传分化系数ΦST=0.256(P<0.001)。一般认为,ΦST≤0.05,种源间存在很小的遗传分化;0.05<ΦST<0.15,种源间存在中等程度遗传分化;ΦST≥0.15,种源间存在明显的遗传分化;本试验的遗传分化系数大于0.25,种源间遗传分化明显[27]。由此可见,花椒种源内存在显著的遗传分化。

表5 花椒种源的AMOVA分析
2.3 花椒种质的群体结构分析
Structure 软件是根据贝叶斯模型对个体间的遗传关系进行聚类分析,对单个样品进行组群分析。由图2可以看出,K=2时ΔK最大(图2),说明这269份花椒样本可以划分为2个类群,与UPGMA聚类结果相似;Structure 软件划分的花椒种源的遗传结构分析结果表明陕西、甘肃2个种源间划分的2个类群的样品数相差不大,山西、云南和四川划分的2个类群的样品数差异明显(图3)。
3 讨论
3.1 种源遗传多样性
遗传多样性是1个居群生存、发展和进化的基础,是生物所携带遗传信息的总和[28]。花椒遗传多样性分析结果显示,陕西花椒的遗传多样性最高(PPB=83.43%,Na=1.834 3,Ne=1.547 7,H=0.310 2,I=0.456 6),云南花椒显示出较低的遗传多样性(PPB=26.04%,Na=1.260 4,Ne=1.155 3,H=0.092 7,I=0.138 7);而且陕西和云南的遗传距离最大(0.206 7),陕西和甘肃的遗传距离最小(0.097 0),不同种源遗传多样性参数差异明显。遗传多样性的变化原因复杂,其本质原因是物种等位基因数目或频率的变化,同时受生殖方式、遗传漂变、基因流、自然选择等因素影响,还包括人为干扰、环境变化及地理隔离等。陕西省区的样本涵盖了至少4个种系的种质,而其他各省区基本上是2~3个种系,陕西区的种质遗传基础比其他各区宽,遗传多样性水平自然偏高。另外,由于各省区花椒种质的重合度高,遗传相似性(一致度)水平通常较高。从地理区域看,陕西与甘肃地理近缘,交互引种现象频繁,基因流影响较大,遗传相似性(一致度)趋高,而云南由于地理隔离,与陕西产区间不存在自然的基因流影响,加上环境条件差异等,遗传相似性自然趋小。而且各种源与陕西之间的遗传距离越大,遗传多样性越小。此外,造成不同种源花椒基因多样性的另一个原因可能是农业上为适应当地的环境而对花椒进行了选育。这一结果表明在农业上对于保留和管理花椒遗传多样性是很重要的,同时为日后培育优良花椒品种提供了良好的基因资源。

图2 Structure 软件分析推定的种群分类数 K 值所对应的 ΔK 散点图

图3 使用 Structure 软件划分的花椒种源的遗传结构
花椒种源的分子方差(AMOVA)分析结果表明遗传多样性主要是由于种源内的变异,且遗传分化显著,因地域环境的不同,花椒长期在这种情况下会产生较大的自然变异,这些多发生在控制数量性状位点上的变异,导致花椒的品质有不同程度的变化[29]。陕西遗传多样性最高的原因一方面是由于农业发展上对花椒进行选育工作增加了种源间基因的多样性,另一方面由于试验所采样本数量最大,涵盖范围广。
与 Feng 等[20]SRAP标记的花椒遗传多样性分析相比,各种源的遗传多样性在本试验中均呈现较低的水平(除陕西),这是由于本试验中搜集品种的遗传背景相对较窄,未能充分代表各种源的多样性和复杂性,因此还需要进一步收集全国不同产区的栽培材料对花椒种质资源的遗传多样性进行全面的评估,为花椒属种质资源的开发和利用奠定基础。
3.2 种源群体遗传结构
在聚类分析中,形态相似、来源于同一地区或相近区域的材料聚在一起,陕西、山西、甘肃的大部分品种聚在一起,云南、四川的大部分品种聚在一起,通过分子标记技术可以进一步确立它们之间的亲缘关系。在33个花椒群体中山西阳泉的花椒与山西忻州的黄金椒之间的遗传距离为0.000,说明二者为同一个品种,即出现 “同种异名”的现象,同时还发现同一种源不同采集地相同品种之间也有一定遗传距离,但是较小,如甘肃天水市秦安县与天水市元龙镇的油椒之间的遗传距离为0.062 3,出现这种结果的原因一方面是由于花椒因长期地域环境不同产生的自然变异,以致花椒的品质有不同程度的变化,另一方面的原因也会因花椒长期的授粉中接受了外来基因。该结果既揭示了各个群体的遗传多样性,也为今后选育优良花椒品种提供了丰富的理论依据,又表明各群体间的遗传距离,为花椒资源的收集、分类和鉴定工作提供了依据。
群体遗传结构分析结果与UPGMA聚类结果相似。5个不同种源的269 份花椒属样品的遗传多样性分析结果表明:陕西与甘肃2个种源的遗传多样性水平和遗传相似性较高,且遗传距离较小,群体结构较为相似,且Structure 分析结果也得出相似的结论,以此推测这 2个种源为2个组群不同程度的混合。本试验分析结果表明:花椒属植物遗传多样性水平高、种质资源培育价值高的优点使花椒具备良好的发展利用前景,SRAP分子标记能有效地揭示花椒属植物间的亲缘关系和遗传多样性,这为农业上选育优良花椒品种提供理论依据,对促进该邻域发展具有重要意义。
但目前,各地对花椒品种的分类与命名多采用历史沿袭下来的俗名。在生产上,同一品种(类型)在不同地区叫法不一样,叫法一样又可能不是同一品种(类型)[30]。目前没有一个权威的、统一的方法对花椒品种进行分类与命名,因此,有关花椒的品种分类与命名还需要进一步的探讨及研究。
[1] 汉素珍,王有科,李 捷,等.甘肃省主产花椒品种ISSR遗传多样性分析[J].甘肃农业大学学报,2011,46(6):46-51.
[2] 张卫明,肖正春.中国辛香料植物资源开发与利用[M].南京:东南大学出版社,2007.
[3] 史劲松,顾龚平,吴素玲,等.花椒资源与开发利用现状调查[J].中国野生植物资源,2003,22(5):6-8.
[4] 魏安智,杨途熙,周 雷.花椒安全生产技术指南[M].北京:中国农业出版社,2012.
[5] 朱晓慧,杨途熙,魏安智.无刺花椒嫁接及其亲和性研究[D].杨凌:西北农林科技大学,2014.
[6] 张卫明.植物资源开发研究与应用[M].南京:东南大学出版社,2005.
[7] 李 洋,魏安智.花椒生物碱和挥发油含量的近红外光谱无损检测[D].杨凌:西北农林科技大学,2012.
[8] Ravindran P N,Nirmal B K.Genetic resources of black pepper[M].New Delhi:Malhotra Publishing House,1994:99-120.
[9] Li G,Quiros C F.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J].Theoretical and Applied Genetics,2001,103(2/3):455-461.
[10] 孙祖霞,刘兆磊,陈素梅,等.荷花SRAP-PCR反应体系的优化与确立[J].南京农业大学学报,2011,34(6):53-58.
[11] 羊杏平,刘 广,侯喜林,等.西瓜核心种质枯萎病抗性与SRAP分子标记的关联分析[J].园艺学报,2013,40(7):1298-1308.
[12] Ferriol M,Picó B,Nuez F.Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers[J].Theoretical and Applied Genetics,2003,107(2):271-282.
[13] 姜树坤,马 慧,刘 君,等.利用SRAP标记分析玉米遗传多样性[J].分子植物育种,2007,5(3):412-416.
[14] 潘庆华,胡铁柱,王 玲,等.稻瘟病菌群体的分子遗传学研究-广东地区特异性宗谱菌株的分子指纹和致病型分析[J].中国农业科学,2004,37(1):57-64.
[15] 王 燕,龚义勤,赵统敏.番茄SRAP-PCR体系优化与品种分子鉴定[J].南京农业大学学报,2007,30(1):23-29.
[16] Pan J S,Wang G,Li X Z,et al.Construction of a genetic map with SRAP markers and localization of the gene responsible for the first-flower-node trait in cucumber(CucumissativusL.) [J].Progress in Natural Science,2005,15(5):407-413.
[17] 雷 剑,柳 俊.一个与马铃薯青枯病抗性连锁的SRAP标记筛选[J].中国马铃薯,2006,20(3):150-153.
[18] 李 晓,杨途熙,魏安智,等.花椒DNA提取方法的研究[J].北方园艺,2011(7):130-132.
[19] 姜 静,杨传平,刘桂丰,等.桦树ISSR-PCR反应体系的优化[J].生态学杂志,2003,22(3):91-93.
[20] Feng Shijing,Yang Tuxi,Liu Zhenshan,et al.Genetic diversity and relationships of wild and cultivatedZanthoxylumgermplasms based on sequence-related amplified polymorphism(SRAP) markers[J].Genetic Resources & Crop Evolution,2015,62(8):1193-1204.
[21] 徐刚标,禹玉婷,申响保.湘鄂西地区珙桐天然群体遗传结构的研究[J].中南林业科技大学学报,2007,27(6):5-9.
[22] Rohlf F J.NTSYSpc:numerical taxonomy and multivariate analysis system,version 2[Z].1998.
[23] Yeh F C,Yang R C,Boyle T,et al.POPGENE version1.31[Z].1999.
[24] Excoffier L,Smouse P E,Quattro J M.Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J].Genetics,1992,131(2):479-491.
[25] Evanno G,Regnaut S,Goudet J.Detecting the number of clusters of individuals using the software Sturcture:a simulation study[J].Molecular Ecology,2005,14(8):2611-2620.
[26] 李仁伟,王 晨,戴思兰.等.菊花品种表型性状与 SRAP 分子标记的关联分析[J].中国农业科学,2012,45(7):1355-1364.
[27] Souza J G,Souza V A,Lima P S.Molecular characterization of Platonia insignis Mart.("bacurizeiro")using inter simple sequence repeat(ISSR)markers[J].Molecular Biology Reports,2012,40(5):3835-3845.
[28] 倪 穗,李纪元,王 强.20个茶花品种遗传关系的ISSR分析[J].林业科学研究,2009,22(5):623-629.
[29] 李晓林,梁国鲁,郭启高,等.国产主要花椒的同工酶分析[J].西南农业大学学报:自然科学版,2004,26(4):445-447,455.
[30] 毕 君,王春荣,赵京献,等.北方花椒主产区种质资源考察报告[J].河北林果研究,2003,18(2):165-168.
Genetic Diversity and Population Structure Analysis ofZanthoxylumGermplasms by SRAP
LI Lixin,YANG Tuxi,WEI Anzhi,FENG Shijing,CHEN Lü,HOU Na
(College of Forestry,Northwest Agriculture and Forestry University,Yangling 712100,China)
To explore the genetic diversity and population structure ofZanthoxylumgermplasm,the genetic diversity among 269 accessions ofZanthoxylumwhich collected from Shaanxi,Shanxi,Yunnan,Sichuan and Gansu Provinces was studied by SRAP Markers.The results showed that 16 pairs of SRAP primes,amplified clear repetitive and intense bands,were selected from 120 combinations.A total of 169 clear bands were amplified,there were 151 polymorphic bands and the percentage of polymorphic bands was 93.8%;The UPGMA clustering showed that the populations were divided into two main groups,Z.armatumDC.andZ.bungeanumMaxim.The highest genetic diversity level was observed within Shanxi Province,the minimum genetic distance was between Shaanxi and Gansu,the maximum was between Shaanxi and Yunnan.The AMOVA showed that the genetic variation was mainly caused by variation of intra-provenances(74%),rather than among provinces(26%);the results of population genetic structure were consistent with the UPGMA clustering and indicated obvious gene flow among provinces.The results provide a basis for the collection,classification and identification ofZanthoxylumresources.
Zanthoxylumgermplasms;SRAP Markers;Genetic diversity;Genetic structure
2016-07-12
国家林业局林业公益性行业科研专项(201304706);西北农林科技大学产业技术集成与示范推广(TGZX2016-08);贵州省林业厅中药现代化项目(黔科合ZY字[2012]3002号)
李立新(1990-),女,河北沧州人,在读硕士,主要从事林木遗传育种研究。
杨途熙(1963-),男,陕西杨凌人,教授,硕士,主要从事林木遗传育种研究。
魏安智(1961-),男,陕西扶风人,教授,博士,博士生导师,主要从事林木遗传育种研究。
S573.03
A
1000-7091(2016)05-0122-07
10.7668/hbnxb.2016.05.018

