基于烟草叶绿体基因组和线粒体基因组SSR标记的烟属植物遗传多样性分析
曾建敏,陈学军,吴兴富,焦芳婵,肖炳光,李永平,童治军
云南省烟草农业科学研究院,烟草行业烟草生物技术育种重点实验室,国家烟草基因工程研究中心,昆明 650021
基于烟草叶绿体基因组和线粒体基因组SSR标记的烟属植物遗传多样性分析
曾建敏,陈学军,吴兴富,焦芳婵,肖炳光,李永平,童治军
云南省烟草农业科学研究院,烟草行业烟草生物技术育种重点实验室,国家烟草基因工程研究中心,昆明 650021
利用本实验室前期开发的烟草叶绿体基因组和线粒体基因组SSR标记,对属于3个烟草亚属、9个组共35份野生烟草种质资源进行了遗传多样性分析。利用20对SSR引物在35份野生烟草材料中共扩增获得176个多态性条带,每对SSR引物可检测的多态性条带数为2~19个,平均为8.8个。35份材料间遗传相似性系数(GS)在0.20~1.00之间,平均为0.57,表明烟属种间的遗传多样性丰富,遗传差异较大,亲缘关系较远。虽然3个亚属间的界限不明显,但在适当的遗传相似性系数条件下,除香甜烟草组外,35份野生烟草按其组可清晰地聚为7类,表明2种细胞器基因组SSR标记适合用于烟草种间进化、分类、遗传分析等方面研究。
野生烟草;遗传多样性分析;简单序列重复标记
烟草在植物分类学上属于茄科(Solanaceae)、烟草属(Nicotiana),其起源于美洲、大洋洲、南太平洋上一些岛屿及近代非洲[1-2]。根据其原产地、植物学形态特征、染色体数目、染色体形态结构、染色体联会特点、种间杂交的可能性、种间杂种育性等,美国学者Goodspeed将烟草属分为黄花烟亚属(Rustica)、普通烟亚属(Tabacum)和碧冬烟亚属(Petunioides)3个亚属、14个组、60个种[3]。随后,人们对Goodspeed的工作进行了大量深入研究并做了修正与扩充,1994年,日本烟草产业株式会社对烟草属66个种进行了系统的分类[4],2004年,Knapp认为烟草属有76个种[5]。而种植的烟草只有2个种,即普通烟草和黄花烟草,因育种工作者过度依赖少数骨干亲本,造成普通烟草(尤其是烤烟)栽培品种间的遗传背景越来越趋同、遗传基础愈来愈狭窄、可利用的种质资源也越来越匮乏[6-7]。
早期,因基因组信息匮乏,在分子水平上对烟草遗传育种的研究进展缓慢,主要局限于随机引物扩增长度多态性(Random Ampli fi ed Polymorphic DNA,RAPD)[8-10]、扩增片段长度多态性(Ampli fi ed Fragment Length Polymorphic,AFLP)[11-13]、 序 列相关扩增多态性标记(Sequence Related Ampli fi ed Polymorphism,SRAP)[6]和区间简单序列重复(Inter-Simple Sequence Repeats,ISSR)[6,14]等非特异性的分子标记。这些非特异性的分子存在标记稳定性差、多态性低、操作复杂、重复性差、标记显性,且应用成本较高等缺点。SSR标记克服以上缺点,但因缺乏足够的烟草基因组信息,直到2007年,首张SSR标记的烟草遗传连锁图谱才公布,使得基于SSR标记的烟草种质资源亲缘关系(遗传多样性)的研究方兴未艾,但这些研究均集中在烟草的栽培品种上[15-19]。作为细胞器的叶绿体和线粒体基因组在植物种属间具有很高的保守性,因此,细胞器基因组SSR不仅具有核基因组SSR标记的共显性、重复性好等固有特点,而且还具有很好的种属通用性[20],继而被广泛应用于物种的起源、进化、分类及种质资源多样性分析等研究领域[21-23]。迄今,基于烟草细胞器基因组SSR标记对野生烟草种质资源的研究在国内外尚未见报道。因此,利用烟草叶绿体基因组和线粒体基因组SSR标记对野生烟草种质资源进行遗传分析,在促进烟草属内各近缘种遗传研究的发展、挖掘和利用烟草野生种的优良基因、拓宽烟草栽培种质的遗传基础等方面就显得十分迫切和重要。
本研究利用本实验室前期公布的烟草细胞器基因组SSR标记[24],对属于3个烟草亚属、9个组的35份野生烟草种质资源的亲缘关系进行研究,旨在为从分子水平上对野生烟草种质资源的研究、利用奠定理论基础。
1 材料与方法
1.1 材料
试验所用的35份野生烟草种质资源由云南省烟草农业科学研究院、中国烟草育种研究(南方)中心提供,其中,黄花烟亚属1个组包含了4份材料(W01~W04);普通烟亚属1个组包含了6份材料(W05~W10);其他25份材料(W11~W35)属于碧冬烟亚属的7个组。35份野生烟草材料的详细信息见表1。
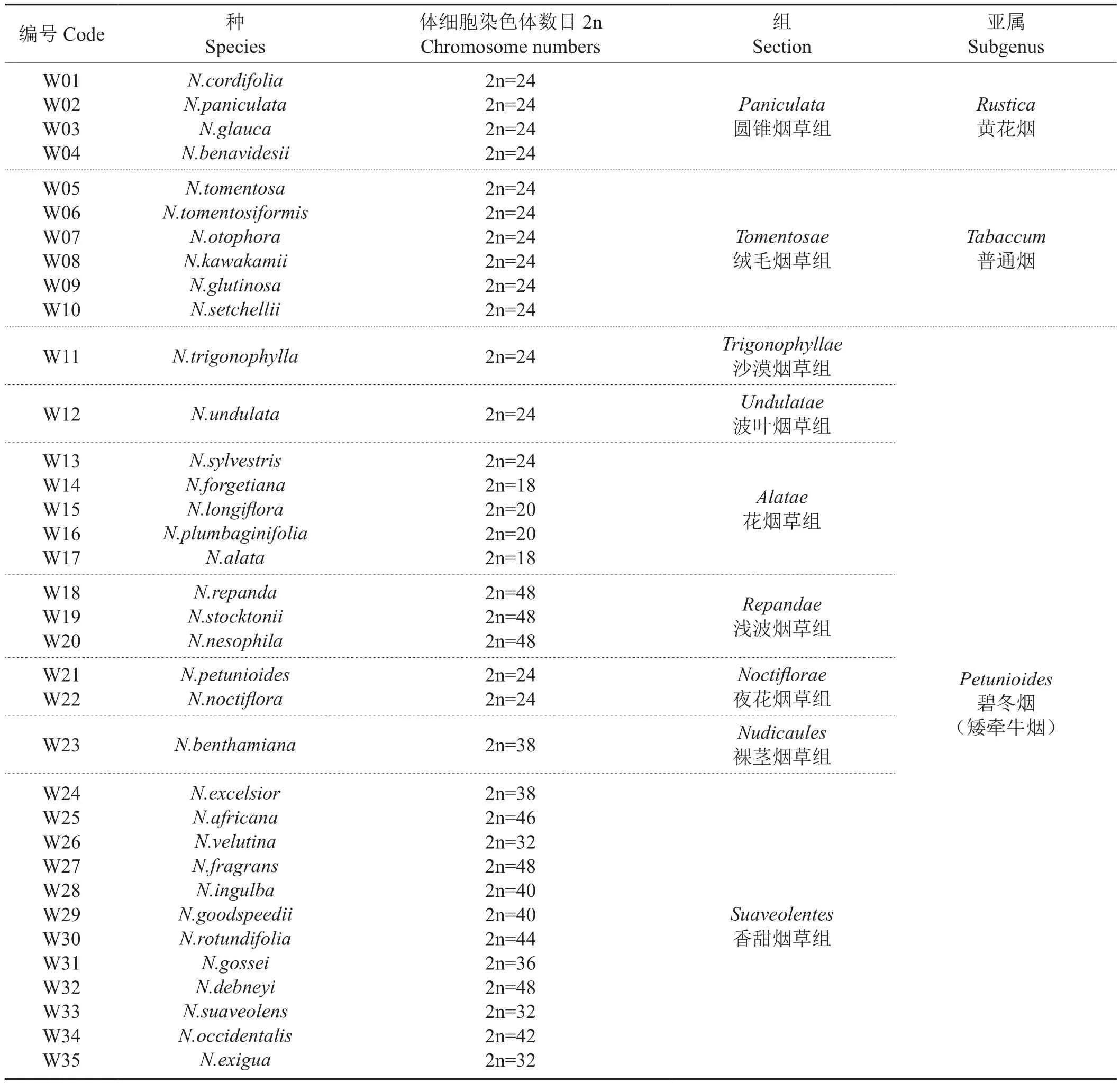
表1 35份供试材料在烟草属的分类信息Tab.1 Classi fi cation information of 35 materials in genus Nicotiana
1.2 方法
1.2.1 烟草基因组DNA提取
35份供试野生烟草材料基因组DNA的提取、纯化和稀释,参照梁景霞等[25]的CTAB大量法略作修改。其中将65℃水浴时间延长至60 min,将离心转速提高至12000 rpm,以提高烟草基因组DNA的得率和纯度。
1.2.2 烟草叶绿体基因组和线粒体基因组SSR引物
在本实验室前期公布的烟草细胞器基因组SSR引物中[24],选取能够扩增出条带清晰、重复性好且在野生烟草中具有较好多态性的SSR引物各10对,引物的详细信息见表2。

表2 烟草叶绿体基因组和线粒体基因组SSR引物序列信息Tab. 2 Information of SSR primer pairs in chloroplast genome and mitochondria genome of Nicotiana
1.2.3 PCR扩增分析
烟草叶绿体基因组和线粒体基因组SSR引物的PCR扩增及扩增产物的6%非变性聚丙烯酰胺凝胶电泳检测,参照李绪英等[24]的方法进行。
1.2.4 数据处理
参照李岩等[26]的方法,对PCR扩增产物的检测结果进行统计、整理。根据Nei等[27]提供的公式计算35份野生烟草材料间的遗传相似系数(Genetic similarity coefficient , GS),利用NTSYSpc Version 2.11软件[28]进行聚类分析,并构建分子进化系统树。
2 结果与分析
2.1 烟草叶绿体基因组和线粒体基因组SSR标记的多态性分析
烟草叶绿体基因组和线粒体基因组SSR引物均能稳定的扩增出清晰、大小与预期片段相符的条带(图1)。20对SSR引物在35份野生烟草材料中共扩增获得176个多态性条带,每对SSR引物可检测的多态性条带数为2~19个,平均为8.8个。其中,10对基于烟草叶绿体基因组开发的SSR引物在35份野生烟草材料中共扩增得到59个多态性条带,每对SSR引物可检测的多态性条带数为2~13个,平均为5.9个。10对烟草线粒体基因组SSR在供试的35份野生烟草中共扩增出117个多态性条带,每对引物可检测3~19个多态性条带,平均为11.7个。可见,基于烟草叶绿体基因组和线粒体基因组序列开发的SSR标记在检测野生烟草基因组遗传多态性上有很高的检出效率。

图1 引物Tcp067在35份野生烟草中的电泳结果Fig. 1 PCR products of SSR primer Tcp067 on non-denaturing PAGE by silver staining in 35 wild Nicotiana species
2.2 35份野生烟草种质的遗传相似性分析
遗传相似性分析结果表明,参试的35份烟草野生资源的遗传多样性丰富,相似性系数在0.20~1.00之间,变化范围较大,平均为0.57(表3)。其中,N.suaveolens(W33)与其他 34份烟草材料间的遗传相似性系数最高,平均为0.86;而N.africana(W25)与其余34份烟草材料间的遗传相似性系数最低,平均为0.23。同属于香甜烟草组(Suaveolentes)的N.gossei(W31)和N.suaveolens(W33)间的遗传相似性系数最大,达到1.00;同属于圆锥烟草组(Paniculata)的N.paniculata(W02)和N.benavidesii(W04),及同属于香甜烟草组(Suaveolentes)的N.occidentalis(W34)与N.gossei(W31) 和N.suaveolens(W33) 间 的 遗 传相似性系数均高达0.98;而N.africana(W25)分别与N.paniculata(W02)、N.benavidesii(W04)、N.tomentosa(W05)、N.tomentosiformis(W06)、N.otophora(W07)和N.kawakamii(W08)间的遗传相似性系数最低,为0.20。

表3 35份野生烟草材料间的遗传相似性系数Tab. 3 Genetic similarity coefficient in 35 wild tobacco materials
2.3 35份野生烟草种质的聚类分析
从UPGMA法聚类结果(图2)看出,供试的35份野生烟草资源并未完全按照经典的三个亚属(黄花烟亚属(Rustica)、普通烟亚属(Tabaccum)和 碧冬烟亚属(Petunioides)进行分类,即在分子水平上,无法将参试的35份野生烟草材料清晰的划分为各自所属的亚属,但各个不同的组(section)却能较好的区分开。在相似性系数为0.50处,可将新发现于非洲的野生烟N.africana(W25)与剩余的34份材料划分开,而剩余的34份材料在适当的遗传相似性系数处,则能很清晰的聚为10个组,但3个亚属间的界限却不明显。在聚类所获得的10个烟草组中,除香甜烟草组(Suaveolentes)外,其余7个组与传统的分组相吻合。

图2 35份野生烟草材料的聚类分析树状图Fig.2 Dendrogram derived from UPGMA cluster analysis of 35 wild tobacco materials
3 讨论
3.1 基于烟草叶绿体基因组和线粒体基因组SSR标记的野生烟草资源研究
在分子水平上对烟草种质资源遗传多样性的研究报道较多,但绝大部分的研究集中在RAPD、AFLP、SRAP和ISSR等非特异性标记对属于普通烟亚属栽培种间的亲缘关系分析[6,8-14]。利用特异性的SSR标记对烟草种质的研究报道始于2007年,之后应用广泛,如烤烟[17]、栽培烟草[18]亲缘关系分析,赤星病抗、感烟草资源区分[22],但核基因组SSR标记在烟草种间的可用率极低[16]。
迄今为止,尚未有利用烟草叶绿体基因组和线粒体基因组SSR标记对野生烟草种质资源进行遗传多样性分析的研究。本研究利用10个烟草叶绿体基因组SSR标记和10个烟草线粒体基因组SSR标记,在35份野生烟草种质资源(35个野生烟草种)中分别扩增出59和117个多态性条带,每个SSR标记可检测的多态性条带数分别为2~13和3~19个,平均为5.9和11.7个,且20个SSR标记在35个野生烟草种间均能进行有效的PCR扩增。两种细胞器基因组SSR标记在35个野生烟草种间的多态率(多态性SSR标记数目/总SSR标记数目×100)及每个多态性SSR标记可检测的多态性条带数差异较大,烟草线粒体基因组SSR标记具有更高的效率,多态率约为叶绿体基因组SSR标记的两倍。虽然叶绿体和线粒体内各自含有一套不同于细胞核且具有相对独立的遗传物质,但绿色植物协同进化过程中,叶绿体能够使维持自身基本功能的许多遗传物质以母性遗传的方式较好保留下来,比线粒体种属间的保守性更强。由此可见,叶绿体基因组和线粒体基因组在生物的种属间均具有很强的保守性,其在烟属种间具有丰富的遗传多样性,这两种细胞器基因组SSR标记也比细胞核基因组SSR标记具有更强的种属通用性,基于烟草细胞器基因组SSR标记在种间或更高分类级别上对烟草遗传育种领域的研究是可行的。
3.2 基于烟草叶绿体基因组和线粒体基因组SSR标记的野生烟草种聚类分析
在分子水平上,对烟草属内66个种的起源、进化及亲缘关系的研究报道已有很多。如Aoki等[29]利用烟草叶绿体matK基因序列证实了烟草祖先种染色体基数为n=12,且起源于南美洲。Chase等[30]利用核糖体DNA转录间隔区(ITS)并结合基因组原位杂交(GISH)方法,对烟草属内66个种进行系统发育及属内杂交种起源研究。Clarkson等[31]利用烟草内含子trnL、间隔子trnL-F和trnS-G、基因ndlF和matK等质体DNA序列的聚类结果表明,N.tabacum起源于南美洲的南部。随后,Clarkson等[32]研究表明,利用低拷贝的核基因序列寻找多倍体的祖先种非常有用。对野生烟草种的聚类分析大多采用 AFLP标记[33]、核基因组SSR标记[16]等。本研究利用烟草叶绿体基因组和线粒体基因组SSR标记[20],对35份野生烟草材料进行分类,但结果与以形态特征和生化表型为基础的系统分类存在一定的出入,同一亚属的野生烟草种并未聚在一类,而同一组(除香甜烟草组)的野生烟草材料却能在适当的遗传相似性系数下清晰的划分在一起。香甜烟草组(Suaveolentes)中的12份材料(W24~W35,见表1)则被划分成3个部分, 即N.velutina(W26) 和N.rotundifolia(W30)为一组,N.fragrans(W27)和N.debneyi(W32)为一组,剩余的8份材料为一组。此外,本属于该组的N.africana(W25)则被单独归为一类,而属于裸茎烟草组(Nudicaules)的N.benthamiana(W23)则被归为香甜烟草组(Suaveolentes)。该结果与Moon等[16]基于烟草核基因组SSR标记的研究结果一致,而与李凤霞等[33]基于AFLP标记的研究结果截然不同。究其原因:1)可能是烟草属3个亚属下的分组在现有的研究中差别较大,以形态特征为基础的传统分类方法将烟草属分为14个组[1,3],而以植物细胞核和质体基因组区域特点为依据的分类方法将烟草属分为13个组[2,4],且组内的成员也存在较大差异。2)可能是烟草属内的各个种多数是通过复杂的组间杂交而形成的[1-5],各个种之间的亲缘关系错综复杂。3)可能是分子标记的不同而导致聚类分析结果的不同。比如AFLP标记,受限制性内切酶种类等选择性因素的制约,不能在DNA水平上全面、真实地揭示出个体间的差异,导致其在聚类分析中的结果不如SSR标记的准确[34-36]。
综上所述,细胞器基因组SSR标记不仅具有核基因组SSR标记的特异、稳定、可重复、共显等固有优点,且还具有较高的种属通用性,因而更适合在种间或更高分类级别上对烟草属的起源、进化、分类及种质资源多样性分析等进行研究;但因其在同一物种内(种内)各品种间的遗传多态性检测效率极低而不适合种内聚类分析研究。
[1]GOODSPEED T H. The Genus Nicotiana[M]. The Chronica Botania Company, 1954:90-116
[2]BALDWIN B G. Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants:an example from the Compositae[J]. Molecular Phylogenetics and Evolution, 1992, 6(1):3-16
[3]GOODSPEED T H. The genus Nicotiana[J].ChronicaBotan ica,1954,16:531-536
[4]Japan tobacco Inc., The genus Niotiana illustrated[M].Japan Tobacco Inc. 1994
[5]Knapp S, Chase MW, Clarkson JJ. Nomenclatural changes and a new section classi fi cation inNicotiana(Solanaceae)[J]. Taxon,2014,52:73–82
[6] 祁建民,梁景霞, 陈美霞, 等. 应用ISSR与SRAP分析烟草种质资源遗传多样性及遗传演化关系[J].作物学报,2012, 38(5):1425-1434 QI Jianmin, LIANG Jingxia, CHEN Meixia, et al. Genetic diversity and evolutionary analysis of tobacco (Nicotiana tabacumL.) germplasm resources based on ISSR and SRAP markers[J], ACTA AGRONOMICA SINICA, 2012, 38(8):1425−1434
[7]王元英,周健. 中美主要烟草品种亲源分析与烟草育种[J].中国烟草学报, 1995, 2(3):11-22 WANG Yuanying, ZHOU Jian. Parentage analysis of major tobacco varieties and tobacco breeding in American and China[J]. Acta Tabacaria Sinica, 1995, 2(3):11-22
[8]DEL PIANO L, ABET M, SORRENTINO C, et al. Genetic variability inNicotiana tabacumandNicotianaspecies as revealed by RAPD procedure[J]. International Contribution to Tobacco Research, 2000, 19:1-15
[9]ARSLAN B, OKUMUS A. Genetic and Geographic Polymorphism of Cultivated Tobaccos (Nicotiana tabacumL.) in Turkey [J].Russian Journal of Genetics, 2006,42(6):667–671
[10]BAI D, REELEDER R, BRANDLE JE. Identification of two RAPD markers tightly linked with theNicotiana debneyigene for resistance to black root rot of tobacco [J].Theor Appl Genet, 1995, 91: 1184-1189
[11]REN N, TIMKO MP. ALFP analysis of genetic polymorphism and evolutionary relationships among cultivated and wildNicotianaspecies [J]. Genome, 2001,44:559-571
[12]JULIO E, VERRIER JL, DORLHAC DE BORNE F.Development of SCAR markers linked to three disease resistances based on AFLP withinNicotiana tabacumL[J].Theor Appl Genet, 2006, 112:335-346
[13]SIVA R K, MADHAV M S, SHARMA R K, et al. Genetic polymorphism of Indian tobacco types as revealed by amplified fragment length polymorphism [J]. CURRENT SCIENCE, 2008, 94(5):633-639
[14]JESSADA D, SORNSUDA S, WILASINEE S, et al.Determination of Local Tobacco Cultivars Using ISSR Molecular Marker [J]. Chiang Mai J. Sci., 2010, 37(2):293-303
[15]BINDLER G, VAN DER HOEVEN R, GUNDUZ I, et al.A microsatellite marker based linkage map of tobacco [J].Theor Appl Genet, 2007, 114:341-349
[16]MOON H. S, NICHOLSON J. S, LEWIS R. S. Use of transferableNicotiana tabacumL. microsatellite markers for investigating genetic diversity in the genusNicotiana[J].Genome, 2008, 51:547-559
[17]MOON H. S, NICHOLSON J. S, HEINEMAN A, et al.Changes in Genetics Diversity of U.S. Flue-Cured Tobacco Germplasm over Seven Decades of Cultivar Development[J]. Crop Science, 2009, 49:498-506
[18]MOON H. S, NIFONG J. M, NICHOLSON J. S, et al.Microsatellite-based Analysis of Tobacco (Nicotiana tabacumL.) Genetic Resources [J]. Crop Science, 2009,49:2149-2159
[19]TONG Z J, JIAO T L, WANG F Q, et al. Mapping of quantitative trait loci conferring resistance to brown spot in flue-cured tobacco (Nicotiana tabacumL.)[J]. Plant Breeding, 2012, 131:335-339
[20]Kovaik A, Fajkus J, Koukalovfi B, et al. Species-specific evolution of telomeric and rDNA repeats in the tobacco composite genome [J]. Theor Appl Genet, 1996, 92:1108-1111
[21]Yukawa M, Tsudzuki T, Sugiura M. The chloroplast genome ofNicotiana sylvestrisandNicotiana tomentosiformis:complete sequencing con fi rms that theNicotiana sylvestrisprogenitor is the maternal genome donor ofNicotiana tabacum[J]. Mol Gen Genomics, 2006, 275: 367-373
[22]ALES K, MARTINA D, YOONG K L, et al. Evolution of rDNA inNicotianaAllopolyploids: A Potential Link between rDNA Homogenization and Epigenetics [J]. Annals of Botany, 2008, 101: 815–823
[23]Claire E M, Ed Newbigin, Pauline Y L. Comparative morphology and phylogeny ofNicotianasectionSuaveolentes(Solanaceae) in Australia and the South Paci fi c [J]. Australian Systematic Botany, 2011, 24: 61-86
[24]李绪英,肖炳光,高玉龙, 等. 烟草叶绿体基因组和线粒体基因组SSR位点分析[J].西北植物学报,2011,31(12):2399-2405 LI Xuying, XIAO Bingguang, GAO Yulong, et al. Analysis of SSR loci in chloroplast genome and mitochondria genome of tobacco (Nicotiana) [J]. Acta Bot Boreal. –Occident Sin, 2011,31(12):2399-2405
[25]梁景霞,祁建民,方平平, 等. 烟草种质资源遗传多样性与亲缘关系的ISSR聚类分析[J].中国农业科学,2008,41(1):286-294 LIANG Jingxia, QI Jianmin, FANG Pingping, et al.Genetic diversity and genetic relatives analysis of tobacco germplasm based on Inter-simple sequence repeat (ISSR)[J]. Scientia Agricultura Sinica, 2008,41(1):286-294
[26]李岩,童治军,焦芳婵, 等. 基于SSR标记对33份烟草材料的聚类分析[J].西北植物学报,2013,33(10):1972-1980 LI Yan, TONG Zhijun, JIAO Fangchan, et al. Cluster analysis of thirty-three tobacco germplasms based on SSR markers[J]. Acta Bot Boreal. –Occident Sin,2013,33(10):1972-1980
[27]NEI M, LI W H. Mathematical model for studying genetic variation in terms of restriction endonueleases[J]. Proc Natl Acad Sci USA, 1979, 76:5269-5273
[28]ROHLF F J. NTSYSpc numerical taxonomy and multivariate analysis system, Version2.0 Exeter Software,Setauket New York.USA, 1998
[29]AOKI S, ITO M. Molecular phylogeny ofNicotiana(Solanaceae) based on the nucleotide sequence of theMatKgene [J]. Plant Biology, 2000, 8(2):316-324
[30]CHASE M W, KNAPP S, COX A V, et al. Molecular systematics, GISH and the origin of Hybrid Taxa inNicotiana(Solanaceae)[J].Annals of Botany,2003, 92:107-127
[31]CLARKSON J J, KNAPP S, GARCIA V F, et al.Phylogenetic relationships inNicotiana(Solanaceae)inferred from multiple plastid DNA regions [J]. Molecular Phylogenetics and Evolution, 2004, 33:75-90
[32]CLARKSON J J, KELLY L J, LEITCH A R, et al. Nuclear glutamine synthetase evolution inNicotiana: Phylogenetics and the origins of allotetraploid and homoploid (diploid)hybrids [J]. Molecular Phylogenetics and Evolution, 2010,55:99-112
[33]李凤霞,王卫锋,王鲁, 等. 烟草属植物遗传多样性和亲缘进化关系的荧光AFLP分析[J]. 中国农业科学,2010,43(12):2418-2427 LI Fengxia, WANG Weifeng, WANG Lu, et al. Fluorescent AFLP analysis of genetic diversity and evolutionary relatives ofNicotianagenus[J]. Scientia Agricultura Sinica,2010,43(12):2418-2427
[34]杨友才, 周清明, 尹晗琪. 利用 RAPD 和 AFLP 标记分析烟草种质资源的遗传多样性[J]. 农业生物技术学报,2006,14 (4): 585-593 YANG Youcai, ZHOU Qingming, YIN Hanqi. Analysis of genetic diversity in tobacco germplasm by RAPDs and AFLPs[J]. Journal of Agricultural Biotechnology, 2006,14(4): 585-593
[35]杨友才, 周清明, 尹晗琪, 等. 烟草种质资源遗传多样性及亲缘关系的AFLP分析[J]. 中国农业科学, 2006,39(11):2194-2199 YANG Youcai, ZHOU Qingming, YIN Hanqi, et al. Studies on genetic diversity and relationship in tobacco germplasms by AFLP analysis[J]. Scientia Agricultura Sinica, 2006,39(11):2194-2199
[36]杜传印, 刘洪祥, 田纪春. 部分烟草种质亲缘关系的AFLP分析[J]. 作物学报, 2006, 32(10):1592-1596 DU Chuanyin, LIU Hongxiang, TIAN Jichun. Phylogenetic analysis of partial tobacco germplasm resources with AFLP markers[J]. ACTA AGRONOMICA SINICA, 2006,32(10):1592-1596
Genetic diversity analysis of genusNicotianabased on SSR markers in chloroplast genome and mitochondria genome
ZENG Jianmin,CHEN Xuejun,WU Xingfu,JIAO Fangchan,XIAO Bingguang, LI Yongping,TONG Zhijun
Yunnan Academy of Tobacco Agricultural Sciences, Key Laboratory of Tobacco Biotechnological Breeding, National Tobacco Genetic Engineering Research Center, Kunming 650021, China
Based on SSR markers in chloroplast genome and mitochondrial genome of tobacco, genetic diversity of 35 wild tobacco germplasm belonging to 3 subgenus and 9 sections was first analyzed in this study. 20 primer pairs were selected from 764 tobacco organelle genomic SSR markers which were previously developed by lab. A total of 176 polymorphic bands were ampli fi ed in 35 wildNicotianaspecies using 20 SSR primers, and the number of polymorphic bands detected by each SSR primer ranged from 2 to 19, with an average of 8.8 polymorphic bands per marker. Genetic similarity coefficient (GS) among the 35 wildNicotianaspecies ranged from 0.20 to 1.00, with an average of 0.57, which indicated the genetic variation of the 35 wildNicotianaspecies was quite large and the genetic diversity was much abundant inNicotianagenus. Except forSuaveolentessection, the 35 wildNicotianaspecies were clustered into 7 categories by an appropriate genetic similarity coefficient, while the boundary of three subgenus was not obvious. Results indicated that two kinds of tobacco organelle genomic SSR markers in this study are suitable for the evolution, classi fi cation, and genetic analysis among di ff erent tobacco species.
wild tobacco; genetic diversity; simple sequence repeat (SSR) markers
曾建敏,陈学军,吴兴富,等. 基于烟草叶绿体基因组和线粒体基因组SSR标记的烟属植物遗传多样性分析[J]. 中国烟草学报,2016, 22(4)
中国烟草总公司项目(110201402002、110201302005);中国烟草总公司云南省公司项目(2014YN04、2013YN09)
曾建敏(1977—),博士,主要从事烟草育种研究,Email:jmzeng@yntsti.com
童治军(1980—),博士,主要从事烟草育种与生物技术研究,Email: tzj861@163.com
2016-04-07
:ZENG Jianmin,CHEN Xuejun,WU Xingfu,et al. Genetic diversity analysis of genusNicotianabased on SSR markers in chloroplast genome and mitochondria genome [J]. Acta Tabacaria Sinica, 2016,22(4)

