重庆烟区青枯雷尔氏菌生化变种及序列变种的特性研究
刘颖,唐元满,张淑婷,郭兵,谷纪涛,丁伟,2
1 西南大学植物保护学院,重庆市北碚区天生路2号 400715;2 重庆烟草科学研究所,重庆市北碚区天生路2号 400715
生物技术
重庆烟区青枯雷尔氏菌生化变种及序列变种的特性研究
刘颖1,唐元满1,张淑婷1,郭兵1,谷纪涛1,丁伟1,2
1 西南大学植物保护学院,重庆市北碚区天生路2号 400715;2 重庆烟草科学研究所,重庆市北碚区天生路2号 400715
为进一步明确重庆烟区烟草青枯菌的遗传及生化特性,采用生化变种分类标准及演化型复合PCR、系统发育进化分析对重庆10个烟草青枯病发生区县青枯雷尔氏菌的种下分化情况进行了研究。结果表明:46株烟草青枯雷尔氏菌均属于生化变种3和亚洲分支菌株(演化型Ⅰ)。内切葡聚糖酶基因(egl)和DNA修复蛋白基因(mutS)部分序列的系统发育学分析表明:46株供试菌株可聚类到青枯雷尔氏菌演化型Ⅰ的3个序列变种,分别为序列变种15、17和44,其中序列变种17和44为优势菌株。表明重庆烟草青枯雷尔氏菌具有一定的遗传分化和地理区域特性。
烟草青枯菌;生化变种;演化型;系统进化分析;内切葡聚糖酶基因;DNA修复蛋白基因
烟草青枯病是由青枯雷尔氏菌(Ralstonia solanacearum;以下简称青枯菌)引起的一种细菌性病害[1]。青枯菌广泛分布于热带、亚热带及温带地区,能侵染54个科450余种植物[2]。烟草青枯病在我国许多烟区,如福建、湖南、云南、贵州、四川、湖北、重庆等地普遍发生[3-4]。近年来,由于环境、气候、种植结构调整等方面的原因,该病的危害范围有逐渐向北和冷凉地区扩展蔓延之势[4]。
青枯菌因其寄主范围、地理分布、致病性以及生理特性的多样性和复杂性而被公认为是多变的复合种(species complex)[5]。根据传统的分类框架青枯菌可分为不同的生理小种(race)和生化变种(biovar),生理小种是依据不同菌株对不同植物种类的致病性差异来划分的,而生化变种则是根据其利用3种双糖和3种己醇的能力差异来分类的[6-9]。随着分子生物学技术的广泛应用,Fegan和Prior(2005年)共同提出了以演化型分类框架来描述青枯菌种以下的差异,该分类框架依次将青枯菌划分为种(Species)、演化型(Phylotype)、序列变种(Sequevar)以及克隆(Clone)4种不同水平的分类单元,并分别建立了与之相对应的鉴定方法[10]。
青枯菌的种水平鉴定采用以lpxC基因为靶标的引物对759/760;演化型(Phylotype)为亚种水平,分为演化型Ⅰ、Ⅱ、Ⅲ、Ⅳ,可由基于ITS区域的演化型特异性复合PCR(Phylotype speci fi c multiplex PCR,Pmx-PCR)鉴定;每个演化型则又是由多个序列变种(Sequevar)组成的,一个序列变种可作为一组在特定基因范围内序列高度保守的菌株集合体,目前用于界定序列变种的基因包括内切葡聚糖酶基因(Endoglucanase,egl)、Ⅲ型分泌系统调节基因(hrpB)和一个编码错配纠正蛋白的基因(mutS)等[11]。演化型分类框架与传统的生理小种及生化变种分类方法相比可以更精确地反映青枯菌这一复合种的地理起源及种内的遗传多样性。
重庆是我国主要的植烟区,烟草种植分布在武陵山区和大巴山区的黔江、石柱、彭水、酉阳、武隆、南川、涪陵、丰都、万州、巫溪、巫山、奉节等12个区县,除巫山和奉节外,其余10个区县均有青枯病发生。本研究分别采用生化变种和演化型分类框架对重庆烟草青枯菌群体进行种下分类研究,旨在了解重庆烟区烟草青枯菌的遗传及生化特性,解释该地区的青枯菌生态多样性,揭示烟草青枯菌在地理环境、生态条件以及种植结构影响下的多样性分化情况,为有针对性的控制病害提供参考依据。
1 材料与方法
1.1 青枯菌株的分离及DNA的提取
供试烟草青枯病菌株由西南大学天然产物农药研究实验室2013年以及2015年在重庆地区采集分离,分别采自黔江、石柱、彭水、酉阳、武隆、南川、涪陵、丰都、万州、巫溪,共46株,供试菌株信息见表1。应用细菌基因组DNA提取试剂盒(TIANGEN,DP302)提取菌株DNA,-20℃保存备用。
1.2 生化变种鉴定
供试菌株生化变种测定参照Hayward的方法[7]。基本培养基包括:NH4H2PO4, 1.0 g·L-1; KCl, 0.2 g·L-1;MgSO4·7H2O, 0.2 g·L-1;蛋白胨, 1.0 g·L-1;溴百里酚蓝, 0.03 g·L-1;琼脂, 1.5 g·L-1(最终用1M NaOH调节pH值到7.1)。分别将3种双糖(乳糖、麦芽糖和纤维二糖)和3种己醇(甘露醇、山梨醇和甜醇)配成10%的溶液,灭菌(3种双糖用过滤法灭菌,3种己醇经121℃蒸气灭菌)后分别加入基本培养基中,使其终浓度为1%。按实验设计将含不同碳水化合物的培养基垂直分布于96孔板上前6孔,不含碳水化合物的培养基作为对照放于垂直的第7、8孔[12]。取200 μL培养基注入每个孔中,每孔接种青枯菌菌悬液5 μL(菌悬液浓度约2 × 109CFU·mL-1),第8孔作为对照不接种,置30℃的恒温培养箱中培养21 d。根据供试菌株对3种双糖和3种己醇的利用情况,即培养基的颜色变化,确定各菌株的生化变种分类归属。
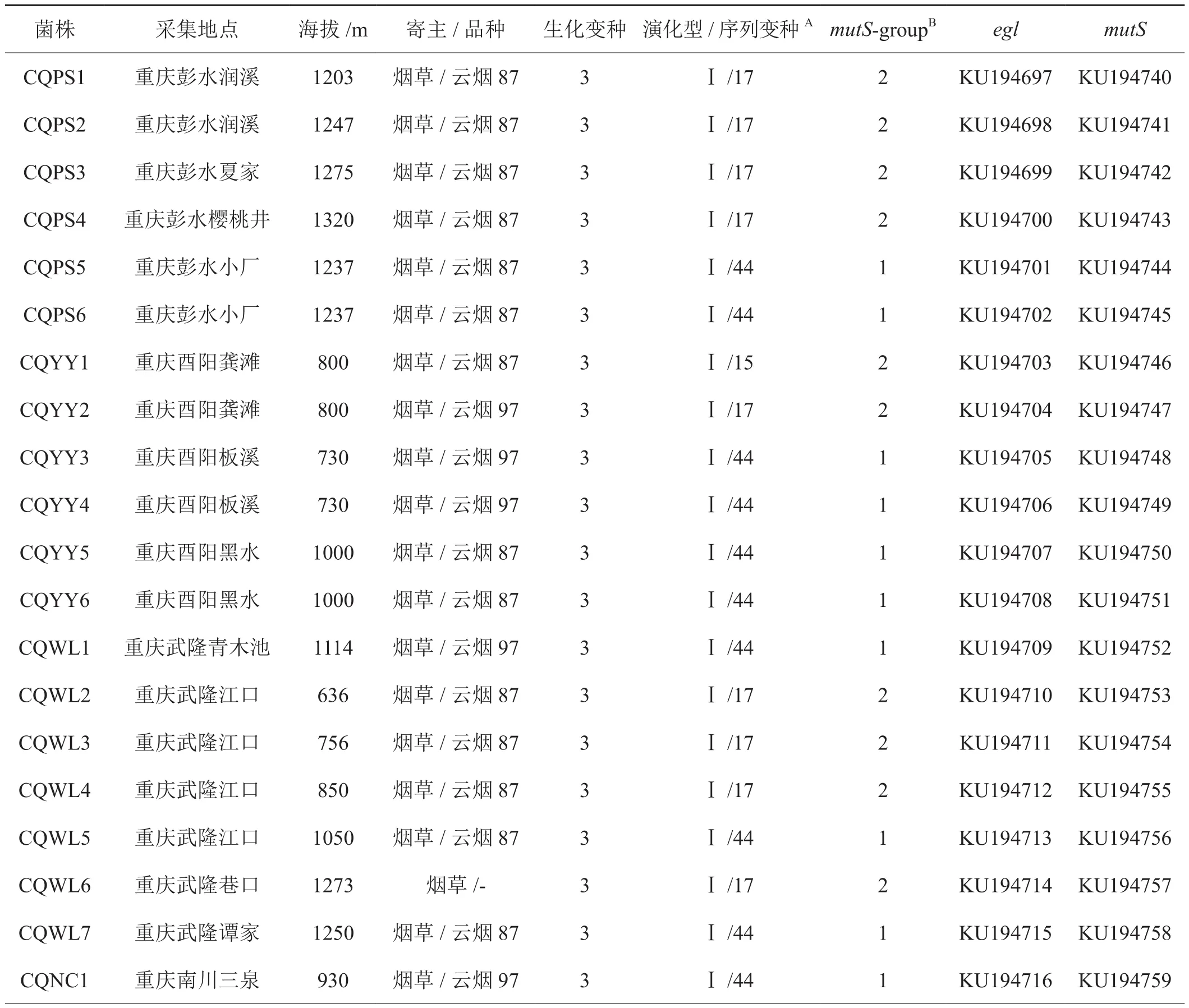
表1 供试烟草菌株相关信息Tab.1 Tobacco Ralstonia solanacearum strains used in the study

续表1
1.3 演化型鉴定
根据演化型分类框架所设计的复合PCR引物对青枯菌菌株进行了演化型鉴定[9]。PCR扩增采用25 μL反应体系:2×Taq PCR MasterMix(TIANGEN,KT201)、 引 物 Nmult:21:1F,Nmult:21:2F和 Nmult:22:InF各 6 pmoles,Nmult:23:AF和Nmult:22:RR 各 18 pmoles,759/760各 4 pmoles, 青枯菌DNA,ddH2O。反应程序为96℃预变性5 min;94℃变性15 s,59℃退火30 s和72℃延伸30 s,30个循环;最后72℃延伸10 min ,4℃保存。取5 μL PCR产物于1%的琼脂糖凝胶中电泳,电压4 V/cm,通过Biorad凝胶成像仪观察结果。实验所用引物均由华大基因科技股份有限公司合成,引物序列见表2。

表2 用于遗传多样性分析的引物序列Tab.2 The primers used for genetic diversity analysis
1.4 部分egl基因扩增
用引物Endo-F和Endo-R(表2)扩增内源葡聚糖酶基因(egl)内部的750 bp片段。egl基因扩增PCR反应体系(25 μl):2×Taq PCR MasterMix(TIANGEN, KT201),青枯菌DNA 50 ng,引物Endo-F和Endo-R各10 pmoles,ddH2O补足。egl扩增的PCR反应程序为96℃预变性9 min;95℃变性1min,64℃退火1 min,72℃延伸2 min,30个循环;最后72℃延伸10 min ,4℃保存。将PCR产物送华大基因科技股份有限公司测序。
1.5 部分mutS基因扩增
用引物mutS-RsF.1570和mutS-RsR.1926(表2)扩增DNA蛋白修复基因mutS,其扩增PCR反应体系(25 μL):2×Taq PCR MasterMix (TIANGEN,KT201),青枯菌DNA 50 ng,引物mutS-RsF.1570和 mutS-RsR.1926各10 pmoles,ddH2O补 足。PCR反应程序为96℃预变性5 min;94℃变性1 min,66℃退火1 min,72℃延伸90 s,35个循环;最后72℃延伸5 min,4℃保存。将PCR产物送华大基因科技股份有限公司测序。
1.6 序列分析
将所测得的供试青枯菌株egl和mutS基因序列与GenBank上已登录的青枯菌核酸序列进行比对(参考序列信息见表3),构建系统发育树。698 bp的egl基因和682 bp的mutS基因的测序结果用Clustal x软件比对后,再用MEGA 5软件进行系统发育分析,采用Jukes and Cantor模型邻接法(neighbor-joining,NJ)构建系统发育树,1000次重复取样作出Bootstrap树状图。序列之间的遗传距离用Kimura-2-parameter方法计算。并将所有的序列提交到GenBank数据库中。

表3 参考序列信息Tab.3 Reference strains used for sequence analysis

续表3
2 结果与分析
2.1 生化变种鉴定结果
测定了46株供试菌株对乳糖、麦芽糖、纤维二糖和甘露醇、山梨醇、甜醇的利用能力(图1)。结果表明,所有的供试菌株均能利用3种双糖和3种己醇,参照生化变种划分标准,重庆地区的烟草青枯菌均属于生化变种3(表1)。

图1 部分菌株生化变种结果展示Fig.1 Results of biovar test of R. solanacearum
2.2 Pmx-PCR的演化型鉴定结果
供试菌株演化型特异性复合PCR结果表明,所有菌株均可同时扩增得到144 bp和281 bp的2条特异性片段,其中281 bp的片段为青枯菌种特异性扩增条带,144 bp的片段为演化型Ⅰ的特异性扩增条带。从而在种和演化型分类单元水平上,揭示出供试菌株属于青枯菌演化型Ⅰ型,即亚洲分支菌株。
2.3 egl基因分析
对供试菌株的egl基因部分序列(698 bp)进行系统发育分析,并从GenBank 上选取32个菌株的egl基因的序列(表3)来确定供试菌株在系统发育结构中的位置。由系统发育分析知,青枯菌被分为4个分支,即4个演化型分支,而我国烟草青枯菌均被聚类到演化型Ⅰ型分支中(图2),这与Pmx-PCR的鉴定结果一致。
供试46株菌株被聚类在演化型Ⅰ型之下的3个分支中,它们分别与序列变种15、17和44对应(图2)。其中,序列变种15的参考菌株为中国番茄菌株PSS358,仅包含了2株菌,分别是酉阳的CQYY1和万州的CQWZ2。序列变种17以分离自中国花生的P11为参考菌株,包含18株供试菌,分别为彭水的CQPS1、CQPS2、CQPS3、CQPS4,酉阳的 CQYY2, 武 隆 的 CQWL2、CQWL3、CQWL4、CQWL6,丰都的CQFD1、CQFD2,黔江的CQQJ4、CQQJ9、CQQJ10、CQQJ11, 石 柱 的 CQSZ-3、CQSZ-4,以及万州的CQWZ1。而序列变种44包含了2个小分支,1个分支以中国烟草青枯菌Tb28和Tb43为参考菌株,共9株菌,其中酉阳2株(CQYY5、CQYY6)、黔江 7株(CQQJ1、CQQJ2、CQQJ3、CQQJ6、CQQJ7、CQQJ8、CQQJ12);1个分支以中国木槿菌株Bd11为参考菌株,共有17株,其中彭水2株(CQPS5、CQPS6)、酉阳2株(CQYY3、CQYY4)、 武 隆 3株(CQWL1、CQWL5、CQWL7)、 南 川 2株(CQNC2、CQNC2)、 涪陵 2株(CQFL1、CQFL2)、黔江3株(CQQJ5、CQQJ13、CQQJ14)、石柱2株(CQSZ-1、CQSZ-2)以及巫溪1株(CQWX-1)。
2.4 mutS基因分析
对供试菌株的mutS基因部分序列(682 bp)进行系统发育分析,并从GenBank 上选取27个菌株的mutS基因的序列(表2)作为参考序列进行比较。由系统发育分析可知,所有的供试菌株可聚到2个分支中,如图3所示,第一分支与egl基因的系统发育图中的序列变种44分支对应。第二分支则与egl基因的系统发育图的序列变种15和17对应。

图2 基于egl基因部分序列的系统发育分析Fig.2 Phylogenetic neighbor-joining tree based on partial sequences of the egl gene

图3 基于mutS基因部分序列的系统发育分析Fig.3 Phylogenetic neighbor-joining tree based on partial sequences of the mutS gene
3 结论与讨论
青枯菌受不同的环境、寄主植物和各种农艺操作的影响,自身不断进化,形成了一个复合种[14]。Fegan和Prior所提出的演化型分类框架表明青枯菌的种下分化与地理起源密切相关[10]。在分析美国东南部的青枯菌多样性时,分离自北卡罗来纳州和佐治亚州的烟草青枯菌均属于生化变种1和演化型Ⅱ(美洲分支)[15]。Wang等在研究中指出中国烟草青枯菌属于生化变种3[16]。对我国青枯菌遗传多样性分析中9株分离自烟草的菌属于演化型Ⅰ[17]。福建烟草青枯菌被鉴定出均属于演化型Ⅰ[18]。总体来看,我国的烟草青枯菌属于演化型Ⅰ(亚洲分支),与美国的烟草青枯菌(演化型Ⅱ即美洲分支)属于不同的地理起源。Wicker等利用多位点序列分析(multilocus sequence analysis, MLSA)追溯了世界范围内的青枯菌的进化历史,发现该世系中存在21个重组事件,而演化型Ⅰ是多态性持续发展的分支之一[19]。重庆地区种下分化情况也属于生化变种3及演化型Ⅰ,并在序列变种上存在分化。
重庆12个烟草种植区县中,除巫溪、巫山和奉节地处大巴山区外,其余9个区县均处于武陵山区。其中,巫山、奉节目前尚未发现青枯病,巫溪县有零星发生,而另外的9个区县均发生青枯病,且较严重。巫山和奉节所种植的烟草品种与重庆其他植烟地并无差异。同时,本研究的结果未发现青枯菌序列变种分化与烟草品种有直接关系,由此推测,烟草品种可能不是青枯菌种下分化的直接原因。而武陵山区与大巴山区的气候、土壤条件等地理区域因子存在明显差异。鄂西烟区也大多属于武陵山区。刘海龙等对湖北地区烟草青枯菌系统发育分析的结果表明,湖北烟草青枯菌菌株属于青枯菌亚洲分支(演化型Ⅰ)的3个序列变种,分别为序列变种15、17和44,其优势菌株也为序列变种17和44[20]。本研究结果与这个结果基本一致。福建和贵州的烟草青枯菌系统发育分析结果证实,福建的烟草青枯菌菌株包含序列变种15、17、34和44,而贵州的供试菌株仅被聚到序列变种17分支中[21]。比起重庆、湖北及靠近武陵山区的贵州,距离较远的福建省所分离的烟草青枯菌菌株除序列变种15、17和44外还存在序列变种34。而其他地区是否存在异同还需进一步验证,因此,对于全国烟草青枯菌分化情况的全面评估是很有必要的。青枯菌与寄主协同进化过程中,地域分布是否是其种内遗传分化的主要驱动力,尚有待于大尺度范围内进一步深入研究。
掌握各地烟草青枯菌的具体分化情况,对今后进一步深入研究青枯菌的系统演化及防治措施具有重要意义。本研究分析了重庆地区的烟草青枯菌生化变种、演化型及系统发育情况,证实重庆地区46株供试菌株均属于生化变种3和演化型Ⅰ;系统发育进化分析结果表明重庆烟草青枯菌可聚类到3个序列变种(序列变种15、17和44)中。为进一步研究影响青枯菌分化的关键因子及其遗传多样性机制提供了相应的依据。
[1]Yabuuchi E, Kosako Y, Yano I, et al. Transfer of twoBurkholderiaand anAlcaligenesspecies toRalstoniagen. nov.:proposal ofRalstoniapickettii(Ralston, Palleroni and Doudoro ff 1973) comb. nov.,Ralstonia solanacearum(Smith 1896)comb. nov. andRalstoniaeutropha(Davis 1969) comb. nov[J].Microbiology Immunology, 1995, 39(11): 897-904.
[2]Wicker E, Grassart L, Coranson-Beaudu R, et al.Ralstonia solanacearumstrains from Martinique (French west indies)exhibiting a new pathogenic potential[J]. Applied and Environmental Microbiology, 2007, 73(21): 6790-6801.
[3]朱朝贤, 王彦亭, 王智发. 中国烟草病害[M]. 北京: 中国农业出版社, 2002: 152-153.ZHU Caoxian, WANG Yanting, WANG Zhifa. Tobacco Diseases of China[M]. Beijing: China Agriculture Press, 2002: 152-153.
[4]孔凡玉. 烟草青枯病的综合防治[J]. 烟草科技, 2003(4): 42-43.KONG Fanyu. Integrated Control of Tobacco Bacterial Wilt Disease[J]. Tobacco science & Technology, 2003(4): 42-43.
[5]Hayward A C. Biology and epidemiology of bacterial wilt caused byPseudomonasSolanacearum[J]. Annual Review of Phytopathology, 1991, 29(1): 65-87.
[6]Buddenhagen I W, Sequeira L, Kelman A. Designation of races inPseudomonasSolanacearum[J]. Phytopathology, 1962, 52(8):726.
[7]Hayward A C. Characteristics ofPseudomonassolanacearum[J].Journal of Applied Microbiology, 1964, 27(2): 265-277.
[8]He L Y, Sequeira L, Kelman A. Characteristics of strains ofPseudomonasSolanacearumfrom China[J]. Plant Disease, 1983,67(12): 1357-1361.
[9]Pegg K G, Moffett M L. Host range of the ginger strain ofPseudomonassolanacearumin Queensland[J]. Animal Production Science, 1971, 11(53): 696-698.
[10]Fegan M, Prior P. How complex is the“Ralstonia solanacearumspecies complex”?[M]// Allen C, Prior P, Hayward A C.Bacterial wilt disease and theRalstonia solanacearumspecies complex. St. Paul, MN: American Phytopathological Society Press, 2005: 449-461.
[11]Prior P, Fegan M. Recent developments in the phylogeny and classi fi cation ofRalstonia solanacearum[J]. Acta horticulturae,2005, 695, 127-136.
[12]Sagar V, Jeevalatha A, Mian S, et al. Potato bacterial wilt in India caused by strains of phylotype I, II and IV ofRalstonia solanacearum[J]. European Journal of Plant Pathology, 2014,138(1): 51-65.
[13]Poussier S, Prior P, Luisetti J, et al. Partial sequencing of the hrpB and endoglucanase genes con fi rms and expands the known diversity within theRalstonia solanacearumspecies complex[J].Systematic and applied microbiology, 2000, 23(4): 479-486.
[14]Genin S, Denny T P. Pathogenomics of theRalstonia solanacearumspecies complex[J]. Annual Review of Phytopathology, 2012, 50: 67-89.
[15]Hong, J C, Norman, D J, Reed, D L, et al. Diversity amongRalstonia solanacearumstrains isolated from the southeastern United States[J]. Phytopathology, 2012, 102(10): 924-936.
[16]Wang Han-cheng, Huang Yan-fei, Wang Jin, et al. Phenotypic Fingerprints ofRalstonia solanacearumBiovar 3 Strains from Tobacco and Tomato in China Assessed by Phenotype MicroArray Analysis[J]. Plant Pathology Journal, 2015, 14(1):38.
[17]Xu J, Pan Z C, Prior P, et al. Genetic diversity ofRalstonia solanacearumstrains from China[J]. European Journal of Plant Pathology, 2009, 125(4): 641-653.
[18]徐进, 顾钢, 潘哲超, 等. 福建烟草青枯菌演化型及生化变种鉴定研究[J]. 中国烟草学报, 2010, 16(6): 66.XU Jin, GU Gang, PAN Zhechao, et al. Phylotype identi fi cation and biovar determination ofRalstonia solanacearumstrains isolated from Fujian province[J]. Acta Tobacaria Sinica, 2010,16(6): 66.
[19]Wicker E, Lefeuvre P, de Cambiaire J C, et al. Contrasting recombination patterns and demographic histories of the plant pathogenRalstonia solanacearuminferred from MLSA[J]. The ISME journal, 2012, 6(5): 961-974.
[20]刘海龙, 黎妍妍, 郑露, 李锡宏, 黄俊斌. 湖北地区烟草青枯菌系统发育分析[J]. 中国烟草科学, 2015, 36(2): 81-86.LIU Hailong, LI Yanyan, ZHENG Lu, LI Xihong, HUANG Junbin. Phylogenetic Analysis of TobaccoRalstonia solanacearumstrains from Hubei Province[J]. Chinese Tobacco Science, 2015, 36(2): 6.
[21]潘哲超, 徐进, 顾钢, 等. 福建及贵州等地烟草青枯菌系统发育分析[J]. 植物保护, 2012, 38(1): 18-23.PAN Zhechao, XU Jin, GU Gang, et al. Phylogeny of tobaccoRalstonia solanacearumstrains from Fujian and Guizhou provinces[J]. Plant Protection, 2012, 38(1): 18-23.
Biovar and sequevar ofRalstonia solanacearumin tobacco growing areas in Chongqing
LIU Ying1, TANG Yuanman1, ZHANG Shuting1, GUO Bing1, GU Jitao1, DING Wei1,2
1 College of Plant Protection, Southwest University, Chongqing 400715, China;2 Chongqing Tobacco Research Institute, Chongqing 400715, China
46 isolates were assessed to explore the diversity of tobaccoRalstonia solanacearumin Chongqing by using biovar, phylotype speci fi c multiplex PCR, and phylogenetic relationships (eglandmutS). Results showed that all tested isolates belonged to biovar 3 and phylotype I. The phylogenetic neighbor-joining tree based on partial sequences ofeglandmutSgenes indicated that the tested isolates were clustered into phylotype I, and were further divided into 3 sequevars, namely sequevars 15, 17, and 44. Sequevar 17 and 44 were predominant.R.solanacearumstrains on tobacco showed genetic differentiation to some extent, which provided basis for studying relationship between classi fi cation and geographic areas.
tobacco;Ralstoniasolanacearum; biovar; phylogenetic analysis;egl;mutS
刘颖,唐元满,张淑婷,等. 重庆烟区青枯雷尔氏菌生化变种及序列变种的特性研究[J]. 中国烟草学报,2016,22(4)
中国烟草总公司重点项目(110201202002);中国烟草总公司重庆市公司重点项目(NY20130501070005)
刘颖(1991—),硕士研究生,研究方向:天然产物农药及植物与病原菌互作, Email: lyying1201@163.com
丁伟(1966—),博士,教授,研究方向:天然产物农药和烟草有害生物系统控制,Email: dwing818@163.com
2015-12-11
:LIU Ying, TANG Yuanman, ZHANG Shuting, et al. Biovar and sequevar ofRalstoniasolanacearumin tobacco growing areas in Chongqing [J]. Acta Tabacaria Sinica, 2016,22(4)

