新型GH5家族β-甘露聚糖酶的基因挖掘及表达鉴定
唐存多,史红玲,蔚晓华,张 梅,唐青海,岳 超,姚伦广,夏 敏,阚云超,*(.南阳师范学院 昆虫生物反应器河南省工程实验室,河南 南阳 4706;.南阳师范学院生命科学与技术学院,河南 南阳 4706;.蓬莱市市场监督管理局,山东 蓬莱 65600)
新型GH5家族β-甘露聚糖酶的基因挖掘及表达鉴定
唐存多1,史红玲1,蔚晓华2,张 梅3,唐青海1,岳 超1,姚伦广1,夏 敏2,阚云超1,*
(1.南阳师范学院 昆虫生物反应器河南省工程实验室,河南 南阳 473061;2.南阳师范学院生命科学与技术学院,河南 南阳 473061;3.蓬莱市市场监督管理局,山东 蓬莱 265600)
为了获得性能优良的新型β-甘露聚糖酶,本研究采用基因挖掘技术从米曲霉(Aspergillus oryzae)RIB40基因组中挖掘到了2 个假定的新型 GH5家族β-甘露聚糖酶基因,分别命名为Aoman5A和Aoman5B。对这两条序列做了相关的生物信息学分析,同时借助pPIC9KM质粒将两个酶的成熟肽编码基因在Pichia pastoris GS115中实现了表达,并对表达产物进行了纯化和鉴定。生物信息学分析的结果表明AoMan5A含有20 个氨基酸残基的信号肽,而AoMan5B含有21 个氨基酸残基的信号肽和12 个氨基酸残基的前导肽;序列比对的结果显示两个酶与目前已报道的序列最高的同源性分别为68%和79%,且AoMan5A的N端还携带有一个1家族的CBM;三维结构预测的结果显示,两者均符合(α/β)8的TIM-桶状结构。表达鉴定的结果表明,在同样表达条件下,reAoMan5A和reAoMan5B上清液的酶活力分别为2.9、12.5 IU/mL;纯化后它们的酶比活力分别为8.3、104.2 IU/mg,前者的最适温度为35 ℃,而后者的最适温度为50 ℃,pH值特性的测定结果表明这两种酶均为酸性酶。硫酸-苯酚法测得reAoMan5A和reAoMan5B的糖含量分别为 25.4%和12.6%,表明这两种重组酶均经过了糖基化修饰。本研究获得了两种新型的β-甘露聚糖酶,比较分析了它们的序列特征,并实现了它们的异源表达,为β-甘露聚糖酶的进一步研究及应用奠定了坚实的基础,也为其他新型酶的基因挖掘提供了可资借鉴的经验。
β-甘露聚糖酶;基因挖掘;生物信息学分析;米曲霉;蛋白表达
β-甘露聚糖酶是一类能够水解甘露聚糖的酶的总称,近年来研究较多的主要是β-1,4-内切甘露聚糖酶[1-2]。基于序列同源性及疏水簇分析,β-甘露聚糖酶可以被归为糖苷水解酶(glycoside hydrolase,GH)第5、26和113家族,而报道最多、研究最广的主要是GH5家族的β-甘露聚糖酶[3-4]。β-1,4-内切甘露聚糖酶在β-1,4-内切葡聚糖酶的协同作用下,可以高效地水解魔芋粉制备低聚甘露糖。迄今为止,尽管已有许多关于生物催化法制备低聚甘露糖的研究,但它的生产成本仍然居高不下。这主要归因于生产所需关键酶的催化活性较低、生产和使用成本较高以及在高温环境下的稳定性较差等[5]。如何获得催化活性高、热稳定性好的优良β-甘露聚糖酶,已成为企业和科研人员当前所面临的巨大挑战,也是降低生物催化法制备低聚甘露糖的生产成本所必须突破的瓶颈[6]。自然界中蕴藏着丰富的未被发掘的新型酶资源,如何去发掘这些未知的酶类并加以适当的改造以满足工业化生产的需求,已渐渐吸引了广大科研工作者的关注[7]。
迄今已有2 000多个微生物基因组信息被公开报道,而且这些数据仍在呈指数增长[8],这些日益丰富的基因组信息为新酶的发掘提供了丰富的资源[9]。“基因挖掘(genome mining)”这一术语已被用于多种领域,主要是指开发基因组信息以发现一些新的过程、靶点和产物[10-11]。基因挖掘技术可以实现从基因组数据库到真实酶数据库的跨越,进一步丰富了可被利用或改造的酶资源[7,12]。近年来,已有许多利用基因挖掘技术发现新酶的报道。Zhu Dunming等[13]借助基因挖掘技术,先搜索到98 个预测的腈水解酶序列,然后结合保守序列比对和氨基酸长度控制等理性的设计,最后成功获得了来源于慢生大豆根瘤菌(Bradyrhizobium japonicum)USDA110的扁桃腈水解酶。Barriuso等[14]利用保守模块搜索、进化树构建及序列同源比对生物信息学分析的手段,从联合基因组研究所(Joint Genome Institute,JGI)公布的128 个真菌基因组中寻找到了6 个具有固醇酯酶或脂肪酶活性的假定蛋白。本研究试图借助基因挖掘技术从已报道的基因组信息中挖掘新型、假定的GH5家族β-甘露聚糖酶基因,并对获得的序列进行相应的生物信息学分析及表达鉴定,为后续的理性改造、高效表达及其应用奠定坚实的基础。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
A. oryzae RIB40菌株购自中国工业微生物菌种保藏中心;E. coli JM109菌株昆虫生物反应器河南省工程实验室保藏;pMD19-T载体购自大连宝生物有限公司;pPIC9KM质粒和Pichia pastoris GS115菌株由江南大学医学院邬敏辰教授馈赠。
1.1.2 试剂
UNIQ-10柱式Trizol总RNA抽提试剂盒 生工生物工程(上海)股份有限公司;DNase I和RevertAid First Strand cDNA Synthesis Kit 美国赛默飞公司;Multi-Copy Pichia Expression Kit Invitrogen公司;Premix Ex Taq Hot Start Version、GXL高保真DNA聚合酶和DNA连接试剂盒 宝生物工程(大连)有限公司;限制性内切酶Xho I、EcoR I和Sal I New England Biolabs公司;角豆胶 美国Sigma公司;质粒小量提取和PCR产物纯化试剂盒 美国Axygen公司;其他试剂均为国产或进口分析纯。
1.1.3 常用培养基
液体种子培养基:魔芋粉5 g/L、玉米粉10 g/L、豆饼粉30 g/L、硫酸铵5 g/L、磷酸二氢钾3 g/L、氯化钙1 g/L、硫酸镁1 g/L,自然pH值,121 ℃灭菌20 min。
液体诱导培养基:由于需要收集菌体提RNA,所以培养基里面尽量不能出现固形物,先将4 g麸皮和6 g豆饼粉加200 mL水煮30 min,纱布过滤后将汁液定容至200 mL;5 g/L魔芋粉、体积分数50%麸皮汁、5 g/L硫酸铵、3 g/L磷酸二氢钾、1 g/L氯化钙、1 g/L硫酸镁,自然pH值(加水时预留出10%的接种量),121 ℃灭菌20 min。LB、YPD、MD、BMGY和BMMY培养基的配制参见Multi-Copy Pichia Expression Kit操作手册。
1.1.4 常用网站及软件
DNAMAN 6.0:简单的序列比对及分析;BioEdit:测序彩图的分析;Oligo 7:引物设计;Geneious 4.7.5:基因或蛋白质序列的存储与管理;Modeller 9.9:蛋白质3-D结构的模拟;PyMOL和Discovery Studio 3.0 Client:蛋白质3-D结构的阅读与分析;CBS Prediction Servers(http://www.cbs.dtu.dk/ services/):信号肽、前导肽及糖基化位点的预测;美国国家生物信息中心(National Center of Biotechnology Information,NCBI)(http://www. ncbi.nlm. nih.gov/)和CAZy(http://www.cazy.org/)数据库:蛋白质、基因序列及蛋白质3-D结构的搜索。
1.2 方法
1.2.1 新型β-甘露聚糖酶的基因挖掘
以研究比较透彻、催化活性较高的GH5家族β-甘露聚糖酶AoMan5A的蛋白序列为查询序列进行BLAST分析,从查询的结果中找出一系列基因组信息来源的且未经表达鉴定、假定的GH5家族β-甘露聚糖酶基因。对这些序列进行开放读码框的预测,并进行序列比对分析,以期在某一微生物基因组中找出2 个目的β-甘露聚糖酶序列,其中一个含有碳水化合物结合结构域(carbohydratebinding module,CBM),另一个不含CBM。本研究最终选定了2 个A. oryzae RIB40基因组中未经表达鉴定的GH5家族的β-甘露聚糖酶为研究对象,2 个基因分别命名为Aoman5A和Aoman5B,对应的酶命名为AoMan5A和AoMan5B。
1.2.2 新型β-甘露聚糖酶的基因克隆
基于上述基因挖掘的结果,分别设计两对特异性引物,引物序列如表1所示。引物委托生工生物工程(上海)股份有限公司合成。采用UNIQ-10柱式Trizol总RNA抽提试剂盒提取A. oryzae RIB40的总RNA,总RNA经DNase I酶处理后用RevertAid First Strand cDNA Synthesis Kit进行cDNA第一条链的合成,然后分别用上述的特异性引物进行目的基因的反转录聚合酶链式反应(reverse transcription polymerase chain reaction,RT-PCR)扩增。扩增的目的片段经电泳纯化回收后,连接到pMD19-T载体上,并转化到宿主菌JM109。经蓝白斑筛选及菌落聚合酶链式反应验证后,阳性克隆子送往生工生物工程(上海)股份有限公司进行测序。克隆及测序的步骤均采用高保真的DNA聚合酶做了3 次平行实验。

表1 新型GHH55 β-甘露聚糖酶基因克隆及表达所需的引物Table 1 Sequences of the primers used for cloning and expression of novel GGHH55 β-mannannaasseess
1.2.3 新型β-甘露聚糖酶序列的生物信息学分析
将上述获得基因序列先利用DNAMAN 6.0软件进行翻译,获得氨基酸序列。然后参照Tang Cunduo等[15]的方法分别对获得的序列进行理论的理化性质、信号肽、前导肽、糖基化位点及三维结构等的预测和分析,为后续的异源表达及酶学特性分析奠定理论基础。
1.2.4 β-甘露聚糖酶基因在毕赤酵母中的表达
基于上述信号肽和成熟肽预测的结果,分别设计表达AoMan5A和AoMan5B成熟肽的引物,引物序列如表1所示,并委托生工生物工程(上海)股份有限公司进行合成。重组毕赤酵母的构建、筛选、表达及产物的纯化参见唐存多等[16]所发表的文献,重组表达质粒的线性化用Sal I进行。
1.2.5 β-甘露聚糖酶酶活性及蛋白分析
采用改进的二硝基水杨酸(dinitrosalicylic acid,DNS)法测定样品β-甘露聚糖酶活性[2]。2.4 mL 0.5 g/100 mL角豆胶溶液(pH 3.6)加入0.1 mL适当稀释的酶液,40 ℃条件下反应10 min,加入2.5 mL DNS试剂,沸水浴中显色7 min,测定OD540nm值。在上述反应条件下,每分钟产生1 μmol还原糖(以甘露糖计)所需的酶量定义为1 个酶活力单位(IU)。采用Bradford法[17]测定蛋白的浓度,以牛血清白蛋白为参照标准。用12.5%的分离胶进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分析[18],并用Quantity One软件计算目的蛋白的表观分子质量。采用硫酸-苯酚法测定纯化后重组酶的糖含量。
1.2.6 温度对β-甘露聚糖酶活性的影响
分别在30~55 ℃条件下,按1.2.5节中的方法测定酶活性,以最高酶活力为100%计算相对酶活力,Topt表示酶的最适温度。将酶分别在25~55 ℃条件下保温1 h后,再在各自的Topt条件下测定其酶活力,以未处理的酶活力为100%计算残留酶活力。
1.2.7 pH值对β-甘露聚糖酶活性的影响
分别在pH值为2.5~7.0的条件下,按1.2.5节中的方法在各自的Topt条件下测定其酶活力,以最高酶活力为100%计算相对酶活力。将酶分别与pH 2.5~7.0的缓冲液按体积比1∶1混合,在4 ℃条件下保温1 h后,再在各自的Topt条件下测定其酶活力,以未处理的酶活力为100%计算残留酶活力。
2 结果与分析
2.1 β-甘露聚糖酶的基因克隆

图1 1 β-甘露聚糖酶基因的RT-PCR扩增PCRFig. 1 RT-PCR amplifi cation of β-mannanase genes
根据1.2.2节中的方法,分别以AoMan5A-F和AoMan5A-R为引物RT-PCR扩增出编码AoMan5A的cDNA片段,以AoMan5B-F和AoMan5B-R为引物RT-PCR扩增出编码AoMan5B的cDNA片段,PCR产物经1%琼脂糖凝胶电泳检测的结果如图1所示。泳道1为AoMan5A产物的电泳图谱,在1 400 bp左右的位置出现了明显的特异性条带,与预期的大小一致;而泳道2为AoMan5B产物的电泳图谱,在1 200 bp左右的位置出现了明显的特异性条带,同样也与预期的大小一致。由于PCR产物均较纯,为了保证回收产物的浓度,因此直接进行了PCR产物纯化回收。回收的产物连接至pMD19-T载体,经蓝白斑筛选和菌落PCR鉴定后送生工生物工程(上海)股份有限公司进行测序。经生工生物工程(上海)股份有限公司测出的序列与基因组中推测的编码序列基本一致,其中Aoman5A的CDS序列为1 392 个碱基,编码463 个氨基酸残基,与基因组数据库中的序列完全一致;而Aoman5B的CDS序列为1 137 个碱基,编码378 个氨基酸残基,与基因组数据库登录的序列存在2 个碱基的差异,并呈现出1 个氨基酸的差异。
2.2 β-甘露聚糖酶序列的生物信息学分析
2.2.1 信号肽及糖基化位点的预测及分析
利用SignalP 4.1服务器[19]对推测出的AoMan5A氨基酸序列进行信号肽预测。结果显示AoMan5A含有一段20 个氨基酸残基的信号肽。利用ProP 1.0 Server预测AoMan5A的前导肽序列,结果显示AoMan5A中无前导肽序列。利用NetNGlyc 1.0 Server对AoMan5A进行潜在的N-连接的糖基化位点预测,结果表明AoMan5A中存在1 个潜在的N-连接的糖基化位点Asn87-Lys88-Ser89。利用NetOGlyc 4.0 Server对AoMan5A进行潜在的O-连接的糖基化位点预测,结果表明AoMan5A中存在26 个潜在的O-连接的糖基化位点。采用同样的方法对AoMan5B的序列也进行信号肽、前导肽及糖基化位点的预测分析。信号肽预测的结果显示AoMan5B中有一段21 个氨基酸残基的信号肽。此外,前导肽预测的结果也表明在AoMan5B中还存在一段12 个氨基酸残基的前导肽。糖基化位点预测的结果显示,AoMan5B中存在1 个潜在的N连接的糖基化位点Asn328-Thr329-Thr330和1 个潜在的O-连接的糖基化位点。
2.2.2 序列的同源比对及分析
利用DNAMAN 6.0软件分别将Aoman5A和Aoman5B的碱基序列翻译成了氨基酸序列,并分别命名为AoMan5A和AoMan5B。将AoMan5A的成熟肽序列进行BLAST分析,发现与其同源性较高的为1 株Aspergillus fumigatus Af293来源的GH5家族β-甘露聚糖酶序列,其同源性为68%;而与AoMan5B的同源性最高的为Aspergillus terreus NIH2624来源的GH5家族β-甘露聚糖酶序列,其同源性为79%。将这2 个序列与本研究得比较透彻的AoMan5A的序列一起进行多序列比对,可以找出它们7 个最为保守的氨基酸残基,分别为R53、N167、E168、H241、Y243、E275和D312,其中E168和E275为2 个假定的Glu催化残基。此外,在AoMan5A的N端存在1 个明显的CBM和连接肽。对CBM作进一步的分析,发现它与CBM1家族的序列同源性较高,与Lu Haiqiang等[20]报道的结果一致。
2.2.3 理论理化性质的预测及分析
利用Protparam服务器分别对AoMan5A和AoMan5B成熟肽的理化性质进行了预测,结果显示它们的理论分子质量分别为48 078.2 D和38 030.8 D,理论等电点pI值分别为5.05和4.64,分子式分别为C2135H3203N559O679S16和C1711H2507N437O536S8;蛋白的不稳定指数分别为25和14.96,均是较稳定的蛋白;另外,它们的脂溶性指数分别为61.26和68.43,总的平均疏水指数分别为-0.454和-0.354,均为亲水蛋白。
2.2.4 高级结构的预测及分析
利用Predict Protein服务器分别预测了AoMan5A和AoMan5B成熟肽的二级结构,结果显示两者均由8 个α-螺旋、8 个β-折叠片和一些无规卷曲组成。以PDB号为3WH9、1QNO和3WFL的3 个3-D结构为模板,利用Modeller 9.9软件[21]分别对AoMan5A和AoMan5B进行多模板的同源建模,并将获得的2个3-D结构用Pymol软件进行比较分析,结果如图2所示。AoMan5A和AoMan5B的氨基酸序列尽管相差较大,但除去AoMan5A中的CBM序列以后,两者的3-D结构非常相似,均为典型的(α/β)8-TIM桶状结构,两者推测的催化残基在空间的位置也能重叠。此外,3-D结构的表面模型显示,2个推测的催化残基恰好位于凹槽的内侧,符合典型的内切酶的特征[22]。这些特征也进一步证明了获得的2个新型β-甘露聚糖酶为GH5家族和GH clan-A超家族的成员[23]。
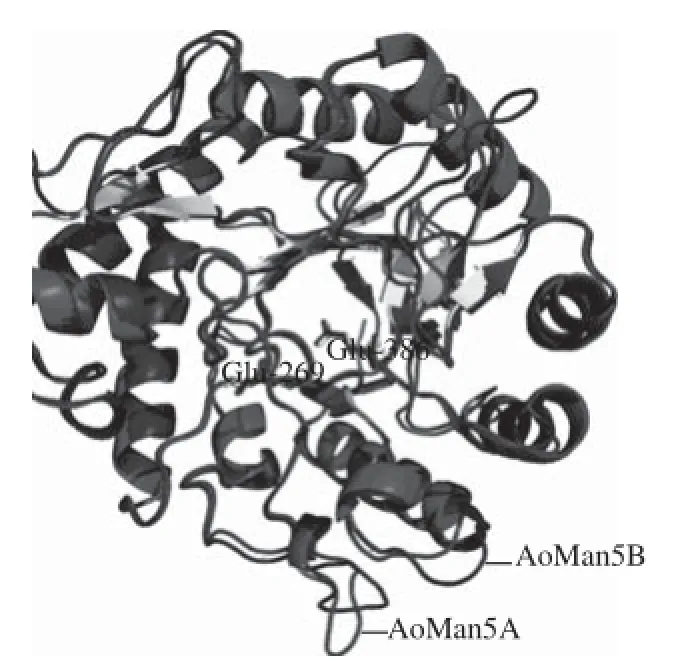
图2 AoMan5A与AoMan5B三维结构的比对Fig. 2 Alignment of 3-D structure between AoMan5A and AoMan5B
2.3 β-甘露聚糖酶基因在毕赤酵母中的表达
按照1.2.4节中的方法将AoMan5A和AoMan5B成熟肽的编码基因整合入了P. pastoris GS115的基因组中,构建出了含有目的基因的重组毕赤酵母GS115/Aoman5A和GS115/Aoman5B。利用5’-AOX和3’-AOX通用引物对GS115、GS115/Aoman5A和GS115/Aoman5B进行菌落PCR鉴定的结果如图3所示。泳道2和3为GS115/ Aoman5A菌落PCR产物的图谱,除了有2 100 bp左右醇氧化酶基因片段外,分别还有一条1 700 bp左右的条带;而泳道4和5为GS115/Aoman5B菌落PCR产物的图谱,除了有2 100 bp左右醇氧化酶基因片段外,分别还有一条1 600 bp左右的条带;这也表明目的基因均已成功整合入酵母基因组中。

图3 毕赤酵母的菌落PCR鉴定Fig. 3 PCR identifi cation of individual bacterial colonies from Pichia pastoris
重组酵母GS115/Aoman5A和GS115/Aoman5B经体积分数1%甲醇诱导96 h,离心后测发酵上清液的酶活力,筛选出重组β-甘露聚糖酶reAoMan5A和reAoMan5B表达水平最高的重组子,筛选重组β-甘露聚糖酶reAoMan5A和reAoMan5B表达水平最高的克隆,其酶活力分别达到2.9 IU/mL和12.5 IU/mL。表达上清液经(NH4)2SO4沉淀、超滤除盐、浓缩及Sephadex G-75凝胶过滤,获得电泳纯的reAoMan5A和reAoMan5B,如图4所示。利用光密度扫描法估算得出纯化后重组酶的纯度大于95%,其表观分子质量分别约为68 kD和41 kD,明显大于它们的理论分子质量。reAoMan5A和reAoMan5B糖含量测定的结果分别为25.4%和12.6%,结合糖基化位点预测的结果,进一步说明这2 种重组酶均经过了糖基化修饰,可以初步确定表观分子质量的偏大是由糖基化所致。在pH 3.6和40 ℃时测得reAoMan5A和reAoMan5B的酶比活力分别为8.3 IU/mg和104.2 IU/mg,在此条件下后者的酶比活力明显高于前者。
2.4 温度对重组酶活力的影响
reAoMan5A和reAoMan5B最适温度(Topt)的测定结果如图5A所示,前者的最适温度为35 ℃,后者为50 ℃;它们热稳定性的结果如图5B所示,reAoMan5A的热稳定性较差,40 ℃保温1 h后残留酶活力仅为10.6%;而reAoMan5B在45 ℃保温1 h后残留酶活力仍能维持在85%以上。

图5 reAoMan5A和reAoMan5B的温度特性分析Fig. 5 Temperature characteristic analysis of the expressed reAoMan5A and reAoMan5B
2.5 pH值对重组酶活力的影响

图6 reAoMan5A和reAoMan5B的pH值特性分析Fig. 6 pH characteristic analysis of the expressed reAoMan5A and reAoMan5B
reAoMan5A和reAoMan5B最适pH值的测定结果如图6A所示,两者的最适pH值均偏酸性,属于酸性β-甘露聚糖酶,前者的最适pH值为4.0,后者的最适pH值为3.5。它们pH稳定性的结果如图6B所示,在pH 3.0~6.0之间均较稳定,残留酶活力都可维持在85%以上。
3 讨 论
随着测序技术的不断发展,微生物基因组测序的成本越来越低,已报道的微生物基因组序列也日趋增多,从海量的微生物基因组信息中挖掘新酶基因将会成为一种非常高效的获取新酶基因的手段,迄今为止也出现了大量的利用基因挖掘技术获得新酶基因的报道[12,24-25]。本研究利用基因挖掘技术从已报道的A. oryzae RIB40基因组序列中挖掘到了2 个GH5家族的β-甘露聚糖酶基因,序列比对的结果显示它们与已报道的序列相似度最高的也分别仅为79%和68%,能够定义为2 个新酶基因。将本课题组克隆出的序列与A. oryzae RIB40基因组中的序列进行了比较,Aoman5A的基因序列与基因组数据库中的序列完全一致,而Aoman5B的基因序列与基因组数据库中的序列存在2 个碱基的差异,并呈现出1 个氨基酸残基的差异。由于本实验过程中所有的逆转录和PCR均是采用高保真的酶,而且每一个克隆都是重复了3 次,因此,碱基的差异基本可以排除是由PCR引入的,而是克隆β-甘露聚糖酶基因所用的米曲霉可能与别人测定基因组的米曲霉的确存在一定的差异。此外,AoMan5A的N端存在一个天然的CBM结构,与Benech等[26]报道的序列结构相似,这对研究天然CBM对β-甘露聚糖酶特性的影响具有很大的帮助。
常用的pPIC9K和pPICZαA质粒由于受酶切位点的限制,均不能在毕赤酵母中实现带有天然N端蛋白的表达[27],因此利用它们表达出的重组酶不利于酶学特性的研究。本研究利用改造过的pPIC9KM质粒能够实现AoMan5A和AoMan5B基因在毕赤酵母中的天然N端表达,研究酶学特性时能够消除N端多余残基对酶学特性的影响。AoMan5A基因在毕赤酵母中的表达量较高,但重组酶reAoMan5A的酶比活力非常低,仅有8.3 IU/mg,而且它的最适温度很低、热稳定性也较差,这可能与CBM的存在有一定关系。因为reAoMan5A中N端的CBM存在着大量的Ser和Gly,它们会使蛋白的柔性增强、热稳定性降低[28]。在下一步的研究中可以考虑将CBM去除或者将它移接至AoMan5A催化域的C端,进一步研究CBM对β-甘露聚糖酶酶学特性的影响。此外,尽管reAoMan5B的酶比活力明显高于reAoMan5A,但它在毕赤酵母中的表达量过低,因此在表达上清液中的酶活力也仅为12.5 IU/mL,明显低于宇佐美曲霉和黑曲霉来源的β-甘露聚糖酶基因在毕赤酵母中的表达水平[2,29],它的表达条件还有待进一步的优化。重组酶的表观分子质量与理论分子质量存在明显的差异,鉴于糖基化位点预测及糖含量测定的结果,可以初步确定分子质量的差异是由糖基化所致。
尽管本研究没能获得高活性和高热稳定性的重组β-甘露聚糖酶,但获得的结果仍能为后续的高效表达、理性改造及其应用奠定一些基础。此外,本研究的成功开展进一步丰富了基因挖掘技术的成功应用的案例,可以为其他酶的基因挖掘提供可资借鉴的经验。
[1] LI Jianfang, WEI Xihuan, TANG Cunduo, et al. Directed modifi cation of the Aspergillus usamii β-mannanase to improv e its substrate affinity by in silico design and site-directed mutagenesis[J]. Journal of Industrial Microbiology and Biotechnology, 2014, 41(4): 693-700. DOI:10.1007/s10295-014-1406-7.
[2] LI Jianfang, ZHAO Shunge, TANG Cunduo, et al. Cloning and functional expression of an acidophilic β-mannanase gene (Anman5A) from Aspergillus niger LW-1 in Pichia pa storis[J]. Journal of Agricultural and Food Chemistry, 2012, 60(3): 765-773. DOI:10.1021/ jf2041565.
[3] CHAUHAN P S, PURI N, SHARMA P, et al. Mannanases: microbial sources, production, properties and potential biotechnol ogical applications[J]. Applied Microbiology and Biotechnology, 2012, 93(5): 1817-1830. DOI:10.1007/s00253-012-3887-5.
[4] TANG Cunduo, LI Jianfang, WEI Xihuan, et al. Fusing a carbohydrate-binding module into the Aspergillus usamii β-mann anase to improve its thermostability and cellulose-binding capacity by in silico design[J]. PLoS ONE, 2013, 8(5): e64766. DOI:10.1371/journal. pone.0064766.
[5] GHOSH A, LUIS A S, BRAS J L A, et al. Thermostable recombinant β-(1,4)-mannanase from C. thermocellum: biochemical ch aracterization and manno-oligosaccharides production[J]. Journal of Agricultural and Food Chemistry, 2013, 61(50): 12333-12344. DOI:10.1021/jf403111g.
[6] 唐存多. β-甘露聚糖酶的基因克隆、分子改造及低聚甘露糖的酶法制备[D]. 无锡: 江南大学, 2013.
[7] 唐存多, 史红玲, 焦铸锦, 等. 生物催化剂发现与改造的研究进展[J]. 中国生物工程杂志, 2014, 34(9): 113-121. DOI:10.13523/ j.cb.20140917.
[8] FURUYA T, KINO K. Genome mining approach for the d iscovery of novel cytochrome P450 biocatalysts[J]. Applied Microbiology and Biotechnology, 2010, 86(4): 991-1002. DOI:10.1007/s00253-010-2450-5.
[9] GONG Jingsong, LU Zhenming, LI Heng, et al. Metagenomic technology and genome mining: emerging areas for exploring novel nitrilases[J]. Applied Microbiology and Biotechnology, 2013, 97(15): 6603-6611. DOI:10.1007/s00253-013-4932-8.
[10] CHALLIS G L. Genome mining for novel natural product discovery[J]. Journal of Medicinal Chemistry, 2008, 51(9): 2618-2628. DOI:10.1021/jm700948z.
[11] WINTER R T, HEUTS D P, RIJPKEMA E M, et al. Hot or not? Discovery and characterization of a thermostable alditol oxidase from Acidothermus cellulolyticus 11B[J]. Applied Microbiology and Biotechnology, 2012, 95(2): 389-403. DOI:10.1007/s00253-011-3750-0.
[12] XIAO Z, WU M, GROSSE S, et al. Genome mining for new alphaamylase and glucoamylase encoding s equences and high level expression of a glucoamylase from Talaromyces stipitatus for potenti al raw starch hydrolysis[J]. Applied Biochemistry and Biotechnology, 2014, 172(1): 73-86. DOI:10.1007/s12010-013-0460-3.
[13] ZHU D, MUKHERJEE C, BIEHL E R, et al. Discovery of a mandelonitrile hydrolase from Bradyrhizobium japonicum USDA110 by rational genome mining[J]. Journal of Biotechnology, 2007, 129(4): 645-650. DOI:10.1016/j.jbiotec.2007.02.001.
[14] BARRIUSO J, PRIETO A, MARTÍNEZ M J. Fungal genomes mining to discover novel sterol esterases and lipases as catalysts[J]. BMC Genomics, 2013, 14(1): 712. DOI:10.1186/1471-2164-14-712.
[1 5] TANG Cunduo, GUO Jing, WU Minchen, et al. Cloning and bioinformatics analysis of a novel acidophilic β-mannanase gene, Auman5A, from Aspergillus usamii YL-01-78[J]. World Journal of Micro biology and Biotechnology, 2011, 27(12): 2921-2929. DOI:10.1007/s11274-011-0775-6.
[16] 唐存多, 李剑芳, 邬敏辰, 等. β-甘露聚糖酶末端残基变体对其酶学特性的影响[J]. 中国生物化学与分子生物学报, 2013(7): 655-660.
[17] BRADFORD M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizin g the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72: 248-254.
[18] LAEMMLI U K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4[J]. Nature, 1970, 227: 680-685.
[19] PETERSEN T N, BRUNAK S, von HEIJNE G, et al. SignalP 4.0: discriminating signal peptides from tran smembrane regions[J]. Nature Method, 2011, 8(10): 785-786.
[20] LU Haiqiang, LUO Huiying, SHI Pengjun, et al. A novel thermophilic end o-β-1,4-mannanase from Aspergillus nidulans XZ3: functional roles of carbohydrate-binding module and Thr/Ser-rich linker region[J]. Applied Biochemistry and Biote chnology, 2014, 98(5): 2155-2163. DOI:10.1007/s00253-013-5112-6.
[21] GAO Shujuan, WANG Junqing, WU Minchen, et al. Engineering hyperthermostability into a mesophilic family 11 xylanase from Aspergillus oryzae by in silico design of N-terminus substitution[J]. Biotechnology and Bioengineering, 2013, 11 0(4): 1028-1038. DOI:10.1002/bit.24768.
[22] PARVIZPOUR S, RAZMARA J, RAMLI A N, et al. Structural and functional analysis of a novel psychrophilic β-mannanase from Glaciozyma antarctica PI12[J]. Journal of Computer-aided Molecular Design, 2014, 28(6): 685-698. DOI:10.1007/s108 22-014-9751-1.
[23] 赵月菊, 薛燕芬, 马延和. β-甘露聚糖酶的结构生物学研究现状和展望[J]. 微生物学报, 2009, 49(9): 1131-1137.
[24] BAN J G, KIM H B, LEE M J, et al. Identifi cation of a vitamin D3-specifi c hydroxylase genes through actinomycetes genome mining[J]. Journal of Industrial Micr obiology and Biotechnology, 2014, 41(2): 265-273.
[25] BAN J G, WOO M W, LEE B R, et al. A novel regiospecifi c cyclosporin hydroxylase gene revealed through the genome mining of Pseudonocardia autotrophica[J]. Journal of Industrial Microbiology and Biotechnology, 2014, 41(5): 879-886. DOI:10.1007/s10295-014-1432-5.
[26] BENECH R O, LI X, PATTON D, et al. Recombinant expression, characterization, and pulp prebleaching property of a Phanerochaete chrysosporium endo-β-1,4-mannanase[J]. Enzyme and Microbial Technology, 2007, 41(6/7): 740-747. DOI:10.1016/ j.enzmictec.2007.06.012.
[27] LI Jianfang, GAO Shujuan, LIU Xiaotong, et al. Modified pPIC9K vector-mediated expression of a family 11 xylanase gene, Aoxyn11A, from Aspergillus oryzae in Pichia pastoris[J]. Annals of Microbiology, 2012, 63(3): 1109-1120. DOI:10.1007/s13213-012-0568-7.
[28] 曾胤新, 蔡明 宏, 俞勇, 等. 微生物低温酶适冷机制研究进展[J]. 中国生物工程杂志, 2003(10): 52-56.
[29] TANG Cunduo, GUO Jing, LI Jianfang, et al. Enhancing expression level of an acidophilic β-mannanase in Pichia pastoris by double vector system[J]. Annals of Microbiology, 2014, 64(2): 561-569. DOI:10. 1007/s13213-013-0689-7.
Gene Mining, Expression and Characterization of Novel GH5 Family β-Mannanases
TANG Cunduo1, SHI Hongling1, YU Xiaohua2, ZHANG Mei3, TANG Qinghai1, YUE Chao1, YAO Lunguang1, XIA Min2, KAN Yunchao1,*
(1. Henan Provincial Engineering Laboratory of Insect Bio-reactor, Nanyang Normal University, Nanyang 473061, China; 2. School of Life Science and Technology, Nanyang Normal University, Nanyang 473061, China; 3. Penglai Market Supervisory Authority, Penglai 265600, China)
In order to obtain novel β-mannanases with excellent performance, two putative novel GH5 family β-mannanase genes were excavated from the Aspergillus oryzae RIB40 genome by genome mining, named as Aoman5A and Aoman5B, respectively. The bioinformatic analysis of the two gene sequences was conducted using corresponding softwares or web server, and the genes encoding two mature peptides were expressed in Pichia pastoris GS115 with the aid of plasmid pPIC9KM, and then the expressed products were purifi ed and identifi ed. The results of bioinformatic analysis showed that AoMan5A contained a signal peptide with 20 amino acid residues, while AoMan5B contained a signal peptide with 21 amino acid residues and a propeptide with 12 amino acid residues. The results of sequence alignment displayed that the sequences of two enzymes had the highest similarity of 68% and 79% with the reported sequences, and the N-terminal of AoMan5A also carried a GH1 family CBM. The results of 3-D structure prediction showed that both of them were in accordance with the (α/β)8TIM-barrel structure. Under the same expression condition, the enzymes activities in supernatants from reAoMan5A and reAoMan5B were 2.9 and 12.5 IU/mL, respectively, with specific activity of 8.3 and 104.2 IU/mg, respectively, after purification. The optimum temperature for the former was 35 ℃, while that for the later was 50 ℃. It turned out that both of them were acidic enzymes. The carbohydrate contents of reAoMan5Aand reAoMan5B were determined to be 25.4% and 12.6% using the phenol sulfuric acid method indicating both to be glycosylated. This study will lay a solid foundation for further research and application of β-mannanases, and also provide a referential guidance for the gene mining of other novel enzymes.
β-mannanase; genome mining; bioinformatic analysis; Aspergillus oryzae; protein expression
10.7506/spkx1002-6630-201611016
Q814
A
1002-6630(2016)11-0090-07
唐存多, 史红玲, 蔚晓华, 等. 新型GH5家族β-甘露聚糖酶的基因挖掘及表达鉴定[J]. 食品科学, 2016, 37(11): 90-96. DOI:10.7506/spkx1002-6630-201611016. http://www.spkx.net.cn TANG Cunduo, SHI Hongling, YU Xiaohua, et al. Gene mining, expression and characterization of novel GH5 family β-mannanases[J]. Food Science, 2016, 37(11): 90-96. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201611016. http://www.spkx.net.cn
2015-08-11
国家自然科学基金青年科学基金项目(31401989);河南省高等学校重点科研计划项目(15A416008)
唐存多(1987—),男,讲师,博士,主要从事工业酶与生物催化研究。E-mail:tcd530@nynu.edu.cn
*通信作者:阚云超(1974—),男,教授,博士,主要从事分子生物学研究。E-mail:kanyunchao@163.com

