不同密度下玉米穗部性状的QTL分析
王 辉 梁前进 胡小娇 李 坤 黄长玲 王 琪 何文昭王红武刘志芳
中国农业科学院作物科学研究所, 北京 100081
不同密度下玉米穗部性状的QTL分析
王 辉 梁前进 胡小娇 李 坤 黄长玲 王 琪 何文昭王红武*刘志芳*
中国农业科学院作物科学研究所, 北京 100081
为研究玉米穗部性状对不同种植密度的遗传响应, 以郑58和HD568为亲本构建的220个重组自交系群体为材料, 于2014年春、2014年冬及2015年春分别在北京和海南进行3个种植密度的田间试验, 调查玉米穗长、穗粗、穗行数、行粒数等表型性状。利用SAS软件计算穗部性状的最优线性无偏估计值(BLUP), 并采用完备区间作图法进行QTL定位。结果表明, 在3个种植密度下共检测到42个QTL, 单个QTL可解释4.20%~14.07%的表型变异。3个种植密度下同时检测到位于第2染色体上控制穗行数的QTL。2个种植密度下同时检测到4个与穗粗、穗行数和行粒数有关的 QTL, 其中第 4染色体上 1个与穗行数有关的主效 QTL, 在低、中种植密度下可分别解释表型变异的10.88%和14.07%。此外, 在第2、第4和第9染色体上检测到3个同时调控不同穗部性状的QTL。研究结果表明玉米穗部性状在不同种植密度下的遗传调控发生变化, 在不同密度下共同检测到的稳定QTL可应用于精细定位或开发玉米耐密性分子标记用于辅助育种。
玉米; 穗部性状; 密度; 数量性状位点(QTL); 最优线性无偏估计(BLUP)
玉米是重要的粮食和饲料作物, 在我国和世界粮食安全中有着不可替代的作用。提高玉米单位面积产量是玉米育种的重要目标之一[1]。为了解释玉米产量相关性状的遗传机制, 前人做了大量的研究。例如, Yan等[2]利用266份F2:3家系为材料及150 个SSR和26个RFLP分子标记进行QTL定位, 共检测到29个与单穗产量、百粒重、穗行数和行粒数有关的 QTL, 单个位点可解释表型变异的 3.70%~ 16.80%, 其中11个位点在多环境中被检测到。Li等[3]以黄早四为共同亲本构建了11个RIL群体, 在6个环境条件下对单株产量、百粒重、粒长、粒宽和粒厚进行考察, 共定位到 146个表型贡献率大于 10% 的 QTL, 其中在所有群体同时检测到的 QTL有 16个。Nikolic等[4]利用包含116份样本的F3群体和234个分子标记进行QTL定位, 共检测到5个与单株产量有关的QTL和40个与产量构成因子相关的QTL,单个性状的 QTL累计可解释表型变异 27.46%~ 95.85%。Liu等[5]利用连锁作图和全基因组关联分析的方法对玉米穗行数进行QTL分析, 检测到的QTL包含了 70%的已知穗行数相关位点, 且具有较大的加性或部分显性效应, 这些QTL可进一步用于玉米的全基因组选择育种研究。Zhang等[6]利用243个家系的永久F2群体, 检测到与玉米籽粒发育有关的97 个QTL和26个条件QTL, 其中8个QTL和6个条件QTL在多个环境中均被检测到, 可能包含了调控籽粒发育的关键基因。近年来, 随着玉米穗部性状研究的不断推进, 部分基因已被克隆。Peter 等[7]在第4染色体上克隆到一个CLAVATA受体蛋白位点FASCIATED EAR2, 该位点的突变导致玉米雌穗分生组织增大, 穗行数增多。Liu等[8]利用近等基因系H12和 H12NX531杂交构建 F2群体, 在第 4染色体Unbranched3 (UB3)基因的下游发现一段约 3 kb的区间通过调控UB3的表达进而调控穗行数的变异。但这些结果仍不足以全面解释玉米产量性状的遗传机制和基因调控网络。
在当前的玉米生产情况下, 合理增加种植密度是提高玉米产量的有效手段。近十几年来育种家通过耐密植育种在提高玉米产量上获得了显著成绩[9-10],与耐密响应相关的遗传研究也逐步展开。Gonzalo 等[11]利用186个RIL家系材料分别在50 000株 hm–2和100 000株 hm–2的种植密度下对玉米产量相关性状进行QTL定位, 共检测到9个与产量有关的QTL,其中5个QTL在2个种植密度下均被检测到。Guo等[12]利用郑单958亲本郑58和昌7-2组建231个F2:3家系和 164个分子标记在 52 500株 hm–2和90 000株 hm–2的种植密度下对玉米产量相关性状进行QTL定位, 共检测到76个QTL, 2个种植密度下均检测到的位点有27个, 单个QTL可以解释的最高表型变异为42.09%。然而种植密度对玉米产量的影响十分复杂, 要理解其遗传特征, 挖掘与耐密响应相关的位点仍需进一步深入研究。本研究揭示种植密度对玉米穗部性状遗传结构的影响。通过比较3个种植密度下检测到的 QTL, 找到不同种植密度下能稳定表达的位点和高密度特异性位点, 为玉米穗部性状精细定位和分子标记辅助耐密性育种提供有利信息。
1 材料与方法
1.1 试验材料
以郑58和HD568为亲本配制组合, 采用单粒传法, 连续自交10代构建成包含220个家系的重组自交系群体(recombination inbred line, RIL)。郑58为国内广泛应用的经典自交系, 属改良 Reid群; HD568由昌7-2改良得到, 属塘四平头群。
1.2 田间试验及性状测定
2014春、2015春在北京、2014冬在海南共 3个环境下, 对试验材料进行穗部性状的表型鉴定。每个环境下均采用裂区设计, 设置低密度(LPD, 52 500株 hm–2)、中密度(MPD, 67 500株 hm–2)、高密度(HPD 82 500株 hm–2) 3个密度, 2次重复, 单行区, 行长3.0 m, 行距0.6 m。全部田间试验的灌溉、施肥、除草及病虫害防治均与当地大田管理一致。植株成熟后, 从每小区收获 4穗用于测量穗长、穗粗、穗行数和行粒数。
1.3 遗传连锁图谱构建
本文图谱为前人所构建[13-14], 即在玉米大喇叭口期取每小区幼嫩叶片, 利用 CTAB法提取 DNA,由中国农业大学国家玉米改良中心利用 MaizeSNP3K芯片完成基因型鉴定[15]。获得亲本及家系基因型数据后: (1)删除双亲无多态性和杂合的标记; (2)删除缺失率和SNP杂合率大于20%的标记; (3)统计单个SNP的最小等位基因频率, 删除等位基因频率小于0.05的标记; (4)应用JoinMap 4.0软件对标记进行卡方测验[16], 删除显著偏分离标记; (5)确定SNP标记后, 再统计每个材料的缺失率(< 20%)和杂合率(< 10%), 利用PLINK对材料进行筛选[17]。筛选后共获得1358个SNP标记, 采用QTL ICI-Mapping V3.2软件构图模块构建遗传图谱[18], 设 LOD阈值为 8,基本步骤为 Grouping-Ordering-Ripping-Outputting, 用 Kosambi函数计算标记间的距离, 遗传图距的单位为厘摩(centiMorgan, cM)[19]。图谱总长1985.21 cM,相邻标记间平均遗传距离为1.46 cM。
1.4 表型数据分析
应用 Microsoft Excel 2013软件整理表型数据,采用SAS软件进行基本统计量分析、方差分析及广义遗传力[20]和最优线性无偏估计值(BLUP)[21]的计算。
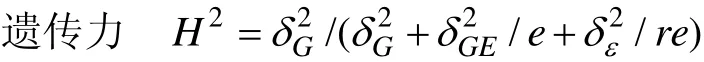
最优线性无偏估计值(BLUP)计算公式:
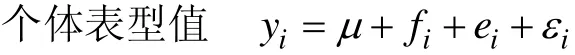
式中, μ为该个体在所有环境下的均值,if为遗传效应, ei为环境效应, εi为随机误差。
1.5 QTL定位方法
利用QTL ICI-Mapping V3.2软件中的完备区间作图法[22]对RIL群体穗部性状进行QTL作图及遗传效应分析, 设作图步长为 1.00 cM, PIN为 0.001, LOD阈值为2.5。
2 结果与分析
2.1 穗部性状表型分析
在3个种植密度下, 亲本郑58和HD568的穗粗、穗长和行粒数的均值随密度增加而减小, 穗行数则无变化规律(表1)。RIL群体中穗部性状呈现丰富的表型变异, 且穗部性状的均值变化规律均随密度增加而减小; RIL群体穗部性状的偏度值和峰度值介于-1 ~ 1之间, 符合数量性状特征, 可进行QTL作图(表1)。
2.2 穗部性状方差分析
如表2所示, 3个种植密度下不同穗部性状家系间和环境间的差异均达到极显著水平。家系与环境互作的差异除低种植密度下的穗粗和穗行数外, 均达到显著水平。邓肯复极差结果表明穗长和行粒数在 3个种植密度间差异显著, 穗粗和穗行数低密度和中密度间差异不显著, 高密度与低、中密度间差异显著(表2)。3个种植密度下各性状的遗传力介于0.54~0.89之间(表2)。
2.3 穗部性状QTL分析

表1 3种种植密度下亲本及RIL群体穗部性状的表现Table1 Performance of ear architectural traits in parents and RIL population under three plant densities
利用SAS软件对2014年春、2014年冬和2015年春得到的穗部性状表型数据进行最优线性无偏估计值(BLUP)计算, 并进行QTL定位。如表3所示, 在3个种植密度下共检测到42个QTL, 分布在除第8染色体外的其他 9条染色体上, 其中低密度下检测到的 15个 QTL, 解释表型变异范围为 4.29%~ 10.88%, 中密度下检测到的13个QTL, 解释表型变异范围为 4.19%~14.07%, 高密度下检测到的 14个QTL, 解释表型变异范围为4.47%~9.77%。多密度下重复检测到的QTL在遗传图谱上的位置如图1所示。

表2 3种种植密度下穗部性状的方差分析Table2 Analysis of variance (ANOVA) for ear architectural traits under three plant densities
2.3.1 穗长 QTL 共检测到 8个, 分布在第 1、第2、第3、第4和第9染色体上, 包括低密度下定位到的3个, 可解释表型变异范围为4.37%~10.50%,中密度下定位到的1个, 可解释表型变异范围6.17%,高密度下定位到的 4个, 可解释表型变异范围为4.30%~9.77% (表3)。在多个密度下未能检测到一致QTL。在低密度下定位到2个控制穗长的主效QTL, qEL2和qEL3, 表型贡献率为10.07%和10.50%; 在高密度下检测到1个控制穗长的位点qEL2, 可解释表型变异 3.48%, 同时该位点也在低密度下被检测到, 与行粒数有关(表3)。
2.3.2 穗粗QTL 共检测到14个, 分布在第1、第2、第3、第4、第5和第9染色体上, 包括低密度下定位到的 5个, 可解释表型变异的 4.74%~ 8.87%, 中密度下定位到的5个, 可解释表型变异的4.20%~8.61%, 高密度下定位到的4个, 可解释表型变异的5.58%~9.05% (表3)。在3个种植密度下未能检测到一致性QTL。在2个种植密度下同时检测到1个位点qED1, 该位点在低密度下的表型贡献率为5.26%, 在中密度下的表型贡献率为5.34% (表3)。在中密度下检测到 1个同时控制穗粗和穗行数的QTL, 可解释穗粗性状表型变异8.53%。在标记区间SNP2707~SNP2712内的不同位置分别检测到高密度下控制穗长和穗粗的位点qEL9和qED9, 可解释穗长表型变异 4.30%, 可解释穗粗表型变异 9.05%(表3)。
2.3.3 穗行数 QTL 共检测到 14个, 分布在第2、第3、第4、第7和第10染色体上, 包括低密度下定位到的4个, 可解释表型变异的4.29%~10.88%,中密度下定位到的 5个, 可解释表型变异的4.34%~14.07%, 高密度下定位到的 5个, 可解释表型变异的4.84%~8.79% (表3)。QTL qERN2在3个种植密度下同时被检测到, 依次分别解释表型变异的7.56%、8.84%和8.79%; QTL qERN3-2和qERN4在低、中2个种植密度下均被检测到, qERN3-2可分别解释表型变异的7.32%和5.58%, qERN4可分别解释表型变异的10.87%和14.07% (表3)。在标记区间SNP1302~SNP1322内检测到同时控制穗行数和穗粗的位点, 高密度下的qERN4和中密度下的qED4,可分别解释表型变异7.67%和8.63% (表3)。
2.3.4 行粒数QTL 共检测到6个, 分布在第1、第2、第5、第6和第7染色体, 包括低密度下定位到的3个, 可解释表型变异的4.78%~8.63%, 中密度下定位到的2个, 可解释表型变异的5.50%和6.23%,高密度下定位到的 1个, 可解释表型变异的 8.06%(表3)。3个种植密度下未检测到一致性QTL。2个种植密度下共同检测到1个行粒数位点qKNR7, 在中、高密度下可分别解释表型变异的6.24%和8.06%(表3)。

表3 多环境条件下穗部性状BLUP预测值QTL定位分析Table3 QTL analysis of ear architectural traits under joint-environments using BLUP predictive value
3 讨论
3.1 玉米穗部性状的遗传基础
玉米穗部性状受到复杂的基因网络调控, 并易受环境影响。准确的表型数据和精密的遗传图谱是挖掘控制玉米穗部性状QTL的保障。基因型和环境间的互作效应对表型值影响显著, 会干扰QTL的定位结果, 本试验以高代RIL群体为材料, 采用2年2点试验, 设置 2次重复等措施, 较好地控制了环境和试验误差, 保证了表型数据的准确性。QTL定位中遗传连锁图谱的标记密度决定着定位区间的大小和准确性, 足够密集的标记可以充分检测到发生在群体内的重组事件。前人研究采用的遗传连锁图谱通常标记较少, 分辨率较低, 如前人所用遗传图谱的平均遗传距离为14.5 cM[2]、13.9 cM[12]、6.8~10.0 cM[3]、6.4 cM[23]、11.53 cM和11.84 cM[24]。标记间距离过大导致部分重组无法被有效检测到, 从而引起表型与基因型的关联偏差, 导致一些QTL不能被检测到, 限制了玉米产量遗传基础的深度解析。随着SNP标记的出现和发展, 越来越多的研究开始采用密集的分子标记构建高密度遗传连锁图谱, Li等[25]采用遗传连锁图谱的平均遗传距离为 1.70~2.10 cM, 秦伟伟等[26]为1.21 cM。本研究中构建的遗传图谱共包含1358个SNP标记, 长度为1985.21 cM, 标记间的平均遗传距离仅为 1.46 cM, 故有效地提高了 QTL作图精密度, 增加了QTL检测的精确性。
本研究在3个种植密度下共检测到42个QTL,在第1、第2、第3、第4和第7染色体定位到的控制穗粗的qED1, 控制穗行数的qERN2、qERN3-2、qERN4和控制行粒数的qKNR7能在多密度下稳定表达。这些位点中, 除 qERN4之外, 可以解释的表型变异均小于 10%。研究这些位点对理解玉米遗传机制有重要作用。本实验与前人结果比较, 吕学高等[27]利用300对SSR分子标记进行QTL定位, 在第1染色体(umc139–bnlg1811)检测到与本文 qED1 (65 Mb~69 Mb)所在标记区间一致的QTL。Liu等[5]利用3个 F2:3群体进行 QTL定位, 分别在第 3染色体(umc2152–umc2048)和第4染色体(bnlg2162–umc1284)上检测到与本文中qERN3-2 (224~225 Mb)和qERN4 (205~215 Mb)位于同一染色体区段的位点, 说明这些区段内含有控制相关性状的基因位点且可靠性较高。此外, Veldboom等[28]、Yan等[2]和Yang等[24]也在第4染色体上定位到与本文的qERN4 (205~215 Mb)在同一染色体区段的位点, 同时, 该位点在本研究中的表型贡献率超过 10%, 可推测该位点是影响玉米穗行数变异的主效位点, 可以用于精细定位和图位克隆。兰进好等[29]利用黄早四和Mo17构建的184 个F2:3家系, 在第7染色体(umc1944–P3936245)检测到与本文中qKNR7 (157~162 Mb)位于相邻染色体区段的位点。此外, 本文结果与Brown等[30]、Liu等[5]、Würschum等[31]、Cai等[32]国内外相关研究比较, 均未发现相同 QTL。本研究在第 2染色体定位到的qERN2 (59~63 Mb), 尚未见报道。
本研究检测到在第2染色体存在同时控制穗长和行粒数的QTL, 在第4染色体存在同时控制穗粗和穗行数的QTL, 在第9染色体上存在同时控制穗长和穗粗的QTL。Tuberosa等[33]认为“一因多效”及控制不同性状的基因间的紧密连锁是导致此类现象的主要原因。
3.2 密度与穗部QTL
环境及QTL与环境互作是影响基因表达的重要因素, 因此在不同环境下的QTL定位结果不尽相同[34-36]。Huang等[37]研究认为QTL对环境表现敏感, 而不同性状的QTL对环境的敏感度不同, 遗传力高的性状在不同环境中更容易被检测到。Tanksley等[38]研究表明, 主效QTL在不同环境中也较容易被检测到。种植密度作为重要的环境因子之一, 同样影响着QTL的定位结果。因此本研究利用多个环境中表型的BLUP数值控制可以有效地降低环境差异对QTL定位结果的影响, 更好地揭示不同种植密度间QTL、数目及效应的差异。
种植密度对QTL定位的影响极其复杂, 其遗传机制尚不清楚。在本研究中, 发现有5个QTL (qED1、qERN2、qERN3-2、qERN4和qKNR7)在多个密度下均被检测到。其中, 除 qED1的加性效应为正值外,其余均为负值, 且加性效应均为中密度下最大, 说明qED1的增效基因来自于母本(郑58), 其余均来自父本(HD568), 且中密度为这些位点表达的最适密度。qKNR7在低密度下未被检测到, 在中密度和高密度下被检测到且贡献率和加性效应值随密度增加而增加。说明该位点对表型的贡献随密度的增加而增加。

图1 穗部性状主效QTL在遗传图谱上的分布Fig.1 Distributions of identified major QTL for ear traits on genetic linkage maps
在本研究中检测到的QTL中大多只在单个密度下被检测到, 且同一性状在3个不同种植密度下检测到的QTL数目差异较大。例如本研究定位的穗部性状中, 在低密度、中密度和高密度分别定位到QTL, 其中控制穗长的为3个、1个和4个; 控制行粒数的为3个、2个和1个。可以推测, 种植密度会影响玉米产量性状的遗传控制; 也暗示着控制不同性状的 QTL可能存在适合各自发挥效应的种植密度。因此发掘在不同密度下同时发挥效应的QTL和在该密度下特定表达的 QTL, 对于分子标记辅助玉米耐密育种具有重要的意义。
4 结论
在不同种植密度下共定位到42个QTL, 其中5个在多个密度下均被检测到。主效位点qERN2在3个种植密度下被定位到且尚未见前人报道, 主效位点qERN4在2个种植密度下被检测到。这些位点为QTL精细作图和图位克隆提供了重要的研究基础。不同种植密度下检测到差异 QTL, 说明玉米穗部性状QTL的表达受种植密度和基因与种植密度互作的影响。以上结果可以在一定程度上解释种植密度对玉米穗部性状影响的遗传机制, 为玉米耐密性育种提供理论依据。
[1] Ku L X, Zhao W M, Zhang J, Wu L C, Wang C L, Wang P A, Zhang W Q, Chen Y H.Quantitative trait loci mapping of leaf angle and leaf orientation value in maize (Zea mays L.).Theor Appl Genet, 2010, 121: 951–959
[2] Yan J B, Tang H, Huang Y Q, Zheng Y L, Li J S.Quantitative trait loci mapping and epistatic analysis for grain yield and yield components using molecular markers with an elite maize hybrid.Euphytica, 2006, 149: 121–131
[3] Li C, Li Y, Sun B, Peng B, Liu C, Liu Z Z, Yang Z Z, Li Q C, Tan W W, Zhang Y, Wang D, Shi Y S, Song Y C, Wang T Y, Li Y.Quantitative trait loci mapping for yield components and kernel-related traits in multiple connected RIL populations in maize.Euphytica, 2013, 19: 303–316
[4] Nikolić A, Anđelković V, Dodig D, Drinić S M, Kravić N, Micić-Ignjatović D.Identification of QTLs for drought tolerance in maize: II.Yield and yield components.Genetika, 2013, 45: 341–350
[5] Liu L, Du Y F, Huo D A, Wang M, Shen X M, Yue B, Qiu F Z, Zheng Y L, Yan J B, Zhang Z X.Genetic architecture of maize kernel row number and whole genome prediction.Theor Appl Genet, 2015, 128: 2243–2254
[6] Zhang Z H, Wu X Y, Shi C N, Wang R N, Li S F, Wang Z H, Liu Z H, Xue Y D, Tang G L, Tang J H.Genetic dissection of the maize kernel development process via conditional QTL mapping for three developing kernel-related traits in an immortalized F2population.Mol General Genet, 2015, 291: 437–454
[7] Peter B, Namiko S N, David J.Quantitative variation in maize kernel row number is controlled by the FASCIATED EAR2 locus.Nat Genet, 2013, 45: 334–337
[8] Liu L, Du Y F, Shen X M, Li M F, Sun W, Huang J, Liu Z J, Tao Y S, Zheng Y L, Yan J B, Zhang Z X.KRN4 controls quantitative variation in maize kernel row number.PLoS Genet, 2015, 11(11): e1005670
[9] Guo J, Su G, Zhang J, Wang G.Genetic analysis and QTL mapping of maize yield and associate agronomic traits under semi-arid land condition.Afr J Biotechnol, 2008, 7: 1829–1838
[10] Ribaut J M, Jiang C, Gonzalez-De-Leon D, Edmeades G O, Hoisington D A.Identification of quantitative trait loci under drought conditions in tropical maize: 2.yield components and marker-assisted selection strategies.Theor Appl Genet, 1997, 94: 887–896
[11] Gonzalo M, Holland J B, Vyn T J, Mclntyre L M.Direct mapping of density response in a population of B73 ×Mo17 recombinant inbred lines of maize (Zea mays L.).Heredity, 2010, 104: 583–599
[12] Guo J, Chen Z, Liu Z, Wang B B, Song W B, Li W, Chen J, Dai J R, Lai J S.Identification of genetic factors affecting plant density response through QTL mapping of yield component traits in maize (Zea mays L.).Euphytica, 2011, 182: 409–422
[13] 刘小刚.玉米茎秆强度 QTL定位研究.中国农业科学院硕士学位论文, 北京, 2014
Liu X G.Quantitative Trait Locus Analysis of Stalk Strength in Maize.MS Thesis of Chinese Academy of Agricultural Sciences, Beijing, China, 2014 (in Chinese with English abstract)
[14] 马飞前.玉米茎秆纤维性状 QTL定位.中国农业科学院硕士学位论文, 北京, 2014
Ma F Q.Mapping of Quantitative Trait Loci (QTL) for Stalk Fiber Traits in Maize.MS Thesis of Chinese Academy of Agricultural Sciences, Beijing, China, 2014 (in Chinese with English abstract)
[15] Ganal M W, Gregor D, Andreas P, Aurélie B, Buckler E S, Alain C, Clarke J D, Graner E M, Hansen M, Joets J, Paslier M-C L, McMullen M D, Montalent P, Rose M, Schön C C, Sun Q, Walter H, Martin O C, Matthieu F.A large maize (Zea mays L.) SNP genotyping array: development and germplasm genotyping, and genetic mapping to compare with the B73 reference genome.PLoS One, 2011, 6(12): e28334
[16] Van Ooijen J: JoinMap4.software for the Calculation of Genetic Linkage Maps in Experimental Populations.Kyazma B V, Wageningen, Netherlands, 2006.p 56
[17] Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira M A R, Bender D, Maller J, Sklar P, Bakker P I W, Daly M J, Sham P C.PLINK: a tool set for whole-genome association and population-based linkage analyses.Am J Human Genet, 2007, 81: 559–575
[18] Wang J K.QTL IciMapping: Integrated Software for Building Linkage Maps and Mapping Quantitative Trait Genes.International Plant and Animal Genome Conference XXI 2013.Scherago International, San Diego, CA, 2013
[19] Kosambi D D.The estimation of map distance from recombination valves.Annu Eugenia, 1994, 12: 172–175
[20] Knapp S, Stroup W, Ross W.Exact confidence intervals for heritability on a progeny mean basis1.Crop Sci, 1985, 25: 192–194
[21] Piepho H P, Möhring J, Melchinger A E, Büchse A.BLUP for phenotypic selection in plant breeding and variety testing.Euphytica, 2008, 161: 209–228
[22] 王建康.数量性状基因的完备区间作图方法.作物学报, 2009, 35: 239–245
Wang J K.Inclusive composite interval mapping of quantitative trait genes.Acta Agron Sin, 2009, 35: 239–245 (in Chinese with English abstract)
[23] Cai L, Li K, Yang X, Li J.Identification of large-effect QTL for kernel row number has potential for maize yield improvement.Mol Breed, 2014, 34: 1087–1096
[24] Yang C, Liu J, Rong T Z.Detection of quantitative trait loci for ear row number in F2populations of maize.Genet Mol Res, 2015, 14: 14229–14238
[25] Li K, Yan J, Li J, Yang X.Genetic architecture of rind penetrometer resistance in two maize recombinant inbred line populations.BMC Plant Biol, 2014, 14: 152
[26] 秦伟伟, 李永祥, 李春辉, 陈林, 吴迅, 白娜, 石云素, 宋燕春,张登峰, 王天宇, 黎裕.基于高密度遗传图谱的玉米籽粒性状QTL定位.作物学报, 2015, 41: 1510–1518
Qin W W, Li Y X , Li C H, Chen L, Wu X, Bai N, Shi Y S, Song Y C, Zhang D F, Wang T Y, Li Y.QTL mapping for kernel related traits based on a high-density genetic map.Acta Agron Sin, 2015, 41: 1510–1518 (in Chinese with English abstract)
[27] 吕学高, 蔡一林, 陈天青, 徐德林, 王伟林, 刘志斋, 王久光.玉米穗部性状 QTL定位.西南大学学报(自然科学版), 2008, 30(2): 64–70
Lü X G, Cai Y L, Chen T Q, Xu D L, Wang W L, Liu Z Z, Wang J G.QTL mapping for ear traits in maize (Zea mays L.).J Southwest Univ (Nat Sci Edn), 2008, 30(2): 64–70 (in Chinese with English abstract)
[28] Veldboom L R, Lee M.Genetic mapping of quantitative trait loci in maize in stress and nonstress environments: I.Grain yield and yield components.Crop Sci, 1996, 36: 1310–1319
[29] 兰进好, 李新海, 高树仁, 张宝石, 张世煌.不同生态环境下玉米产量性状QTL分析.作物学报, 2005, 31: 1253–1259
Lan J H, Li X H, Gao S R, Zhang B S, Zhang S H.QTL analysis of yield components in maize under different environments.Acta Agron Sin, 2005, 31: 1253–1259 (in Chinese with English abstract)
[30] Brown P J, Upadyayula N, Mahone G S, Tian F, Bradbury P J, Myles S, Holland J B, Flint-Garcia S, McMullen M D, Buckler E S, Rocheford T R.Distinct genetic architectures for male and female inflorescence traits of maize.PLoS Genet, 2011, 7: 1276–1280
[31] Würschum T.Mapping QTL for agronomic traits in breeding population.Theor Appl Genet, 2012, 125: 201–210
[32] Cai L C, Li K, Yang X H, Li J S.Identification of large-effect QTL for kernel row number has potential for maize yield improvement.Mol Breed, 2014, 34: 1087–1096
[33] Tuberosa R, Salvi S, Sanguineti M C, Landi P, Maccaferri M, Conti S.Mapping QTLs regulating morpho-physiological traits and yield: case studies, shortcomings and perspectives in drought-stressed maize.Ann Bot, 2002, 89: 941–963
[34] Zhuang J Y, Lin H X, Lu J, Qian H R, Hittalmani S, Huang N, Zheng K L.Analysis of QTL environment interaction for yield components and plant height in rice.Theor Appl Genet, 1997, 95: 799–808
[35] Chen J, Zhu J.Genetic effects and genotype ×environment interactions for cooking quality traits in indica-japonica crosses of rice (Oryza sativa L.).Euphytica, 1999, 109: 9–15
[36] Shi C H, He C X, Zhu J, Chen J G.Analysis of genetic effects and genotype ×environment interaction effects for apparent quality traits of indica rice.Chin J Rice Sci, 1999, 13: 179–182 (in English with Chinese abstract)
[37] Huang N, Angeles E R, Domingo J, Magpantay S, Singh S, Zhang G, Kumaravadivel N, Bennett J, Khush G S.Pyramiding of bacterial blight resistance genes in rice: marker-assisted selection using RFLP and PCR.Theor Appl Genet, 1997, 95: 313–320
[38] Tanksley S D, Ahn N, Causse M, Coffman R, Fulton T, McCouch S R, Second G, Tai T, Wang Z, Wu K, Yu Z.RFLP mapping of the rice genome.In: Rice Genetics II.Los Banos, Laguna: IRRI, 1991.pp 435–442
QTL Mapping for Ear Architectural Traits under Three Plant Densities in Maize
WANG Hui, LIANG Qian-Jin, HU Xiao-Jiao, LI Kun, HUANG Chang-Ling, WANG Qi, HE Wen-Zhao, WANG Hong-Wu*, and LIU Zhi-Fang*
Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China
To identify genetic factors of ear architectural traits response to plant density, we developed a recombination inbred line (RIL) mapping population with 220 families from a cross between two maize inbred lines, Zheng 58 and HD568.The filed experiments were performed in 2014 and 2015 seasons of Beijing and Hainan.The ear architectural traits including ear length, ear diameter, ear row number and kernel number per row were evaluated under three plant densities in each environment.With the BLUP value estimated by SAS software, QTLs for ear architectural traits were detected by inclusive composite interval mapping (ICIM) using Windows QTL ICI-Mapping software.In total, 42 QTLs were detected under three plant densities, each QTL explained phenotypic variation ranging from 4.20% to 14.07%.One QTL related to ear row number on chromosome 2 was repeatedly detected under three plant densities.Four QTLs related to ear diameter, ear row number and kernel number per row were commonly detected under two plant densities, among them an ear row number QTL was located on chromosome 4 with explained 10.88% and 14.07% of phenotypic variance under plant density of 52 500 plants ha–1and 67 500 plants ha–1.In addition, we found three QTLs for different ear architectural traits on chromosomes 2, 4, and 9 simultaneously.This study revealed the genetic mechanisms of ear architectural traits changed under different plant densities.The QTLs stably expressed under different plant densities can be applied in fine mapping and marker assisted selection in density tolerance breeding of maize.
Maize; Ear architectural traits; Plant density; QTL; BLUP
10.3724/SP.J.1006.2016.01592
本研究由国家重点基础研究发展计划(973计划)项目(2014CB138200), 北京市科技计划项目(D141100005014003)和中国农业科学院科技创新工程项目资助。
This study was supported by the National Basic Research Program of China (2014CB138200), the Program of Beijing Municipal Science and Technology (D141100005014003), and the Agricultural Science and Technology Innovation Program (ASTIP) of CAAS.
*通讯作者(Corresponding authors): 刘志芳, E-mail: liuzhifang@caas.cn; 王红武, E-mail: wanghongwu@caas.cn
联系方式: E-mail: 1300272654@qq.com
稿日期): 2016-03-03; Accepted(接受日期): 2016-06-20; Published online(
日期): 2016-07-04.
URL: http://www.cnki.net/kcms/detail/11.1809.S.20160811.1623.024.html

