小五台山蟹蛛DNA条形码分子鉴定
何静超,胡岚岚,郭晨辉,张锋
(河北大学生命科学学院,河北保定 071002)
小五台山蟹蛛DNA条形码分子鉴定
何静超,胡岚岚,郭晨辉,张锋
(河北大学生命科学学院,河北保定071002)
为了探讨DNA条形码技术在蟹蛛科蜘蛛物种鉴定中的可行性,本研究基于线粒体COI基因序列,使用邻接法和贝叶斯法构建系统发育树,ABGD(automatic barcode gap discovery)软件对样本进行划分,对小五台山25种蟹蛛110个样本进行DNA条形码分子鉴定.结果表明:邻接法和贝叶斯法构建的系统发育树聚类结果与ABGD软件划分结果以及形态分类鉴定结果相一致.据此笔者认为DNA条形码作为一种有效的分子鉴定工具可以应用到蟹蛛科蜘蛛物种鉴定中.
蟹蛛科;DNA条形码;系统发育树;ABGD
科学准确的物种鉴定是深入开展生物多样性、环境监测、生态学、进化生物学、人口健康和医学等多学科研究和利用的前提[1].传统的分类学研究对标本的完整性要求较高,且需要通过长时间的专业训练才能培养出精通于特定类群的分类学专家.除此之外,由于形态学鉴定存在局限性并且分类学家队伍也在不断缩减,使得进化分类学的发展面临着巨大的挑战[2].
Hebert等[3-4]于2003年提出利用一段较短的DNA序列作为分子标记可以精准地进行物种鉴定,此技术称为DNA条形码技术.同时他们认为动物的DNA条形码可以用线粒体基因细胞色素C氧化酶I(COI)5′端一段长度为658 bp的DNA序列表示.
到目前为止,DNA条形码技术已应用到大部分动物物种鉴定中[5-13],其中包括鸟类、鱼类、昆虫、哺乳类、两栖类、兽类、浮游和海洋小型底栖动物等.该片段不仅可在物种水平上对绝大多数动物类群进行鉴定,还能够准确地区分近缘类群,同时也是鉴定新种与发现隐存种的良好工具,并用于快速构建不同分类阶元的系统发育关系[14].因此,COI基因已被公认为动物DNA标准条形码片段并得到广泛应用.但是由于DNA条形码的COI基因位于线粒体上,而线粒体是母系遗传,因此在鉴定存在杂交的生物类群时存在缺陷[15],对于存在不完全支序演化与基因渗入现象的生物类群,DNA条形码难以区分[16].例如:蝇属中大约有60%的物种无法用COI基因进行鉴别,因为一些种的条形码是一样的;在对珊瑚虫的研究中发现COI基因在不同的物种间变化极小,因此也不能成功地鉴别该物种[1].Sperling利用COI基因分析昆虫,结果显示:至少有1/4的昆虫物种很难被区分[17].而蜘蛛类此方面的报道甚少,因此,利用COI基因对蜘蛛进行物种鉴定的可行性有待于证明.
蟹蛛科Thomisidae是蜘蛛目中的第7大科[18],物种多样性丰富,体型变化较大.传统的蟹蛛分类鉴定工作主要依据成熟标本的外生殖器官来进行分类,即雄蛛的触肢器和雌蛛的外雌器.但对于外形相近的隐存种的鉴别、多组形态上相近物种的雌雄配对、同一物种受分布区域及生境变化引起的种内差异以及发育未成熟物种的鉴定等问题,单纯依靠进化分类学方法较难解决.有鉴于此,本研究选用分布于小五台山的10属25种蟹蛛作为实验材料,利用线粒体COI基因,进行基于系统发育树和距离方法的DNA条形码分析,探索了DNA条形码技术对蟹蛛科物种鉴定的准确性,以探索建立快速高效的蜘蛛目物种分类的新方法.
1 材料与方法
1.1标本采集及形态鉴定
本研究选用的110头蜘蛛标本均由本研究室成员于2013年6~9月和2014年6~9月在河北小五台山(东经114°50′~115°15′,北纬39°40′~40°10′)采得(表1).
标本采集后保存于体积分数为95%的酒精中,由本研究室成员进行了形态学种类鉴定,并保存于河北大学博物馆(MHBU).

表1 所用标本信息Tab.1 Thomisidae samples used in this study

续表1 Continue tab.1
1.2DNA的提取、PCR扩增及测序
取样本一侧步足,在液氮中研磨后使用天根生化科技(北京)有限公司的TIANamp血液/组织/细胞基因组DNA提取试剂盒提取总DNA.
对于提取的总DNA使用通用引物LCO 1490 (5′-GGTCAACAAATCATAAAGATATTGG-3′)和HCO 2198 (5′-TAAACTTCAGGGTGACCAAAAAATCA-3′)[19]进行扩增.PCR体系为25 μL:MasterMix 12.5 μL,LCO 1490(10 μmol/L)HCO 2198(10 μmol/L)各0.8 μL,ddH2O 8.9 μL, DNA模板2.0 μL.PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,48 ℃退火40 s,72 ℃延伸1 min,33~35个循环;72 ℃延伸7 min.
用质量分数1%琼脂糖凝胶电泳检测PCR扩增产物,将条带清晰的PCR样品送金维智生物科技有限公司(北京)进行双向测序.测序结果首先用Chromas软件判断峰图质量,并对每个碱基逐一进行人工校对,然后用seqman软件对双向测序结果进行拼接后,以Fasta格式输出.
1.3系统发育分析
1.3.1系统发育分析
经校对的序列以园蛛科的1条COI序列为外群,用Clustal X[20]进行比对,用MEGA 6.05[21]中的Kimura双参数模型(Kimura-2-parameter,K2P)[22]计算种内和种间遗传距离,采用邻接法构建系统发育树,自展检验值(Bootstrap)设置为1 000,得到NJ树上各分支节点的支持率.使用 MrBayes v3.2.1[23],以园蛛科的1条相应序列为外群,构建各单倍型的 Bayesian 系统发育树,替代模型为GTR+I.贝叶斯分析包含5×104generations 的运算,以运算得到的后验概率来标示系统发育树各分支的置信度.
1.3.2BACODING GAP 分析
基于K2P模型使用MEGA6.05计算种内及种间遗传距离并在EXCEL中进行距离统计,根据统计结果画出频率分布直方图,进行“barcoding gap”[24]分析.
1.3.3ABGD划分
基于遗传距离使用ABGD(automatic barcode gap discovery)软件[25]对样本进行划分,划分在同一组的样本被认为是同一种.将111个样本的基因序列的fasta文件在线提交到ABGD网站(http://wwwabi.snv.jussieu.fr/public/abgd/),其中种内差异先验值P(prior intraspecific divergence)设置为0.001到0.1, 最小相对gap 宽度值X(minimum relative gap width)设置为1.0,将样本划分结果与形态鉴定结果逐一进行比较.
2 结果及分析
2.1系统发育树的聚类分析
以样本的总DNA为模板,将经PCR扩增、公司测序得到的110条长度为658 bp的COI序列提交到中国生命条形码数据门户中心(http://www.barcodeoflife.cn/).
根据形态学鉴定,110头蟹蛛标本最终被鉴定为10属25种.分别采用邻接法和贝叶斯法,以1种园蛛的COI序列为外群构建NJ系统发育树和BI系统发育树,聚类结果如图1所示,圆花叶蛛Synemaglobosum和其他种的蜘蛛形成姐妹群,每个种聚类明显,没有出现物种交叉现象,并且均各自聚成一个单系群,且各单系分支的支持率均超过60%.
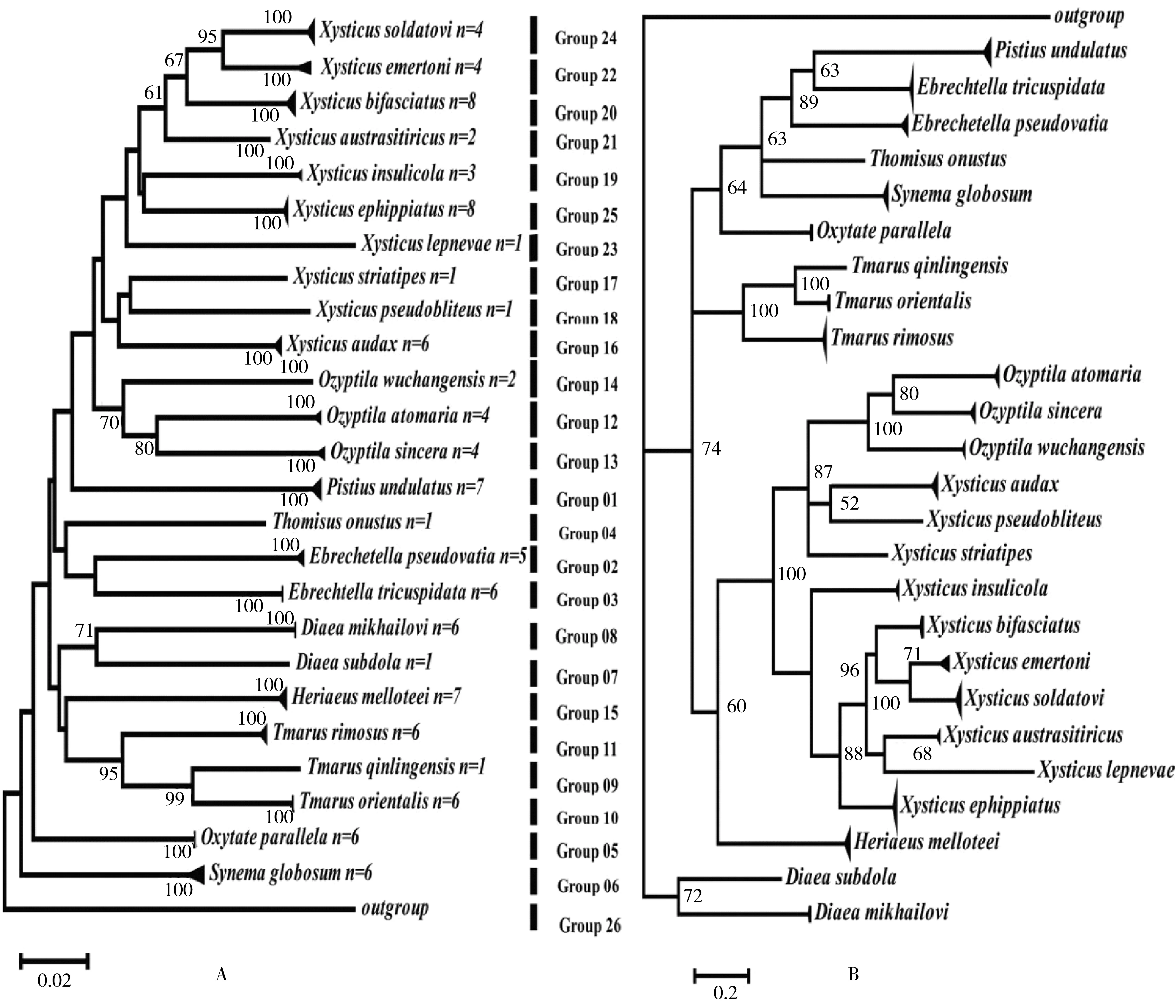
A.NJ 系统发育树; B.BI 系统发育树; n.序列条数 group、ABGD自动分组结果.图1 基于COI序列构建的小五台山蟹蛛的系统发育树Fig.1 Phylogenetic tree of the spiders of thomisidae based on COI gene
2.2DNA条形码的GAP分析
使用MEGA6.05基于K2P模型进行“barcoding gap”分析,做频率直方图(图2),种内遗传距离为0~0.5%,平均为0.15%;种间遗传距离为5.25%~23.06%,平均为13.97%;存在明显的GAP区,即种内的最大值小于种间的最小值,说明所用蟹蛛科数据能够很好地区分种内和种间.
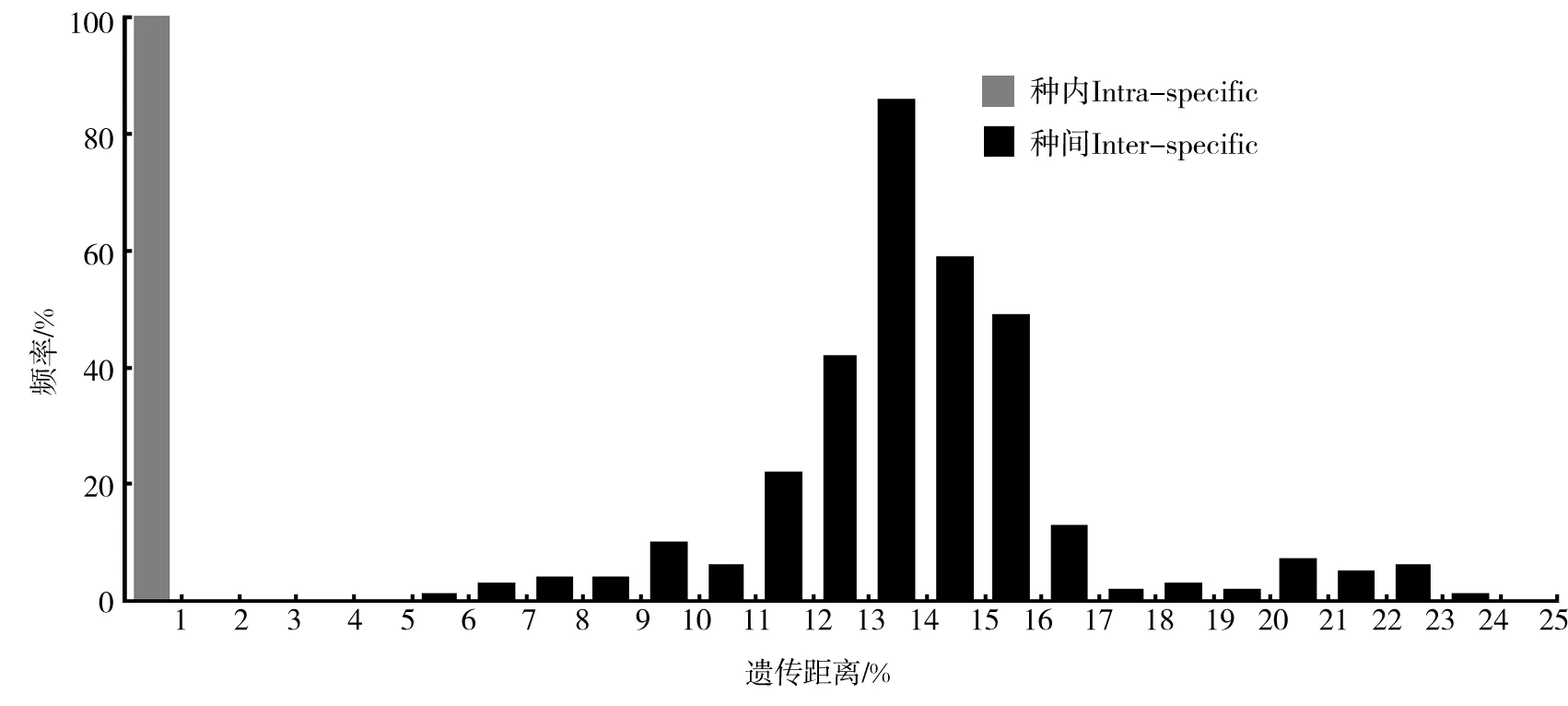
图2 蟹蛛科COI序列遗传距离分析Fig.2 Analysis of genetic distance of COI gene from Thomisidae taxa
2.3基于距离的分类分析
用ABGD软件对111个样本(包含外群)进行划分,结果包括初始划分和递归划分2种情况(图3):初始划分较稳定,111个样本被分成26种;递归划分把111个样本分成1~36组,但是在很大的P值范围内都维持在26组.将初始划分结果与进化分类学鉴定结果进行比较,发现初始划分中每个分组的种与形态种均呈现一一对应的关系,基于样本遗传距离的ABGD方法鉴定25种蟹蛛的成功率为100%.
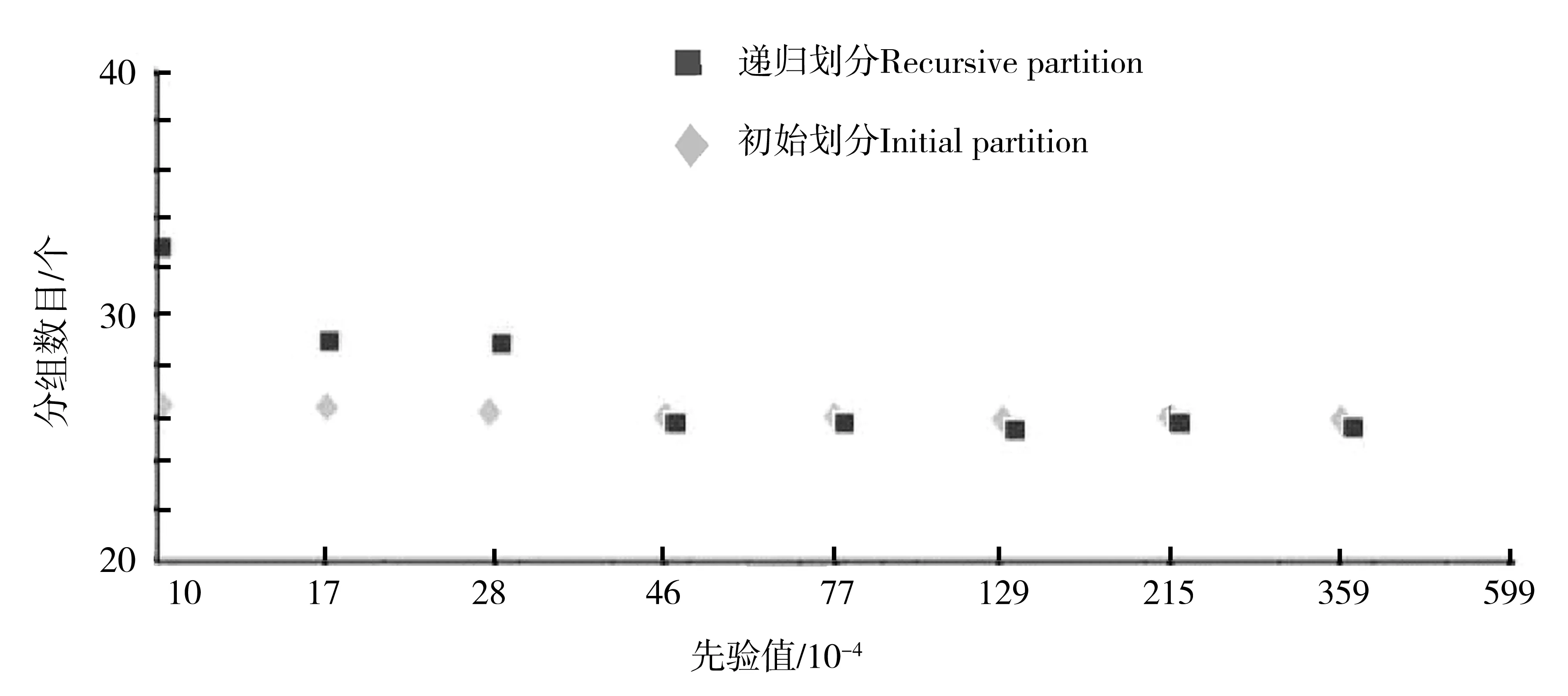
图3 ABGD划分结果Fig.3 Automatic partition results of ABGD
3 讨论
本研究利用通用引物对河北小五台山的110头蟹蛛进行了COI基因序列的扩增,利用邻接法和贝叶斯法分别构建了系统发育树,结果显示所有的种均各自聚成了一个单系群.对种内和种间遗传距离分析可得,种间平均遗传距离(13.97%)是种内平均遗传距离(0.15%)的93.1倍,符合种间差异是种内差异的10倍[3]的DNA条形码有效性的检测标准.因此,COI序列在蜘蛛目蟹蛛科的鉴定中具有较高的准确性,可以作为鉴定小五台山蟹蛛的DNA条形码.
在某些类群中由于线粒体基因置换速率不同,出现了种内和种间遗传距离出现重叠现象,导致难以界定种内和种间差异的阈值标准[2].ABGD方法能成功解决上述难题,因为ABGD是基于一定范围的先验值P自动的探测给定数据集的“barcoding gap”,在一定程度上能降低阈值定义的人为因素,减少了基于距离分类的主观性,并且拥有很高的准确性.在本研究中显示出ABGD的分组结果与形态分种结果完全一致.由于DNA条形码技术能够快速准确地鉴别物种,并能发现新种与隐存种而被广泛应用.目前这种技术也广泛地应用到蛛形纲动物中,如Zhang等[26]于2014年利用COI和其他基因片段来研究洞穴蜘蛛隐存种的多样性;Huber等[27]于2009年利用形态与分子结合的方法对幽灵蛛科的Tainonia属进行了修订.Franzini 等[28]利用COI和H3基因片段构建系统发育树并且与形态学相结合的方法发现了园蛛科的新种.Zhang等[29]利用COI及其他基因片段研究了跳蛛科斑蛛亚科的分子系统学、分歧时间和生物地理分布,结果显示采用的263头标本通过贝叶斯法、最大似然法和简约法构建的系统发育树中支持斑蛛亚科为单系的有85属,进而对斑蛛亚科进行了属级修订.
不可否认,DNA数据具有很强的可靠性,但如果只依靠DNA数据而忽略其他因素,得到的仅仅是序列相似性模式,而不是进化模式.因此,用DNA条形码技术进行分析时不能脱离形态学手段.除此以外,由于单分子标记的局限性,应该将其他基因片段与COI一起作为DNA条形码的标记基因,以适应物种鉴定的需要[30].
虽然条形码技术受到一些专家的质疑,但DNA信息的丰富性和独一无二的可重复性,以及在动物、植物和微生物中已经取得的丰富的研究成果,明显推动了生物分类学的发展.事实上,DNA 条形码与进化分类学相辅相成,彼此促进,共同推动生物分类和系统学研究[31].
[1]程希婷,王爱民,顾志峰,等.DNA条形码研究进展[J].基因组学与应用生物学, 2011,30(6):748-758.
CHENG Xiting,WANG Aimin,GU Zhifeng,et al.Current progress of DNA barcoding[J].Genomics and Applied Biology,2011,30(6):748-758.
[2]肖金花,肖晖,黄大卫.生物分类学的新动向——DNA条形编码[J].动物学报,2004,50(5):852-855.
XIAO Jinhua,XIAO Hui,HUANG Dawei.DNA barcoding:new approach of biological taxonomy[J].Acta Zoologica Sinica,2004,50(5):852-855.
[3]HEBERT P D N,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B:Biological Sciences,2003,270:313-321.DOI:10.1098/rspb.2002.2218.
[4]HEBERT P D N,RATNASINGHAM S,DEWAARD J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceedings of the Royal Society B:Biological Sciences,2003,270:S96-S99.DOI:10.1098/rsbl.2003.0025.
[5]KWONG S,SRIVATHSAN A,MEIER R.An update on DNA barcoding:low species coverage and numerous unidentified sequences[J].Cladistics,2012,28:639-644.DOI:10.1111/j.1096-0031.2012.00408.
[6]HEBERT P D N,STOECKLE M Y,ZEMLAK T S,et al.Identification of birds through DNA barcodes[J].PLoS Biology,2004,2:1657-1663.DOI:10.1371/journal.pbio.0020312.
[7]WARD R D,ZEMLAK T S,INNES B H,et al.DNA barcoding Australia’s fish species[J].Proceedings of the Royal Society B:Biological Sciences,2005,360:1847-1857.DOI:10.1001/archneurol.2008.523.
[8]HAJIBABAEI M,JANZEN D H,BURNS J M,et al.DNA barcodes distinguish species of tropical Lepidoptera[J].Proceedings of the National Academy of Sciences,USA,2006,103:968-971.DOI:10.1073/pnas.0510466103.
[9]CRAFT K J,PAULS S U,DARROW K,et al.Population genetics of ecological communities with DNA barcodes:an example from New Guinea Lepidoptera[J].Proceedings of the National Academy of Sciences,USA,2010,107:5041-5046.DOI:10.1073/pnas.0913084107.
[10]MURPHY R W,CRAWFORD A J,BAUER A M,et al.Cold Code:the global initiative to DNA barcode amphibians and nonavian reptiles[J].Molecular Ecology Resources,2013,13:161-167.DOI:10.1111/1755-0998.12050.
[11]CHE J,CHEN H M,YANG J X,et al.Universal COI primers for DNA barcoding amphibians[J].Molecular Ecology Resources,2012,12:247-258.DOI:10.1111/j.1755-0998.2011.03090.x.
[12]何锴,王文智,李权,等.DNA条形码技术在小型兽类鉴定中的探索:以甘肃莲花山为例[J].生物多样性,2013,21(2):197-205.
HE Kai,WANG Wenzhi,LI Quan,et al.DNA barcoding in surveys of small mammal community:a case study in Lianhuashan,Gansu Province,China[J].Biodiversity Science,2013,21(2):197-205.
[13]李超伦,王敏晓,程方平,等.DNA条形码及其在海洋浮游动物生态学研究中的应用[J].生物多样性,2011,19(6):805-814.
LI Chaolun,WANG Minxiao,CHENG Fangping,et al.DNA barcoding and its application to marine zooplankton ecology[J].Biodiversity Science,2011,19(6):805-814.
[14]ARDURA A,POLA I G,GINUNO I,et al.Application of barcoding to Amazonian commercial fish labeling[J].Food Research International,2010,43(5):1549-1552.DOI:10.1016/j.foodres.2010.03.016.
[15]WILL K W,MISHLER B D,WHEELER Q D.The perils of DNA barcoding and the need for integrative taxonomy[J].Syst Biol,2005,54(5),844-851.DOI:10.1080/10635150500354878.
[16]MORITZ C.CICERO C,DNA barcoding:Promise and pitfalls[J].Plos Biology,2004,2(10):e354.DOI:10.1371/journal.pbio.0020354.
[17]SPERLING F.DNA Barcoding:Deusex machina[J].Society,2003,45(4):1-4.
[18]World Spider Catalog(2015).World Spider Catalog[J/OL].Version 16.0.Natural History Museum Bern.[2015-01-22],http://wsc.nmbe.ch.
[19]FOLMER O,BLACK M,HOEH W,et al.DNA primer for amplification of mitochondrial cyctorome coxidase subunit I from diverse metazoan invertebrates[J].Mol Mar Biotechnol,1994,3(5):294-199.
[20]THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876-4882.DOI:10.1093/nar/25.24.4876.
[21]TAMURA K,STECHER G,PETERSON D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0.[J].Molecular Biology and Evolution,2013,30:2725-2729.DOI:10.1093/molbev/mst197.
[22]KIMURE M A.Simple method for estimating evolutionary rates of base substitution through comparative studies of nucleotide sequences[J].Journal of Molecular Evolution,1980,16:111-120.DOI:10.1007/BF01731581.
[23]HUELSENBECK J P,RONQUIST F.MrBayes:Bayesian inference of phylogenetic trees[J].Bioinformatics,2001,17:754-755.DOI:10.1093/bioinformatics/17.8.754.
[24]MEYER C P,PAULAY G.DNA barcoding:error rates based on comprehensive sampling[J].PLoS Biology,2005,3(12):e422.DOI:10.1371/journal.pbio.0030422.
[25]PUILLANDRE N,LAMBERT A,BROUILLET S,et al.ABGD,automatic barcode gap discovery for primary species delimitation[J].Mol Ecol,2011,21(8):1864-1877.DOI:10.1111/j.1365-294X.2011.05239.x.
[26]ZHANG Y Y,LI S Q.A spider species complex revealed high cryptic diversity in South China caves[J].Molecular Phylogenetics and Evolution,2014,79:353-358.DOI:10.1016/j.ympev.2014.05.017.
[27]BERNHARD A,JONAS J.Increased samp ling blurs morpho logical and molecular species limits:revision of the Hispanio lan endemic spider genus Tainonia (Araneae:Pholcidae)[J].Astrin Invertebrate Systematics,2009,23:281-300.DOI:10.1071/IS09017.
[28]FRANZINI P Z N.Combined analyses of genetic and morphological data indicate more than one species of Cyrtophora (Araneae:Araneidae) in South Africa[J].Internationnal Journal of Modern Biological Research 2013,1:21-34.
[29]ZHANG J X,MADDISON W P.Molecular phylogeny,divergence times and biogeography of spiders of the subfamily Euophryinae (Araneae:Salticidae)[J].Molecular Phylogenetics and Evolution,2013,68:81-92.DOI:10.1016/j.ympev.
[30]刘勇,宋毓,李晓宇.基于线粒体COI基因的DNA条形码技术在昆虫分子鉴定中的应用[J].植物检疫,2010,2(24):46-50.
[31]宁淑萍,颜海飞,郝刚,等.植物DNA条形码研究进展[J].生物多样性,2008,16(5):417-425.DOI:10.3724/SP.J.1003.2008.08215.
NING Shuping,YAN Haifei,HAO Gang,et al.Current advances of DNA barcoding study in plants[J].Biodiversity Science,2008,16(5):417-425.DOI:10.3724/SP.J.1003.2008.08215.
(责任编辑:赵藏赏)
Molecular identification of crab spiders from Xiaowutai Mountains,China with DNA barcoding
HE Jingchao,HU Lanlan,GUO Chenhui,ZHANG Feng
(College of Life Sciences,Hebei University,Baoding 071002,China)
In order to explore the feasibility of DNA barcoding in the species identification of crab spiders,the COI gene of 110 thomisid samples belonged to 25 species in Xiaowutai Mountains,China,was applied; the phylogenetic trees were reconstructed by neighbor-joining (NJ) and Bayesian Inference (BI) methods,and with ABGD(automatic barcode gap discovery) software to divide the samples.The results showed that the samples belonging to the same species clustered in one monophyly and all species were successfully distinguished by the phylogenetic trees,and the groups automatically defined by ABGD perfectly matched the groups defined by morphological characters,and accorded with phylogenetic trees.We believe DNA barcoding can be used as an effective tool in the molecular identification of crab spiders.
Thomisidae; DNA barcoding; phylogenetic trees; ABGD
10.3969/j.issn.1000-1565.2016.03.011
2015-09-12
科技部科技基础性工作专项基金资助项目(2012FY110803);河北省自然科学基金资助项目(C2014201041)
何静超(1989-),女,河北保定人,河北大学在读硕士研究生.E-mail:hejingchao2014@163.com
张锋(1971-),男,河北蔚县人,河北大学教授,博士生导师,主要从事蛛形动物学研究.
E-mail:zhangfeng@hbu.cn
Q959.2262
A
1000-1565(2016)03-0286-07

