儿童粪便中四环素抗性大肠杆菌的分离与鉴定*
徐先林,欧刚卫
(遵义医学院 生物化学教研室,贵州 遵义 563099)
临床医学研究
儿童粪便中四环素抗性大肠杆菌的分离与鉴定*
徐先林,欧刚卫
(遵义医学院 生物化学教研室,贵州 遵义563099)
目的 分离、鉴定无四环素使用史儿童粪便中四环素抗性菌株。方法 采用含150 μg/mL四环素营养琼脂培养基分区划线法从儿童粪便样本中分离、纯化四环素抗性细菌; 通过观察菌落外观形态特征、16S rRNA基因测序法、生理生化试验、梅里埃全自动微生物分析仪鉴定细菌。结果 从儿童粪便样本中随机分离出20例细菌, 鉴定结果显示均为大肠杆菌。结论 在未接触四环素儿童粪便中大肠杆菌是携带四环素抗性的细菌之一。
儿童;四环素;16S rRNA基因;分离;鉴定
肠道是一个巨大的微生物生态系统, 菌群种类复杂, 由1000到1150种细菌组成[1], 菌群结构稳定对人体健康至关重要。研究表明, 直接或间接使用抗生素都会破坏环境中菌群平衡并致抗性基因出现[2-3]。四环素不良反应和副作用较大, 已多年禁止使用于儿童疾病治疗[4], 但仍作为饲料添加剂以及促生长剂广泛应用于水产、家禽和家畜养殖[5]。四环素具有高度水溶性和低挥发性导致其可以稳定存在于环境[6], 通过食物链等方式进入人体肠道, 造成抗生素及抗性基因蓄积于人体肠道。前期实验研究结果表明, 受试儿童虽无明确四环素使用史, 然而大多数粪便样本检测出四环素抗性基因tet(B)[7]。抗性基因在肠道菌群间广泛传播可能导致菌群稳定结构破坏, 可导致潜在致病菌过分繁殖, 破坏肠粘膜屏障从而引发多种疾病。而对于儿童生长发育的潜在影响仍有待研究。
分离肠道细菌并鉴定抗生素抗性菌株, 对于初步了解肠道内抗性菌株分布情况及探讨抗性基因的携带与传播方式有着重要意义。目前, 关于四环素抗性细菌探讨主要集中在环境土壤或者动物粪便样本, 但对于人体肠道菌群中四环素抗性细菌现状有待研究。我国儿童已经禁止使用四环素多年, 是探讨环境抗生素对儿童肠道菌群抗生素抗性菌及相关抗性基因的分布和携带状态、对肠道菌群的种类和分布以及对儿童健康的影响十分理想的群体。本实验采用四环素选择性培养基分纯并鉴定儿童粪便样本中四环素抗性大肠杆菌, 以探讨儿童肠道菌群中大肠杆菌四环素抗性基因携带状况, 为进一步研究环境抗生素对儿童健康影响提供实验基础。
1 材料与方法
1.1样本遵义市健康儿童粪便样本, 无四环素使用史。粪便采集后, 迅速置于-80 ℃保存,并尽快进行后续实验。
1.2方法
1.2.1四环素抗性细菌分纯取新购置的营养琼脂培养基粉末按说明书配制, 121 ℃灭菌15 min, 待温度降至50 ℃加入四环素盐酸盐溶液, 使四环素终浓度为150 μg/mL[8], 迅速摇匀后倾倒平板, 4 ℃保存备用; 从-80 ℃冰箱中取出粪便样本于室温中解冻, 用灭菌接种环蘸取少许样本采用分区划线法接种于新鲜配制的含四环素营养琼脂平板上, 置于37 ℃培养箱中培养24 h; 随机挑取平板上菌落20个, 分区划线接种于新平板上, 37 ℃培养24 h; 连续传代3次后, 观察并记录各菌落外观特征。
1.2.216S rRNA基因测序鉴定
1.2.2.1引物[9]上游引物(27F): 5′-AGAGTTTGATCCTGGCTCAG-3′,下游引物(1492R): 5′-TACGGTTACCTTGTTACGACTT-3′, 引物由生工生物工程(上海)股份有限公司合成。
1.2.2.216S rRNA基因PCR模板制备用灭菌枪头挑取纯化单菌落菌体少许加入100 μL无菌水中, 涡旋混匀后, 沸水浴10 min, 稍冷后12 000 rpm离心1 min, 上清液作为模板直接用于16S rRNA基因PCR扩增。
1.2.2.3PCR扩增体系上游引物(10 pmol/μL) 1 μL、下游引物(10 pmol/μL) 1 μL、PCR Master Mix (2×) 25 μL、模板(空白对照以灭菌水代替) 5 μL和灭菌水18 μL。
1.2.2.4PCR扩增条件94 ℃预变性5 min,94 ℃ 变性45 s, 56 ℃退火45 s, 72 ℃延伸90 s, 30个循环, 72 ℃延伸10 min;4 ℃保存扩增产物。
1.2.2.5电泳取PCR扩增产物5 μL进行1%琼脂糖凝胶电泳。恒压120 V, 电泳35 min后用凝胶成像系统观察结果。
1.2.2.6测序和比对根据电泳结果将能清晰分辨出目的条带且具有一定亮度的PCR产物送生工生物工程(上海)股份有限公司武汉测序部测序。16S rRNA基因大小约为1 500 bp, 采用双向测序。
将测序结果利用SeqMan软件去除测序图谱两端峰形不规则的序列, 拼接双向测序结果, 查找并修正其中拼接错误; 对于测序失败和双向测序结果不能正确拼接的细菌, 需要再次PCR扩增16S rRNA基因, 重新测序和拼接。
将测序结果进行Blast(Basic Local Alignment Search Tool)在线序列比对分析用作细菌鉴定, 方法参照文献[10]。
1.2.3革兰氏染色、生理生化试验及运动性检测对分离的20株细菌进行革兰氏染色、生理生化试验及运动性检测, 方法参照《常见细菌系统鉴定手册》第二部分[11]。
1.2.4梅里埃VITEK-2全自动微生物分析仪鉴定将培养18~24h的细菌用无菌稀释液(0.45%~0.5% NaCl, pH4.5~7.0) 2.5 mL调节至0.50~0.63个麦氏比浊单位, 连接革兰氏阴性细菌鉴定卡, 上机并按照梅里埃全自动微生物分析仪说明书进行操作, 10h后查看鉴定结果。
2 结果
2.1菌株菌落外观形态特征从含四环素的营养琼脂平板上随机分离20个菌落, 传代3次, 编号为1~20, 各菌株在营养琼脂平板上呈现灰白色, 菌落小, 表面光滑, 边缘整齐的菌落外观形态。
2.216S rRNA测序鉴定结果采用通用引物27F和1492R扩增细菌16S rRNA基因, 扩增片段长度为1 500 bp左右, 特异性好, 可用作测序[10](见图1); 测序图谱采用chromas软件分析, 信号强且无套峰, 测序结果可靠。将双向测序结果利用SeqMan软件去除测序图谱两端峰形不规则序列, 拼接双向测序结果, 查找并修正其中拼接错误, 将各菌株16S rRNA基因约1265 bp序列导入NCBI网站进行Blast在线序列比对, 以同源性高低的顺序从上到下排列, 取最大分值(max score)最高且E值为0的菌株作为鉴定结果[10]。最大分值越大, 同源性越高; E值表示在数据库搜索时与期望值随机匹配的可能性, E=0表示匹配不是随机产生的, E值越小置信度越高[10]。部分16S rRNA基因序列在线比对最大分值最高、相似性大于99%且E值=0的细菌不只一种, 需要结合其它方法得出最终结论, 表1列出了每株细菌Blast比对后按同源性高低排序前两位细菌。
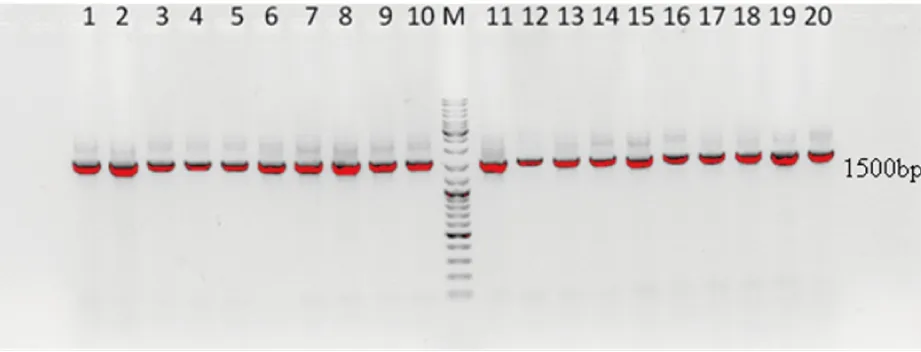
图1 20株细菌16S rRNA基因PCR扩增电泳结果
表120株细菌的16S rRNA基因序列分析鉴定结果
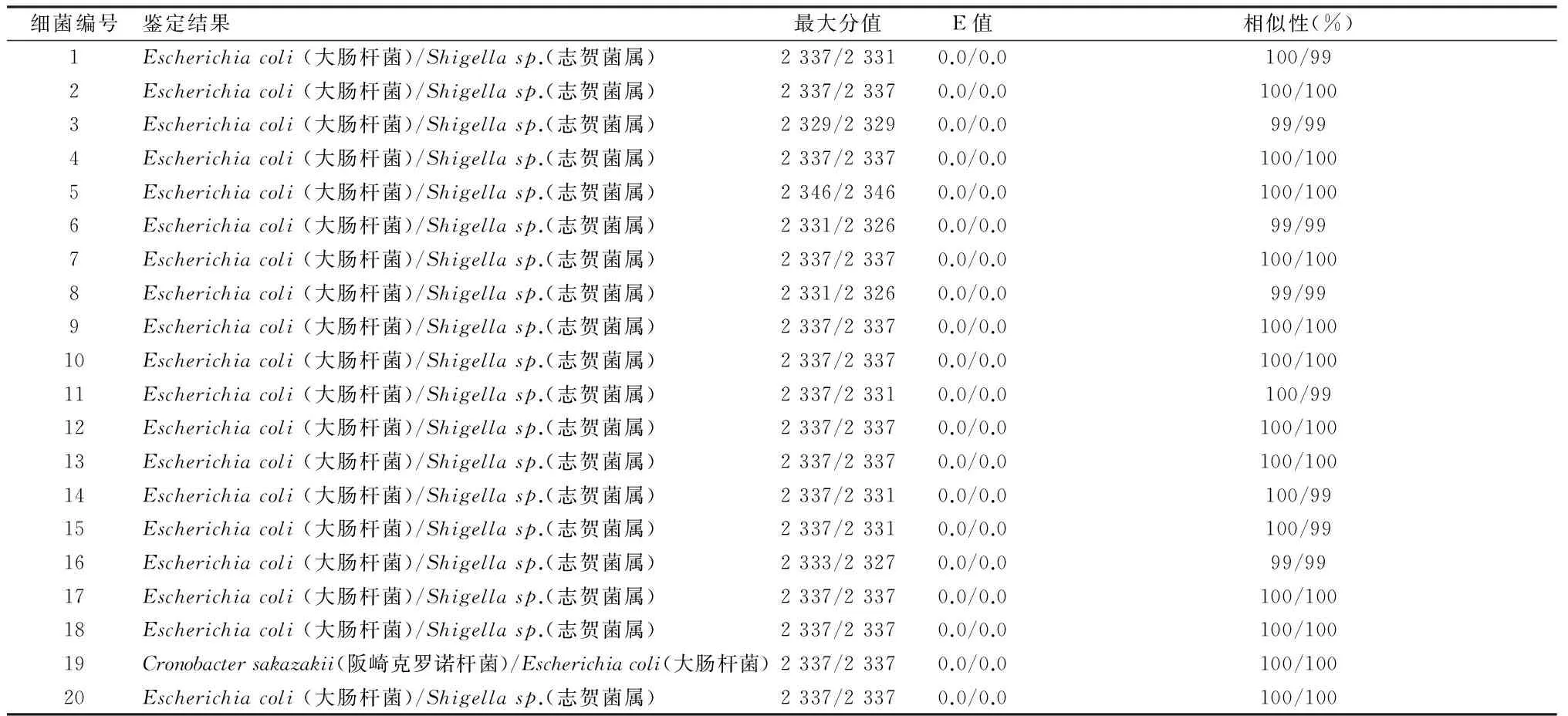
细菌编号鉴定结果最大分值E值相似性(%)1Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23310.0/0.0100/992Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/1003Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2329/23290.0/0.099/994Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/1005Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2346/23460.0/0.0100/1006Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2331/23260.0/0.099/997Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/1008Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2331/23260.0/0.099/999Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/10010Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/10011Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23310.0/0.0100/9912Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/10013Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/10014Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23310.0/0.0100/9915Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23310.0/0.0100/9916Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2333/23270.0/0.099/9917Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/10018Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/10019Cronobactersakazakii(阪崎克罗诺杆菌)/Escherichiacoli(大肠杆菌)2337/23370.0/0.0100/10020Escherichiacoli(大肠杆菌)/Shigellasp.(志贺菌属)2337/23370.0/0.0100/100
2.3革兰氏染色、生理生化试验及细菌运动性试验结果20株细菌均为G-杆菌, 运动性呈阴性, 生理生化试验结果见表2。生理生化试验结果参照《常见细菌系统鉴定手册》[11]和《伯杰氏细菌鉴定手册(第8版)》[12]标准, 所分离细菌与大肠杆菌特征最相符。志贺菌属细菌发酵葡萄糖不产气, 不发酵乳糖; 阪崎克罗诺杆菌甲基红试验阴性、伏普试验阳性(见表2)。
表220株细菌生理生化试验、革兰氏染色、运动性试验结果
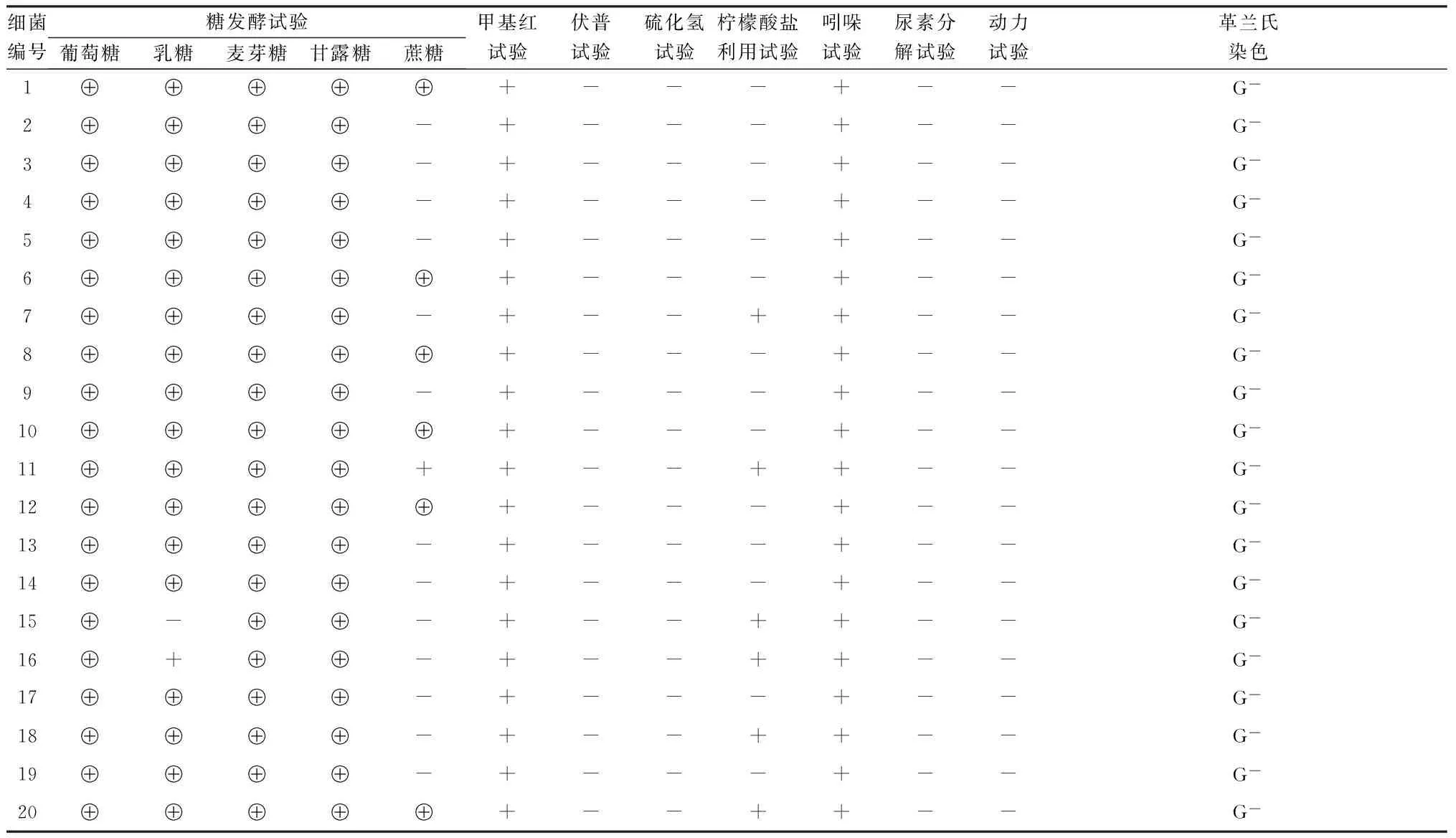
细菌编号糖发酵试验葡萄糖乳糖麦芽糖甘露糖蔗糖甲基红试验伏普试验硫化氢试验柠檬酸盐利用试验吲哚试验尿素分解试验动力试验革兰氏染色1⊕⊕⊕⊕⊕+---+--G-2⊕⊕⊕⊕-+---+--G-3⊕⊕⊕⊕-+---+--G-4⊕⊕⊕⊕-+---+--G-5⊕⊕⊕⊕-+---+--G-6⊕⊕⊕⊕⊕+---+--G-7⊕⊕⊕⊕-+--++--G-8⊕⊕⊕⊕⊕+---+--G-9⊕⊕⊕⊕-+---+--G-10⊕⊕⊕⊕⊕+---+--G-11⊕⊕⊕⊕++--++--G-12⊕⊕⊕⊕⊕+---+--G-13⊕⊕⊕⊕-+---+--G-14⊕⊕⊕⊕-+---+--G-15⊕-⊕⊕-+--++--G-16⊕+⊕⊕-+--++--G-17⊕⊕⊕⊕-+---+--G-18⊕⊕⊕⊕-+--++--G-19⊕⊕⊕⊕-+---+--G-20⊕⊕⊕⊕⊕+--++--G-
+表示阳性; -表示阴性; ⊕表示产气产酸。
2.4梅里埃全自动微生物分析仪鉴定结果采用梅里埃全自动微生物分析仪对20株细菌进行鉴定(见表3), 结果显示20株细菌均为大肠杆菌, 鉴定率(可能性)>86.78%, 置信度为可接受(acceptable identification)至极好(excellent identification)。综合上述结果可以确定该20株细菌都为大肠杆菌。
表3梅里埃VITEK-2全自动微生物分析仪鉴定结果
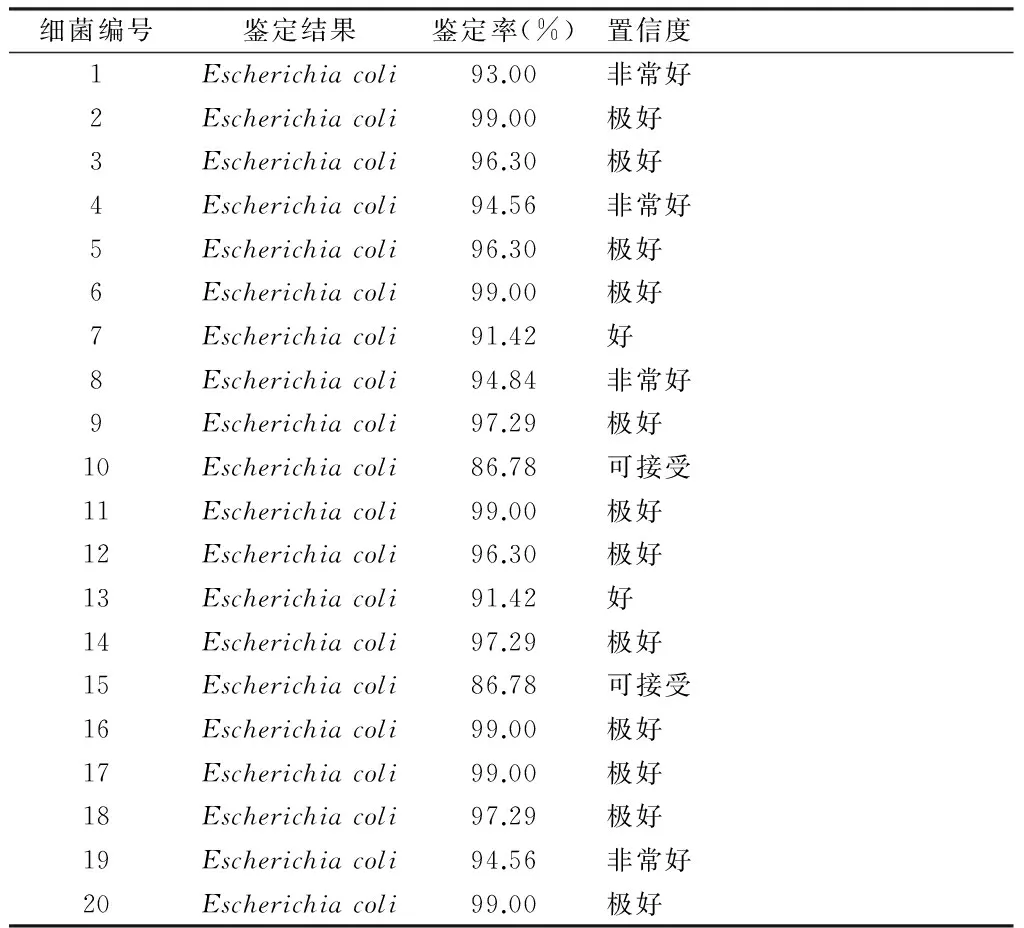
细菌编号鉴定结果鉴定率(%)置信度1Escherichiacoli93.00非常好2Escherichiacoli99.00极好3Escherichiacoli96.30极好4Escherichiacoli94.56非常好5Escherichiacoli96.30极好6Escherichiacoli99.00极好7Escherichiacoli91.42好8Escherichiacoli94.84非常好9Escherichiacoli97.29极好10Escherichiacoli86.78可接受11Escherichiacoli99.00极好12Escherichiacoli96.30极好13Escherichiacoli91.42好14Escherichiacoli97.29极好15Escherichiacoli86.78可接受16Escherichiacoli99.00极好17Escherichiacoli99.00极好18Escherichiacoli97.29极好19Escherichiacoli94.56非常好20Escherichiacoli99.00极好

图2 细菌进化树分析
2.516S rRNA基因序列同源性分析从NCBI中查找大肠杆菌(E.coli, gi:174375)、志贺菌属(Shigellasp. CH-35, gi:928241852)和阪崎克罗诺杆菌(Cronobactersakazakiistrain E601, gi: 118498 806) 16S rRNA基因序列, 并与20株细菌16S rRNA基因进行序列比对, clustalx 1.83软件分析结果显示它们具有相似的保守区和多态区, 序列相似度高。采用MEGA5.0构建系统发育树(见图2), 分离的20株大肠杆菌(编号1~20)分布在2个类群; 大肠杆菌、志贺菌和阪崎克罗诺杆菌进化关系较近, 位于同一个分类群中。
3 讨论
抗生素过度使用导致肠道菌群结构改变以及抗性基因增多已经引起全球高度关注。人体肠道细菌种类繁多且密度大, 是抗生素抗性基因储存和转移的良好场所, 有利于抗性基因在肠道细菌间水平传播; 健康的肠道菌群主要由有益或者无害细菌组成, 在肠腔形成一个天然保护屏障抵御病原菌入侵, 保护和促进肠上皮细胞发育, 产生短链脂肪酸等, 对人体健康发挥着重要作用。尤其在儿童阶段, 肠道菌群促进机体免疫系统发育的作用与远期健康密切相关。抗性基因转入致病菌或条件致病菌并大量繁殖极易导致稳定的菌群结构破坏, 引发肥胖、过敏等多种疾病。菌群中抗性基因的出现除了源于直接的抗生素筛选压力外, 还可能来自于环境中[7,13]。四环素是一种广谱抗生素, 已多年不使用于儿童群体, 探讨其肠道菌群对抗生素抗性基因的携带和传播具有特殊意义。
细菌16S rRNA基因序列由保守区和可变区组成, 两者互相交错排列, 采用通用引物27F和1492R扩增产物包含16S rRNA基因所有的可变区, 适用于亲缘关系不同的细菌鉴定。在未直接接触四环素的儿童群体中, 用16S rRNA基因检测方法发现肠道菌群中普遍存在四环素抗性基因分布[7], 但四环素抗性基因存在于何种细菌仍不清楚。本实验通过四环素抗性培养筛选, 其抗性菌株经16S rRNA基因全序列测定后通过Blast比对尚不能得到精确鉴定细菌, 其原因在于大肠杆菌与志贺菌或大肠杆菌与阪崎克罗诺杆菌无法区分(见表1)。有研究表明, 志贺菌未被排除在大肠杆菌种系发育群之外, 它们可能具有相同的进化起源[14]; 而从NCBI中查找大肠杆菌、志贺菌和阪崎克罗诺杆菌16S rRNA基因序列, 经相似性比对分析显示它们序列同源性高, 在构建的系统发育树中位于同一个分类群(见图2)。基于大肠杆菌、志贺菌和阪崎克罗诺杆菌16S rRNA基因序列相似度极高, 单纯的16S rRNA基因测序法鉴定存在不确定性, 而且细菌16S rRNA基因多拷贝现象[15-16]、水平转移问题[17]、异化现象[18]及PCR扩增等实验操作过程中的诸多问题[18]都可能影响鉴定结果, 还需其他方法补充或验证鉴定结果。
本研究首先通过16S rRNA测序法确定未知细菌与数据库中细菌的相似性, 初步判断细菌种类; 再以生理生化试验结果加以验证; 最后结合全自动微生物分析仪得出准确鉴定结果。综合基因型与表型分析, 避免了单一方法错误判读, 结果可靠, 20株细菌鉴定均为大肠杆菌。肠道菌群复杂, 厌氧及兼性厌氧菌居多[19], 在目前条件下, 能够通过培养分离的肠道微生物只占极少数, 且不同培养条件以及不同培养基分离到的细菌不尽相同[20]。当采用脑心浸液琼脂、血平板等不同培养基, 运用厌氧罐及延长培养时间可培养出更多细菌, 如肠球菌、 双歧杆菌等(结果未展示)。实验中以四环素选择性培养基分离的20株大肠杆菌, 经四环素K-B纸片法药敏试验确定均为四环素抗性菌(结果未展示), 有利于深入研究抗性大肠杆菌携带四环素抗性基因现状及传播方式。研究结果表明, 在未直接接触四环素的儿童肠道菌群中,抗四环素抗性基因存在于大肠杆菌中, 但其抗性基因的种类、携带方式、传播以及对儿童健康的影响仍有待研究。
[1] Eckburg P B, Bik E M, Bernstein C N, et al. Diversity of the human intestinal microbial flora[J]. Science, 2005, 308(5728): 1635-1638.
[2] Jernberg C, Löfmark S, Edlund C, et al. Long-term impacts of antibiotic exposure on the human intestinal microbiota[J]. Microbiology, 2010, 156(11): 3216-3223.
[3] Mitsou E K, Kirtzalidou E, Pramateftaki P, et al. Antibiotic resistance in faecal microbiota of Greek healthy infants[J]. Benef Microbes, 2010, 1(3): 297-306.
[4] Sánchez A R, Rogers R S, Sheridan P J. Tetracycline and other tetracycline-derivative staining of the teeth and oral cavity[J]. Int J Dermatol, 2004, 43(10): 709-715.
[5] Kim K R, Owens G, Kwon S I, et al. Occurrence and environmental fate of veterinary antibiotics in the terrestrial environment[J]. Water, Air, & Soil Pollution, 2011, 214(1): 163-174.
[6] Daghrir R, Drogui P. Tetracycline antibiotics in the environment: a review[J]. Environ Chem Lett, 2013, 11(3): 209-227.
[7] 文雯, 徐先林, 欧刚卫. 多种抗生素抗性基因在儿童肠道菌群的携带情况[J]. 遵义医学院学报, 2013, 36(2): 113-115.
[8] Narisawa N, Haruta S, Arai H, et al. Coexistence of antibiotic-producing and antibiotic-sensitive bacteria in biofilms is mediated by resistant bacteria[J]. Appl Environ Microbiol, 2008, 74(12): 3887-3894.
[9] Moreno C, Romero J, Espejo R T. Polymorphism in repeated 16S rRNA genes is a common property of type strains and environmental isolates of the genus Vibrio[J]. Microbiology, 2002, 148(Pt 4):1233-1239.
[10] 朱飞舟, 陈利玉, 陈汉春. 16S rRNA基因序列分析法鉴定病原细菌[J]. 中南大学学报: 医学版, 2013, 38(10): 1035-1041.
[11] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 66-398.
[12] 布坎南, 吉本斯. 伯杰细菌鉴定手册[M]. 中国科学院微生物研究所《伯杰细菌鉴定手册》翻译组,译. 第8版. 北京: 科学出版社, 1984: 382-462.
[13] Mitsou E K, Kirtzalidou E, Pramateftaki P, et al. Antibiotic resistance in faecal microbiota of Greek healthy infants[J]. Benef microbes, 2010, 1(3): 297-306.
[14] 王林, 徐建国. 志贺菌与大肠杆菌进化关系的研究进展[J]. 疾病监测, 2005, 20(5): 275-277.
[15] Větrovsky T, Baldrian P. The variability of the 16S rRNA gene in bacterial genomes and its consequences for bacterial community analyses[J]. Plos One, 2013, 8(2): e57923.
[16] 阎永伟, 张德民. 原核生物16S rRNA基因多重拷贝及其序列异化[J]. 生物学杂志, 2013, 30(4): 63-66.
[17] Tian R M, Cai L, Zhang W P, et al. Rare events of intragenus and intraspecies horizontal transfer of the 16S rRNA gene[J]. Genome Biol Evol, 2015, 7(8): 2310-2320.
[18] 刘驰, 李家宝, 芮俊鹏, 等. 16S rRNA基因在微生物生态学中的应用[J]. 生态学报, 2015, 35(9): 2769-2788.
[19] O'Hara A M, Shanahan F. The gut flora as a forgotten organ[J]. Embo Rep, 2006, 7(7): 688-693.
[20] O'Sullivan D J. Methods for analysis of the intestinal microflora[J]. Curr Issues Intest Microbiol, 2000, 1(2): 39-50.
[收稿2016-05-04;修回2016-06-12]
(编辑:王福军)
Isolation and identification of tetracycline resistantE.coliin children intestinal microbiota
XuXianlin,OuGangwei
(Department of Biochemistry,Zunyi Medical University, Zunyi Guizhou 563099,China)
Objective To isolate and identify the tetracycline resistant strains in fecal of children without tetracycline treatment.Methods The strains were cultivated and isolated by nutrient agar plates with tetracycline (150 μg/mL). The bacterial strains were identified by the characteristics of bacterial colonies, 16S rRNA gene sequencing, bacterial physiological and biochemical tests and BioMerienx fully-automatic germ analysis system respectively.Results Twenty strains ofEscherichiacoliwere isolated and identified.ConclusionE.coliis one of the tetracycline resistant bacteria in fecal samples of children which without tetracycline treatment.
children; tetracycline; 16S rRNA gene; isolation; identification
贵州省科技厅基金资助项目(NO:黔科合J字[2009]2301);遵义医学院博士启动金资助项目(NO:2010F-391)。
欧刚卫,男,博士,教授,硕士生导师,研究方向:肠道菌群与免疫,E-mail:gangwei.ou@zmc.edu.cn。
R372
A
1000-2715(2016)04-0387-05

