小麦5DL上基于SNP序列的新SSR标记的开发
陈芳,李文静,邓志英,田纪春,陈建省
作物生物学国家重点实验室;山东省作物生物学重点实验室、小麦品质及分子育种研究室,山东泰安271018
小麦5DL上基于SNP序列的新SSR标记的开发
陈芳,李文静,邓志英*,田纪春*,陈建省
作物生物学国家重点实验室;山东省作物生物学重点实验室、小麦品质及分子育种研究室,山东泰安271018
本文旨在利用粗山羊草的物理图谱及其基因组序列数据库开发普通小麦5DL上Xbarc320-Xwmc215区段与SNP紧密连锁的SSR标记(SNP-SSR)。对AT5D4910-AT5D5010之间的90个SNP位点进行了序列延伸和本地Blast,利用SSRHunter软件查找SNP位点附近的SSR位点,并利用Primer5设计引物。结果共发现109个SSR位点。对其中的72个位点设计引物,得到了47个5DL上Xbarc320-Xwmc215区段的SNP-SSR标记。对新开发的标记,利用22个小麦品种及人工合成小麦材料、中国春缺体四体及DH群体进行了有效性、多态性的检测及染色体定位。结果表明,这些标记均能扩增出稳定清晰的带型,并且均定位与小麦5D染色体上;其中四个标记Xtdc11、Xtdc31、Xtdc38、Xtdc44整合到了豫麦57与花培3号群体的5DL图谱上。
SNP-SSR;标记开发;小麦5D染色体
小麦A、D基因组测序工作的初步完成[1,2]及D基因组物理图谱的构建[3]为开发物理位置确定的SSR标记提供了帮助。小麦上很多重要的性状都定位在5DL上Xbarc320-Xwmc215区段附近,可知该区段存在十分重要的基因。因此,开发此区段的标记对进一步精细定位及克隆[4,5]、种质资源多样性检测[6-8]和分子辅助育种[9,10]等具有重要的作用和意义。
小麦是异源六倍体,基因组结构复杂,每个基因组上存在的分子标记的密度也有较大差异。通过前人构建的分子遗传图谱发现A、B基因组上的分子标记较多,而D基因组各个染色体上的标记较少,尤其是4D和5D染色体[11-15],这也为这些染色体上控制重要性状的基因的克隆带来了较大的困难。因此,开发4D和5D染色体上新的分子标记不仅能够加密饱和分子遗传图谱,而且对克隆控制重要性状(如矮败不育性状、抽穗期等性状)的基因奠定良好的基础。在各类分子标记中,基于PCR的SSR (Simple sequence repeat)标记极其丰富,且广泛分布于基因组中。因具有超变异性及位点专一性、共显性和多等位基因等优越性得到广泛应用[16,17]。但由于受小麦基因组测序困难的限制[18],以往开发的SSR标记只能计算出遗传位置,没有确定的物理位置,这不利于基因的精细定位及克隆。本课题组对有关主要农艺性状、产量、品质等性状的QTL定位发现[19-21],Xbarc320-Xwmc215染色体区段附近存在控制多个重要性状的“QTL簇”。Sang Y等发现关于维管束的主效QTL位点qTVB-5D、qLVB-5D、qL/S-5D都在标记Xwmc215-Xbarc345之间[19];张坤普等在Xwmc215-Xdm63之间定位到关于籽粒产量、穗粒数、总小穗数、小穗着生密度的主效QTL,而在Xbarc320-Xwmc215之间定位到可育小穗数的QTL[20];Zhao L等在Xbarc320-Xwmc215之间定位到关于籽粒和面粉蛋白质含量的QTL。其他研究者也发现了这种现象,瑶琴等在Vrn-D1附近检测到关于抽穗期、株高、穗粒数的QTL位点[22];Pestsova EG等在5DL标记Xgwm292及Xgwm982附近定位到关于开花期、小穗数和粒重的QTL[23];Wang G等在Xwmc215-Xcfd29之间定位到关于籽粒硬度、容重、籽粒蛋白含量的QTL[24];尤其是Zhang K定位到的抽穗期主效QTL-Qhd5D,其贡献率最高达53.19%[25]。但是,已定位的这些主效QTL还未被精细定位及克隆。因此,本研究利用贾继增等[2]发表的D基因组序列和罗明成等[3]构建的D基因组物理图谱及网站http://wheat-urgi.versailles.inra.fr/Seq-Repository/BLAS开发5DL上Xbarc320-Xwmc215区段附近基于SNP序列新的SSR标记,一方面可为此区段的遗传图谱的加密提供新的分子标记,另一方为该区段附近主效QTL的精细定位和克隆奠定基础。
1 材料与方法
1.1序列的来源
根据Xbarc320-Xwmc215在遗传图谱上的位置[26]确定此区段在物理图谱(http://probes.pw.usda.gov/ WheatDMarker/)上的大体位置为AT5D4910-AT5D5010(区段内的Contig序列由美国戴维斯分校罗明成教授提供),此区段有90个SNP标记,用这些SNP位点的延伸序列,约500 bp,对D基因组测序数据库[2]进行本地blast或网站http://wheat-urgi.versailles.inra.fr/Seq-Repository/BLAST提供的小麦基因组序列数据库进行比对,根据相似度≥99%的条件,得到几十kb的序列片段。以Xtdc38为例,用物理图谱上的SNP位点AT5D4993的Contig对建立的D基因组数据库进行Blast,找到相匹配的序列是KD546761[2]。
1.2SSR位点的查找
利用SSRHunter软件查找二、三、四核苷酸3种类型的SSR,识别标准为:重复序列长度≥20 bp,即重复次数分别大于或等于10、7、5。对完全型、不完全型、复合型三种SSR进行查找,获得SSR位点。
1.3SSR引物设计
利用Primer 5.0软件根据SSR的侧翼区域设计引物。引物设计的主要参数为:SSR序列的开始和结束位置分别距5′和3′端不少于20 bp;引物长度18~25 bp;引物GC含量40%~60%;退火温度Tm值50~65℃,而且上游和下游引物的Tm值相差不大于5℃;PCR扩增产物长度100~400 bp;得分80分以上,尽量避免引物二级结构的出现;引物设计完成以后由上海英潍捷基贸易有限公司合成。引物命名为Xtdc加序号,如Xtdc1。
1.4试验材料
1.4.1普通小麦品种(系)材料山农19、山农20、衡6632、衡6503、衡4568、衡观76、科农1006、科农2009、郑麦0856、郑麦7698、西农529、西农157、泛7030、10繁27、花培3号、豫麦57。
1.4.2人工合成小麦材料SDAU1、SDAU2、SDAU3、SDAU4、SDAU5。
1.4.3中国春缺体四体材料N5A-T5B、N5B-T5A、N5D-T5A。
1.4.4DH群体(亲本为花培3号和豫麦57)共168个系。
1.5DNA提取、SSR扩增和电泳
采用CTAB法提取基因组DNA,用全式金公司的2×EasyTaq PCR SuperM ix进行PCR扩增,产物用8%聚丙烯酰胺凝胶电泳,0.2%硝酸银染色检测。
1.6遗传图谱构建
在花培3号和豫麦57之间有多态性的新SSR引物分别在DH群体的168个株系的DNA中进行PCR扩增,记录基因型。利用软件Mapmaker/Exp3.0的“try”命令将其整合到DH群体的5DL区段的分子标记遗传连锁图谱上,设定LOD≥3.0。用MapChart软件绘制图谱。
2 结果
2.1SSR的分布特征
对AT5D4910-AT5D5010的90条SNP延伸序列blast,搜索SSR位点,共发现109个SSR位点。其中二、三、四核苷酸重复基元各有67个,34个,8个,各占61.47%,31.19%,7.34%。
在109个SSR中二核苷酸基元TC/AG或CT/GA出现频率最高,共37个,占SSR总数的33.94%。AC/GT或CA/TG共17个,占15.60%。AT/TA(占11%)也有较高的出现频率;三核苷酸基元TTC/AAG出现频率最高,占SSR总数的11.93%;其次是CCT/GGA、CAT/GTA、CGG/GCC,出现频率分别为3.67%,3.67%,2.75%,其他类型出现频率较低;四核苷酸基元类型出现的频率均较低(表1)。
对于重复次数,二核苷酸基元的重复次数类型多、跨度大。其中GA/CT或AG/TC的重复次数类型最多,跨度最大,为16种,从重复10次到50次;其次是AC/TG或CA/GT,重复次数类型为12种,重复11次到38次;AT/TA或TA/AT也有较大的重复次数跨度,有9种重复类型,重复次数为10~30次。三核苷酸基元的重复次数类型和跨度小于二核苷酸。其中TTC/AAG或CTT/GAA有10种重复类型,重复次数8~38次,重复次数类型最多,跨度最大;其次是CCT/GGA,CAT/GTA(表1)。
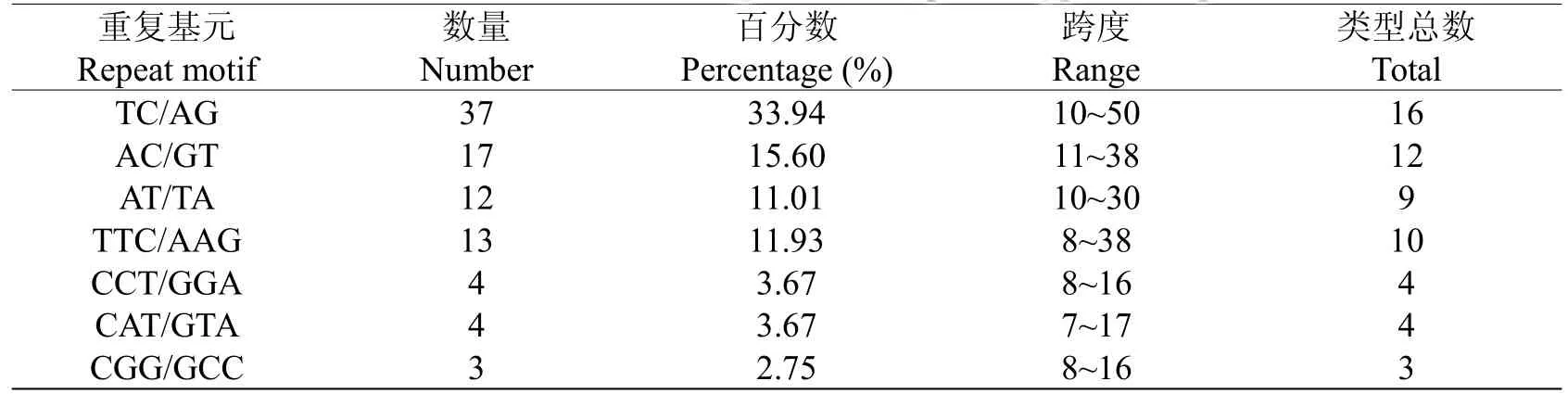
表1 主要重复基元类型及跨度分布Table1Distribution of SSR according tomotif sequence typeand repeatnumber
2.2SNP-SSR引物设计及其有效性
将发现的109个SNP-SSR位点,用Primer5对其中的72个位点设计合成了引物(因有些SSR引物得分较低,所以未设计引物)。对22个不同的小麦品种及个人工合成六倍体材料的DNA进行PCR扩增,并用8%的PAGE胶对扩增产物进行了电泳检测分析,发现47对引物对可以扩增出稳定清晰的带型(表2)。同时,用3个中国春缺体材料(N5A-T5B、N5B-T5A、N5D-T5A)验证SNP-SSR所在染色体的位置,发现这些新SNP-SSR标记均在5D染色体上有扩增(图1)。
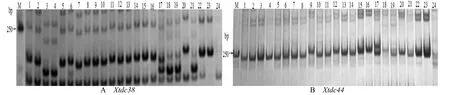
图1 SNP-SSR引物Xtdc38和Xtdc44 PCR扩增产物结果Fig.1 The PCR amp lified bandsof the SNP-SSR primer pairs Xtdc38 and Xtdc44
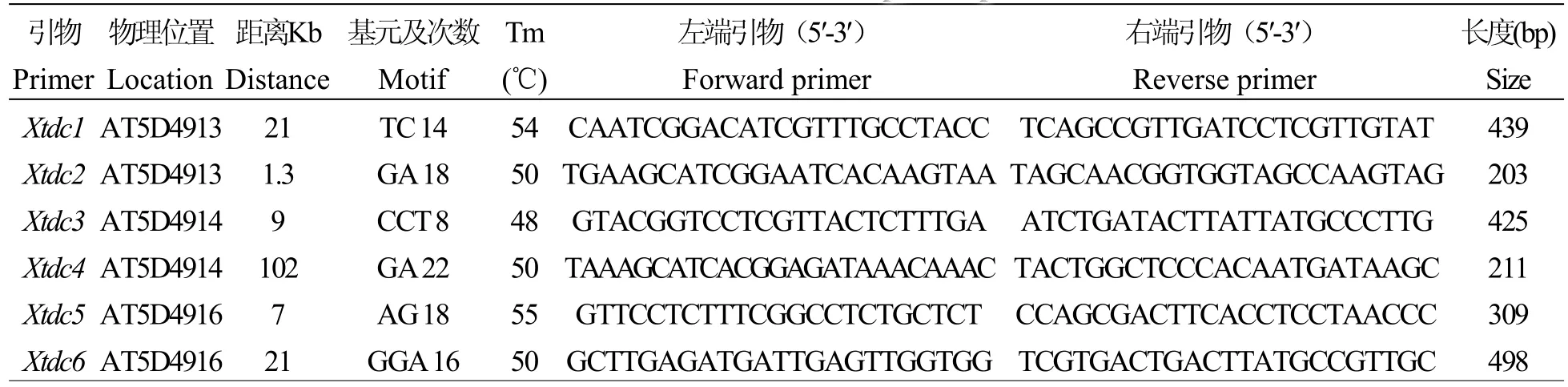
表2 47个小麦新SNP-SSR引物Table 2 47wheat SNP-SSR primer pairs
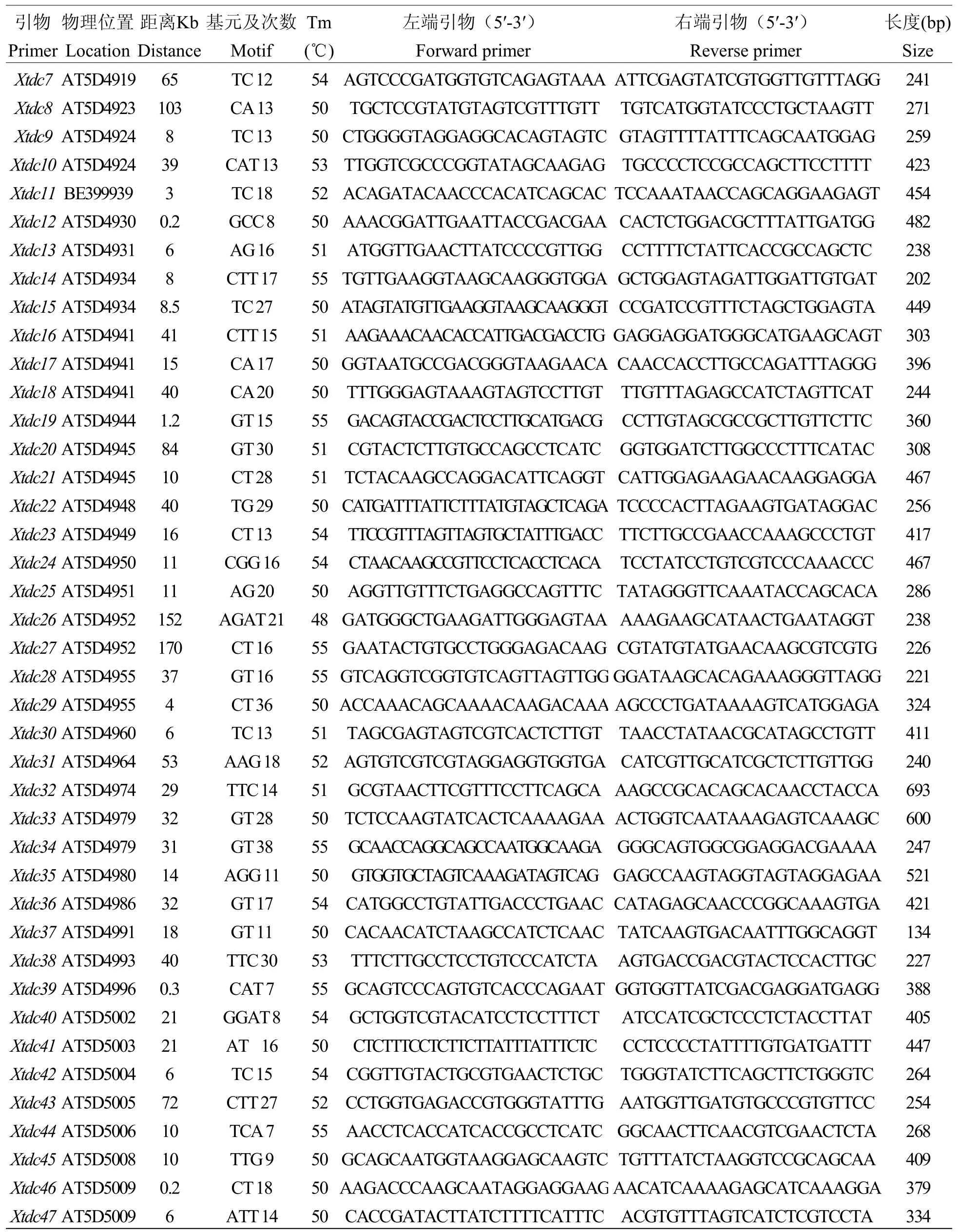
物理位置指与新SSR标记紧密连锁的SNP标记的位置。距离Kb指新标记与对应的SNP标记之间的距离。Location is the location of SNP linked new SSRmarker,Distance is distance between the new SSRmarker and corresponding SNP.
2.3遗传作图
用豫麦57与花培3号构建的含有168个家系的DH群体对47对新设计的引物进行多态性检测,其中有4个(Xtdc11、Xtdc31、Xtdc38、Xtdc44)(图2)在豫麦57与花培3号中存在多态性,将其整合到本实验室已构建遗传连锁图谱[26]的5DL染色体上(图3)。
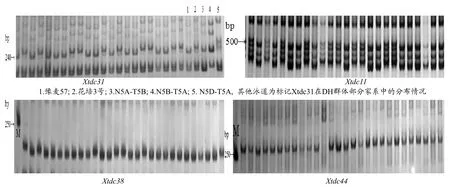
图2 四个SNP-SSR新标记在DH群体部分家系中的分布Fig.2 The resultsof PCR amp lification in some linesof DH population using four SNP-SSR new markers
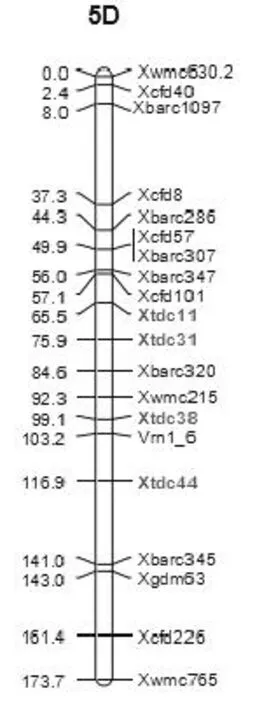
图3 新SNP-SSR标记在5D遗传图谱上的位置Fig.3 Location of SNP-SSRmakers in geneticmap of 5D
3 讨论
小麦的基因组研究远远落后于水稻、玉米、大麦等作物,因为小麦是异源六倍体,基因组结构复杂。近年来虽然构建了大量的高密度遗传图谱,但由于基因组测序工作的困难,大部分标记的物理位置并不确定,小麦重要基因的精细定位及克隆进展缓慢。2013年我国完成了小麦A、D基因组的测序工作[1,2],同时罗明成等构建了小麦D基因组物理草图[3]。尽管目前小麦基因组数据库还不够完善,但这些成果为开发物理位置确定的SSR标记提供了很大的帮助。之前因EST和cDNA大规模测序的开展,开发了大量的EST-SSR引物[27-29]。但是这些EST-SSR标记没有具体的物理位置,只能根据中国春缺体或遗传群体来确定它们的大体位置。本研究根据罗明成等建立的小麦D基因组物理图谱,结合小麦目前的基因组序列数据库,得到47个5DL上区段Xbarc320-Xwmc215附近与SNP紧密连锁的SSR标记,命名为SNP-SSR标记,并用中国春缺体验证了它们的位置。由于这些新的SSR标记与罗明成等所建物理图谱上的SNP标记紧密连锁,具有相应的物理位置,因此,为此区段内重要基因的精细定位及克隆和分子育种等提供了方便。
关于SSR类型的出现频率,前人有报道EST中SSR的三核苷酸重复出现频率最高,而本研究中二核苷酸和三核苷酸重复出现频率最高,其中二核苷酸高于三核苷酸,这可能是因为SSR标记设计时的序列来源不同;其次本文查找SSR的标准为重复序列长度≥20 bp,而前人的标准为18 bp,且18 bp长度的重复序列中三核苷酸重复出现频率高于二核苷酸重复。在水稻、玉米、大豆、高粱中二核苷酸重复基元出现频率最多的都是GA/CT[30];在大麦中二核苷酸重复基元AG/TC和AC/TG出现频率较高[31]。在水稻、玉米、大麦中三核苷酸CCG/GGC和AGG/TCC出现频率较高[30,31]。本研究发现的109 个SSR位点中,二核苷酸重复出现频率最高,其中以GA/CT或AG/TC居多,占SSR总数的33.94%;此外,AC/GT或CA/GT也有较高频率。三核苷酸基元TTC/AAG出现频率最高,占SSR总数的11.93%。这与前人的研究结果一致。
NicotN等开发的小麦EST-SSR标记为二核苷酸SSR多态性较高[32]。本研究中二核苷酸基元的重复次数类型多、跨度大,其中GA/CT或AG/TC的重复次数及类型最多,跨度最大,为16种,从重复10次到50次。与前人结果相一致。
由于SSR标记使用方便、多态性丰富和位点专一等优势,前人已将SSR标记广泛应用于构建小麦遗传连锁图谱。Chu等将410个SSR标记定位在小麦DH群体的遗传图谱上[18];Elangovan等用202个SSR标记构建了一个小麦RIL群体的遗传图谱[19];Janice等用由402个家系构成的DH群体,构建了一个遗传图谱,包括268个SSR标记[21];Li等构建了含有250个SSR标记的小麦遗传图谱,并对面包加工品质进行了QTL定位。但是这些SSR标记的位置是根据遗传群体的分子数据计算出来的遗传位置,其物理位置并不清楚,甚至在不同的图谱中同一标记的顺序不同。而本研究开发的47个SNP-SSR标记具有确切的物理位置,且发现其中有4个SNP-SSR标记在DH群体的亲本豫麦57与花培3号间存在多态性,将其定位在5DL上,其中Xtdc31和Xtdc38分别在Xbarc320的上方和Xwmc215的下方,将Xbarc320 和Xwmc215标记卡在物理图谱AT5D4964和AT5D4993之间,物理距离为12 Mb,确定了Xbarc320-Xwmc215间的基因组序列,为进一步开发此区段内其他类型的标记,如CAPS、dCAPS、STS标记,提供相应的基因组序列依据。
本研究公开了利用SNP开发与SNP紧密连锁的SNP-SSR分子标记的方法,该方法能够快速大量的筛选与SNP紧密连锁的SNP-SSR分子标记,对丰富染色体分子标记的多态性、构建高密度的遗传图谱及其克隆控制重要性状如抽穗期的基因具有重要的意义。
通过本研究所述方法获得的与SNP紧密连锁的SNP-SSR分子标记可应用于品种多态性的筛选、高密度遗传图谱的构建及重要产量和品质性状的基因的精细定位和克隆,能够极大缩短研究周期,加快速度,降低成本,具有操作简单,成本低廉,周期短的优点,适于推广应用,为控制小麦重要性状基因的克隆提供了技术支持,提高了效率和质量。
4 结论
开发了47个5DL上区段Xbarc320-Xwmc215附近新的SNP-SSR标记,将其中4个标记Xtdc11、Xtdc31、Xtdc38和Xtdc44整合到已构建的遗传连锁图谱上;初步确定了区段Xbarc320-Xwmc215物理位置在AT5D4964-AT5D4993之间,其物理距离为12Mb,为该区段内主要基因的精细定位和克隆奠定了基础。
致谢:小麦人工合成六倍体材料及中国春缺体DNA由付道林教授提供,本地blastn的安装及使用由倪飞博士指导帮助,再此特表感谢。
[1]Ling HQ,Zhao HC,Liu DC.Draftgenomeof thewheatA-genomeprogenitor Triticum urartu[J].Nature Letter,2013,496:87-90
[2]Jia JZ,Zhao HC,Kong XY.Aegilops tauschii draftgenome sequence reveals a gene repertoire forwheatadaptation[J]. Nature Letter,2013,496:91-95
[3]Luo MC,Yong QG,Frank MY.A 4-gigabase physicalmap unlocks the structure and evolution of the complex genome of Aegilops tauschii,thewheatD-genome progenitor[J].PNAS,2013,110(19):7940-7945
[4]LiSS,Jia JZ,WeiXY.A intervarietalgeneticmapandQTLanalysisforyield traitsinwheat[J].MolecularBreeding,2007,20:167-178
[5]Hao YF,Liu AF,Wang YH.A new allele of Pm4 located on chromosome 2AL in wheat[J].Theoretical and Applied Genetics,2008,117:1205-1212
[6]Reif JC,Zhang P,Dreisigacker S.Wheatgenetic diversity trends during domestication and breeding[J].Theoreticaland Applied Genetics,2005,110:859-864
[7]Wang HY,Wang XE,Chen PD.Assessment of genetic diversity of Yunnan,Tibetan,and Xinjiang wheat using SSR markers[J].Journalof Geneticsand Genomics,2007,34:623-633
[8]Gupta PK,Rustgi S,Sharma S.Transferable EST-SSRmarkers for the study of polymorphism and genetic diversity in bread wheat[J].MolecularGeneticsand Genomics,2003,270:315-323
[9]John D,William AB,Gene DL.A comparison of marker-assisted and phenotypic selection for high grain protein content in springwheat[J].Euphytica,2006,152:117-134
[10]Huang XQ,Cöster H,Ganal MW.Advanced backcross QTL analysis for the identification of quantitative trait loci alleles from w ild relatives ofwheat(Triticum aestivum L.)[J].Theorand ApplGenet,2003,106:1379-1389
[11]Chu CG,Xu SS,Friesen J.Whole genomemapping in awheatdoubled haploid population using SSRsand TRAPsand the identification of QTL foragronomic traits[J].MolBreeding,2008,22:251-166
[12]Elangovan M,RaiR,Dholakia BB.Molecular geneticmapping of quantitative trait lociassociated w ith loaf volume in hexapoid wheat(Triticum aestivum)[J].Journalof Cereal Science,2008,47:587-598
[13]Wang RX,Hai L,Zhang XY.QTLmapping for grain filling rate and yield-related traits in RILs of the Chinese w inter wheatpopulation Heshangmai×YU8679[J].Theor ApplGenet,2009,118:313-325
[14]Janice L,Cuthbert D,Somers J.Molecularmapping of quantitative trait loci for yield and yield components in spring wheat(Triticum aestivum L.)[J].TheorApplGenet,2008,117:595-608
[15]Li Y,Song Y,Zhou R.Detection of QTLs for bread-making quality in wheat using a recombinant inbred line population[J].PlantBreeding,2009,128:235-243
[16]PowellW,Machray GC,Provan J.Polymorphism revealed by simple sequence repeats[J].Trends in Plant Science,1996,1:215-222
[17]GuptaPK,Balyan HS,SharmaPC.M icrosatellitesin plants:Anew classofmolecularmarkers[J].CurrentScience,1996,70:45-54
[18]Rachel B,Manuel S,Matthias P.Analysis of the bread wheat genome using whole-genome shotgun sequencing[J]. Nature,2012,419:705-710
[19]Sang Y,Deng ZY,Tian JC.QTL for the vascular bundle system of the uppermost internode using a doubled haploid population of two elite Chinesewheatcultivars[J].PlantBreeding,2010,129:605-610
[20]张坤普,徐宪斌,田纪春.小麦籽粒产量及穗部相关性状的QTL定位[J].作物学报,2009,35(2):270-278
[21]Zhao L,Zhang KP,Tian JC.A comparison of grain protein content QTLs and flour protein content QTLs across environments in cultivated wheat[J].Euphytica,2010,174:325-335
[22]姚琴,周荣华,潘昱名,等.小麦品种偃展1号与品系早穗30重组自交系群体遗传连锁图谱构建及重要农艺性状的QTL分析[J].中国农业科学,2010,43(20):4130-4139
[23]Pestsova EG,Börner A,Röder MS.Development and QTL assessment of Triticum aestivum-Aegilops tauschii introgression lines[J].TheorApplGenet,2006,112:634-647
[24]Wang G,Leonard JM,Ross AS.Identification of genetic factors controlling kernel hardness and related traits in a recombinant inbred population derived from a soft 3‘extra-soft'wheat(Triticum aestivum L.)cross[J].Theor Appl Genet,2012,124:207-221
[25]Zhang K,Tian J,Zhao L.Detection of quantitative trait loci for heading date based on the doubled haploid progeny of two elite Chinesewheatcultivars[J].Genetica,2009,135:257-265
[26]Zang KP,Zhao L,Tian JC.A genetic map constructed using a doubled haploid population derived from two elite chinese commonwheatvarieties[J].Journalof Integrative PlantBiology,2008,50(8):941-950
[27]Eujayl I,Sorrells ME,Baum M,et al.Isolation of EST-derived m icrosatellite markers for genotyping the A and B genomesofwheat[J].Theorand ApplGenet,2002,104:399-407
[28]Gao LF,Jing RJ,Huo NX,etal.One hundred and one new microsatellite lociderived from ESTs(EST-SSRs)in bread wheat[J].Theoreticaland Applied Genetics,2004,108:1392-1400
[29]Zhang LY,Bernard M,Leroy P,et al.High transferability of bread wheat EST-derived SSRs to other cereals[J]. Theoreticaland Applied Genetics,2005,111:677-687
[30]Li LZ,Wang JJ,Guo Y,et al.Development of SSRmarkers from ESTs of gramineous species and their chromosome location onwheat[J].Progress in Natural Science,2008,18:1485-1490
[31]Thiel T,M ichalek W,Varshney RK,et al.Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley(Hordeum vulgare L.)[J].Theorand ApplGenet,2003,106:411-422
[32]Nicot N,Chiquet V,Gandon B,et al.Study of simple sequence repeat(SSR)markers from wheatexpressed sequence tags(ESTs)[J].Theorand ApplGenet,2004,109:800-805
Development of Wheat SNP-SSR Markers on 5DL Chromosome
CHEN Fang,LIWen-jing,DENG Zhi-ying*,TIAN Ji-chun*,CHEN Jian-sheng
Group ofWheatQualityand MolecularBreeding/Key Laboratory ofCrop Biology ofShandong Province;State Key Laboratory ofCrop Biology,Taian 271018,China
The objective of this study was to develop SNP-SSR markers on Xbarc320-Xwmc215 of 5DL chromosome for wheatby using the physicalmap of Aegilops tauschii and the database of wheat D genome sequence.SSR Hunter software was used to search SSR loci based on SNP sequences and then SNP-SSR primer pairs were designed by using Primer 5.0 software.The validity of primer pairswas detected using 22 wheat varieties and synthetic hexaploid wheatmaterials.These markersweremapped using DH population and Chinese Spring Nullisomic Tetrasomic lines.109 SSRswere found among 90 SNP extended sequences and local Blast between AT5D4910-AT5D5010.Based on these SSR sequences,72 SNP-SSR primer pairs were designed.47 new SNP-SSR markers on Xbarc320-Xwmc215 of 5DL were obtained.Four of those loci Xtdc11,Xtdc31,Xtdc38,and Xtdc44were integrated into thewheat5DLgeneticmap using DH population.
SNP-SSR;developmentofmarkers;wheat5D chromosome
S33
A
1000-2324(2016)04-0494-07
2014-07-24
2014-10-09
国家自然科学基金(31301315);山东省自然科学基金(ZR2013CM 004);作物生物学国家重点实验室开放课题(2013KF06);和山东省农业良种工程(鲁农良种字[2013]207号和[2014]96号)
陈芳(1988-),女,山东泰安人,在读研究生,研究方向为小麦分子标记与分子设计育种.E-mail:1172898805@qq.com
Authors for correspondence.E-mail:deng868@163.com;E-mail:jctian@sdau.edu.cn

