福建东方蜜蜂线粒体DNA的遗传变异和遗传多样性
周姝婧, 朱翔杰, 徐新建, 吴显达, 周冰峰
(福建农林大学蜂学学院,福建 福州 350002)
福建东方蜜蜂线粒体DNA的遗传变异和遗传多样性
周姝婧, 朱翔杰, 徐新建, 吴显达, 周冰峰
(福建农林大学蜂学学院,福建 福州 350002)
为了研究福建东方蜜蜂的遗传变异和遗传多样性,在全省采集9个样点,共424群东方蜜蜂样本,进行线粒体DNA tRNAleu-COⅡ序列分析.结果表明,tRNAleu-COⅡ片段序列包括93 bp的非编码区和259 bp的COⅡ编码区.在整个352 bp序列中,共检测到23个多态位点和27种单倍型,平均单倍型多样度为0.484 3,平均核苷酸差异数为0.687,平均核苷酸多样度为0.001 93.福建东方蜜蜂各样点间不存在遗传分化,总体遗传分化系数为0.003,各样点的基因流参数为49.75~249.75.
东方蜜蜂; 种群; 线粒体DNA; 遗传变异; 遗传多样性
遗传多样性是种群适应环境变化的基础,与适合度显著相关[1].遗传多样性是种群进化的潜力,是全球生物多样性保护的3个优先内容之一[2],是制定物种和种群保护方案的依据.种群遗传结构是研究种群间遗传分化和基因渗入检测的基础[3-4],也是理解生物地理分布规律的重要手段[5-6].
福建省位于中国东南沿海,山地丘陵面积约占全省土地总面积的90%,森林覆盖率为62.96%,居全国首位[7].福建省的养蜂历史悠久,蕴涵了丰富的东方蜜蜂(Apiscerana)资源,是东方蜜蜂模式标本的采集地[8].东方蜜蜂是福建最主要的饲养蜂种,个体耐寒性强,6.5 ℃的气温就能出巢采集[9],能够充分利用枇杷、八叶五加和野桂花(柃属植物)等主要冬季蜜源[10-11].
线粒体DNA是单倍体,母系遗传,无重组,具有高突变率,是一种有效检测遗传多样性和种群遗传结构的方法[12-14].tRNAleu-COⅡ片段是蜜蜂属昆虫mtDNA中所特有的,是研究线粒体遗传的重要标记[15-17].前人在进行福建东方蜜蜂mtDNA遗传多样性的研究时,测定6只样本共检测到2种单倍型[16,18],其中一种为中国最常见的单倍型Japan1[18],另一种为单倍型Laos1[16].对东方蜜蜂遗传变异和遗传多样性的认识,还需要加大样本量进行全面深入的研究.本试验在福建全省采集蜜蜂样本,进行了线粒体DNA tRNAleu-COⅡ片段分析,旨在全面了解福建东方蜜蜂的线粒体遗传变异和遗传多样性.
1 材料与方法
1.1样本采集
样本采自福建省的光泽、政和、宁德、龙栖山、尤溪、福州、武平、永定和南靖等9个样点(图1),共采集424群.采样地理位置为:东经116°05′~119°33′,北纬24°33′~27°47′.每群蜜蜂取1只测定,总样本量为424只蜜蜂个体(表1).
1.2数据分析
采用目的片段tRNAleu-COⅡ序列进行分析,引物、PCR反应体系和反应程序参照文献[12]的方法.PCR产物测序由上海英骏生物技术有限公司完成,测序仪为ABI PRISM 3730.本试验得到的序列用Clustal X软件进行序列比对,并与测序图核对校准.有变异的个体进行重复测序,验证其准确性.
采用DnaSP 5.0软件计算福建东方蜜蜂各样点的核苷酸差异数、核苷酸多样度和单倍型多样度[19];采用Mega 6.0软件计算各样点间的遗传分化系数,并以此参数为基础计算基因流参数;采用DnaSP 5.0和Arlequin 3.5.2.2软件计算Tajimas′D、Fu and Li′ sD、Fu and Li′sF和P[19-20];采用DnaSP 5.0绘制错配分布图,检验福建东方蜜蜂是否经历过扩张.
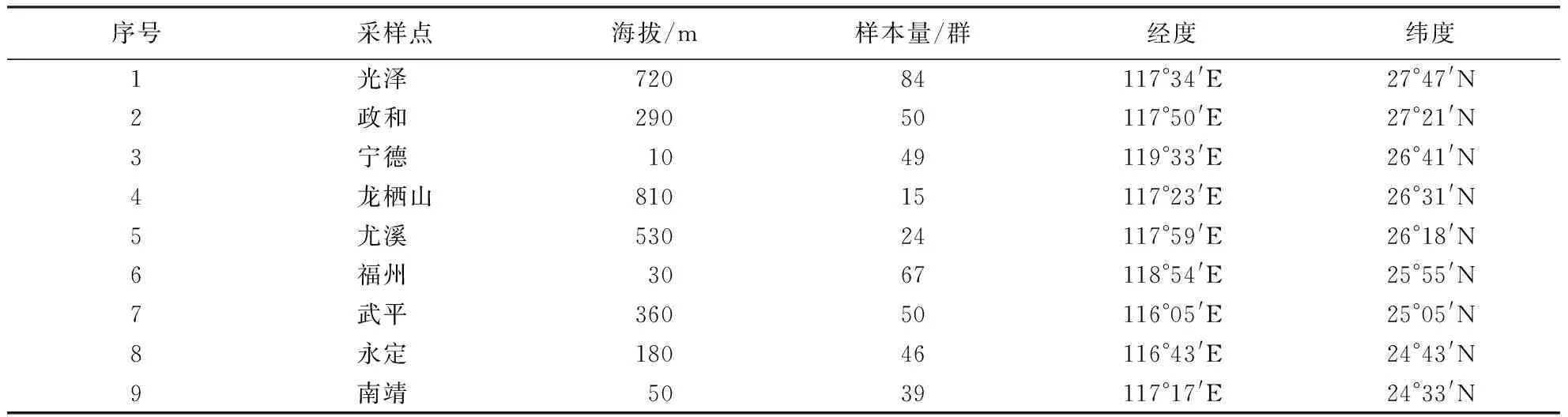
表1 福建东方蜜蜂样本信息
2 结果与分析
2.1遗传变异
PCR产物为一条352 bp的序列片段,包括一个93 bp的非编码区和一个259 bp的部分COⅡ基因编码区(图 2).检测序列为碱基A和T的富集区,在非编码区的A和T分别占非编码区序列长度的48.92%和37.74%,在部分COⅡ编码区的A和T分别占编码区序列长度的37.93%和47.40%.
本试验共检测到23个变异位点.其中,非编码区变异位点13个,占该区序列长度的13.98%;COⅡ编码区变异位点10个,占3.86%.1个简约信息位点位于第80位置;其他变异位点均为单一多态位点.19个位点发生转换,其中,A-G之间的转换有10个,分别发生在第13、22、34、49、53、62、80、81、117和288位置;T-C之间的转换有8个,分别发生在第37、39、45、132、144、237、282和297位置;A-C之间的转换有1个,发生在第226位置.4个位点发生颠换,均为A-T之间的颠换,分别在第16、80、210和261位置;1个位点发生缺失,发生在第56位置;在第80位置同时发生颠换和转换.
2.2单倍型多样性
本试验共检测到单倍型27种,分别为Acmt01001~Acmt01004、Acmt01010~Acmt01012、Acmt01014~Acmt01016、Acmt01018、Acmt01020~Acmt01022、Acmt01029~Acmt01035和Acmt01038~Acmt01043,NCBI登录号分别为HM461260~HM461263、HM461269、HQ186273~HQ186274、HQ186276~HQ186278、HQ186280、JN157775~JN157777、JN157784~JN157790和JN157793~JN157798.从254个样本中检测到单倍型Acmt01001,占总样本量的59.90%;单倍型Acmt01012、Acmt01002和Acmt01022分别检测到42、38和23只样本,分别占总样本量的9.91%、8.96%和5.42%;其余21种单倍型的检测频率较小,其中,17种单倍型在多个样点出现比例小,发现的个体数为2~7只,6种单倍型仅在一个样本中检测到(表2).
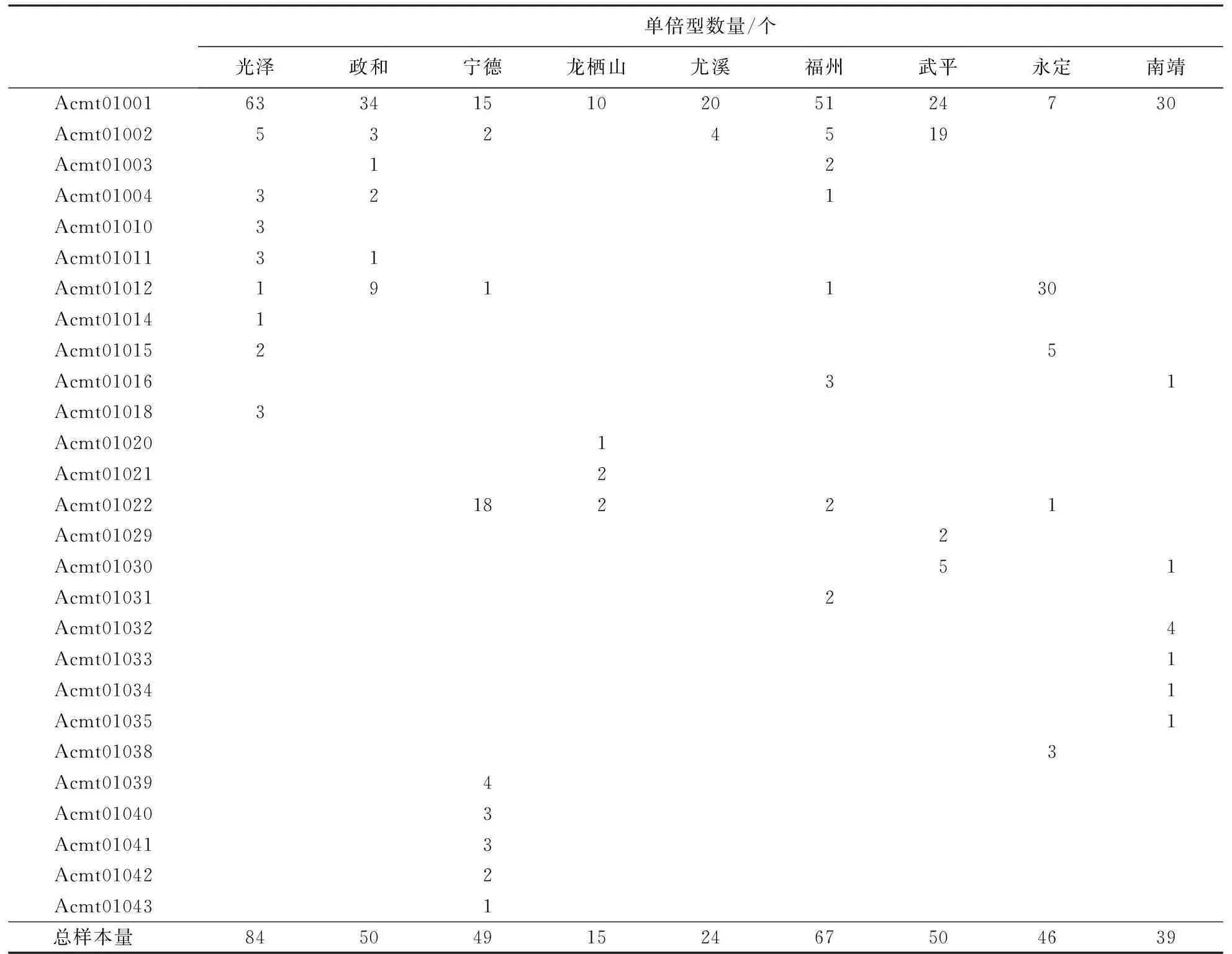
表2 福建东方蜜蜂单倍型分布
福建东方蜜蜂的平均单倍型多样度为0.484 3,平均核苷酸差异数为0.687,平均核苷酸多样度为0.001 93.各采样点东方蜜蜂的单倍型数量、单倍型多样度、核苷酸差异数、核苷酸多样度和核苷酸变异位点数存在较大差异(表3).宁德东方蜜蜂的遗传多样性指标最高,3个体现遗传多样性指标均明显高于其他样点;尤溪仅发现2种单倍型,遗传多样性指标最低.
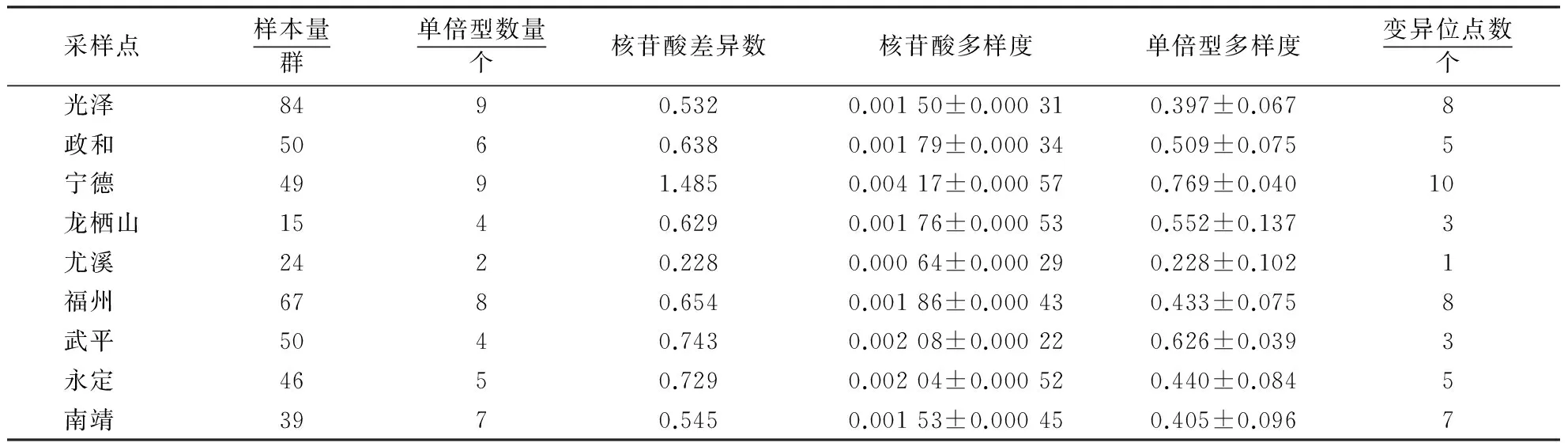
表3 福建各样点东方蜜蜂遗传多样性指标
2.3种群分化与遗传结构
福建东方蜜蜂的总体遗传分化系数为0.003.宁德、永定两个样点与其他样点间的遗传分化系数最大(0.003~0.005);其他样点间的两两遗传分化系数均在0.003以下.根据遗传分化系数计算出的基因流参数为49.75~249.75(表4).
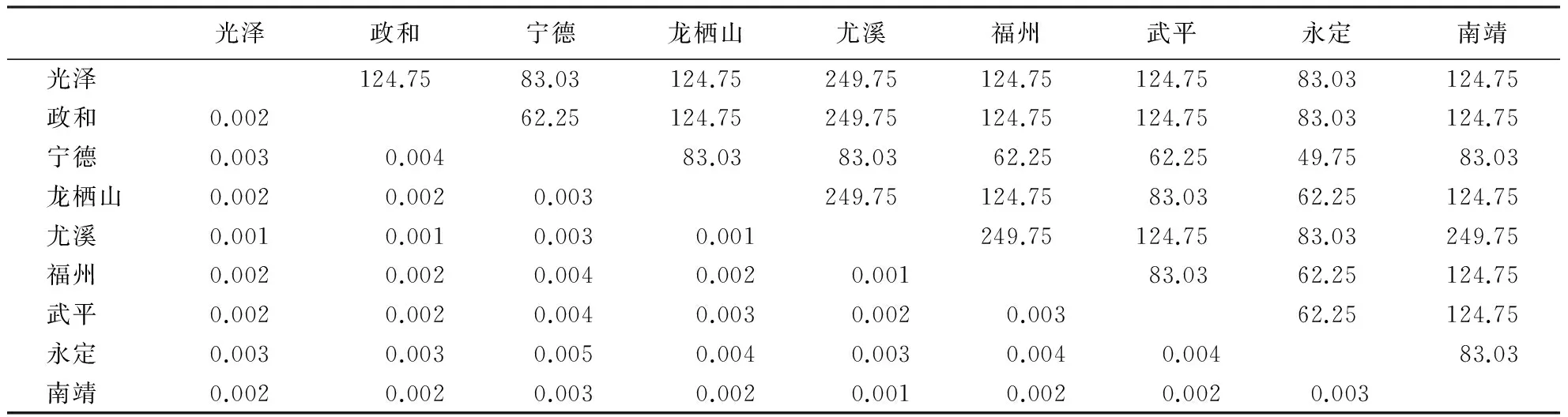
表4 福建东方蜜蜂9个样点间的遗传分化系数和基因流参数1)
1)对角线下的数值为遗传分化系数,对角线上的数值为基因流参数.
福建省大部分采样点的东方蜜蜂线粒体DNA遗传结构相似,以Acmt01001为主要单倍型(表2).除永定和宁德外,几乎所有采样点都以Acmt01001为主要单倍型,占总样本量的48.00%~83.33%.永定以Acmt01002为主要单倍型,占永定样本量的65.22%;宁德以Acmt01001和Acmt01022为主要单倍型,分别占宁德样本量的30.61%和36.73%(表2).
2.4种群历史动态
对福建东方蜜蜂种群进行中性检验,所有中性检验结果均为负数,并显著偏离中性检验.Tajima′D为-2.113 86,差异极显著(P<0.01);Fu and Li′sD为-2.084 90,Fu and Li′sF为-2.541 22,差异均为显著(P<0.01).错配分布曲线为单峰,背离期望值的假定模型(图3).
3 讨论
本试验明确了福建东方蜜蜂线粒体DNA的遗传变异和遗传多样性.福建省各采样点间的遗传分化系数均小于0.005,基因流参数大于49.75,表明分布在福建省的东方蜜蜂未发生遗传分化,属于同一个种群[21-22].在遗传多样性的检测中,检测到的单倍型比前人发现更为丰富.建立在424个蜂群大样本量研究的基础上,在tRNAleu-COⅡ序列中共发现27种单倍型,其中有8种单倍型在其他地区也检测到.单倍型Acmt01001、Acmt01002、Acmt01011、Acmt01022和Acmt01030在海南检测到[23];单倍型Acmt01015在长白山检测到[24];单倍型Acmt01001~Acmt01004在浙江温州检测到[17].这些单倍型信息为东方蜜蜂种群遗传学、亚种分类、系统发育、起源和进化及生物地理学等研究提供了重要的基础数据.
福建的生态环境良好,蜜粉源丰富,东方蜜蜂饲养量占全省饲养蜂群数的2/3以上,在山区有大量的野生东方蜜蜂资源[11].在适宜的栖息生境下,种群数量大,遗传多样性高[1,25-26].正常情况下,福建东方蜜蜂遗传多样性应处于较高的水平.但福建东方蜜蜂遗传多样性的检测结果并没有预期丰富,线粒体遗传多样性水平仅高于长白山小种群,低于其他大部分地区[23-24].这些研究结果说明,在蜜蜂的饲养过程中,不当的人工育王严重影响东方蜜蜂的遗传多样性.因此,保护当地东方蜜蜂资源应引起重视,建议在福建环境良好的深山区建立东方蜜蜂自然保护区,区内促进原始饲养,扩大种群数量,禁止人工育王,避免人为改变种群遗传结构,保护遗传多样性.
永定和宁德的东方蜜蜂具有特殊的线粒体遗传结构,这两个采样点的样本均采自活框饲养蜂群,通过人工育王更换新蜂王.在养蜂技术相对较好的地区,人工育王多选用少数生产性能好的蜂群作为种用群,这种育王方式会导致线粒体遗传结构快速改变及遗传多样性急剧下降.
福建东方蜜蜂发生过种群扩张,且近期仍处于种群增长过程.福建东方蜜蜂内部有较大的基因流、较高频率的共享单倍型Acmt01001以及错配分布的单峰曲线,这些都说明福建东方蜜蜂经历过种群扩张[27].中性检验结果显示,福建东方蜜蜂的Tajima′D、Fu and Li′sD和Fu and Li′sF均为负数,并显著偏离中性检验,也表明种群在近期有扩张的趋势,而不是处于稳定状态[28-30].
致谢:在样本采集中得到王光其、颜复志、郑友义、廖文新、曾清泉、吴克倾、周春其、黄秉正、黄建勇、龚建军、汪明、林水海、林裕忠、李典章、王福灿、罗守忠、陈修汉、黄于勇、裘尚维和郑智华等的帮助.在此表示感谢!
[1] FRANKHAM R, BALLOU J D, BRISCOE D A. Introduction to Conservation Genetics [M]. 2nd ed. Cambridge, UK: Cambridge University Press, 2010.
[2] MCNEELY J A, MILLER K R, REID W V. Conserving the World′s Biological Diversity [M].Washington: IUCN, 1990.
[5] COLLET T, FERREIRA K M, ARIAS M C, et al. Genetic structure of Africanized honeybee populations (ApismelliferaL.) from Brazil and Uruguay viewed through mitochondrial DNA COⅠ-COⅡ patterns [J]. Heredity, 2006,97(5):329-335.
[6] RADLOFF S E, HEPBURN C, HEPBURN H R, et al. Population structure and classification ofApiscerana[J]. Apidologie, 2010,41(6):589-601.
[7] 杜秀荣,唐建军.中国地图集[M].北京:中国地图出版社,2004.
[8] 杨冠煌.中华蜜蜂[M].北京:中国农业科技出版社,2001.
[9] 周冰峰,许正鼎.蜜蜂低温采集活动的研究[J].中国蜂业,1988(5):7-9.
[10] 龚一飞,张其康.蜜蜂分类与进化[M].福建:福建科学技术出版社,2000.
[11] 陈耀春.中国蜂业[M].北京:农业出版社,1993.
[12] GARNERY L, CORNUET J M, SOLIGNAC M. Evolutionary history of the honey beeApismelliferainferred from mitochondrial DNA analysis [J]. Molecular Ecology, 1992,1(3):145-154.
[13] PALMER M R, SMITH D R, KAFTANOGLU O. Turkish honeybees: genetic variation and evidence for a fourth lineage ofApismelliferamtDNA [J]. Journal of Heredity, 2000,91(1):42-46.
[14] SMITH D R, HAGEN R H. The biogeography ofApisceranaas revealed by mitochondrial DNA sequence data [J]. Journal of the Kansas Entomological Society, 1996,69(4 supplement):294-310.
[15] CORNUET J M, GARNERY L, SOLIGNAC M. Putative origin and function of the intergenic region between COⅠand COⅡofApismelliferaL. mitochondrial DNA [J]. Genetics, 1991,128(2):393-403.
[16] TAN K, WARRIT N, SMITH D R. Mitochondrial DNA diversity of ChineseApiscerana[J]. Apidologie, 2007,38(3):238-246.
[17] XU X, ZHU X, ZHOU S, et al. Genetic differentiation betweenApisceranaceranapopulations from Damen Island and adjacent mainland in China [J]. Acta Ecologica Sinica, 2013,33(3):122-126.
[18] 姜玉锁,赵慧婷,姜俊兵,等.中国境内不同地理型东方蜜蜂线粒体DNA tRNAleu-COⅡ基因多态性研究[J].中国农业科学,2007,40(7):1 535-1 542.
[19] LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 2009,25(11):1 451-1 452.
[20] FU Y X, LI W H. Statistical tests of neutrality of mutations [J]. Genetics, 1993, 133(3): 693-709.
[21] SOARES A A F, BASSI D, MANGOLIN C A, et al. Evidence of high gene flow between samples of horseweed (Conyzacanadensis) and hairy fleabane (Conyzabonariensis) as revealed by isozyme polymorphisms [J]. Weed Science, 2015, 63(3): 604-612.
[22] WRIGHT S. Evolution and the Genetics of Population [M]. Chicago and London: University of Chicago Press, 1978.
[23] 周姝婧,徐新建,朱翔杰,等.海南中华蜜蜂线粒体DNA的遗传多样性[J].福建农林大学学报(自然科学版),2012,41(2):170-175.
[24] 于瀛龙,周姝婧,徐新建,等.长白山中华蜜蜂(Apisceranacerana)遗传多样性分析[J].福建农林大学学报(自然科学版),2013,42(6):643-647.
[25] PETIT R J, ITZIAR A, JACQUES-LOUIS D B, et al. Glacial refugia: hotspots but not melting pots of genetic diversity [J]. Science, 2003,300:1 563-1 565.
[26] ZENG Q Q, HE K, SUN D D, et al. Balancing selection and recombination as evolutionary forces caused population genetic variations in golden pheasant MHC class I genes [J]. BMC Evolutionary Biology, 2016,16:1-15.
[27] KVIST L, MARTENS J, HIGUCHI H, et al. Evolution and genetic structure of the great tit (Parusmajor) complex [J]. Proceedings of the Royal Society B-Biological Sciences, 2003,270:1 447-1 454.
[28] FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection [J]. Genetics, 1997,147(2):915-925.
[29] HU Z M, UWAI S, YU S H, et al. Phylogeographic heterogeneity of the brown macroalgaSargassumhorneri(Fucaceae) in the northwestern Pacific in relation to late Pleistocene glaciation and tectonic configurations [J]. Molecular Ecology, 2011,20(18):3 894-3 909.
[30] GUO S S, ZHANG G R, GUO X Z, et al. Genetic diversity and population structure ofSchizopygopsisyounghusbandiRegan in the Yarlung Tsangpo River inferred from mitochondrial DNA sequence analysis [J]. Biochemical Systematics and Ecology, 2014,57:141-151.
(责任编辑:施晓棠)
Genetic variation and genetic diversity ofApisceranafrom Fujian province based on mitochondrial DNA sequence analysis
ZHOU Shujing, ZHU Xiangjie, XU Xinjian, WU Xianda, ZHOU Bingfeng
(College of Bee Science, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China)
To examine the genetic variation and diversity ofApisceranain Fujian province, sequence analysis of mitochondrial DNA fragments of tRNAleu-COⅡ in honeybees was conducted. These honeybees were sampled from 424 colonies from 9 cities cross Fujian province. The sequence was 352 bp at length, consisting of a 93 bp non-coding region and a 259 bp partial COⅡ coding region. A total of 23 polymorphic sites and 27 haplotypes were found in 424 sequences. Haplotype diversity and nucleotide difference averaged at 0.484 3 and 0.687, with nucleotide diversity of the samples being 0.001 93. The overall genetic differentiation coefficient was 0.003, with gene flow ranging from 49.75 to 249.75. To summarize, no population differentiation was detected among honeybees across Fujian province.
Apiscerana; population; mitochondrion DNA; genetic variation; genetic diversity
2016-03-03
2016-04-19
现代农业蜂产业技术体系建设专项资金资助项目(CARS-45-KXJ11);福建省自然科学基金资助项目(2010J05043).
周姝婧(1987-),女,博士研究生.研究方向:蜜蜂生态学和蜜蜂种群遗传学.Email:sjzhou118@gmail.com.通讯作者周冰峰(1958-),男,教授,博士生导师.研究方向:蜜蜂生态学和蜜蜂种群遗传学.Email:bingfengfz@126.com.
S891
A
1671-5470(2016)03-0310-06
10.13323/j.cnki.j.fafu(nat.sci.).2016.03.012

