复杂性状遗传CC小鼠
——肿瘤研究新平台
李克娟,高 苒
(北京协和医学院比较医学中心,中国医学科学院医学实验动物研究所,北京100021)
复杂性状遗传CC小鼠
——肿瘤研究新平台
李克娟,高苒
(北京协和医学院比较医学中心,中国医学科学院医学实验动物研究所,北京100021)
癌症是由于机体细胞失去正常调控过度增殖而引发的一系列复杂性状疾病的统称,其本质上是一种基因疾病。用于传统肿瘤小鼠模型的重组近交(RI)品系由于只起源于两个近交祖系,因此规模小且等位基因多样性较少,在统计功效上存在局限性。复杂性状遗传(Collaborative Cross mice, CC)小鼠是由来自八个原始种系近交产生的数百种基因型不同的小鼠品系,能够体现不同小鼠亚种的遗传学变异,具有高遗传多样性,大种群规模等重要特征。它可以模拟人类复杂性状疾病中不同人群对病因或治疗方法个体易感性的差异,为促进复杂性状疾病易感基因筛查、基因功能发现等方面的研究提供了一个更好的研究工具与信息平台。本文就CC小鼠在医学应用中的研究现状、挑战性与局限性、以及其在肿瘤研究中的应用前景进行综述。
复杂性状遗传小鼠(CC小鼠);遗传多样性;个体易感性;肿瘤
癌症是由于多种致癌因素激活原癌基因或(和)灭活肿瘤抑制因子,使细胞发生转化并无限增殖的一类基因组疾病的统称[1]。癌症已成为世界范围内高发病率和高死亡率的主要原因之一。根据世界卫生组织(WHO)2014年发布的数据显示,2012年全世界共新增1400万癌症病例并有820万人死亡[2]。其中,中国新增307万癌症患者并造成约220万人死亡,分别占全球总量的21.9%和26.8%。据Siegel[3]最新研究报道,种族对肿瘤的发病率具有非常显著的影响。在所有癌症种类中,黑色人均比白色人种拥有更高的癌症发病率和死亡率(肾癌的死亡率除外),几乎是亚太裔人种的2倍。此外,性别对癌症的发病率也有显著影响,与女性相比男性的泌尿系统癌症发病率和死亡率均是女性的2倍左右。大量流行病学研究与临床观察表明大部分癌症(约90%~95%)都是由环境因素导致的[4],其余的5%~10%则是由遗传因素导致的[5]。但是对于相同的致癌风险为什么有些人更加易感仍然是一个谜。另一方面,为什么有些肿瘤患者对同一药物表现出更好的敏感性或者对治疗方案表现出更好的治疗效果,对此临床上也没有给出科学的解释。例如,我们知道白血病是一种常见的恶性程度极高的肿瘤,曾经一度是被认为是一种“无药可救”的疾病。随着2012年第一例白血病患者的治愈,免疫疗法CAR-T给白血病患者带来了希望,但是同样是白血病患者,CAR-T疗法却也不能达到100%的治愈效果,而白血病临床分型非常复杂,如何在这些分型中找到针对CAR-T疗法有效的分型对于白血病的治疗是否成功尤为重要。为此在第15届爱丁堡全球小鼠基因峰会上,美国橡树岭国家实验室(Oak Ridge National Laboratory)提出了复杂性状遗传(Collaborative Cross,CC)小鼠育种计划,并建立了复杂性状联盟(Complex Trait Consortium,CTC)组织[6],用于选择培育一种更适用于研究复杂性状或复杂病因疾病的研究工具,以模拟人群基因多样性进行复杂形状的相关研究[7]。
1 CC小鼠
CC小鼠的理念背后是为了充分开发RI小鼠的潜能,并将这种潜能发挥到极致。众所周知,RI小鼠是由两个无关高质近交系进行杂交,用其F2代进行全同胞兄妹交配20代以上而育成的新近交系,其近交系数达98.6%以上。RI具有双亲近交系特性和重组后新近交品系特性,大大推动了遗传学,肿瘤学,免疫学和行为学等学科的发展。RI小鼠因为其具备来源清楚,个体间均一性高,组织相容性高,重复性好等优点,到目前为止已成为肿瘤病因学,肿瘤药理学等研究的理想和经典模型并且被广泛应用。但是RI小鼠由于受到遗传多样性(genetic variability)与映射能力(mapping power)的局限也存在很多不足。遗传多样性的不足是因为RI小鼠仅由两个亲本品系杂交而来,并且这些亲本品系往往起源于共同的祖系,更加限制了遗传多样化。而映射能力取决于测试过程中可以获取的品系。常用的基因片段的分辨率通常在2~4 cM(~ 4至8 Mb),但是传统的RI小鼠在后基因组时代不足以提供有效的候选基因进行分析。第一个问题可以通过使用8个原始品系,其中包含来自不同大陆的野生型品系,来代替原来的2个亲本品系来解决。第二个问题则可以通过产生大量的RI品系来解决。CC小鼠的概念的提出正好弥补了这些不足,所选的8个原始品系使得CC小鼠的遗传多样性最大化,其中包括三种刚刚完成基因组测序的实验用近交系:A/J,C57BL/6J和129S1/SvImJ,以及来自日本和新西兰被用作自身免疫与肥胖模型的两个品系NOD/LtJ和NZO/HiLtJ,除此之外,使用来自小鼠3个Mus亚种的野生近交系作为补充,他们分别是来自捷克布拉格的PWK/PhJ(Mus.musculus.musculus),来自美国巴尔的摩的WSB/EiJ(Mus.musculus.domesticus),以及来自泰国的CAST/Ei(Mus.musculus..castaneus)[8, 9]。他们的遗传物质的多样性可以基本覆盖小鼠全基因组的90%[10],且亲缘关系相对较远,可以保证CC品系遗传位点的分散性[11]。
CC小鼠的育种过程如图1所示,八种原始品系作为G0代,首先互相交配形成56种可能的G1代杂交组合,然后,56个G1品系再分别进行交配得到1680种可能的G2代杂交组合,G2代品系再次进行交配可以获得8!(40 320)种组合可能的G3代杂交小鼠。通过G3:F1代进行数代的同胞近交,大概在G3:F20时获得充分近交的子代,最终繁育形成独立的CC小鼠品系。每个CC小鼠品系都将随机获得八个原始品系小鼠所有的遗传变异,这种独一无二的基因重组模式称为“漏斗型育种”(breeding funnel)。CC小鼠在繁殖过程中的控制和最小随机化选择将目前存在于这些自交系中的自然遗传变异进行充分重组,使得该实验种群的每个重组位点都是独一无二的,可提供足够的资源来研究单基因附近的数量性状位点(QTL)[6, 13]。其结果将是一个具高遗传多样性的大种群规模的独特品系,为近交系小鼠的表型表达带来巨大的遗传变异潜能[14, 15]。
CC小鼠项目最初在杰克逊实验室(The Jackson Laboratory)运作,而后将G3:F1代运至3个育种机构,包括美国田纳西州的橡树岭国家实验室(Oak Ridge National Laboratory),后来被迁移到北卡罗来纳大学教堂山分校(CC-UNC)[7];肯尼亚内罗毕的国际家畜研究中心(The International Livestock Research Institute),后来被迁移至以色列的特拉维夫大学(Tel Aviv University) (CC-TAU)[16];以及澳大利亚佩斯的西澳医学研究中心/西澳大学(Western Australia Institute for Medical Research/University of Western Australia/Geniad Ltd.) (CC-GND)[12]。目前,在上述3个机构共育有约200个CC品系[17]。
2 CC小鼠的研究及其医学应用
CC种群平衡的等位基因频率,均匀分布的重组位点和表型分布都优于其他实验资源[18-20]。经过十几年的发展,CC小鼠已被用于遗传学,传染病学和免疫学等研究领域并取得了突破性研究进展。例如基因定位,Ram等[21]利用120个CC小鼠品系的毛色设计了一个原理循证研究,不论使用SNP-Based法或者一种基于QTL定位法,调节六种毛色表型的四种基因均被成功定位。CC小鼠作为一种新型的动物模型在医学研究中的优势也日益凸显,目前已经有两个利用CC小鼠成功构建疾病模型的案例,一个是溃疡性结肠炎[22],另外一个是糖尿病视网膜病变[23]。同时,对于大规模不同遗传背景的小鼠品系在群体层面对各种生理和病理表型进行全面分析,可揭示各种表型与潜在的遗传变异、基因之间,以及基因与环境之间的相互作用,为人类复杂性疾病的研究和理解提供更多的证据。Rasmussen等[24]利用47个CC小鼠品系感染埃博拉病毒,发现19%的小鼠品系能够抗病毒并且能够在两周内恢复体重,11%的小鼠品系对病毒有部分抗性,70%的小鼠品系感染病毒后病死率超过半数以上。不同品系的小鼠表现出的症状也有差异,有的小鼠只出现肝脏炎症,有的小鼠则表现出凝血障碍等埃博拉病毒感染的的标志性症状。这些小鼠很好地模拟了人群患病的不同病理过程。研究人员还比较了最易感和最不易感的两种CC小鼠的基因表达模式,发现内皮细胞酪氨酸激酶基因Tiel和Tek的转录水平有显著差异,而这两种酶与凝血功能有关。研究表明埃博拉感染的严重程度与宿主的遗传背景有很大关系,而不能单从病毒方面进行解释。此外,Ferris[25], Leist[26], 以及Xiong 等[27]人分别将H1N1亚型流感病毒,H3N2亚型流感病毒以及SARS流感病毒感染CC小鼠的8个原始品系,均通过品系间的表型差异揭示了宿主易感背后的遗传学机理。此外,Durrant等[28]在真菌感染实验中使用了4种近交系小鼠(BALB/CJ,DBA/2J,C3H/HeJ,C57BL/6J)和66个CC小鼠品系定位了烟曲霉菌易感的候选基因。与此类似,Vered等[29]使用上述4种近交系小鼠和73个CC小鼠品系通过评价腹腔内接种克雷伯氏肺炎杆菌后的存活时间差异证实了不同CC小鼠品系感染细菌后存在表型多样性。
具有高度遗传多样性的CC小鼠无疑也为肿瘤这一复杂性状疾病的研究提供了一个绝佳的机会。Ferguson等[30]首次将CC小鼠应用在肿瘤研究上,他们评价了50个CC小鼠黑色素瘤的特征,其中包括色素痣,色素瘤的发病时间及其多样性,解剖位置偏好,色素痣转化为黑色素瘤以及其转移的时间。研究表明不同CC小鼠品系间表现出了广泛的差异(图2)。此外,研究还发现,紫外线辐射暴露加剧了大多数小鼠品系的新生仔色素痣和黑色素瘤的形成,但是仍有少部分CC小鼠品系并未表现出该现象。这表明CC小鼠体内的遗传变异将有助于解释对阳光暴晒(皮肤癌的主要环境致癌因素)的个体敏感性。由于在一个小鼠模型里面CC小鼠的遗传变异带来了引人注目的表型多样性,黑色素瘤相关的内表型对比给我们提供了有关致癌的机制,例如黑色素瘤发病率是否依赖于预先存在的色素痣细胞的密度。已有的小鼠模型已被用于研究基因突变在肿瘤发生过程中的功能作用。CC小鼠模型将在下一阶段进一步研究生物变异或者遗传变异如何最大程度地影响肿瘤的形成与发病机制。
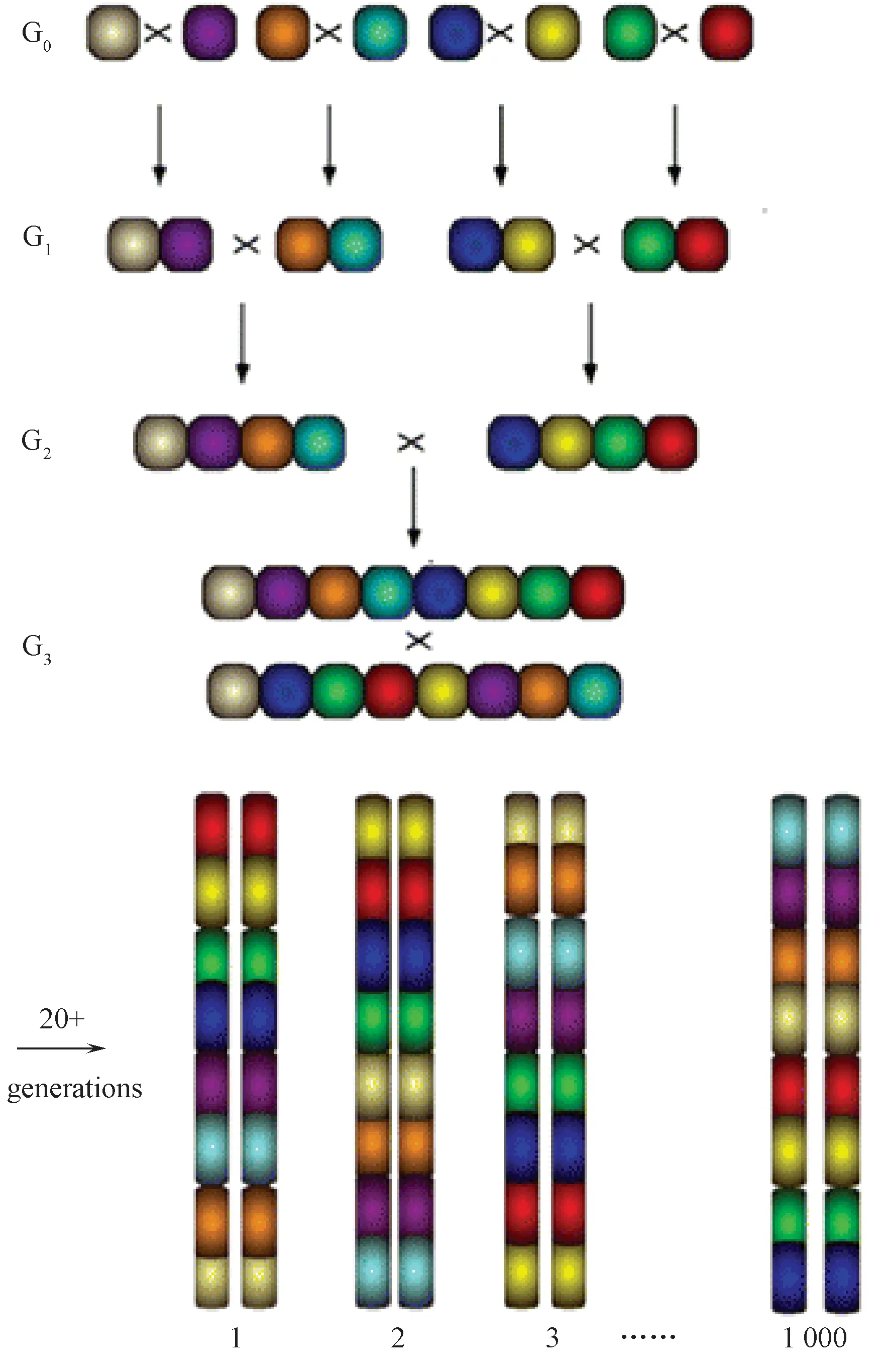
图1 CC小鼠育种设计方案。8个原始品系被定义为G0并且它们的基因组构成分别使用8种颜色来代替[12]Fig.1 Breeding strategy to generate a single breeding “funnel.” The eight founder strains are designated as G0 and their genomes are represented by different colors.
3 CC小鼠的局限性

图2 紫外线照射条件下不同CC小鼠品系色素痣及黑色素瘤的数目差异 [30]注:(A)色素痣;(B)黑色素瘤。Fig.2 Average number of nevoid plaques and melanomas per mouse for each CC strain background.Note. (A) nevoid plaques; (B) melanomas.
CC小鼠相关的报道主要集中在病毒细菌和真菌等感染性疾病的研究中,为了更好地利用CC小鼠来研究人类复杂性状疾病,需要解决当前存在的某些局限性,因为近交动物经常会出现健康问题,生殖能力也会下降,据报道,CC小鼠在近交传代5代后,有90%的小鼠将不再繁殖[31],因此寻求更好地方法做好CC小鼠的繁殖与保种工作显得尤为重要。同时,利用CC小鼠作为动物模型进行研究时,需要使用70个品系以上才能基本消除不同品系带来的假阳性结果,而目前的研究报道很少能够达到这一品系数目的要求,使得QTL分辨率达不到基因水平[32]。如何创造更好的实验条件来实现大规模群体实验来弥补这一缺陷,为更加精准的定位易感基因,研究复杂性状疾病也是开展CC小鼠研究亟待解决的问题之一。
4 CC小鼠在肿瘤研究中的应用前景
CC小鼠在肿瘤领域的研究刚刚起步,目前仅在黑色素瘤的易感性表型差异上面有相关研究报道。与此同时,CC小鼠作为一种新型研究工具与信息平台,将越来越受到肿瘤研究者们的关注及应用。研究者可以利用CC小鼠资源进行肿瘤的病因学研究,用以模拟不同人群对致癌因子的敏感差异,加上系统遗传学方法对肿瘤易感基因进行筛查;还可以利用CC小鼠模拟评价不同人群对现有抗肿瘤药物的药敏药效以及药理作用,寻找药物靶向基因;此外,CC小鼠因其具有丰富的性状差异与遗传多样性,可以帮助我们理解类似前面所述的为什么不同白血病患者对CAR-T疗法的不同反应的问题,并且可以帮助我们找到CAR-T疗法的易感表型和易感基因,类似的,其他有效疗法也可以更加准确的定位相关表型或者相关基因,实现精准治疗。这一切都表明CC小鼠将在肿瘤领域掀起一股划时代的具有革命意义的研究风潮,将为实现精准的分子诊断及精准医疗提供更多的临床前指导数据。
[1]Garraway LA, Verweij J, Ballman KV. Precision oncology: an overview [J]. J Clin Oncol, 2013, 31(15):1803-1805.
[2]World Health Organization (WHO). World Cancer Report 2014. Chapter 1.1 [M]. ISBN 9283204298.
[3]Siegel R, Miller KD, Jemal A. Cancer statistics, 2015 [J]. CA Cancer J Clin, 2015, 65, 5-29.
[4]Manton KG, Akushevich I, Kravchenko J. Cancer mortality and morbidity patterns in the US population: An interdisciplinary approach [M]. Springer Science & Business Media. 2008.89-140.
[5]Anand P, Kunnumakara AB, Sundaram C,etal. Cancer is a preventable disease that requires major lifestyle changes [J]. Pharm Res, 2008, 25 (9):2097-2116.
[6]Threadgill D, Churchill G. Ten Years of the Collaborative Cross [J]. Genetics, 2012, 2 (2):153-156.
[7]Chesler EJ, Miller DR, Branstetter LR,etal. The Collaborative Cross at Oak Ridge National Laboratory: developing a powerful resource for systems genetics [J]. Mamm Genome, 2008, 19 (6):382-389.
[8]Churchill G, Airey D, Allayee H,etal. The Collaborative Cross, a community resource for the genetic analysis of complex traits [J]. Nat Genet, 2004, 36 (11): 1133-1137.
[9]Broman KW. The genomes of recombinant inbred lines [J]. Genetics, 2005, 169 (2):1133-1146.
[10]Roberts A, Pardo-Manul de-Villena F, Wang W,etal. The polymorphism architecture of mouse genetic resources elucidated using genome-wide resequencing data: implications for QTL discovery and systems genetics [J]. Mamm Genome, 2007, 18 (6-7):473-481.
[11]Yang H, Wang JR, Didion JP,etal. Subspecific origin and haplotype diversity in the laboratory mouse [J]. Nat Genet, 2011, 43 (7):648-655.
[12]Morahan G, Balmer L, Monley D. Establishment of ‘‘The Gene Mine‘’: a resource for rapid identification of complex trait genes [J]. Mamm Genome, 2008, 19 (6):390-393.
[13]Threadgill DW, Hunter KW, Williams RW. Genetic dissection of complex and quantitative traits: from fantasy to reality via a community effort [J]. Mamm Genome, 2002, 13 (4):175-178.
[14]Lander ES, Schork NJ. Genetic dissection of complex traits [J].Science 1994, 265: 2037-2048.
[15]Paterson AH. Molecular dissection of quantitative traits: progress and prospects [J]. Genome Res, 1995, 5 (4):321-333.
[16]Iraqi FA, Churchill G, Mott R. The Collaborative Cross, developing a resource for mammalian systems genetics: a status report of the Wellcome Trust cohort [J].Mamm Genome, 2008, 19:379-381.
[17]Morgan AP, Welsh CE. Informatics resources for the Collaborative Cross and related mouse population [J]. Mamm Genome, 2015, 26 (9-10):521-539.
[18]Soller M, Iraqi FA. The Collaborative Cross - A next generation mouse genetic resource population for high resolution genomic analysis of complex traits [J]. Livestock Science, 2014, 166 (4):19-25.
[19]Aylor DL, Valdae W, Foulds-Mathes W,etal. Genetic analysis of complex traits in the emerging collaborative Cross [J]. Genome Res, 2011, 21 (8):1213-1222.
[20]Philip VM, Sokoloff G, Ackert-Bicknell CL,etal. Genetic analysis in the Collaborative Cross breeding population [J]. Genome Res, 2011, 21 (8):1223-1238.
[21]Ram R, Mehta M. Balmer L,etal. Rapid identification of major-effect genes using the collaborative cross [J]. Genetics, 2014, 198 (1):75-86.
[22]Rogala AR, Morgan AP, Christensen AM,etal. The collaborative Cross as a resource for modeling human disease: CC011/Unc, a new mouse model for spontaneous colitis [J]. Mamm Genome, 2014, 25 (3-4):95-108.
[23]Weerasekera LY, Balmer LA, Ram R,etal. Characterization of retinal vascular and neural damage in a novel model of diabetic retinopathy [J]. Invest Ophthalmol Vis Sci, 2015, 56 (6):3721-3730.
[24]Rasmussen AL, Okumura A, Ferris MT,etal. Host genetic diversity enables Ebola hemorrhagic fever pathogenesis and resistance [J]. Science, 2014, 346 (6212):987-991.
[25]Ferris MT, Aylor DL, Bottomly D,etal. Modeling host genetic regulation of influenza pathogenesis in the collaborative cross [J]. PLoS Pathog, 2013, 9 (2):e1003196.
[26]Leist SR, Pilzner C, van den Brand JM,etal. Influenza H3N2 infection of the collaborative cross founder strains reveals highly divergent host responses and identifies unique phenotype in CAST/EiJ mice [J]. BMC Genomics, 2016, 17 (1):143.
[27]Xiong H, Morrison J, Ferris MT,etal. Genomic profiling of collaborative cross founder mice infected with respiratory viruses reveals novel transcripts and infection-specific gene and isoform expression [J]. G3 (Bethesda). 2014, 4 (8):1429-1444.
[28]Durrant C, Tayem H, Yalcin B,etal. Collaborative Cross mice and their power to map host susceptibility toAspergillusfumigatusinfection [J]. Genome Res, 2011, 21 (8):1239-1248.
[29]Vered K, Durrant C, Mott R,etal. Susceptibility toKlebsiellaPneumonaieinfection in collaborative cross mice is a complex trait controlled by at least three loci acting at different time points [J]. BMC Genomics, 2014, 15:865.
[30]Ferguson B, Ram R, Handoko HY,etal. Melanoma susceptibility as a complex trait: genetic variation controls all stages of tumor progression [J]. Oncogene, 2015, 34 (22): 2879-2886.
[31]Schmidt CW. Diversity outbred: a new generation of mouse model [J]. Environ Health Perspect, 2015, 123 (3):A64-67.
[32]Flint J, Mott R. Applying mouse complex-trait resources to behavioural genetics [J]. Nature, 2008, 456 (7223):724-727.
Collaborative Cross mice as a novel platform in cancer research
LI Ke-juan, GAO Ran
(Institute of laboratory animal sciences, CAMS & PUMC, Beijing 100021, China)
Cancer is a general term of a series of complex traits of the disease triggered by the body cells losing their normal regulation of excessive proliferation, which essentially is a genetic disease. Recombinant inbred strain (RI) mouse generated from one pair of founders has been widely used in traditional tumor model. However, RI has many limitations on the statistic efficiency because of the small scale and lacking of allele diversity. The Collaborative Cross (CC) was designed to generate hundreds of recombinant inbred lines by 8 divergent strains of mice. CC mice embody a tremendous amount of natural genetic variation in different sub-strains of mouse and the single nucleotide polymorphism is four times of the traditional experimental mice. The high-genetic diversity and large scale population enables CC mice simulates the differences of individual susceptibility to the pathogeny or the therapies,thus provides a better research tool and information platform for expediting discovery of genes and genes function in human complex traits diseases. This review summarizes our current knowledge of this field, including methodologic aspects, applications, challenges and limitations, and utilization for cancer research.
Collaborative Cross; Genetic diversity; Individual sensitivity; Cancer
中国医学科学院基本科研业务费专项资助项目(DWS201402)。
李克娟(1987-),女,博士,研究方向:肿瘤模型构建。E-mail:likejuan@cnilas.org。
高苒(1980-),女,副研究员,研究方向:肿瘤模型构建。E-mail:gaoran26@hotmail.com。
专题研究
R-33
A
1671-7856(2016) 08-0025-05
10.3969.j.issn.1671-7856.2016.08.000
2016-07-05

