关节病型银屑病与强直性脊柱炎易感基因关联研究
杨 青 刘 红 付希安 于永翔 于功奇 屈丽娜 张福仁
关节病型银屑病与强直性脊柱炎易感基因关联研究
杨青刘红付希安于永翔于功奇屈丽娜张福仁
目的:对关节病型银屑病与强直性脊柱炎的易感基因进行关联分析 ,以期发现共同的易感基因。方法:以379例关节病型银屑病(PsA)、595例寻常型银屑病(PsV)及806例健康对照为样本,以Sequenom MassARRAY系统为平台,对全基因组关联研究发现的强直性脊柱炎的9个易感基因SNP位点进行基因分型和数据分析。结果: ERAP1基因(rs27037,P=6.66×10-5,OR:1.43)、21q22.2(rs2242944,P=1.07×10-3,OR:0.73)及IL23R基因(rs1004819,P=4.58×10-3,OR:1.28)与PsA相关。ERAP1(rs27037,P=1.56×10-4,OR:1.35)与PsV相关。ERAP1基因对于PsA和PsV的患病风险无差异。IL23R基因(rs1004819)及21q22.2(rs2242944)在PsA和PsV患病风险上存在中等程度的异质性(I2值分别为57.41和71.20),但P值无明显差异(>0.05)。IL23R基因(rs11209032,P=1.57×10-3,OR: 1.52)与PsA脊柱炎相关。结论:ERAP1基因、21q22.2区域及IL23R基因是PsA与强直性脊柱炎共有的易感基因。
关节病型银屑病; 易感基因; 易感位点; 强直性脊柱炎
关节病型银屑病(psoriatic arthritis,PsA)不仅引起皮肤的炎症,还可导致脊柱和外周关节疼痛、肿胀、活动受限及关节畸形,是一种严重危害健康的顽固性疾病。虽然国内外研究已成功发现银屑病的多个易感基因,引起银屑病患者关节损害和畸形的易感基因尚未明确[1]。以往研究证实临床表现相近疾病间具有共同的易感基因。例如,PsA与类风湿关节炎均与TNFAIP3、TNIP1基因相关[2,3]。而银屑病关节炎与
强直性脊柱炎在临床表现、组织病理学等方面均有一定共性[4]。本研究拟以GWAS研究发现的强直性脊柱炎多个易感基因SNP位点为候选研究位点[5,6],以PsA患者为样本,以寻常型银屑病患者(psoriasis vulgaris,PsV)和健康志愿者为对照,以Sequenom MassARRAY系统为平台进行SNP分型和关联分析,以期发现共有的易感基因,为阐明该病的发病机制、研制与强直性脊柱炎共同作用靶点的药物奠定基础。
1 材料和方法
1.1研究样本 本实验的样本包括PsA患者379例,PsV患者595例和正常对照806例。PsA采用国际公认的CASPAR诊断标准[7]诊断。PsV为就诊时具有典型寻常型银屑病皮损,并经过全面临床查体,无任何关节症状和体征,亦无其他自身免疫病的患者。正常对照入选标准:需同时满足以下条件:①身体健康;②本人无银屑病、自身免疫性疾病(包括一、二、三级亲属)、系统性疾病;③家族中无上述疾病患者。所有入选的PsA及PsV患者均来自我院皮肤科门诊,经两名高级职称的医师确诊;正常对照为健康志愿者。在所有对象知情同意并签字后进行资料的收集。对所有对象抽取外周静脉血3~5 mL,编号排序后应用Qiagen DNA抽提试剂盒提取DNA。
1.2SNP的选择 以国外发表的强直性脊柱炎GWAS研究为基础[5,6],以原文献中P<10-5为界,优先选择OR值相对较大的SNP位点。在Hapmap数据库中查询该SNP等位基因在中国人群最小等位基因频率(Minor Allel Frequency,MAF)>0.05。且能够在Sequenom平台进行分型,并在引物设计中相互兼容。依上述原则,最终选择9个非MHC(Major Histocompatibility complex,主要组织相容性抗体)的SNPs在试验样本中分型进行验证分析。
1.3基因分型 利用高通量、自动化的Sequenom MassARRAY系统(San Diego,California,USA),对选择的SNP位点进行分型。基本步骤:①将DNA样本进行待测位点的PCR扩增;②将PCR产物进行虾碱性磷酸酶处理(SAP反应);③加MassEXTEND®延伸引物进行单碱基延伸反应;④将反应产物加入SpectroCHIP共结晶;⑤放入MALDI-TOF质谱仪进行检测;⑥质谱图经MassARRAY分型软件分析处理,出分型报告。质量控制:对于call rate≥90%、符合遗传平衡定律、分型聚类图清晰的SNP及分型成功的样本进行统计分析。
1.4数据分析 利用Plink 1.07软件对分型成功并通过质控的SNPs在病例组(PsA、PsV组)和对照组做Cochran-Armitage trend检验计算其关联性。多重logistic回归分析用于检测区域内的信号的独立性。检验水准α以5.6×10-3(即以0.05除以9,即SNP数目进行校正)。采用异质性检验方法(I2and p-values of Q statistic)比较PsA与PsV的异质性。依据文献,异质性的程度主要以I2值大小进行判断,小于25%认为无异质性,25%~50%为存在轻度异质性,50%~75%为中度异质性,75%以上为存在较强异质性。鉴于PsA是一异质性疾病,有些病例的关节表现似强直性脊柱炎,有些则与类风湿性关节炎类似,本研究将PsA患者按照脊柱是否受累分为脊柱炎型和外周关节炎型2组进行亚型分析和型别间异质性分析。
2 结果
2.1研究对象一般情况 本研究所用样本情况分析结果见表1。各组年龄、性别基本匹配,PsA组年龄略大。所有样本均来自山东,均为汉族。PsA患者中有186例(49.1%)患者伴有脊柱受累(其中单纯脊柱受累患者37例,脊柱和外周关节同时受累患者193例),其余193例(50.9%)患者仅伴有外周关节受累。
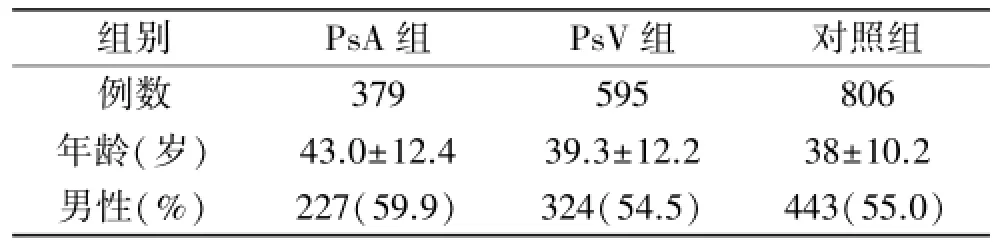
表1 样本基本信息
2.2统计结果
2.2.1PsA与正常对照比较见表2 在379例PsA和806例正常对照中对9个通过质控的SNPs进行Cochran-Armitage trend检验,共发现3个强直性脊柱炎相关的SNPs与PsA显著关联,分别是ERAP1基因rs27037(p=6.66×10-5,OR:1.43)和rs27434(P=1.98 ×10-4,OR:072)、21q22.2区域的rs 2242944(P=1.07 ×10-3,OR:0.73)、IL23R基因rs1004819(P=4.58× 10-3,OR:1.28)。此外,IL23R基因rs11209032(P= 2.75×10-2,OR:1.22)及2p15区域rs10865331(P= 7.00×10-3,OR:1.28)与PsA提示关联(P<0.05)。其中ERAP1基因rs27434与rs27037具有一定程度连锁不平衡(r2值为0.58)。利用logistic回归方法,控制rs27037的遗传学效应后,rs27434显著性消失(P值降为0.1)。通过logistic回归,控制IL23R基因rs1004819的遗传学效应后,rs11209032的p值降为0.65,提示两者为同一信号。既往研究报道ERAP1基因(rs27524、rs151823)、IL23R(rs12131065、rs2201841)与PsV及PsA均相关[1,8-10]。通过Haploview软件和hapmap数据库查询,本次研究的ERAP1基因rs27037、rs27434与rs27524、rs151823具有较强的连锁不平衡(r2值为0.58~0.93)。而IL23R基因rs1004819与既往报道的rs12131065的连锁不平衡程度较低(r2为0.02),与rs2201841则存在一定程度连锁不平衡(r2为0.465)。查阅国内外文献未见PsA与21q22.2区域的相关性文献报道。
2.2.2PsV与正常对照比较见表2 在595例PsV和806例正常对照进行Cochran-Armitage trend检验,共发现2个强直性脊柱炎相关的SNPs与PsV显著关联,分别是ERAP1基因rs27037(P=1.56×10-4,OR: 1.35)和rs27434(P=2.87×10-4,OR:0.76)。利用logistic回归方法,控制rs27037的遗传学效应后,rs27434显著性消失,表明2个SNP为同一关联信号。2p15区域的rs10865331(P=2.55×10-2,OR:1.19)与PsV提示关联(P<0.05),该位点与银屑病的相关性未见文献报道。
2.2.3PsA与PsV异质性分析(见表2) 采用异质性检验方法对与PsA显著相关和提示相关的6个SNP(P<0.05)在PsA和PsV的发病风险进行异质性分析。异质性的程度主要以I2值大小进行判断,同时结合Q检验的P值进行判断,但在样本较小的情况下,P值往往呈假阴性,而I2值更加敏感。结果显示位于IL23R基因的rs11209032和rs1004819,以及21q22.2的rs2242944在PsA和PsV患病风险上存在中等程度的异质性(I2值分别为63.22%、57.41%和71.2%),但Q检验的P值无显著性差异(>0.05)。
2.2.4脊柱炎型、外周关节炎型PsA亚型关联分析及异质性分析见表3 其将PsA患者按照脊柱是否受累分为脊柱炎型PsA及外周关节炎型PsA进行亚型分析,并对发病风险进行异质性分析。列入分析的脊柱炎型PsA患者共186例(包括37例单纯脊柱受累者和149例脊柱和外周关节同时受累者),外周关节型患者为无脊柱受累而仅有外周关节受累的PsA患者193例。结果显示位于IL23R的rs11209032与脊柱炎型PsA相关(P=1.57×10-3,OR:1.52,而在外周关节炎型PsA中未发现具有阳性意义(P=5.53× 10-1,OR:1.07)。这两组异质性分析结果显示该SNP在组间存在较强异质性(I2值76.68%,P=0.04)。
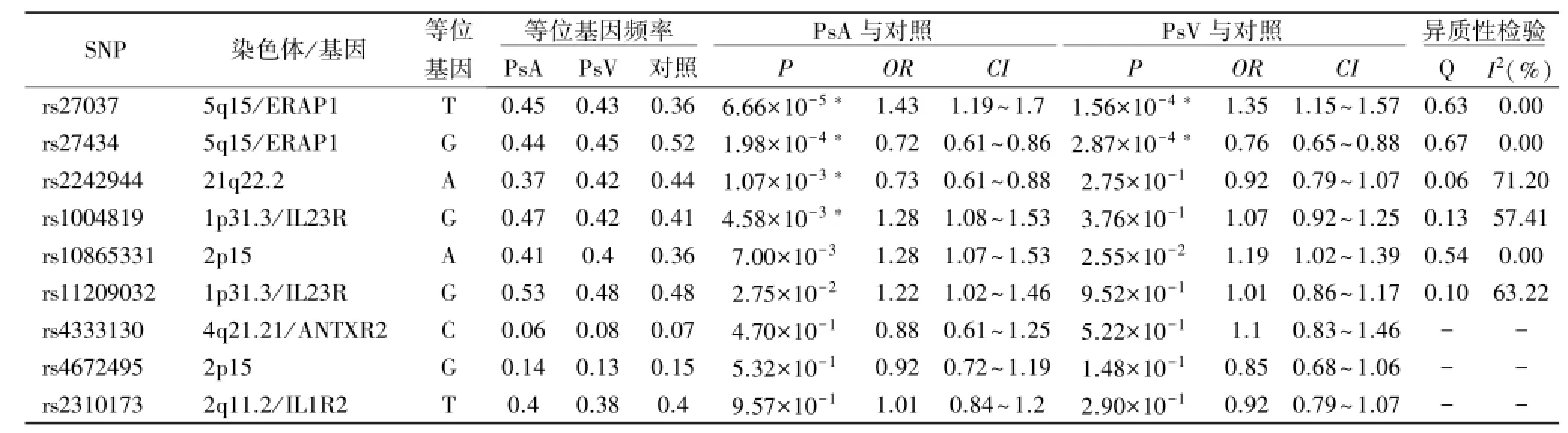
表2 PsA、PsV和正常对照样本关联分析及异质性检验结果

表3 脊柱炎型、外周关节炎型PsA和正常对照样本关联分析及异质性检验结果
3 讨论
近年来随着GWAS研究广泛开展和疾病易感基因的发现,多种临床表型近似或者免疫相关的疾病间共有易感基因的现象逐渐引起关注[2,3,6]。鉴于PsA与强直性脊柱炎在临床表型上、组织病理的相似性,及既往已有某些易感基因交叉存在的证据,本研究以GWAS研究发现的强直性脊柱炎的易感基因位点作为PsA候选位点进行研究,发现染色体21q22.2区域是PsA和强直性脊柱炎共有的易感位点,与PsV无关。在山东汉族人群证实了ERAP1基因是PsA、PsV和强直性脊柱炎共有的易感基因,IL23R是PsA和强直性脊柱炎共有的易感基因。通过对PsA进行临床亚型分析,发现IL23R与脊柱炎型PsA相关。
本次研究的易感位点中与PsA关联信号最强的是ERAP1基因(rs27037,P=6.66×10-5,OR:1.43),该基因位点与PsV亦显著相关,异质性分析显示其在两组间无异质性差异,证实了ERAP1基因是PsA、PsV及强直性脊柱炎共有的易感基因。ERAP1(Endoplasmic reticulum aminopeptidase 1,内质网氨基肽酶)基因编码一种γ干扰素诱导的氨基肽酶。类似多数MHC I类分子,ERAP1倾向于水解带有疏水性C羧基末端的氨基酸,其在疏水的C羧基末端9-16个氨基酸活性残基结合过程中具有独特的分子作用,这提示ERAP1是抗原提呈过程中不可或缺的重要分子基础。ERAP1在内质网中发挥着生物学功能,将多肽抗原修剪到合适的长度结合到银屑病易感位点MHC I类分子上[8]。既往研究发现ERAP1基因(rs27524和rs151823)亦与寻常型银屑病相关[8,9],且本团队以往研究发现rs27524与PsA相关[10]。这两个位点(rs27524和rs151823)与本次研究的AS易感基因位点rs27037和rs27434具有较强的连锁不平衡(r2值为0.58~0.93),这提示强直性脊柱炎和银屑病在ER-AP1基因的易感性具有同源性。
本研究发现的另一与PsA显著关联的强直性脊柱炎易感位点位于21q22.2区域(rs2242944,P=1.07 ×10-3,OR:0.73)。该SNP位点在PsV与对照的等位基因频率相近,无统计学差异。提示21q22.2区域存在PsA和强直性脊柱炎的共有易感基因位点,而与PsV无关。在该区域与rs2242944最近的已知基因是距离82kb的PSMG1基因。研究发现该区域亦与儿童炎症性肠病相关,与之最强关联SNP是rs2836878[11]。该位点与rs2242944连锁不平衡程度较强(r2=0.6,D=1)。该研究发现炎症性肠病患者结肠组织PSMG1基因表达增强,认为该基因可能是炎症性肠病的易感基因[11]。PsA、炎症性肠病性关节炎、强直性脊柱炎三者的临床表现具有很多共性,如常伴有脊柱炎、骶髂关节炎、附着点炎,且HLA-B27阳性率高、常伴虹膜睫状体炎、主动脉根扩张、尿道炎等伴发疾病,所以在风湿科都属于血清阴性脊柱关节病的范畴[4]。这些疾病可能有共同的遗传学背景。然而在强直性脊柱炎外周血单核细胞PSMG1基因的表达与对照相比并无明显差异,且与rs2242944亦无关联性[5]。鉴于该基因的生物学功能与关节炎无明显相关性,PSMG1基因可能并非疾病的真正易感基因,21q22区域可能存在某些非编码RNA序列或者某个尚未发现的基因参与关节病变的发生过程。
本研究中IL23R基因(rs1004819,P=4.58×10-3,OR:1.28)与PsA相关,而与PsV无关。亚型分析显示其关联信号主要来源于脊柱炎型PsA患者(rs11209032,P=1.57×10-3,OR:1.52)。这提示IL23R基因变异可能参与了PsA的脊柱炎和强直性脊柱炎的发病过程的某个环节。既往研究曾报道IL23R与PsA和PsV均相关[12]。近期一项PsA的GWAS研究显示该基因在PsA与PsV均相关,但与PsA相关性更强[2]。本研究选择的2个SNP与既往报道的银屑病易感位点不同(r20.02~0.47),且本研究所用样本量的检验效能有限,因此尚不能排除其与PsV的相关性。IL23R基因编码IL-23受体的一个亚单位,位于T细胞和自然杀伤细胞(NK细胞)上。IL23R通过与IL23结合而参与固有免疫和适应性免疫过程。而IL23对于Th17细胞的分化、维持和增生具有重要的作用[14]。此外,抗p40单克隆抗体对银屑病和PsA的疗效,亦印证了IL12和IL23在银屑病皮损和关节炎症发病中的作用[13]。
综上所述,本研究发现21q22.2区域(rs2242944)为这两种疾病共有易感基因位点,在山东汉族人群证实了PsA与强直性脊柱炎具有共同的易感基因ERAP1基因、IL23R基因,为炎症性关节病发病机制的阐明、共同作用药物的研制,及PsA患病类型风险预测和早期诊断奠定了基础。
[1]Chandran V.The genetics of psoriasis and psoriatic arthritis[J].Clin Rev Allergy Immunol,2013,44(2):149-156.
[2]Stuart PE,Nair RP,Tsoi LC,et al.Genome-wide association analysis of psoriatic arthritis and cutaneous psoriasis reveals differences in their genetic architecture[J].Am J Hum Genet,2015,97(6):816-836.
[3]Uddin M,Sturge M,Rahman P,et al.Autosome-wide copy number variation association analysis for rheumatoid arthritis using the WTCCC high-density SNP genotype data[J].J Rheumatol,2011,38(5):797-801.
[4]O'Neill T,Silman AJ.Psoriatic arthritis.Historical background and epidemiology[J].Baillieres Clin Rheumatol,1994,8(2):245-261.
[5]Australo-Anglo-American Spondyloarthritis Consortium(TASC),Reveille JD,Sims AM,et al.Genome-wide association study of ankylosing spondylitis identifies non-MHC susceptibility loci[J].Nat Genet,2010,42(2):123-127.
[6]Wellcome Trust Case Control Consortium,Australo-Anglo-American Spondylitis Consortium(TASC),Burton PR.Association scan of 14,500 nonsynonymous SNPs in four diseases identifies autoimmunity variants[J].Nat Genet,2007,39(11):1329-1337.
[7]Taylor W,Gladman D,Helliwell P,et al.Classification criteria for psoriatic arthritis:development of new criteria from a large international study[J].Arthritis Rheum,2006,54(8): 2665-2673.
[8]Genetic Analysis of Psoriasis Consortium&the Wellcome Trust Case Control Consortium.A genome-wide association study identifies new psoriasis susceptibility loci and an interaction between HLA-C and ERAP1[J].Nat Genet,2010,42(11):985-990.
[9]Sun LD,Cheng H,Wang ZX,et al.Association analyses identify six new psoriasis susceptibility loci in the Chinese population[J].Nat Genet,2010,42(11):1005-1009.
[10]Yang Q,Liu H,Qu L,et al.Investigation of 20 non-HLA(human leucocyte antigen)psoriasis susceptibility loci in Chinese patients with psoriatic arthritis and psoriasis vulgaris[J].Br J Dermatol,2013,168(5):1060-1065.
[11]Kugathasan S,Baldassano RN,Bradfield JP,et al.Loci on 20q13 and 21q22 are associated with pediatric-onset inflammatory bowel disease[J].Nat Genet,2008,40(10): 1211-1215.
[12]Filer C,Ho P,Smith RL,et al.Investigation of association of the IL12B and IL23R genes with psoriatic arthritis[J]. Arthritis Rheum,2008,58(12):3705-3709.
[13]McKeage K.Ustekinumab:a review of its use in psoriatic arthritis[J].Drugs,2014,74(9):1029-1039.
[14]Suzuki E,Mellins ED,Gershwin ME,et al.The IL-23/IL -17 axis in psoriatic arthritis[J].Autoimmun Rev,2014,13(4):496-502.
(收稿:2016-01-04)
Susceptibility loci of ankylosing spondylitis in patients with psoriatic arthritis
YANG Qing,LIU Hong,FU Xi'an,YU Yongxiang,YU Gongqi,QU Lina,ZHANG Furen. Shandong Provincial Institute of Dermatology and Venereology,Jinan 250022,China
Corresponding author:Zhang Furen,E-mail:zhangfuren@hotmail.com
Objective:To determine the susceptibility loci of ankylosing spondylitis found by GWAS in patients with psoriatic arthritis(PsA).Methods:Nine susceptibility SNPs of ankylosing spondylitis were genotyped in 379 patients with PsA,595 patients with psoriasis vulgaris(PsV)and 806 healthy controls by Sequenom MassARRAY.Results:ERAP1(rs27037,P=6.66×10-5,OR:1.43),chromosome 21q22.2(rs2242944,P=1.07×10-3,OR:0.73)and IL23R(rs1004819,P=4.58×10-3,OR:1.28)were significant association with susceptibility of PsA.ERAP1(rs27037,P=1.56×10-4,OR:1.35)associated with PsV. There was no heterogeneity of ERAP1(rs27037)between PsA and PsV groups and there was a heterogeneity of chromosome 21q22.2(rs2242944)and IL23R(rs1004819)between them.IL23R(rs11209032)was found to be associated with axial PsA(P=1.57×10-3,OR:1.52).Conclusion:ERAP1,21q22.2 domain and IL23R are common susceptibility genes of both PsA and ankylosing spondylitis.
psoriatic arthritis;susceptibility gene;susceptibility loci;ankylosing spondylitis
山东省优秀中青年科学家科研奖励基金计划(编号:BS2013YY010)
山东省皮肤病性病防治研究所,济南,250022
张福仁,E-mail:zhangfuren@hotmail.com

