白芨SSR引物筛选及群体遗传多样性研究
黎 君,杨 恒,周天华,2*
(1 陕西理工大学 生物科学与工程学院,陕西汉中 723001; 2 陕南秦巴山区资源生物综合开发协同创新中心,陕西汉中 723001)
白芨SSR引物筛选及群体遗传多样性研究
黎君1,杨恒1,周天华1,2*
(1 陕西理工大学 生物科学与工程学院,陕西汉中 723001; 2 陕南秦巴山区资源生物综合开发协同创新中心,陕西汉中 723001)
白芨(BletillastriataRchb.)为中国珍稀濒危植物,重要的药用植物。该研究基于Illumina测序技术构建白芨基因组文库和微卫星文库,设计白芨微卫星引物,用白芨4个野生种群80个个体对引物进行多态性检测,应用4个白芨近缘种中进行引物的通用性检测,并在此基础上分析了白芨的遗传多样性和遗传分化,以探讨白芨的遗传结构和进化潜能。结果表明:(1)白芨基因组中微卫星片断丰富,共检测出17 841条微卫星片断。基于白芨微卫星库对100个位点设计了引物对,经PCR扩增和检测筛选出能够稳定扩增的多态性位点20个,每个位点的复等位基因数(Na)在2~6之间,平均为3.85;20对引物的大部分能够在4个白芨近缘种中成功扩增。(2)白芨在物种水平均有较高的遗传多样性(Na=3.85,I=1.07,H=0.614 7),白芨种群遗传分化强烈(Gst=0.43),居群间的基因流较弱(Nm= 0.867 6),居群聚类分析结果均表明地理距离较近的居群具有较近的遗传关系。
白芨;微卫星;遗传多样性;居群;引物筛选
白芨(BletillastriataRchb. f.)是兰科多年生草本植物,生长在海拔100~3 200 m的常绿阔叶林下,栎树林、路边草或岩石缝中。主要分布于四川、云南、陕西、湖北、甘肃等省份,全国各地有少量栽培。花紫红色或粉红色,可供观赏。白芨块茎含白芨胶质(粘液质)、淀粉、挥发油、葡萄糖等。以干燥块茎入药,味苦、甘、涩,性微寒,归肺、肝、胃经,具有收敛止血,消肿生肌的功能,主要用于治疗咳血、吐血、外伤出血、疮疡肿毒、皮肤皲裂、肺结核咳血、溃疡病出血等[1-2]。此外,白芨还是优良的天然粘合剂,可作为工业糊料,用于浆丝绸、棉纱、精表装帧等。
白芨分布范围较广,野生资源比较丰富,但随着医药行业对白芨的进一步开发利用,市场需求量逐年增加,其野生资源遭到过度采挖,生态环境也因多种原因受到严重破坏,药材的后续供应已成为一个严重的问题,因此,白芨的人工栽培已成为解决白芨资源匮乏的必然选择,要选育出品种优良白芨,以及其真实性的检测成为当务之急。近几年来,中国学者在白芨的药理、有效成分和组织培养技术等方面开展了大量的研究,但对白芨遗多样性的研究尚属空白。
简单序列重复(SSR, Simple Sequence Repeat),又称微卫星DNA Microsatel-lite DNA(Litt and Luty 1989)[3],是一类由1~6个碱基组成的基序串联重复而成的DNA序列。不同等位基因间的重复数存在丰富的差异,且重复序列两侧多是相对保守的单拷贝序列,可以通过设计特异引物,对基因组总DNA进行PCR扩增,扩增片段的长度多态性可用作分子标记。微卫星标记多态性丰富,重复性好,标记多呈共显性,在基因组中分散分布,现已证明微卫星DNA存在于绝大多数真核生物基因组中[4],因此已广泛应用于遗传图谱构建、品种指纹图谱绘制、品种纯度检测及目标性状分子标记筛选等领域。特别在人类及哺乳动物的分子连锁图谱中微卫星标记已成为最重要的遗传标记之一,目前植物基因组中微卫星标记研究也非常活跃[5]。植物SSR的分离及克隆先在几个热带树种[6]上展开,其后扩展到大豆、水稻、小麦、油菜、西红柿、甜菜等作物上[7]。
目前国内对白芨种质资源的研究局限于理化水平、组培技术等方面,尚未深入到分子领域,可供使用的SSR引物很少,相关的分子标记也很少,难以满足对白芨的遗传多样性分析,资源评价以及混伪品鉴定的需要。本研究基于第二代测序技术建立白芨基因文库,开发白芨的微卫星(SSR)引物,并应用该分子标记技术对该物种进行群体遗传分析,并提出保护策略,为白芨这种重要资源植物的保护提供理论支撑。
1 材料与方法
1.1实验材料
本研究选取白芨(B.striata)的新鲜块茎为实验材料。分别于2014~2015年采自陕西岚皋(LG)、陕西镇巴(ZB)、四川汉源(HY)和云南玉溪(YX)4个白芨自然分布区(表1),用于白芨引物开发和物种遗传多样性分析。此外,本研究还选取采自陕西留坝和云南的黄花白芨(Bletillaochracea)、小白芨(Bletillaformosana)、华白芨(Bletillasinensis)和绶草(Spiranthessinensis)4个近缘种(表1)来检测引物的通用性。野外采集到新鲜叶片放入自封袋中加硅胶干燥,带回实验室在-80 ℃超低温冰箱中保存备用,并记录样品的海拔、经纬度、生境等信息。所有样品的凭证标本保存于陕西理工学院生物科学与工程学院植物分子生物学实验室。
1.2方法
1.2.1白芨DNA提取采用改良2×CTAB法[8]提取白芨4个野生居群80份个体的基因组DNA,记录DNA浓度和A260/A280吸光比值并用浓度为1%琼脂糖凝胶检测DNA完整性,凝胶成像仪拍照观察并记录结果。
1.2.2白芨微卫星文库的建立提取白芨全基因组DNA,构建白芨基因组文库,采取Illumina二代测序技术获取大量100 bp短序列片段。
采用CLC Genomics Workbench[9]软件进行片段组装,设置过滤参数为:片段长度大于299 bp,序列平均覆盖率为25倍,初步筛选出适合微卫星位点开发的潜在序列集合。设置一定阈值,初步筛选出适合微卫星位点开发的潜在序列集合。基于筛选出的序列比对,导出一致性序列,以fasta格式保存成单一文件。以SciRoKo v3.3[10]软件扫描上述文件,统计出微卫星位点情况,包括各类重复位点数量、比例,对应原始序列名称等信息。二代测序建库工作由北京NOVOGENE生物科技公司代理完成。
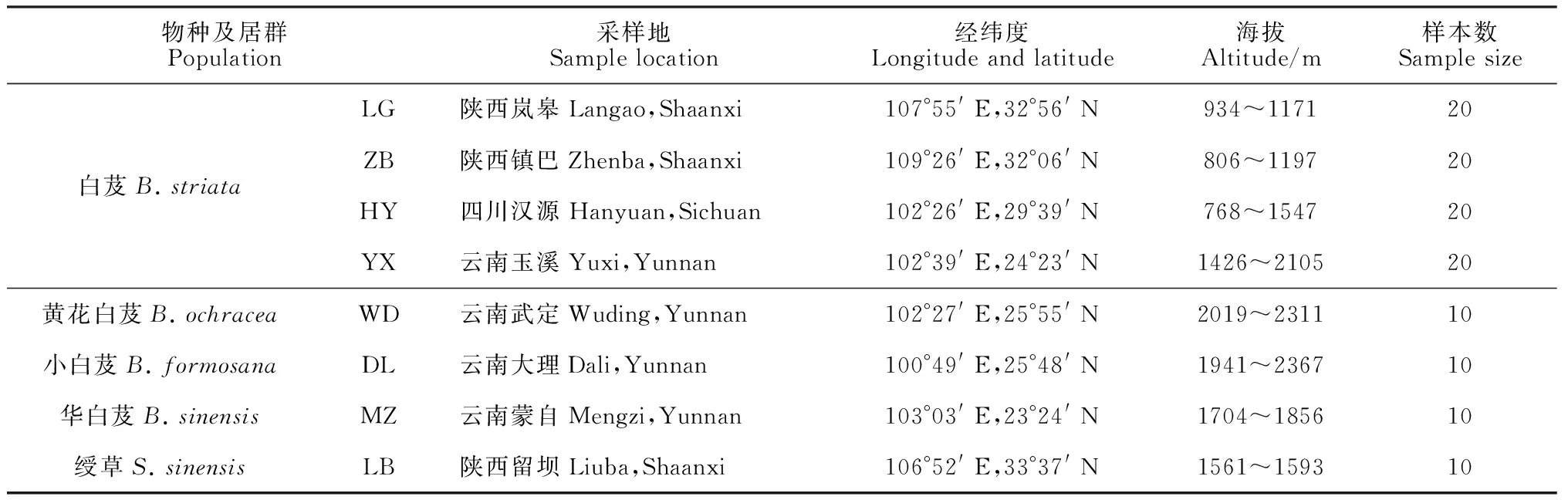
表1 白芨及其近缘种居群采样基本数据
1.2.3白芨SSR引物设计采用Oligo 7[11]软件设计SSR引物100对。利用软件设计引物的时候必须遵循以下基本原则:①引物最好在模板cDNA的保守区内设计;(2)引物长度一般在15~30 bp之间;(3)引物的GC含量在40%~60%之间,Tm值最好接近72;(5)引物自身及引物之间不应存在互补序列;(6)引物5′端及3′端ΔG的绝对值应该小于4;(7)扩增产物的单链不能形成二级结构;(8)引物3′端不能选择A,最好选择T;选取软件综合评分在95分以上的35对送往北京奥科鼎盛生物科技有限公司进行引物合成。
1.2.4PCR扩增与聚丙烯酰氨凝胶电泳检测PCR总反应体系为20 μL,包括DNA模板50 ng,2×Mix(天根生物科技)10 μL,正反引物各50 ng,ddH2O至20 μL。PCR反应在PCR扩增仪(Bio-Rad S1000TM)上按以下程序运行:首先94 ℃预变性3 min;94 ℃变性30 s,退火温度下45 s,72 ℃下延伸45 s,35个循环;最后72 ℃延伸7 min,4 ℃保存。
完成PCR循环48 h内,取2 μL PCR产物在8%非变性聚丙烯酰胺凝胶上恒电压160 V电泳14 h。用NaOH银染方法进行染色显影。各种溶剂配方和体积为:银染液,AgNO31 g,去离子水500 mL;显影液, NaOH 15 g,甲醛2 mL,去离子水500 mL。银染程序为:去离子水漂洗30 s,银染30 min,去离子水漂洗30 s,在显影液中轻摇至显带。显影完成之后,用凝胶成像系统(Gel DOCTM XR+)采集图像并进行数据统计分析。
1.2.5引物筛选及通用性检测用35对引物对4个白芨居群的DNA进行检测,采用聚丙烯凝胶电泳检测,电泳结束后,按照NaOH银染方法进行染色显影,并拍照保存。从35对引物中筛选出20对条带比较明显,且和预期长度相近的SSR引物。使用这些引物对白芨的4个近缘种进行聚丙烯酰氨凝胶电泳检测,观测扩增产物及目标产物的杂合度。
1.2.6白芨种群的遗传多样性分析使用Excel 2007和Bio-rad quantity软件对电泳后的凝胶图像进行条带统计,采用人工读带的方式,记录主要带型,忽略弱杂带。应用Powermarker 3.25[12]软件,计算各引物的等位基因数目(Na),期望杂合度(He),观测杂合度(Ho),多态性信息含量(PIC)等(表2)。应用POPGENE Version 1.32[13]软件,分析各种群的平均复等位基因数(Na)、遗传杂合度(Ne)、Nei’s遗传多样性指数(H)、Shannon指数和遗传分化系数(Fst)等(表3)。应用GenALEx 6.3软件[14]进行AMOVA分析、各种群地理距离和遗传距离的相关性分析及居群主相关性分析。此外还使用采用MEGA5.0软件基于各居群的遗传距离进行UPGMA聚类。
2 实验结果
2.1白芨微卫星文库
二代测序共返回40 715 734条125 bp配对序列,采用CLC Genomics Workbench软件进行组装得到576 466条拼接序列,用于后续分析;采用该软件进行片段组装,设置过滤参数为:片段长度大于299 bp,序列平均覆盖率为25倍,初步筛选出适合微卫星位点开发的潜在序列集合,得到拼接序列数23 906条。虽然上述筛选后的组装序列平均覆盖率均达到25倍,但部分数据内部覆盖率不均匀,局部覆盖率较低。在此情况下,去除内部覆盖率低于5倍的区域,并将一致性序列分为若干“亚片段”,以增加最终用于微卫星开发母序列的可靠性。最终,得到序列17 841条。基于筛选出的序列比对,导出一致性序列,以fasta格式保存成单一文件。以SciRoKo v3.3软件扫描上述文件,统计出微卫星位点情况,包括各类重复位点数量、比例,对应原始序列名称等信息。

2.2微卫星引物多态性分析
利用POPGENE Version软件对筛选出的白芨微卫星位点进行群体遗传多样性分析,得到了等位基因数以及观察杂合度和期望杂合度等群体多态性信息。由表2和表3可知,20对扩增成功的微卫星引物中检测到2~6个不等的等位基因数,平均等位基因数目为3.85个。多态性信息含量范围为0.357 2~0.722 9。这些微卫星引物的Ho为0.350 0~0.700 0,平均值为0.485 6;He为0.468 5~0.765 3,平均值为0.618 5。就单个引物位点而言,通常其He总是大于Ho[15]。通过分析Ho和He值,说明各个群体之间存在较丰富的遗样性。
2.3群体遗传多样性分析
筛选出的20对白芨微卫星引物能够成功扩增样品的DNA(图1);这些引物扩增产物的等位基因数目为2~6个,观察杂合度范围为0.350 0~0.700 0,期望杂合度范围为0.468 5~0.765 3,通过AMOVA分析得出,Gst为0.43,说明有57%变异来自于种群内,有43%变异来自于种群间,种群间存在着显著的遗传分化。Mantel Test检测结果(表4)表明,白芨居群之间的遗传距离与其相应的地理距离无显著的正相关关系(r=0.294 3,P< 0.01)。
2.4基于SSR标记的聚类分析
用软件进行聚类分析,构建UPGMA聚类树(图2)显示,4个白芨野生居群在遗传上有明显的差异,地理距离近的LG居群和ZB居群进化关系较近,而YX居群和其他3个居群进化关系较远。使用GenAlEx 6.3 软件进行居群主相关性分析,结果(图3)与UPGMA聚类分析的结果完全一致,4个白芨野生居群完全分开。

表4 白芨各居群间Nei’s遗传距离(下三角)和
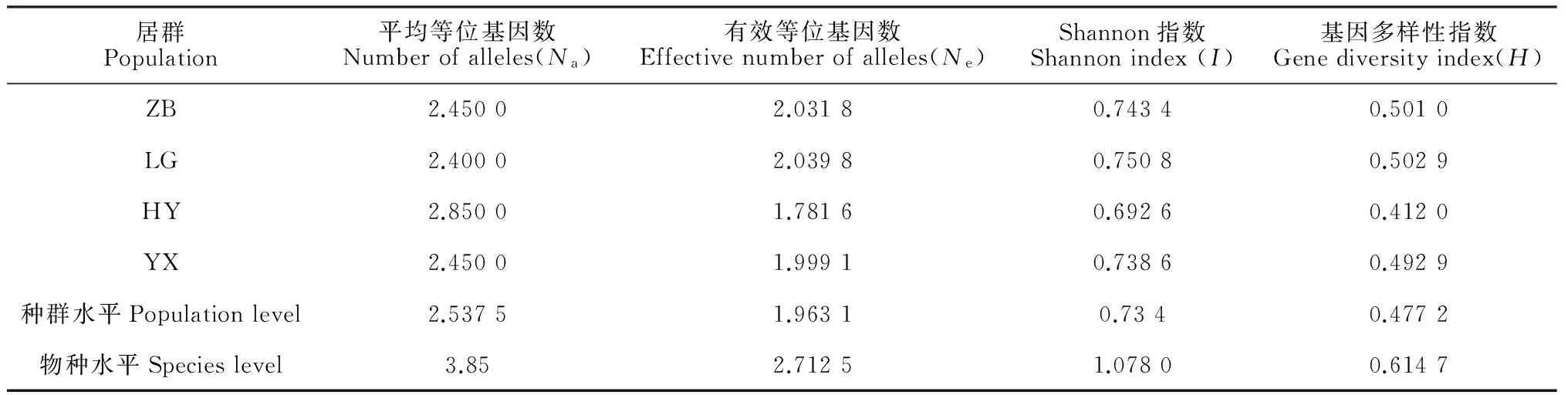
表3 白芨居群间基因流及遗传分化参数
注: 居群代号同表1;下同
Note: Population codes are same as in Table 1; The same as below

M.PUC-18;1~20.HY居群个体图1 引物Bjssr14对四川汉源(HY)居群的SSR扩增1~20.Samples of HY populationFig. 1 SSR amplification products generated by the primer pairs Bjssr14 for HY(Sichuan hanyuan)population

图2 基于Nei’s遗传距离的白芨自然居群的UPGMA聚类树Fig. 2 UPGMA dendrogram of B. striata populations using Nei’s unbiased genetic distances

图3 基于GD的白芨自然居群主相关性分析Fig. 3 PCA analysis of B. striata populations using genetic distances
2.5引物通用性检测
通过筛选得到20对能够稳定扩增且多态性丰富的白芨微卫星引物,使用这些引物对采集的4个白芨近缘种(华白芨、小白芨、黄花白芨和绶草)样本进行扩增,利用聚丙烯酰胺凝胶电泳对扩增结果进行检测。结果表明,华白芨、小白芨和黄花白芨种群中,20对引物均能检测出等位基因(表5),供试的40份样品中30份能够检测出条带,因此,筛选出的引物可以在白芨属植物中通用,在兰科其他属中也有一定的通用性。
3 讨 论
本研究通过对白芨4个野生居群的聚丙烯酰氨凝胶电泳实验,筛选出20对稳定性好,多态性信息位点丰富的SSR引物。对80份白芨野生种质资源的实验结果分析表明,20对引物共获得77个等位变异点,平均每对引物3.85个,表明筛选出的微卫星引物多态性丰富。多态性信息含量(PIC)范围为0.357 2~0.722 9,平均值为0.547 9,说明本研究构建的SSR标记体系对于4个白芨野生居群遗传多样性的分析十分有效。因为重复序列和包含SSR位点的侧翼序列在物种间具有保守性, 所以通常一个物种的微卫星引物在同科不同属或者更近物种间具有适用性[16]。提高微卫星利用率的一个办法是一旦微卫星位点在某一物种中分离并测序, 它就可被用于近缘种的分析[17]。本研究设计筛选的白芨SSR引物能够在4个白芨近缘种共40份样品中成功扩增,说明SSR引物通用性良好。因此,筛选出的白芨SSR引物能够被用于白芨群体遗传学分析的研究,同时也能够用于白芨属其他近缘物种的群体遗传学研究。这对于利用SSR分子标记对全国范围白芨野生种质资源的分析与评价,提供了科学依据。
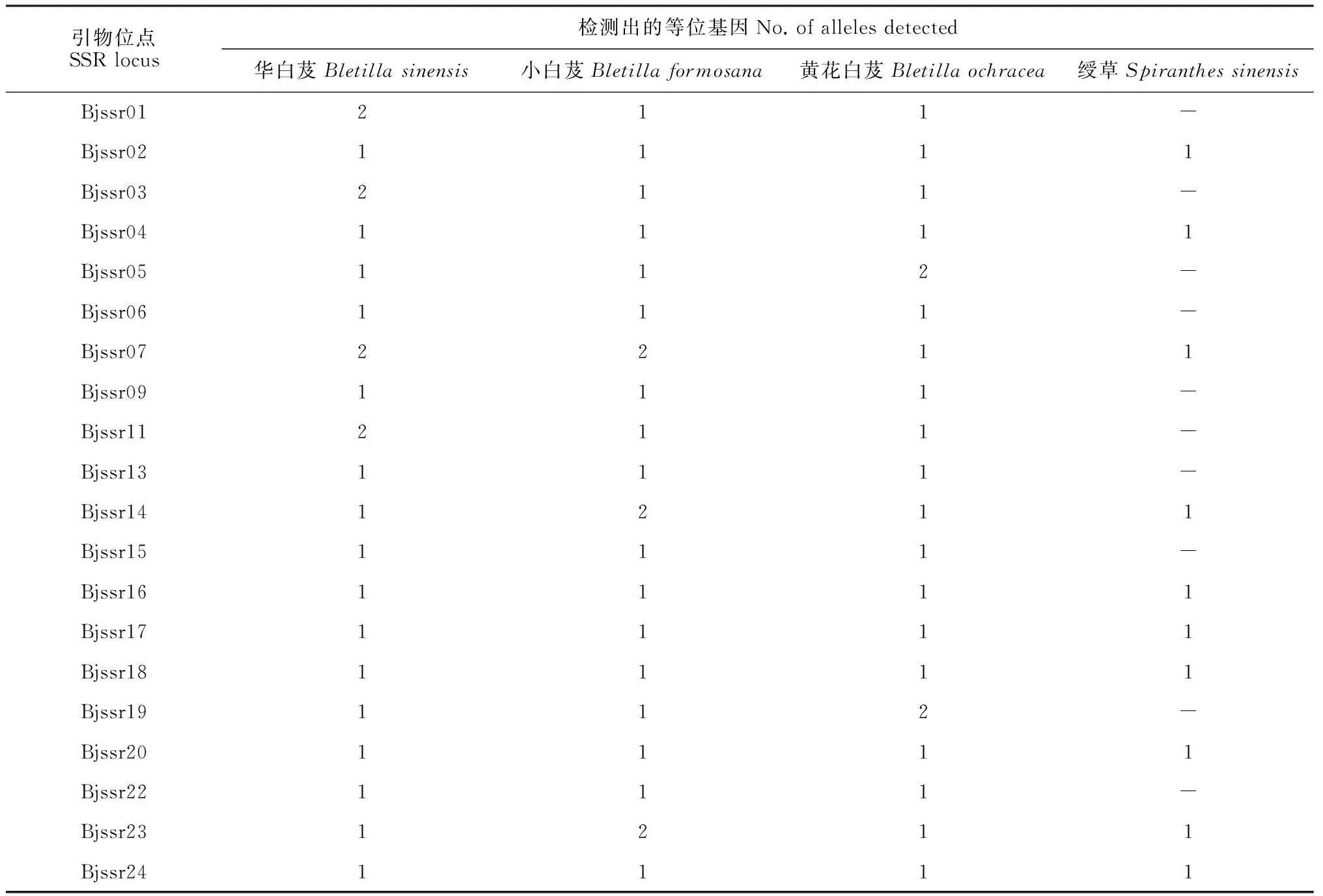
表5 20对引物对4个白芨近缘种的通用性检测(n=10)
注: n.每种群个体数;1.观测到纯合子;2.观测到杂合子
Note: n.Number of individuals; 1.Observed homozygote; 2.Observed heterozygote
自然界中,生物所表现出来的遗传多样性,其在本质上就是DNA的差异。因此通过分子手段来分析物种或群体遗传多样性更为直接,也更为准确。它直接分析遗传物质本身,是分析生物的基因型而非表现型,排除了环境因素给药材鉴定带来的干扰,也不受样品形态和材料来源的影响,比传统的方法更准确可靠。它不仅能区分相近物种,而且可以检测外型极为相似的同种不同变种、变型、品系、化学型乃至个体之间的DNA 的细微变异。本研究利用SSR分子标记技术,发现白芨在物种水平上,观察杂合度范围为0.350 0~0.700 0,期望杂合度范围为0.468 5~0.765 3,多态性信息含量范围为0.357 2~0.722 9,通过AMOVA分析显示,有57%变异来自于种群内,有43%变异来自于种群间,说明白芨在物种水平上的遗传多样性非常丰富。
有研究表明,在地理分布上受限的植物具有较低的遗传多样性,相反,分布范围越广的物种具有越高的遗传多样性[18-20]。白芨在物种水平上具有较高的遗传多样性,可能与白芨属植物的生活史和自然分布有关。白芨属植物大多分布在海拔100~3 200 m的常绿阔叶林下,栎树林、路边草或岩石缝中。该属植物属于起源比较晚的物种类别,分布区域较广,在四川、云南、陕西、湖北、甘肃、安徽等地的山区均有分布,并且多省均有大面积种植。一个物种对环境变化的适应能力主要取决于该物种是否具有丰富的遗传多样性,因此,虽然由于人为过度采挖以及自然变迁的影响导致白芨的自然资源稀少,但是该属植物频繁的基因交流使其具有丰富的遗传变异,使其能够很好地生存下来。
白芨(BletillastriataRchb. f.)是中国二级保护植物,也是重要的药用植物,野生资源枯竭,亟需保护。白芨在人工栽培条件下繁殖速度很慢,繁殖系数极低,尽管近几年对白芨的组织培养研究较多,在一定程度上缓解了需求增长与资源危机的矛盾,但因药材道地性观念的影响而造成的野生白芨与栽培白芨市场价格的悬殊,导致了人们对野生白芨的过度采挖,加上兰科植物的种子萌发率极低,自然繁殖困难,所以白芨野生资源较稀少,已被列人国家珍稀濒危保护植物名录,因此野生白芨种质资源的有效保育已刻不容缓。近年来,SSR 分子标记技术已被广泛应用于药用植物的遗传多样性分析[21-23]。本实验运用 SSR 分子标记技术,对白芨的野生居群进行遗传多样性分析,结果显示,白芨居群具有较丰富的遗传多样性水平,从DNA水平上验证了白芨居群丰富的遗传变异能力,说明白芨具有较强的进化潜能,为后续白芨属植物资源的保护、利用和评价奠定了良好基础。
[1]马立祥, 赵甍, 毛子军, 等. 不同氮素水平下增温及CO2升高综合作用对蒙古栎幼苗生物量及其分配的影响[J]. 植物生态学报, 2010, 34(3): 279-288.
MA L X, ZHAO M, MAO Z J,etal. Effects of elevated temperature and (CO2) under different nitrogen regimes on biomass and its allocation inQuercusmongolicaseedlings[J].ChineseJournalofPlantEcology, 2010, 34(3): 279-288.
[2]扬春澍. 药用植物学[M]. 上海: 上海科学技术出版社, 1998: 321-327.
[3]TAUTZ D. Hyper variability of simple sequences as ageneral source for polymorphic DNA markers[J].NucleicAcidsResearch, 1989, 17: 6 463-6 471.
[4]LITT M, LUTY J A. A hypervariable microsatellite revealed by invitro amplification of dinucleotide repeat within the cardiac muscle actin gene[J].AmericanJournalofHumanGenetics, 1989, 44: 397-401.
[5]TAUTZ D, RENZ M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes[J].NucleicAcidsResearch, 1984, 12: 4 127-4 138.
[6]陈佩度. 作物育种生物技术[M]. 北京: 中国农业出版社, 2001: 134.
[7]POWELL W, MACHRAY G C, PROVAN J. Polymorphism revealed by simple sequence repeat[J].TrendsinPlantScience, 1996, 70: 45-54.
[8]周天华. 中国特有属植物——马蹄香(Saruma henryi Oliv.)的分子谱系地理学与遗传多样性研究[D]. 西安: 西北大学, 2010.
[9]朱大强, 李存, 陈斌, 等. 四种常用高通量测序拼接软件的应用比较[J]. 生物信息学, 2011, 9(2); 106-110.
ZHU D Q, LI C, CHEN B,etal. Comparison of the widely used high-throughput sequencing assembly software[J].ChinaJournalofBioinformatics, 2011, 9(2); 106-110.
[10]Robert Kofler, Christian Schlötterer, Tamas Lelley. SciRoKo: A new tool for whole genome microsatellite search and investigation[J].Bioinformatics, 2007. 23(13):1 683-1 685.
[11]张新宇, 高燕宁. PCR引物设计及软件使用技巧[J]. 生物信息学, 2004, 2(4): 15-18.
ZHANG X Y,GAO Y N. To design PCR primers with Oligo 6 and Primer Premier 5[J].ChinaJournalofBioinformatics, 2004, 2(4): 15-18.
[12]LIU K, MUSE S V. PowerMarker: an integrated analysis environment for genetic marker analysis[J].Bioinformatics.2005,21(9):2 128-2 129.
[13]YEH F C, YANG R C, BOYLE T B J,etal. POPGENE, the User-friendly Shareware for Population Genetic Analysis[M]. Molecular biology and biotechnology centre, University of Alberta, Canada, 1997:997.
[14]PEAKALL R, SMOUSE P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research[J].MolecularEcologyNotes, 2005, 6(1); 288-295.
[15]李忠虎, 刘晓东, 王小琦, 等. 党参微卫星引物筛选及群体遗传多样性研究[J]. 中草药, 2013, 44(2); 210-214.
LI Z H, LIU X D, WANG X Q,etal. Microsatellite primer screening and population genetic diversity ofCodonopsispilosula[J].ChineseTraditionalandHerbalDrugs, 2013, 44(2); 210-214.
[16]陈怀琼,隋春,魏建, 等. 植物SSR引物开发策略简述[J]. 分子植物育种, 2009,7(4): 845-851.
CHEN H Q, SUI C, WEI J,etal. Summary of strategies for developing SSR primer[J].MolecularPlantBreeding, 2009, 7(4):845-851.
[17]王丽, 赵桂仿. 植物不同种属间共用微卫星引物的研究[J]. 西北植物学报,2005, 25(8): 1 540-1 546.
WANG L, ZHAO G F. Micro-satellite primers shared by different plant species and genera[J].ActaBotanicaBoreali-OccidentaliaSinica. 2005, 25(8):1 540-1 546.
[18]HAMRICK J L, GODT M J W. Allozyme diversity in plant species[C]// BROWN A H D, CLEGG M T,KAHLER A L, WIER B S, eds. Plant Population Genetics, Breeding, and Genetic Resources. Sunderland, Mass: Sinauer: Sinauer Associates, 1990: 43-63.
[19]KARRON J D. Patterns of genetic variation and breeding systems in rare plant species[C]// FALK D A, HOLSINGER K E. eds. Genetic and Conservation of Rare Plants. USA: Oxford University Press, NY, 1991, 87- 98.
[20]HAMRICK J L, GODT M J W. Conservation genetics of endemic plant species[C]// AVISE J C, HAMRICK J E. Conservation Genetics: Case Histories From Nature. New York: Chapman and Hall, 1996, 281-304.
[21]李凤岚, 马小军. 药用植物分子遗传图谱研究进展[J]. 中草药, 2008, 39(1): 129-133.
LI F L, MA X J. Advances in studies on molecular linkage map in medicinal plants[J].ChineseTraditionalandHerbalDrugs. 2008, 39(1): 129-133.
[22]葛淑俊, 孟义江, 李广敏, 等. 我国药用植物遗传多样性研究进展[J]. 中草药, 2006, 37(10): 1 584-1 589.
GE S J, MENG Y J, LI G M,etal. Research progress on genetic diversity in Chinese medicinal plants[J].ChineseTraditionalandHerbalDrugs, 2006, 37(10): 1 584-1 589.
[23]吴天姝, 梁翠, 李宏博, 等. 人参单一基因微卫星标记的分析[J]. 中国农业科学, 2011, 44(13): 2 650-2 660.
WU T S, LIANG C, LI H B,etal. Development of unigene derived microsatellite (UGMS) markers in Panax ginseng[J].ScientiaAgriculturaSinica, 2011, 44(13): 2 650-2 660.
(编辑:宋亚珍)
Microsatellite Primer Screening and Population Genetic Diversity ofBletillastriata(Thunb.) Rchb. f.
LI Jun1,YANG Heng1,ZHOU Tianhua1,2*
(1 College of Biological Science and Engineering, Shaanxi Sci-Tech University,Hanzhong Shaanxi 723001, China;2 Qinling-Bashan Mountains Bioresources Comprehensive Development Collaborative Innovation Center,Hanzhong, Shaanxi 723001, China)
Bletillastriata(Thunb.) Rchb. f. is an endangered plant in China with important medicinal value. In this study, the genomic sequence library and microsatellite library ofB.striatawas set up based on Illumina sequencing. SSR primers were designed and carried out to test their polymorphism with 80 individuals from 4 wild populations. Four related species ofB.striatawere used to investigated their cross-taxa transferability. In addition, genetic diversity and genetic differentiation of the species were also investigated. The results showed that: (1) Microsatellite sequences were abundance in the genome ofB.striata, 17 841 loci were detected in total.100 pair of primers were designed, 20 of them performed high polymorphism after PCR and polyacrylamide gelelectrophoresis, the alleles ranged from 2 to 6 with average of 3.85 per locus. The cross-taxa transferability was also examined in 4 related species, most of 20 loci were proved amplifiable in these species. (2) The study revealed that the genetic diversity ofB.striatawas high (Na=3.85,I=1.07,H=0.614 7), genetic differentiation was prominent among populations (Gst=0.43). The gene flow among populations was limited (Nm=0.867 6), the adjacent populations performed tight genetic relationship as revealed by cluster analyses of populations. The reasons for genetic construction and conservational strategies of the species were also discussed in this study.
Bletillastriata(Thunb.) Rchb. f.; microsatellite; genetic diversity; population; primer screening
1000-4025(2016)07-1343-08
10.7606/j.issn.1000-4025.2016.07.1343
2016-04-15;修改稿收到日期:2016-06-15
陕西省自然科学基础研究计划(2015JM3115);秦巴山区生物资源综合开发协同创新中心2016年度自然科学基金(QBXT-Z(P)-15-10);陕西理工学院2015年研究生创新基金(SLGYCX1524);陕西省科技厅项目(2015KTTSSF01-02)
黎君(1990-),男,在读硕士研究生,主要从事植物分子生态学研究。E-mail:lijun5525@163.com
周天华,硕士生导师,主要从事植物资源学研究。E-mail:444721689@qq.com
Q346+.5;Q789
A

