加拿大棘球绦虫的基因分型与分子流行病学研究进展
李双男,闫鸿斌,李 立,娄忠子,付宝权,2,殷 宏,2,贾万忠,2
加拿大棘球绦虫的基因分型与分子流行病学研究进展
李双男1,闫鸿斌1,李立1,娄忠子1,付宝权1,2,殷宏1,2,贾万忠1,2
1.中国农业科学院兰州兽医研究所/家畜疫病病原生物学国家重点实验室/农业部兽医公共卫生重点实验室甘肃省动物寄生虫病重点实验室, 兰州7300462.江苏省动物重要疫病与人兽共患病防控协同创新中心,扬州225009
摘要:通过比较加拿大棘球绦虫不同基因型的线粒体基因组,尤其是其中的cox1和nad1基因核苷酸序列的差异性,了解加拿大棘球绦虫各基因型的分子遗传标记特征与变异情况,阐明加拿大棘球绦虫基因型在棘球属中的分类地位、命名和进化关系。另外,本文通过对加拿大棘球绦虫各基因型的分子流行病学特征、研究意义、未来研究方向等进行综述,为从事该领域研究的学者和临床工作者提供丰富的分子流行病学信息或资料,并为棘球蚴病分子流行病学调查、预警预报和综合防治策略的制定等提供理论依据和指导。
关键词:加拿大棘球绦虫;基因型;分子遗传标记;线粒体基因;分类学;流行病学
Supported by the Gansu Provincial Key Project of Scientific and Technical Supporting Projects (No. 1203NKDA039), the Special Fund for Agro-scientific Research in the Public Interest (Nos. 200903036-07, 201303037, 201503-047), the Science Fund for Creative Research Groups of Gansu Province(No. 1210RJIA006), and the NBCITS MOA (No. CARS-38)
细粒棘球绦虫(Echinococcusgranulosus)隶属于绦虫纲、带科、棘球属(Echinococcus),其中绦期幼虫—细粒棘球蚴是引起人和家畜棘球蚴病的病原体。由于地理分布上的差别和对宿主适应性的不同,细粒棘球绦虫在长期的进化过程中,其遗传物质发生了变异和进化[1]。细粒棘球绦虫种内变异现象突出,其分类和命名正发生着显著的变化。一些新的分子生物学和流行病学数据表明,先前命名的细粒棘球绦虫虫株或基因型(如G1—G10)中的一些必须被单独视为物种[2-3]。造成这种分类与命名变化的原因除了遗传距离上的差异以外,主要是在同一地区这些类群保持着遗传稳定性和宿主特异性方面的差异[4]。研究人员通过对分离株样品线粒体细胞色素c氧化酶亚基1(Cytochrome c oxidase subunit 1,cox1)和NADH脱氢酶亚基1(NADH dehydrogenase subunit 1,nad1)以及核糖体RNA基因(rDNA)的部分片段序列、线粒体基因组(mitochondrial genome, mt genome)序列等分子遗传标记特征,成功地对细粒棘球绦虫不同虫株进行了基因分型、进化关系分析、重新分类等。有学者建议将细粒棘球绦虫G1—G3基因型定名为细粒棘球绦虫狭义种(其中G1就是常见的绵羊株),而G4—G10型定名为细粒棘球绦虫广义种,其中G4定名为马棘球绦虫(E.equinus),G5定名为奥氏棘球绦虫(E.ortleppi),G6—G10定名为加拿大棘球绦虫(E.canadensis)[5]。这种依据寄生虫的基因型特征的分类与命名对明确其在包括人在内的中间宿主之间的传播方式与开展分子流行病学调查上非常重要。本文主要通过综述加拿大棘球绦虫的基因分型、各基因型的分子遗传标记、宿主范围、对人和动物的致病力、流行病学意义等方面的差异,从而给棘球蚴病流行病学调查、预警预报、综合防控措施的制定、诊断试剂和疫苗的研发等提供重要依据和指导意义。
1基因分型的分子鉴定方法
目前已有多种分子生物学方法用于研究细粒棘球绦虫的变异研究与基因分型,其方法主要有限制性片段长度多态性、聚合酶链式反应连接的限制性片断长度多态性、随机扩增多态性DNA、扩增片断长度多态性、单链构象多态性、DNA序列分析、微卫星DNA等,本文主要针对加拿大棘球绦虫的常用分类研究方法作简要概述。
1.1聚合酶链式反应连接的限制性片断长度多态性(PCR-RFLP)PCR-RFLP的原理是用一对特异的引物对基因组DNA某一段序列进行PCR扩增,扩增产物用限制性内切酶消化,经琼脂糖凝胶电泳分离和溴化乙锭染色后,在紫外光下进行检测。与常规的RFLP相比,该方法具有快速简便、成本较低等优点。Rosenzvit等[6]对细粒棘球绦虫进行rDNA的PCR-RFLP分析,首次发现G6基因型对人体具有感染性。
1.2DNA序列分析DNA序列中含有丰富的进化信息,不同的生物种类存在不同的DNA序列,且具有稳定性。G7基因型具有独特的DNA特征,可与其他基因型相区别。G7基因型主要寄生于猪的肝脏、肺脏,其他内脏少见,对猪有高度感染力,对羊感染力低。Okamoto等[7]对多个棘球绦虫虫株cox1基因部分片段(391 bp)的核苷酸序列进行了对比研究,结果发现其种间核苷酸序列差异超过12%。Bowles等[8]通过mtDNA的nad1基因序列分析和rDNAITS1(核糖体RNA基因的内转录间隔区1,ITS1)的PCR-RFLP分析,鉴定出了一种新的基因型即G8。利用此方法,Lavikainen等[9]在芬兰发现了另一种新的基因型G10。分子生物学技术的发展极大地促进了细粒棘球绦虫虫株鉴定的深入研究。随着科技的发展,鉴定虫株变异情况的方法也日益增多,研究人员可以根据需要选择快捷简便的分型方法或者遗传变异分析方法。使用核DNA的调查多涉及核基因组区域,比如Bowles和McManus等人[8]对ITS1的研究,Bart等人[10]使用编码抗原B/1、BG1的基因DNA探针以及肌动蛋白III基因等,Moro等[11]对伸长因子1α的研究以及Scott和McManus[12]对随机扩增多态性DNA标记的研究等。此外,使用线粒体基因进行遗传变异的分析,大多涉及到cox1和nad1基因序列的测定[13-14],聚合酶链反应-单链构象多态性或者PCR-RFLP分析以及12S rRNA、atp6基因乃至线粒体全基因组的序列分析[15-19]。这些方法都被证明能有效分析棘球绦虫的虫株变异和进行基因分型。随着科技的发展,用于鉴别虫株遗传变异分析和基因分型的方法也日益增多,研究人员可以根据需要选择快捷简便的方法。
2G6—G10基因型的分子遗传标记特征
2.1线粒体基因cox1和nad1基因部分序列比较
目前有许多分子遗传标记用于加拿大棘球绦虫基因分型的研究,但是对cox1和nad1基因片段部分核苷酸序列的研究应用最广泛。G6(骆驼株)、G7(猪株)和G9(猪株)基因型之间以及G8(鹿株)和G10(麋鹿株)基因型之间核苷酸序列相似性很高。G6最早是由Wachira等人在骆驼和山羊体内发现并命名的[20]。G6(GenBank登录号:AB208063)、G7(AB235847)、G8(AB235848)和G10(AB745463)cox1和nad1基因核苷酸序列间的差异性比较见表1。
表1加拿大棘球绦虫各基因型cox1和nad1序列差异性比较
Tab.1Differences of the cox1 and nad1 sequences among E. canadensis genotypes

基因型(虫株)Genotype(strain)骆驼株(G6)Camelstrain(G6)猪株(G7)Pigstrain(G7)鹿株(G8)Cervidstrain(G8)麋鹿株(G10)Fennoscandiancervidstrain(G10)骆驼株(G6)Camelstrain(G6)-0.0060.0320.021猪株(G7)Pigstrain(G7)0.003-0.0350.027鹿株(G8)Cervidstrain(G8)0.0330.035-0.029麋鹿株(G10)Fennoscandiancervidstrain(G10)0.0200.0220.035-
注:“-”线之上的数据为nad1基因核苷酸序列比较的结果,而“-”线之下的数据为cox1基因核苷酸序列比较的结果。
Note: Dates above the line "-" are the results ofnad1 gene sequences comparisons, and dates under the line "-"are the results ofcox1 gene sequences comparisons.
由表1可知,G6—G10基因型线粒体序列差异不大,cox1序列和nad1序列相似性均达95.6%以上。尽管如此,加拿大棘球绦虫的分类地位仍有争议,从表中也可看出G6与G7基因型cox1序列和nad1序列相似性最大。Thompson认为G6和G7应被分为加拿大棘球绦虫中间型[21]。但是,这种命名似乎不太合适,因为对加拿大棘球绦虫中间型的原始描述没有涉及中间宿主的特异性[22]。目前还没有任何关于中间型G6或G7的形态、遗传、生态研究。因此,基因型G6—G10的比较遗传学研究是加拿大棘球绦虫重新分类的前提,也是明确不同论文间差异的依据[15,21,23-25]。至于Scoot等人关于G9基因型的报道至今还尚未被证实,因为至今尚没有机会对其进行更深入的研究[26-27]。就总体研究来看,目前的证据支持棘球绦虫的分离株G6、G7、G8和G10基因型是独立进化谱系,因此将其合并为加拿大棘球绦虫有一定的依据。
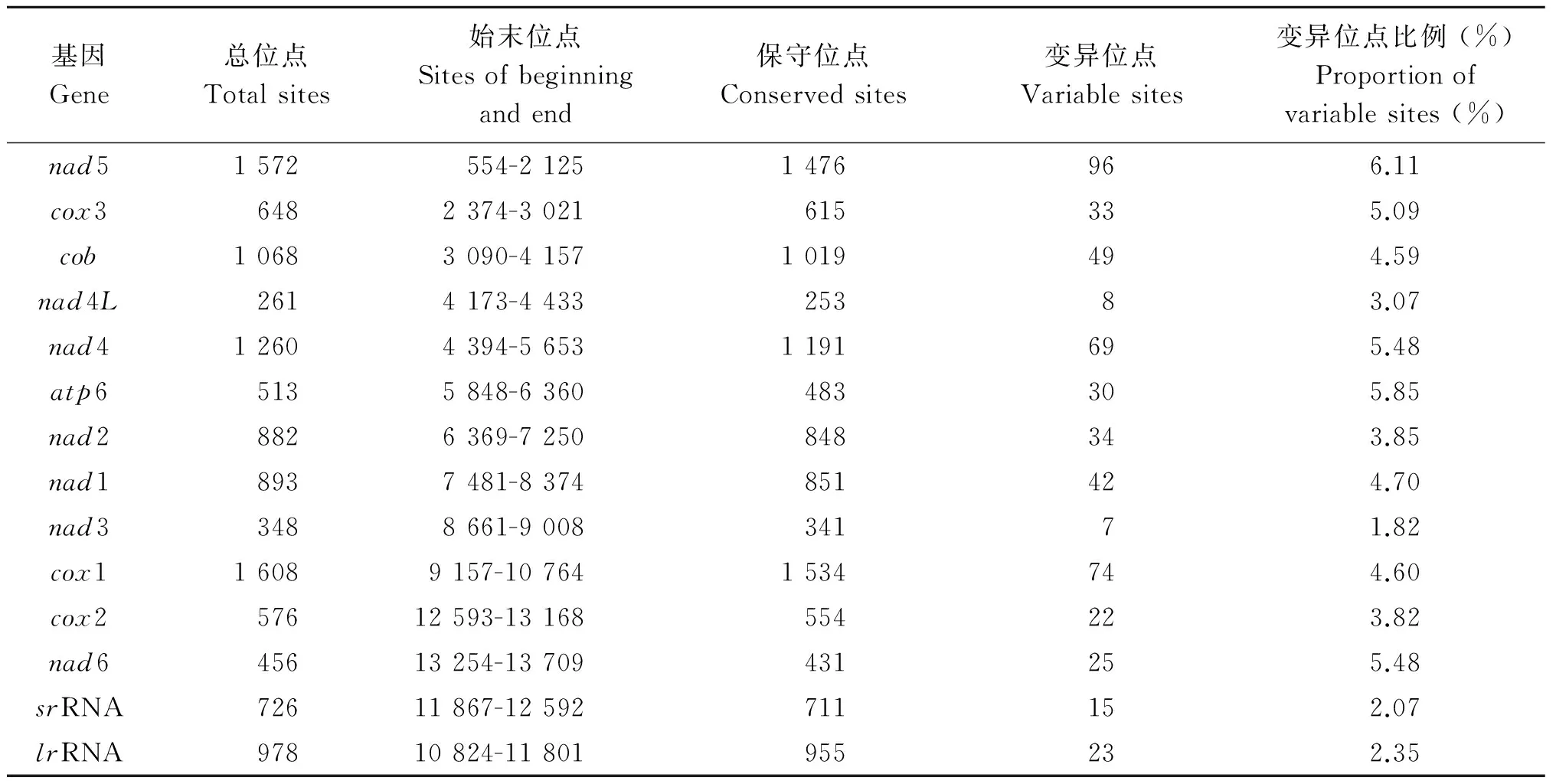
2.2线粒体基因组序列的比较除了cox1和nad1基因片段部分核苷酸序列的研究外,我们从NCBI数据库中下载加拿大棘球绦虫线粒体基因组的全部基因,并对每个基因变异位点进行分析,其基因组序列变异位点比较见表2。
表2加拿大棘球绦虫线粒体基因组序列的比较
Tab.2Comparison of mt genome sequences among E. canadensis genotypes
基因Gene总位点Totalsites始末位点Sitesofbeginningandend保守位点Conservedsites变异位点Variablesites变异位点比例(%)Proportionofvariablesites(%)nad51572554-21251476966.11cox36482374-3021615335.09cob10683090-41571019494.59nad4L2614173-443325383.07nad412604394-56531191695.48atp65135848-6360483305.85nad28826369-7250848343.85nad18937481-8374851424.70nad33488661-900834171.82cox116089157-107641534744.60cox257612593-13168554223.82nad645613254-13709431255.48srRNA72611867-12592711152.07lrRNA97810824-11801955232.35
对G6—G10基因型线粒体基因组序列比较,从基因变异位点分析结果可以看出,nad3基因的保守性最高,变异位点的比例仅为1.82%;srRNA、lrRNA、nad4L、cox2、nad2等5个基因的变异位点的比例介于2%~4%之间;cob、cox1、cox3、nad1、nad4、nad6、atp6等7个基因的变异位点的比例介于4%~6%之间,其中atp6的差异达5.85%;nad5基因变异位点的比例最高,达到6.11%。因此,nad5和atp6等基因可以作为备选的分子标记用于棘球属绦虫的遗传学研究中,这为合理利用其生物资源提供了依据和指导。
3G6—G10基因型分子流行病学意义
对棘球绦虫进行准确基因分型,目的在于明确诊断结果,便于收集和分析病原体传播途径和对人体易感基因型的信息与数据。Sweatman[28]认为加拿大棘球绦虫广泛分布于有狼出没的加拿大其他地区,而加拿大的沿海诸岛和纽芬兰被报道狼已灭绝。至于北极群岛地区,因恶劣的环境条件以及低密度的中间宿主形成了加拿大棘球绦虫在狼和蹄类动物间传播的天然屏障。对于人体棘球蚴病例,加拿大棘球绦虫G6—G10均可不同程度感染人[14,26,29-31]。G6—G10基因型的宿主及地理分布见下表(表3)。
表3加拿大棘球绦虫分子流行病学资料汇总
Tab.3Summary of molecular epidemiology of E. canadensis

虫株/基因型Strain/Genotype中间宿主Intermediatehost终末宿主Definitivehost地理分布Distribution参考文献References骆驼株(G6)Camelstrain(G6)骆驼、山羊、绵羊、牛、人Camels,Goats,Sheep,Cattle,Human犬、狼Dogs,Wolves中东、非洲、中国、阿根廷、加拿大、蒙古、俄罗斯MiddleEast,Africa,China,Argentina,Canada,Mongolia,Russia[29,32]猪株(G7)Pigstrain(G7)猪、野熊、海狸、牛、人Pig,Wildbear,Beaver,Cows,Human犬、狼Dogs,Wolves欧洲(匈牙利、波兰、斯洛伐克、意大利)、前苏联、南非、加拿大、巴西、葡萄牙、蒙古、墨西哥、中国Europe(Hungary,Poland,Slovakia,Ita-ly),theformerSovietUnion,SouthAfri-ca,Canada,Brazil,Portugal,Mongolia,Mexico,China[14,33-34]鹿株(G8)Cervidstrain(G8)驼鹿、马鹿Moose,Reddeer狼,犬Dogs,Wolves南美、欧亚大陆、加拿大、俄罗斯、美国SouthAmerica,Eurasia,Canada,Russia,theUnitedStates[30,35-36]猪株(G9)Pigstrain(G9)猪Pig犬Dogs波兰、加拿大Poland,Canada[26]麋鹿株(G10)Fennoscandiancer-vidstrain(G10)驼鹿、驯鹿、马鹿Moose,Reindeer,Reddeer狼,犬Dogs,Wolves加拿大、芬兰、瑞典、爱沙尼亚、蒙古、俄罗斯、美国、中国Canada,Finland,Sweden,Estonia,Mon-golia,Russia,theUnitedStates,China[30-32]中间型(G6/G7)Intermediatestrain(G6/G7)猪Pig野熊Wildbears法国France[37]
由表3可知,G6—G10复合群有着广泛的宿主类型和地理分布。与其他细粒棘球绦虫不同,加拿大细粒棘球绦虫主要分布在加拿大,有调查表明,在包虫病记录的93头鹿中,42%是麋鹿,37%是驼鹿,14%是北美驯鹿,6%是白尾长耳鹿和骡鹿。这些感染动物中,有83%只在肺中检测到包囊,8%在肺和肝脏都能检测到包囊,3%只在肝脏中检测到包囊,6%在其他器官中检测到包囊[29]。这些数据的记录可以有助于我们全面了解加拿大细粒棘球绦虫的生态、遗传多样性、基因型与致病性。根据加拿大棘球绦虫感染记录,相比于有蹄动物,驼鹿、麋鹿和驯鹿因在狼多的地方生存而有较高的发病率和感染强度,被认为是加拿大细粒棘球绦虫的适宜中间宿主[30-31,38]。包括气候变化、迁移、宿主灭绝以及土地利用变化在内的诸多因素影响加拿大棘球绦虫的发病率和地域分布[29]。有报道表明加拿大棘球绦虫可以中度感染人类[4,15,21,29,38-39]。最近分子研究发现加拿大棘球绦虫引起的包虫病病例在骆驼等牲畜,包括牛,猪和山羊在内的地区分布广泛,尤其在蒙古和西伯利亚,包虫病是常见的疾病[40]。
有关加拿大棘球绦虫感染人类的情况见表2所示。其中G6基因型被列为人类感染包虫病除G1基因型(普通绵羊株)外的第二个常见基因型,关于人类感染G6基因型的报道最早来自于阿根廷地区。到目前为止,有7个国家报道人感染G7型(表3),有12个国家的其他5个物种可以作为其中间宿主。G7基因型的传播似乎主要限于某些地区的中心(其中包括波兰和波罗的海和东欧,其中最常见的中间宿主是猪)。一般来说,在全世界范围内G7型在人或者动物的感染不像G1一样普遍。尽管只有少数人类包虫病呈现明显的特征,G7基因型仍被认为是来自波兰和斯洛伐克的唯一感染人的包虫病例[27,41]。经鉴定在波兰,奥地利和前南斯拉夫人感染G7型病例分别占100%(30/30)、92.0%(23/25)和33.3%(9/27)[29,52]。最近,非洲首例感染人类的G7基因型病例在南非发现[42]。另外有报道发现G8基因型感染人类的病例[43],流行病学数据也表明G8基因型传染人类[30]。至于G10基因型感染人类的病例也于2010年和2014年在蒙古和西伯利亚,2013年于俄罗斯报道出现过[40,44-45],在乌兰巴托国际病理学中心证实的人类包虫病例中,通过分析其cox1序列确定这些病例主要感染的是加拿大棘球绦虫(72.1%)或是棘球绦虫狭义种(27.9%),而感染加拿大棘球绦虫的病例中,以G6和G7株居多。通过数据分析表明加拿大棘球绦虫感染儿童较为普遍。调查的儿童病例中感染加拿大棘球绦虫的比例为100%,乌兰巴托的16个包虫病患者中13例为加拿大棘球绦虫,感染率为81.3%,而这13个疾病中有9名儿童,占69.2%,该研究首次证明该病的感染与年龄有关但具体原因尚不明确[40]。
在北纬和一些土著地区,生活在驼鹿和狼并存的区域附近的人感染包虫病仍然是一个公共健康问题[46]。最近报道,加拿大囊性包虫病的年总发病率估计为每百万人中有0.72例感染。然而,由于临床病例难以确诊,这样的感染率很有可能被低估[47]。在布加勒斯特,罗马尼亚分离的60株细粒棘球绦虫样本中,发现有一例加拿大棘球绦虫病例来自于人[48],加拿大棘球绦虫的传播和分布可能会因气候和景观变化而加快,加之旅游和贸易日益全球化,国家对这类寄生虫的检测将对人和动物的健康发挥重要作用[49]。
4我国的流行情况
包虫病是在中国感染人类的主要寄生虫病之一。在我国,四川、新疆等地也有报道发现G6,也有研究结果表明G3、G6基因型存在于青海省的牦牛和绵羊,所分离出来的G6型与哈萨克斯坦、伊朗(骆驼)分离株nad1基因序列完全一样。
目前已有27个省、自治区、直辖市报道过人类包虫病例,中国西部和西北部是主要流行区[50-51]。黑龙江省在我国东北,虽然不是包虫病的主要流行区,但是自从1958年首次报道人类感染包虫病病例以来,尤其是近几年,医院包虫病患者数量一直在增加。事实上早在2000年我国黑龙江省就已经被报道有4.5%的猪感染包虫。根据之前的关于棘球蚴病分子流行病学数据的总结,猪是G7型的主要中间宿主,在猪中占的比例最高为82.8%。在这之前我国未出现人感染加拿大棘球绦虫G7型的报道,这可能是由于最近二十年不断增加的国际贸易和牲畜贸易及进口犬的数量不断增加[53]。随着研究的进一步深入,我国的黑龙江省也首次报道在病人体内检测到G10基因型[54],但患者除了轻度的上腹部不适外,并无任何显著的临床表现,该报道的发布进一步表明了加拿大细粒棘球绦虫的地域分布远比预期的广泛,G10基因型病例可能会随着人口的增长扩展到新的地理区域(特别是自然栖息地),因为在这些地方人们直接或间接接触野生肉食动物的机会大大增加。此外,即使是在我国,越来越多的国际旅行也为棘球绦虫的复杂变种提供潜在的可能性。为了确认加拿大棘球绦虫G10基因型的真正传播模式,有必要在未来于野生动物宿主中检测该寄生虫,确认其生活史或者循环传播模式[54]。
5在疫苗和诊断制剂研制与开发上的意义
随着现代社会的发展,人口流动增加,宠物(犬)和经济动物(狐狸)养殖数量增加,导致加拿大棘球绦虫感染的增多。世界许多国家均投入大量的人力、物力、财力,通过各种途径开展棘球蚴病的防治研究,疫苗防治可能是彻底控制该病流行的最有效的方法之一。EG95蛋白、EgM家族、抗原B、EgA31、GST、EF1等都是具有发展前景的候选疫苗抗原,其中以EG95蛋白制成的基因工程亚单位疫苗,无论是实验动物的多样性还是试验地域的广泛性都优于其他抗原。
对于不同基因型的细粒棘球绦虫的EG95基因的研究发现,可以从加拿大棘球绦虫的G6/G7基因型中扩增出与细粒棘球绦虫狭义种G1/G2基因型EG95-1序列相似性为97.5%的基因(EG95-a1)。EG95-a1的氨基酸序列与EG95-1只有7个位点不同,且EG95-a1比EG95-1多一个与糖基化有关的N-X-S/T位点序列,二级结构多一个β片层,这些变化都说明EG95-a1可能与EG95-1的构象不同,也就是说EG95-1疫苗对加拿大棘球绦虫G6/G7基因型的交叉保护作用可能减少或消失[55-56]。最近的研究也显示来自G6型的EG95蛋白与免疫G1型EG95蛋白的绵羊产生的抗体不能全部结合[57]。因此,对细粒棘球绦虫进行分型定种对疫苗和诊断试剂的研制和开发是十分必要的。
6未来研究方向的展望
G6—G10基因型在终末宿主如犬或狼体内的感染情况尚不十分清楚,这需要今后进一步对不同地理区域终末宿主体内的G6—G10基因型进行调查与分析,以了解终末宿主在人体包虫病传播中所扮演的角色。气候环境的变化,动物的迁移以及全球化速度加快等因素都可能造成G6—G10基因型的传播,通过研究中间宿主的生活状态及遗传学背景,进一步分析G6—G10基因型在流行病学上的重要性和意义,为包虫病防控措施的制定提供指导。
由于G6—G10复合群均有感染人的病例,因此其感染途径与流行不可忽视。通过搜集不同地区不同宿主的样品,研究其保护性抗原,分析比较不同表达系统生产的重组抗原制成高效新型疫苗,用于控制包虫病的暴发也十分必要。
目前对G6—G10基因型的深入研究还不充分,它们的定种问题仍存在争议,其分类学研究仍需要深入进行。因此,应该用多种分子遗传学标记积累信息,扩充除cox1、nad1等线粒体基因外的其他信息,以获取更多遗传变异信息,为分类学研究及分子流行病学的调查提供科学依据和理论指导。
参考文献:
[1] Thompson RCA, Lymbery AJ. The nature, extent and significance of variation within the genusEchinococcus[J]. Adv Parasitol, 1988, 27: 209. DOI:10.1016/S0065-308X(08)60356-5
[2] Thompson RCA, McManus DP. Towards a taxonomic revision of the genusEchinococcus[J]. Trends Parasitol, 2002, 18(10): 452-457. DOI: 10.1016/S1471-4922(02)02358-9
[3] Jenkins DJ, Romig T, Thompson RCA. Emergence/re-emergence ofEchinococcusspp.—a global update[J]. Int J Parasitol, 2005, 35(11): 1205-1219. DOI: 10.1016/j.ijpara.2005.07.014
[4] Romig T, Dinkel A, Mackenstedt U. The present situation of echinococcosis in Europe[J]. Parasitol Int, 2006, 55: S187-S191. DOI: 10.1016/j.parint.2005.11.028
[5] Nakao M, Yanagida T, Okamoto M, et al. State-of-the-artEchinococcusandTaenia: phylogenetic taxonomy of human-pathogenic tapeworms and its application to molecular diagnosis[J]. Infect Genet Evol, 2010, 10(4): 444-452. DOI: 10.1016/j.meegid.2010.01.011
[6] Rosenzvit MC, Zhang LH, Kamenetzky L, et al. Genetic variation and epidemiology ofEchinococcusgranulosus in Argentina[J]. Parasitology, 1999, 118(5): 523-530. DOI:10.1017/S0031182099004035
[7] Okamoto M, Bessho Y, Kamiya M, et al. Phylogenetic relationships withinTaeniataeniaeformisvariants and other taeniid cestodes inferred from the nucleotide sequence of the cytochromec oxidase subunit I gene[J]. Parasitol Res, 1995, 81(6): 451-458. DOI: 10.1007/BF00931785
[8] Bowles J, McManus DP. Rapid discrimination ofEchinococcusspecies and strains using a polymerase chain reaction-based RFLP method[J]. Mol Biochem Parasitol, 1993, 57(2): 231-239. DOI: 10.1016/0166-6851(93)90199-8
[9] Lavikainen A, Lehtinen MJ, Meri T, et al. Molecular genetic characterization of theFennoscandiancervid strain, a new genotypic group (G10) ofEchinococcusgranulosus[J]. Parasitology, 2003, 127(3): 207-215. DOI: 10.1017/S0031182003003780
[10]Bart JM, Bardonnet K, Elfegoun MCB, et al.Echinococcusgranulosusstrain typing in North Africa: comparison of eight nuclear and mitochondrial DNA fragments[J]. Parasitology, 2004, 128(2): 229-234. DOI: 10.1017/S0031182003004359
[11] Moro PL, Nakao M, Ito A, et al. Molecular identification ofEchinococcusisolates from Peru[J]. Parasitol Int, 2009, 58(2): 184-186. DOI: 10.1016/j.parint.2009.01.005
[12] Scott JC, McManus DP. The random amplification of polymorphic DNA can discriminate species and strains ofEchinococcus[J]. Trop Med Parasitol, 1994, 45(1): 1-4.
[13] Bowles J, Blair D, McManus DP. Genetic variants within the genusEchinococcusidentified by mitochondrial DNA sequencing[J]. Mol Biochem Parasitol, 1992, 54(2): 165-173. DOI: 10.1016/0166-6851(92)90109-W
[14] Bowles J, McManus DP. NADH dehydrogenase 1 gene sequences compared for species and strains of the genusEchinococcus[J]. Int J Parasitol, 1993, 23(7): 969-972. DOI: 10.1016/0020-7519(93)90065-7
[15] Nakao M, McManus DP, Schantz PM, et al. A molecular phylogeny of the genusEchinococcusinferred from complete mitochondrial genomes[J]. Parasitology, 2007, 134(05): 713-722. DOI: 10.1017/S0031182006001934
[16] Zhang L, Gasser RB, Zhu X, et al. Screening for different genotypes ofEchinococcusgranulosuswithin China and Argentina by single-strand conformation polymorphism (SSCP) analysis[J]. Transact R Soc Trop Med Hyg, 1999, 93(3): 329-334. DOI: 10.1016/S0035-9203(99)90043-3
[17] Zhang L, Eslami A, Hosseini SH, et al. Indication of the presence of two distinct strains ofEchinococcusgranulosusin Iran by mitochondrial DNA markers[J]. Am J Trop Med Hyg, 1998, 59(1): 171-174.
[18] Dinkel A, Njoroge EM, Zimmermann A, et al. A PCR system for detection of species and genotypes of theEchinococcusgranulosus-complex, with reference to the epidemiological situation in eastern Africa[J]. Int J Parasitol, 2004, 34(5): 645-653. DOI: 10.1016/j.ijpara.2003.12.013
[19] Xiao N, Qiu J, Nakao M, et al.Echinococcusshiquicusn. sp., a taeniid cestode from Tibetan fox and plateau pika in China[J]. Int J Parasitol, 2005, 35(6): 693-701. DOI: 10.1016/j.ijpara.2005.01.003
[20] Wachira TM, Bowles J, Zeyhle E, et al. Molecular examination of the sympatry and distribution of sheep and camel strains ofEchinococcusgranulosusin Kenya[J]. Am J Trop Med Hyg, 1993, 48(4): 473-479.
[21] Thompson RCA. The taxonomy, phylogeny and transmission ofEchinococcus[J]. Exp Parasitol, 2008, 119(4): 439-446. DOI: 10.1016/j.exppara.2008.04.016
[22] Le TH, Pearson MS, Blair D, et al. Complete mitochondrial genomes confirm the distinctiveness of the horse-dog and sheep-dog strains ofEchinococcusgranulosus[J]. Parasitology, 2002, 124(01): 97-112. DOI: 10.101710.1017/S0031182001008976[23]Lavikainen A, Haukisalmi V, Lehtinen MJ, et al. A phylogeny of members of the familyTaeniidaebased on the mitochondrialcox1 andnad1 gene data[J]. Parasitology, 2008, 135(12): 1457-1467. DOI: 10.1017/S003118200800499X
[24] Moks E, Jogisalu I, Valdmann H, et al. First report ofEchinococcusgranulosusG8 inEurasiaand a reappraisal of the phylogenetic relationships of ‘genotypes’ G5-G10[J]. Parasitology, 2008, 135(5): 647-654. DOI: 10.1017/S0031182008004198
[25] Saarma U, Jogisalu I, Moks E, et al. A novel phylogeny for the genusEchinococcus, based on nuclear data, challenges relationships based on mitochondrial evidence[J]. Parasitology, 2009, 136(3): 317-328. DOI: 10.1017/S0031182008005453
[26] Scott JC, Stefaniak J, Pawlowski ZS, et al. Molecular genetic analysis of human cystic hydatid cases from Poland: identification of a new genotypic group (G9) ofEchinococcusgranulosus[J]. Parasitology, 1997, 114(1): 37-43. DOI:10.1017/S0031182096008062
[27] Pawiowski Z, Stefaniak J. The pig strain ofEchinococcusgranulosusin humans: a neglected issue?[J]. Genomics, 2003, 31: 38-42. DOI: 10.1016/S1471-4922(03)00199-5
[28] Sweatman GK. Distribution and incidence ofEchinococcusgranulosusin man and other animals with special reference to Canada[J]. Canadian J Public Health, 1952: 480-486.
[29] Schneider R, Gollackner B, Schindl M, et al.EchinococcuscanadensisG7 (pig strain): an underestimated cause of cystic echinococcosis in Austria[J]. Am J Trop Med Hyg, 2010, 82(5): 871-874. DOI: 10.4269/ajtmh.2010.09-0639
[30] Thompson RCA, Boxell AC, Ralston BJ, et al. Molecular and morphological characterization ofEchinococcusin cervids from North America[J]. Parasitology, 2006, 132(3): 439-447. DOI:10.1017/S0031182005009170
[31] Schurer J, Shury T, Leighton F, et al. Surveillance forEchinococcuscanadensisgenotypes in Canadian ungulates[J]. Int J Parasitol Parasit Wildlife, 2013, 2: 97-101. DOI: 10.1016/j.ijppaw.2013.02.004
[32] Ito A, Chuluunbaatar G, Yanagida T, et al.Echinococcusspecies from red foxes, corsac foxes, and wolves in Mongolia[J]. Parasitology, 2013, 140(13): 1648-1654. DOI: 10.1017/S0031182013001030
[33] Rojas CAA, Romig T, Lightowlers MW.Echinococcusgranulosussensulatogenotypes infecting humans-review of current knowledge[J]. Int J Parasitol, 2014, 44(1): 9-18. DOI: 10.1016/j.ijpara.2013.08.008
[34] Rodriguez-Prado U, Jimenez-Gonzalez DE, Avila G, et al. Genetic variation ofEchinococcuscanadensis(G7) in Mexico[J]. Am J Trop Med Hyg, 2014, 91(6): 1149-1153. DOI: 10.4269/ajtmh.14-0317
[35] Schurer JM, Gesy KM, Elkin BT, et al.EchinococcusmultilocularisandEchinococcuscanadensisin wolves from western Canada[J]. Parasitology, 2013, 141: 159-163. DOI: 10.1017/S0031182013001716
[36] Bryan HM, Darimont CT, Hill JE, et al. Seasonal and biogeographical patterns of gastrointestinal parasites in large carnivores: wolves in a coastal archipelago[J]. Parasitology, 2012, 139(6): 781-790. DOI: 10.1017/S0031182011002319
[37] Umhang G, Richomme C, Hormaz V, et al. Pigs and wild boar in Corsica harborEchinococcuscanadensisG6/7 at levels of concern for public health and local economy[J]. Acta Tropica, 2014, 133: 64-68. DOI: 10.1016/j.actatropica.2014.02.005
[38] Knapp J, Nakao M, Yanagida T, et al. Phylogenetic relationships withinEchinococcusandTaeniatapeworms (Cestoda: Taeniidae): an inference from nuclear protein-coding genes[J]. Mol Phylogenet Evol, 2011, 61(3): 628-638. DOI: 10.1016/j.ympev.2011.07.022
[39] Simsek S, Kaplan M, Ozercan IH. A comprehensive molecular survey ofEchinococcusgranulosusin formalin-fixed paraffin-embedded tissues in human isolates in Turkey[J]. Parasitol Res, 2011, 109(2): 411-416. DOI: 10.1007/s00436-011-2269-8
[40] Ito A, Dorjsuren T, Davaasuren A, et al. Cystic echinococcoses in Mongolia: molecular identification, serology and risk factors[J]. PLoS Negl Trop Dis, 2014, 8(6): e2937. DOI: 10.1371/journal.pntd.0002937
[41] Turcekova L, Snabel V, D'Amelio S, et al. Morphological and genetic characterization ofEchinococcusgranulosusin the Slovak Republic[J]. Acta Tropica, 2003, 85(2): 223-229. DOI: 10.1016/S0001-706X(02)00229-2
[42] Mogoye BK, Menezes CN, Wong ML, et al. First insights into species and genotypes ofEchinococcusin South Africa[J]. Vet Parasitol, 2013, 196(3): 427-432. DOI: 10.1016/j.vetpar.2013.03.033
[43] McManus DP, Zhang L, Castrodale LJ, et al. Short report: molecular genetic characterization of an unusually severe case of hydatid disease in Alaska caused by the cervid strain ofEchinococcusgranulosus[J]. Am J Trop Med Hyg, 2002, 67(3): 296-298.
[44] Jabbar A, Narankhajid M, Nolan MJ, et al. A first insight into the genotypes ofEchinococcusgranulosusfrom humans in Mongolia[J]. Mol Cellular Probes, 2011, 25(1): 49-54. DOI: 10.1016/j.mcp.2010.11.001
[45] Nakao M, Yanagida T, Konyaev S, et al. Mitochondrial phylogeny of the genusEchinococcus(Cestoda:Taeniidae) with emphasis on relationships amongEchinococcuscanadensisgenotypes[J]. Parasitology, 2013, 140(13): 1625-1636. DOI: 10.1017/S0031182013000565
[46] Himsworth CG, Skinner S, Chaban B, et al. Multiple zoonotic pathogens identified in canine feces collected from a remote Canadian indigenous community[J]. Am J Trop Med Hyg, 2010, 83(2): 338-341. DOI: 10.4269/ajtmh.2010.10-0137
[47] Gilbert NL, Dare OK, Libman MD, et al. Hospitalization for trichinellosis and echinococcosis in Canada, 2001-2005: the tip of the iceberg?[J]. Canadian J Public Health, 2010: 337-340.
[48] Piccoli L, Bazzocchi C, Brunetti E, et al. Molecular characterization ofEchinococcusgranulosusin south-eastern Romania: evidence of G1-G3 and G6-G10 complexes in humans[J]. Clin Microbiol Infect, 2013, 19(6): 578-582. DOI: 10.1111/j.1469-0691.2012.03993.x
[49] Jenkins EJ, Castrodale LJ, de Rosemond SJC, et al. Tradition and transition: parasitic zoonoses of people and animals in Alaska, northern Canada, and Greenland[J]. Adv Parasitol, 2013, 82: 33-204. DOI: 10.1016/B978-0-12-407706-5.00002-2[50] ALIMU MMT, Yan WL. The geographic distribution, endangerment, prevention and control strategies of Hydatid disease[J]. Xinjiang Med J, 2011, 2:37. DOI: 10.3969/j.issn.1001-5183.2011.02.035 (in Chinese)
阿里木·马木提, 严卫丽. 包虫病地理分布, 危害及防治策略[J]. 新疆医学, 2011, 2: 37. DOI:10.3969/j.issn.1001-5183.2011.02.035
[51] Grosso G, Gruttadauria S, Biondi A, et al. Worldwide epidemiology of liver hydatidosis including the Mediterranean area[J]. World J Gastroenterol, 2012, 18(13): 1425. DOI: 10.3748/wjg.v18.i13.1425
[52] Dybicz M, Gierczak A, Dabrowska J, et al. Molecular diagnosis of cystic echinococcosis in humans from central Poland[J]. Parasitol Int, 2013, 62(4): 364-367. DOI: 10.1016/j.parint.2013.03.005
[53] Zhang T, Yang D, Zeng Z, et al. Genetic characterization of human-derived hydatid cysts ofEchinococcusgranulosussensu lato in Heilongjiang Province and the first report of G7 genotype ofE.canadensisin humans in China[J]. PLoS One,2014, 9(10): e109059. DOI: 10.1371/journal.pone.0109059
[54] Yang D, Zhang T, Zeng Z, et al. The first report of human-derived G10 genotype ofEchinococcuscanadensisin China and possible sources and routes of transmission[J]. Parasitol Int, 2015, 64(5): 330-333. DOI: 10.1016/j.parint.2015.05.001
[55] Chow C, Gauci CG, Vural G, et al.Echinococcusgranulosus: variability of the host-protective EG95 vaccine antigen in G6 and G7 genotypic variants[J]. Exp Parasitol, 2008, 119(4): 499-505. DOI: 10.1016/j.exppara.2008.01.004
[56] Rojas CAA, Gauci CG, Nolan MJ, et al. Characterization of theeg95 gene family in the G6 genotype ofEchinococcusgranulosus[J]. Mol Biochem Parasitol, 2012, 183(2): 115-121. DOI: 10.1016/j.molbiopara.2012.02.005
[57] Alvarez Rojas CA, Gauci CG, Lightowlers MW. Antigenic differences between the EG95-related proteins fromEchinococcusgranulosusG1 and G6 genotypes: implications for vaccination[J]. Parasit Immunol, 2013, 35(2): 99-102. DOI: 10.1111/pim.12009
DOI:10.3969/j.issn.1002-2694.2016.04.015
通讯作者:贾万忠,Email:jiawanzhong@caas.cn
中图分类号:R383
文献标识码:A
文章编号:1002-2694(2016)04-0392-08
Corresponding author:Jia Wan-zhong, Email: jiawanzhong@caas.cn
收稿日期:2015-10-29修回日期:2016-01-25
Genotyping and molecular epidemiology ofEchinococcuscanadensis
LI Shuang-nan1, YAN Hong-bin1, LI Li1, LOU Zhong-zi1, FU Bao-quan1,2,YIN Hong1,2, JIA Wan-zhong1,2
(1.KeyLaboratoryofVeterinaryParasitologyofGansuProvince/KeyLaboratoryofZoonosesofAgricultureMinistry/StateKeyLaboratoryofVeterinaryEtiologicalBiology/LanzhouVeterinaryResearchInstitute,CAAS,Lanzhou730046,China;2.JiangsuCo-innovationCenterforPreventionandControlofImportantAnimalInfectiousDisease,Yangzhou225009,China)
Abstract:Through aligning the diversity of mitochondrial genome, especially the nad1 and cox1 nucleotide sequences of E. canadensis, recent development studies on genotyping and molecular phylogeny and variation of E. canadensis was summarized. Based on these data, the taxonomic status, nomenclature and evolutionary relationships of E. canadensis genotypes in the genus Echinococcus were determined. Also, advance and future direction of research on E. canadensis were discussed. This review will provide researchers and clinicians in this field with rich molecular epidemiological information or data, and lay theoretical foundation for molecular epidemiological investigations, warning and forecasting, and strategy formulation for comprehensive prevention and control of echinococcosis.
Keywords:Echinococcus canadensis; genotyping; molecular genetic markers; mitochondrial gene; taxonomy; epidemiology
甘肃省科技重大专项(No. 1203NKDA039)、公益性行业农业科研专项(Nos. 200903036-07、201303037、201503-047)、甘肃省科技创新团队项目(No. 1210RJIA006)和国家肉牛牦牛产业技术体系项目(No. CARS-38)联合资助

