不同钾水平下花魔芋差异表达基因分析
曾黎琼韩蓉蓉,,段玉云陈 玲文亦芾李娥贤姜 飞,
(1云南省农业科学院生物技术与种质资源研究所,云南省农业生物技术重点实验室,农业部西南作物基因资源与种质创制重点实验室,云南昆明 650223;2云南农业大学动物科学技术学院,云南昆明 650201;3西南大学动物科技学院,重庆 400715)
不同钾水平下花魔芋差异表达基因分析
曾黎琼1韩蓉蓉1,2,3段玉云1陈玲1文亦芾2李娥贤1姜飞1,2
(1云南省农业科学院生物技术与种质资源研究所,云南省农业生物技术重点实验室,农业部西南作物基因资源与种质创制重点实验室,云南昆明 650223;2云南农业大学动物科学技术学院,云南昆明 650201;3西南大学动物科技学院,重庆 400715)
摘 要:为了探索魔芋对不同水平钾素营养的反应机制,提取施用低、中、高水平硫酸钾5 d后的富源花魔芋叶片总RNA,合成了cDNA,采用优化的cDNA-SRAP技术体系分析了在低钾、中钾、高钾水平下富源花魔芋叶片的差异表达基因,并对其进行了生物信息学分析。结果显示:29对引物组合共扩增出507条基因片段,其中差异片段126条。相对于中钾处理,低钾、高钾处理的差异片段包括抑制型表达片段19条,诱导型表达片段14条,上调表达型片段29条,下调表达型片段64条。选取表达量高的13条差异片段进行回收测序,并在NCBI上进行BLAST比对,共比对出9条差异片段的同源序列,其中4条与已知功能基因(谷氨酰胺酰-tRNA 合成酶、β-ATP合成酶、GTP结合蛋白、核苷酸还原酶)有较高同源性,5条与假定基因或预测基因同源性较高,剩余序列未找到同源序列,可能是一些新的魔芋与钾营养吸收利用相关的基因,具体功能还有待于进一步研究。
关键词:富源花魔芋;cDNA-SRAP;基因表达;钾水平
曾黎琼,女,研究员,专业方向:农作物生物技术,E-mail:liqiongzeng@sina.com
魔芋(Amorphophallus konjac)是天南星科魔芋属多年生草本植物,球茎富含葡甘露聚糖,是我国重要的经济作物,在食品、医药、环保、化工、建筑等领域有着广泛的用途。富源花魔芋是云南魔芋生产中的主栽品种,段玉云等(2015)研究表明,富源花魔芋是一种喜钾作物,对钾素营养有较强的吸收能力,钾素营养水平影响其产量和品质。
SRAP (sequence-related amplified polymorphism)即相关序列扩增多态性,该技术操作简便、重复性好,近年来广泛应用于植物遗传多样性分析、分子遗传图谱构建、重要性状基因定位、亲缘关系鉴定、分子标记辅助选育等方面(Yves et al.,2010;Muhammad et al.,2011;卢超 等,2014)。cDNASRAP技术目前已在甘蔗(吴建明 等,2010)、蜡梅(赵凯歌 等,2012)、棉花(陈沙沙 等,2013)、甘蓝(张云霞 等,2014)等作物的基因表达分析中得到应用,成为了一种新的转录基因组学研究工具。本试验以云南魔芋主栽品种富源花魔芋为材料,利用cDNA-SRAP技术研究在低钾、高钾胁迫下展叶期富源花魔芋基因表达差异性,为筛选和鉴定富源花魔芋钾高效吸收利用基因、认识魔芋钾营养吸收利用的分子机制以及魔芋的生物技术育种奠定基础。
1 材料与方法
1.1试验材料
供试富源花魔芋:采自云南省农业科学院生物技术与种质资源研究所温室,种植方式为盆栽。试验药剂:RNAprep Pure多糖多酚植物总RNA提取试剂盒、FastQuant cDNA第一链合成试剂盒、Marker DL 2000购于天根生化科技(北京)有限公司;dNTP、Taq聚合酶购于Thermo公司;引物(表1)由生工生物工程(上海)股份有限公司合成,其他一般试剂为国产分析纯。主要试验仪器:离心机(Eppendorf 5417R),DNA扩增仪(Biometra Tprofessional PCR仪),电泳仪(北京六一DYY-6C型号),凝胶成像系统(Syngene GeneGenius),紫外分光光度计(Thermo NanoDrop 2000分光光度计)。
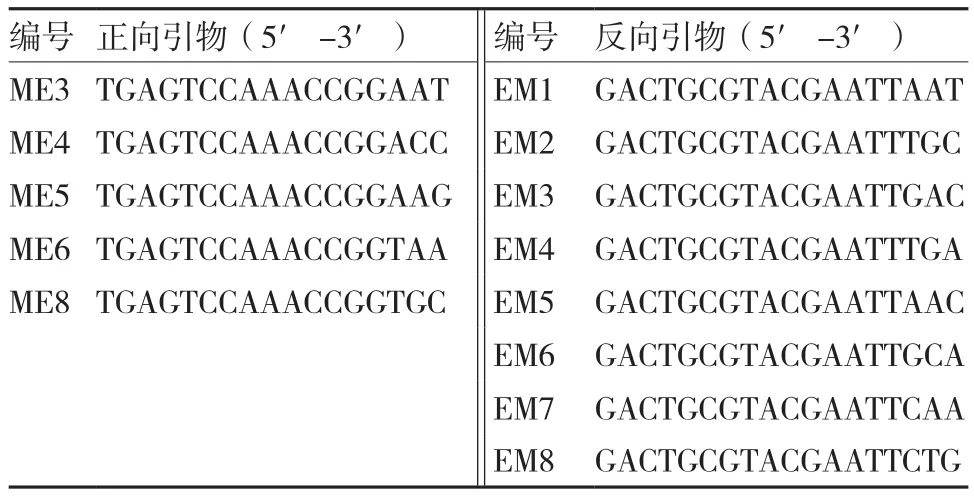
表1 引物序列
1.2试验方法
试验于2014年4月20日在云南省农业科学院生物技术与种质资源研究所温室中进行富源花魔芋盆栽种植,根据段玉云等(2015)对富源花魔芋不同时期植株含钾量及其对钾肥利用效率的分析研究结果,本试验选择在富源花魔芋生长(基质培)至展叶期时,浇施高(K2SO4施用量为6.6 g·株-1)、中(K2SO4施用量为4.4 g·株-1)、低(K2SO4施用量为2.2 g·株-1)不同水平硫酸钾,每处理重复3次,每重复5株,5 d后每个处理分别采集5株植株复叶的中部叶片,液氮速冻后于-70 ℃保存。
1.2.1RNA的提取和cDNA的合成 采用RNAprep Pure多糖多酚植物总RNA提取试剂盒提取花魔芋叶片总RNA,利用超微紫外分光光度计测定RNA浓度,用浓度为1%的琼脂糖凝胶电泳分析其纯度;用FastQuant cDNA第一链合成试剂盒合成cDNA第一链。严格按照说明书进行操作。
1.2.2cDNA-SRAP反应体系的建立 采用单因素变量分析方法分别对cDNA用量、Mg2+浓度、引物浓度、dNTP浓度和Taq酶用量进行cDNA-SRAP反应体系的优化,优化时选用ME4/EM4引物组合。在20 μL的反应体系中:cDNA用量梯度设为50、100、150、200 ng;Mg2+浓度梯度设为1.5、2.0、2.5、3.0 mmol·L-1;引物浓度梯度设为0.2、0.3、0.4、0.5 μmol·L-1;dNTP浓度梯度设为0.15、0.20、0.25、0.30 mmol·L-1;Taq酶用量分别为0.5、1.0 、1.5、2.0 U。cDNA-SRAP PCR扩增反应程序为:94 ℃ 4 min;94 ℃ 45 s,33 ℃ 45 s,72 ℃ 45 s 5个循环;94 ℃ 45 s,50 ℃ 45 s,72 ℃ 45 s 30个循环;最后72 ℃延伸5 min。反应结束后4 ℃保存,扩增产物采用2%琼脂糖凝胶电泳检测。
1.2.3花魔芋差异表达基因的cDNA-SRAP分析
用优化的cDNA-SRAP反应体系对低钾、中钾、高钾3个处理的富源花魔芋样品进行分析。cDNASRAP PCR扩增反应程序同1.2.2。扩增产物采用6%聚丙烯酰胺凝胶进行电泳检测。
1.2.4差异片段的克隆、测序分析 将差异片段从聚丙烯酰胺凝胶上切下回收,加入30 μL ddH2O,室温放置24 h后取10 μL作为模板再次进行PCR扩增,所用引物和程序与最初反应相同。采用1%琼脂糖凝胶检测产物是否与原片断大小相同,选取与原片段大小相同的目的条带的PCR产物送生工生物工程(上海)股份有限公司进行测序。
2 结果与分析
2.1花魔芋RNA的质量和cDNA质量
花魔芋RNA的琼脂糖凝胶电泳结果显示(图1):低钾、中钾、高钾3种处理样品的RNA 28S、18S带型清晰整齐、完整性好,经测定RNA的OD260/280值均在1.8~2.0之间,说明提取的RNA纯度高、质量好。

图1 花魔芋RNA琼脂糖凝胶电泳检测结果
用cDNA第一链合成试剂盒合成了花魔芋cDNA第一链,1%琼脂糖凝胶电泳检测结果显示:低钾、中钾、高钾3种处理样品在100~2 000 bp之间均有弥散带,说明合成的cDNA质量好。
2.2花魔芋cDNA-SRAP反应体系的建立
根据单因素变量分析法优化cDNA-SRAP反应体系结果可知,花魔芋最佳的cDNA-SRAP反应体系(20 μL)为:cDNA模板用量150 ng,Mg2+浓度为2.0 mmol·L-1,引物浓度为0.3 μmol·L-1,dNTP浓度为0.25 mmol·L-1,Taq酶用量为1.0 U。
2.3不同钾水平下花魔芋的差异表达基因分析
以低钾、中钾、高钾3种处理样品合成的cDNA为模板,用建立的最佳反应体系进行SRAPPCR扩增。如图2所示,分别以ME4/EM3、ME4/EM4、ME4/EM5、ME4/EM6为引物,低钾、中钾、高钾3种处理共扩增出差异条带13条。试验所用的29对引物组合共扩增出507条片段,其中差异片段126条。相对中钾处理,低钾、高钾处理的差异片段包括抑制型表达片段19条,诱导型表达片段14条,上调表达型片段29条,下调表达型片段64条。

图2 花魔芋cDNA-SRAP聚丙烯酰胺凝胶电泳检测结果
2.4差异表达基因片段的序列分析
选取条带亮、表达量高的13条差异片段进行回收测序,测序结果在NCBI上进行BLAST同源性比对分析,共比对出9条差异片段的同源序列(表2),其中4条差异表达核苷酸序列与已知功能基因(谷氨酰胺酰-tRNA 合成酶、β-ATP合成酶、GTP结合蛋白、核苷酸还原酶)有较高同源性,5条序列与假定基因或预测基因同源性较高,剩余序列未找到同源序列,可能是一些新的魔芋与钾营养相关基因,具体功能有待进一步研究。
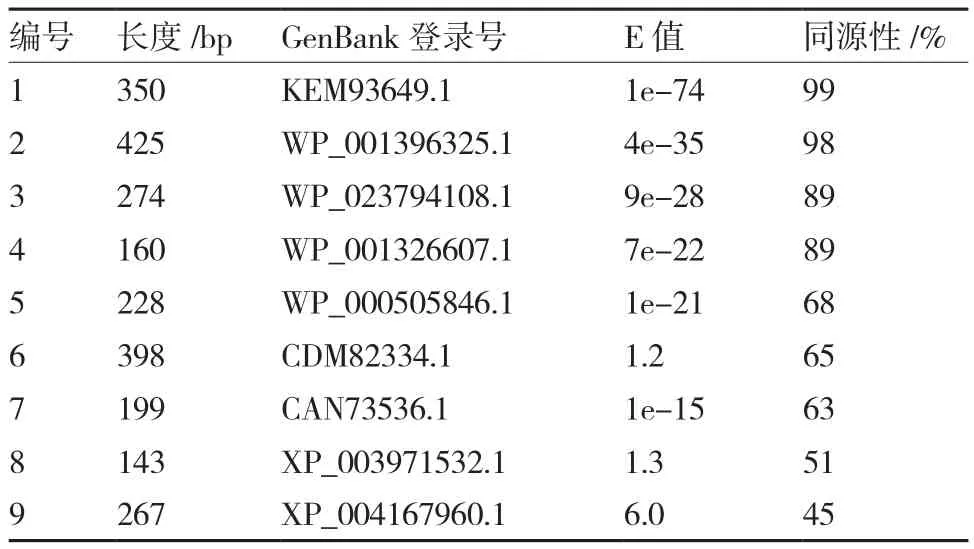
表2 花魔芋差异表达基因片段的同源性分析
3 结论与讨论
cDNA-SRAP是一种无需任何序列信息即可进行差异表达研究的新技术,通过一对引物对开放读码框(ORFs)进行扩增,正向引物为17个碱基,其中的CCGG序列目的是与位于富含GC区域的开放读码框中的外显子进行特异结合;反向引物为18个碱基,其中的AATT序列可以对富含AT的内含子区域和启动子区域进行扩增。SRAP产生共显性标记的效率远大于其他标记技术,而且步骤较少,操作相对简单,成本较低,花费时间少。cDNA-SRAP技术在棉花(陈沙沙 等,2013)、甘蔗(吴建明 等,2010)、甘蓝(张云霞 等,2014)、蜡梅(赵凯歌 等,2012)等作物基因表达分析中的应用,证明了此技术是行之有效的。
富源花魔芋是云南魔芋生产上的主栽品种。段玉云等(2015)的研究发现,展叶期富源花魔芋对适量钾素营养有很好的吸收利用效率。施用适量钾素营养有利于提高叶片叶绿素、可溶性蛋白和可溶性糖含量,增强了叶片SOD、 CAT、 POD活性,降低了魔芋叶片MDA含量,对魔芋抗氧化酶系统以及膜脂过氧化系统有一定的生理效果,增强了魔芋植株抵抗逆境和抗衰老的能力,进而影响了叶片的光合作用和同化产物的合成、转化和运输,从而获得了魔芋的高产(待发表)。本试验利用优化的cDNA-SRAP技术最终得到花魔芋最佳cDNA-SRAP反应体系(20 μL):cDNA模板150 ng、Mg2+浓度为2.0 mmol·L-1、引物浓度0.3 μmol·L-1、dNTP浓度0.25 mmol·L-1、Taq酶用量1.0 U,并初步分析了展叶期施用不同量硫酸钾5 d后富源花魔芋叶片的差异表达基因,表明不同钾水平下富源花魔芋的基因表达存在差异,这些差异表达基因的具体功能有待于进一步分析研究。
下一步可以通过与转录组测序等分析结果的比较筛选以及生物信息学的分析研究,结合RTPCR、RACE等技术对得到的新候选基因转录本进行时空表达研究和全长序列克隆,为认识魔芋钾吸收利用的分子机制和魔芋钾高效吸收利用基因的发掘奠定基础。
参考文献
陈沙沙,樊洪泓,关蕾,林毅,蔡永萍,钱森和,孙旭.2013.光质对离体诱导棕色棉纤维基因表达影响的cDNA-SRAP分析.棉花学报,26(1):34-40.
段玉云,董坤,王片吉,谢勇,李萍仙,卢俊,李胜,曾黎琼,潘开华.2015.富源花魔芋对不同硝酸钾施用量的吸收利用.西南农业学报, 28(4):1697-1701.
卢超,张美德,何银生,刘海华,艾伦强.2014.SRAP技术在植物遗传育种中的应用.现代农业科技,(16):18-20.
吴建明,李杨瑞,王爱勤,杨柳,杨丽涛.2010.利用cDNASRAP分析赤霉素诱导甘蔗节间伸长的差异表达.中国农业科学,43(19):3937-3944.
张云霞,宋立晓,曾爱松,高兵,严继勇.2014.利用cDNASRAP分离结球甘蓝抗黑腐病相关基因的研究.华北农学报,29(5):29-32.
赵凯歌,王文颖,陈龙清.2012.用cDNA-SRAP技术分离蜡梅花发育不同时期差异表达基因片段.华中农业大学学报,31(6):693-698.
Muhammad Y,Andrew C J,Renata R M.2011.Musa genetic diversity revealed by SRAP and AFLP.Molecular Biotechnology,47(3):189-199.
Yves C,Jean C,Annick B,Réal M,Serge L.2010.SRAP polymorphisms associated with superior freezing tolerance in alfalfa (Medicago sativa spp.sativa).Theoretical and Applied Genetics,120(8):1611-1619.
Differential Genes Expression Analysis of Amorphophallus konjac Plants Growing under Different Potassium Levels
ZENG Li-qiong1,HAN Rong-rong1,2,3,DUAN Yu-yun1,CHEN Ling1,WEN Yi-fu2,LI E-xian1,JIANG Fei1,2
(1Biotechnology & Germplasm Resources Institute,Yunnan Academy of Agricultural Sciences,Key Lab of Agricultural Biotechnology of Yunnan Province,Key Lab of Southwestern Crop Gene Resource and Germplasm Innovation,Ministry of Agriculture,Kunming 650223,Yunnan,China;2College of Animal Science and Technology,Yunnan Agricultural University,Kunming 650201,Yunnan,China;3College of Animal Science and Technology,Southwest University,Chongqing 400715,China)
Abstract:In order to investigate the response mechanism of Amorphophallus konjac plants growing under different potassium levels,the total RNA of the leaves of Amorphophallus konjac Fuyuan on the fifth day after potassium sulfate(low,medium and high level)was applied.Then cDNA was synthesized,and the differentially expressed genes of Amorphophallus konjac Fuyuan leaves under low,medium and high potassium sulfate were amplified by optimized cDNA-SRAP,and their bioinformatics was analyzed.The results showed that 507 gene segments were amplified by 29 pairs primer combinations,including 126 differentially expressed gene segments.Compared with the medium potassium level,19 inhibited expressed fragments,14 induced expressed fragments,29 up-regulated expressed fragments,and 64 down-regulated expressed fragments were found in the low and high potassium levels.After recycling 13 differentially expressed gene segments of good quantity,9 homologous sequences matched with them in NCBI.Except for 4 nucleotide sequences matching with higher homology to the known function genes (glutaminyl-tRNA synthetase,β-ATP synthase,YchFGTP-binding protein YchF,ribonucleotide reductase) and 5 nucleotide sequences matching with higher homology to the assumption genes or the predict genes,other differentially expressed gene segments were not found,which might be some new genes of Amorphophallus konjac Fuyuan associated with potassium nutrition uptake.The specific function of the new genes will be further studied and analyzed in the future.
Key words:Amorphophallus konjac Fuyuan;cDNA-SRAP;Gene expression;Potassium level
收稿日期:2015-10-14;接受日期:2016-02-26

