猪ITGB-5基因外显子多态性及其与仔猪腹泻的关联性研究
牛晓艳(山西省农科院畜牧兽医研究所,山西 太原 030032)
猪ITGB-5基因外显子多态性及其与仔猪腹泻的关联性研究
牛晓艳
(山西省农科院畜牧兽医研究所,山西 太原 030032)
摘 要:采用PCR—测序的方法对猪ITGB-5基因所有17个外显子进行扩增测序,经多重比对后找到该基因内的SNPs。对该基因外显子内所有SNPs进行Snap-shot分型并与黏附表型进行关联分析。结果表明:ITGB-5外显子内共发现10个SNPs,其中有3个SNPs关联分析结果呈现显著,分别位于该基因的5号及13号外显子。结合该基因的生物学功能以及本试验的结果表明:ITGB-5可能是与仔猪腹泻相关的基因。
关键词:仔猪腹泻;ITGB-5基因;Snap-shot分型;关联分析
仔猪腹泻是导致畜牧业损失的重要原因之一,由于仔猪腹泻造成的损失占到畜牧业总产值的20%左右,腹泻造成的死亡占仔猪总死亡数的40%左右。仔猪腹泻常常导致饲料报酬率下降,生长发育迟缓,产生僵猪等。在所有腹泻当中,由ETEC(产肠毒素大肠杆菌)引起的腹泻是最为常见的一种,这种腹泻发生的机理是以大肠杆菌和宿主小肠上皮细胞的黏附为前提,所以可以表达受体蛋白的个体为易感个体,不表达受体蛋白的个体为抗性个体。根据大肠杆菌表达菌毛的种类,又分为F4ab,F4ac和F4ad三种类型。ETEC粘附于猪小肠上皮细胞后,释放毒素,导致肠道内电解质平衡紊乱,从而导致腹泻。
猪ITGB-5基因位于猪13号染色体31q41区段。ITGB-5属于整合素家族成员,编码细胞表面糖蛋白,主要功能是参与免疫细胞的黏附作用。该基因在生物进程中具有多种功能,包括细胞生长和创伤修复时的细胞迁移,细胞变异和细胞凋亡,调节潜在的一些肿瘤细胞的转移和癌细胞的扩散等。ITGB-5通常以二聚体的形式存在,包括α和β亚基,该异二聚体优先结合在细胞黏附分子上,构成细胞外基质的成分。该基因已经徐如海[1]等经抑制消减杂交筛选技术而得到,可作为研究仔猪腹泻的候选基因之一。
本研究基于PCR-测序技术发现猪ITGB-5基因外显子的SNPs,通过Snap-shot技术对该基因所有外显子内的SNPs进行分型,再经卡方检验获得其与黏附表型之间的关联性。为进一步研究该基因和仔猪腹泻之间的关系提供实验依据。
1 材料与方法
1.1实验材料
1.1.1试验动物 试验所采用的动物个体来源于中国农科院畜牧所。是由大白,长白和松黑三个品种所组成的群体,该群体为两代半同胞家系,包括366头子代和77头父母代个体。根据黏附表型,选取6头黏附个体(case:Y)以及6头非黏附个体(control:NY)作为测序反应的试验样本,case组与control组之间无亲缘关系。
1.1.2试验试剂 PCR引物购自上海生工生物工程技术有限公司;超纯dNTP,PCR反应Buffer(10X),Taq酶,Marker DL2000,6X上样缓冲液均购自Invitrogen公司;双蒸水,II型琼脂糖为实验室自备。
SNP基因型分型:Snapshot委托上海捷瑞生物公司进行。
用于测量黏附表型的菌株分别为:195(F4ab,C83901,O8;K87,猪源),200(F4ac,C83907,O149:K91,猪源),216(F4ad,C83923,O8:K87,猪源),238(C83286,O38:K99,牛源,阴性对照菌)。以上菌株均购自中国兽药监察所。
1.1.3引物设计 通过软件Primer 3.0对ITGB-5基因的外显子区域进行在线引物设计,用于PCR反应的引物如下表所示:

表1 ITGB-5引物序列
1.2试验方法
1.2.1黏附表型测定 三个品种小肠上皮细胞与大肠杆菌菌毛之间的黏附情况通过显微镜观察后记录[2]。根据小肠上皮细胞吸附大肠杆菌的个数,分为黏附(Y)和非黏附(NY)两种类型。
黏附标准采用单个小肠上皮细胞:黏附多于5个细菌为黏附。
1.2.2DNA提取 取大小为1 g的耳组织样本,放入Eppendorf管中剪碎。耳组织DNA提取过程采用《分子克隆试验指南》中所提供的酚-氯仿法[3],提取获得的DNA用双蒸水溶解后,将样品在50 ℃下放置1小时,灭DNA酶。
1.2.3PCR反应体系和条件 PCR反应体系:DNA模板(50~75 ng/ul):1.5 ul;上游引物(10 μmol/L):0.5 ul,下游引(10 μmol/L):0.5 ul;dNTP(10 μmol/L):1.6 ul;Buffer:2.0 ul;Taq酶(5 U/ul):0.4 ul;ddH2O:14.1~14.6 ul。
PCR反应条件:94 ℃预变性5 min,94 ℃预变性30 s,55 ℃~65 ℃退火30~45 s,72 ℃延伸30~45 s,72 ℃终末延伸7 min~10 min,20 ℃保存。
1.2.4测序比对 将PCR产物收集后送至北京三博远志公司进行测序分析。测序结果用Chromas软件判读。部分片段(F1,F3,F6,F9)由于片段内存在高GC含量的区域(在70%以上),普通测序反应无法实现,采用克隆测序的方法进行。
质粒小提:质粒小提采用试剂盒进行。
克隆测序:将PCR产物切胶回收后与T-easy载体连接,并转化感受态细胞,将培养获得的阳性菌落送公司进行克隆测序。
测序结束后将所获得的试验个体各外显子的序列采用DNAMAN进行多序列比对,获得各外显子内SNPs和片段缺失的信息。
1.2.5Snapshot分型
对选定区段的DNA扩增样本进行混池后采用多重PCR进行分型。加入荧光染料后,在ABI3730核酸检测仪上进行测序分析,收集荧光信号,并对分型结果进行总结。
1.3统计分析
统计F4ab和F4ac黏附(Y)以及非黏附(NY)个体在群体内的基因型频率。对各SNPs在群体中进行卡方检验,检验统计量为:自由度df=k-1,即自由度df=3+3-1=5,经检验获得ITGB-5内显著的SNPs。
2 实验结果
2.1黏附表型检测
经显微镜观察,得到两组个体的体外黏附结果,如表2所示:

表2 Case-control设计所选择的样本
2.2测序结果
2.2.1ITGB-5基因各外显子内的突变 对ITGB-5进行测序后结果如下表所示:
由测序结果可以看出,在ITGB-5基因的5号,6号,7号以及11,13,16号和17号外显子内,均发现一些SNPs,在17号外显子内还发现了一个SEXA1的酶切位点。
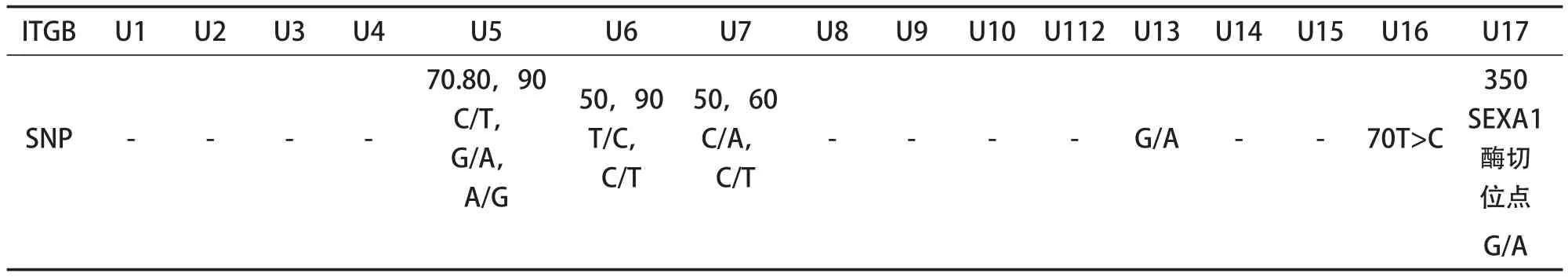
表3 ITGB-5外显子测序结果
2.2.2酶切位点 通过在线的分子生物学软件(http://rna.Lundberg.gu.se/cuttter2/)对ITGB-5基因外显子进行酶切位点预测,结果发现,该基因外显子5,6,7,11,13,16内的突变位点为非酶切位点,而17号外显子中存在酶切位点SEXAI。进一步分析这些酶切位点和黏附表型间的关系,检测了7个黏附型个体和7个非黏附型个体,但这些个体的酶切基因型和黏附表型之间并没有关联关系,所以认为试验检测到的酶切位点并非因果突变。
在第17号外显子中存在SEXA1酶切位点,其PCR和酶切结果如下图所示,酶切序列(5'…A/ CCWGGT…3'/3'…TGGWCC/A…5'),经分析,这个突变不是影响基因功能的突 变:

图1 ITGB-5基因17号外显子SEXA1酶切位点
2.3ITGB-5外显子内SNPs的卡方检验结果
采用卡方检验的方法对ITGB-5内所发现的9个SNPs进行分析,取显著性阈值X2(5)0.05=11.07,X2(5)0.01= 15.09。卡方检验结果如表4所示。

表4 对于ITGB-5内各SNPs的卡方检验结果
由卡方检验结果可知:F4ac检验结果均不显著,F4ab号外显子的2个SNPs与黏附表型存在显著关联(P<0.05),13号外显子的1个SNP与F4ab黏附表型存在极显著关联(P<0.01)。但这些突变与腹泻之间的关系,还需要进一步的基因功能验证。
3 讨论
3.1ITGB-5可能是导致黏附的候选基因
ITGB-5蛋白由α和β两个亚基组成,这两个亚基在宿主细胞表面表达,该蛋白是由整合素所编码的,玻璃体蛋白Vn可以和整合素的β亚基结合,而大肠杆菌又可以和Vn结合,它们共同构成复合体通过小肠上皮细胞侵入宿主细胞,形成包涵体的形式。在这个过程中,整合素发挥了重要的桥梁作用。
ITGB-5在细胞内具有多重功能,该蛋白属于跨膜蛋白家族成员,可以和RGD序列相结合。该序列是整合素蛋白所特有的序列,包含了一系列的氨基酸序列,整合素属于跨膜受体,可以结合在RGD氨基酸的多肽当中。整合素介导了细胞分化,迁移,扩增,形态改变以及细胞死亡过程。与整合素结合的配体氨基酸包括Arg-Gly-Asp序列,这个序列可以通过整合素蛋白进行识别,而该蛋白是RGD依赖的蛋白,该整合素蛋白是B3整合素家族的成员。Maki ISHII等检测了整合素家族的两个成员在小鼠性腺细胞中的表达模式,并认为它们和该基因中的RGD序列有关,该研究检测了ITGB-5在不同日龄,主要是10.5~13.5天内的表达模式,结果表明该基因在性腺细胞中广泛表达[4]。R.Pasqualini等研究了ITGB-5在不同组织中的表达模式,也证明了ITGB-5与器官发育,炎症,组织修复以及细胞的稳态维持有关,黏附过程是通过玻璃体蛋白以及纤维蛋白所介导的[5,6]。上述这些研究都说明了ITGB-5在细菌侵入细胞过程中的介导作用,但除此之外,ITGB-5还具有信号转导等一系列功能。通过对该基因外显子序列的测序分析发现了9个SNPs,其中5号外显子内的2个SNPs和13号外显子内的1个SNPs与黏附表型存在显著关联。
ITGB-5和MUC4具有相似的结构、基因大小和功能,并在染色体上处于相近位置[7]。但对于它们的功能以及黏附表型与腹泻关系的研究还停留在基本的统计分析阶段,并不能真正的揭示其与腹泻的关系。

图5 Vn与整合素结合,侵染宿主细胞的过程
3.2与腹泻有关的其他基因
黏液素家族成员(MUC4,MUC13,MUC20)等是细胞膜上表达的蛋白,Jorgensen等2003,2006年所发表的文章中认为MUC4是导致黏附以及腹泻的重要候选基因,该基因7号内含子中存在一个XbaI的酶切位点,通过RFLP,在相关群体中得到验证,并在表型为黏附及非黏附的个体中进行关联分析,该关联分析可以得到基本吻合的结果[8]。该突变被认为是一个可能的分子标记并在2004年申请了专利。但MUC4本身属于一个多能性基因,在消化道表面表达,形成黏液,该基因所表达的蛋白可以形成屏障,防止病原微生物侵入消化道。除此之外,该基因还具有多种功能,并与胰腺癌的发生有关,在人类中,该基因的mRNA具有多个转录本。而黏液素家族的系列基因MUC13,MUC20等都具有类似功能。MUC4位于消化道和呼吸道的内表面,一般由消化道和呼吸道内表面的腺体所分泌,它可以维持细胞内环境的稳态以及维持细胞的生存。其结构和生化组成可以为细胞表面提供保护并且使细胞处于稳定的内环境中,不仅如此,MUC4还可以防止细胞的癌变[9]。
对于MUC13和MUC20,国内黄路生等对该基因cDNA序列内所存在的突变进行了测序研究。并在MUC13 2 679 bp的cDNA序列内发现三个突变(c.576C>T,c.908C>G,c.935A>C)。并且比较了猪和人该基因序列的差异,对于550 bp的cDNA进行了纯化以及扩增的过程,并通过3'以及5'RACE,获得该基因的全长以及基因内部的各个功能元件。并通过UTRSCAN寻找该基因内部的SNPs。通过RTPCR,检测了该基因在不同组织中的表达情况,并且在黏附以及非黏附两组个体中找到位于外显子内的多态(SNPs)以及进行传递不平衡研究(TDT)[10]。
对于MUC20,该研究小组也对该基因内的多态进行了研究,研究方法与MUC20相似,通过扩增得到该基因全长的mRNA,在ORF处鉴别出两个18 bp片段的缺失,该区段包含一个1 527 bp的开放阅读框,并编码532个氨基酸。RT-PCR分析表明,该基因在肾脏,前列腺,上皮以及膀胱中的表达量最高。SNP分析显示,该基因内存在7个多态,分别是内含子5中g.191C>T,以及外显子6中c.1 600C>T,通过TDT分析可以得到这两个多态与黏附表型之间存在显著的关联关系[11]。
4 结论
上述研究通过对ITGB-5基因外显子的测序和突变分析,在基因的外显子部分区段(外显子5,6,7 和11,13,16和17)检测得到突变,其中17号外显子350 bp处存在SEXAI酶切位点。通过Snap-shot分型后发现3个SNPs与F4ab的黏附表型之间具有显著关联,但这些突变是否是与仔猪腹泻相关的因果突变,还有待进一步试验证实。从生物学功能上分析,ITGB-5很可能是影响黏附的候选基因。本研究为从分子水平研究仔猪腹泻的机制提供试验依据。
参考文献:
[1]徐如海,胡锦平,翁经强等.抑制消减杂交筛选仔猪ETEC K88ab/ac受体候选基因[C].中国动物遗传育种研究进展——第十五次全国动物遗传育种学术讨论会论文集.
[2]李玉华.仔猪大肠杆菌F4受体基因的精细定位.2006博士学位论文.
[3]J.萨姆布鲁克,D.W.拉塞尔著.[M].分子克隆实验指南,科学出版社.2003年.
[4]Maki I,T.W.Tay,Masamichi K et.al.Expression parttern of avB3 and avB5 Integrin mrna in mouse fetal gonads.[J]. Journal of Reproduction and development.Vol 52,No 3.2006.
[5]Pasqualini.R,Bodorova J,Ye S et.al.A study of the structure,function and distribution of B5 integrins using novel anti-B5 monoclonal antibodies.[J].Journal of Cell Science 105,101-111,1993.
[6]Memmo.L.M & McKeown-Longo.P.The alpha v beta 5 integrin functions as an endocytic receptor for vitronecin.[J].Cell Sci.111,425-433.1998.
[7]Antonio R,Jacoben M.J.,Joller D.et.al.The receptor locus for Escherichia coli F4ab/F4ac in the pig maps distal to the MUC4-LMLN region.[J].Mammalian Genome.Nov.2010.
[8]Jorgensen C.B.,Cirera S.,Anderson S.I.et.al.Linkage and comparative mapping of the locus controlling susceptibility towards E.coli F4ab/ac Diarrhoea in pigs.[J].Cytogenetic Genome Research.102:157-162,2003.
[9]Hollingsworth M.A.and Swanson B.J.Mucins in cancer:protection and control of the cell surface.[J].Nature.Review Cancer.Jan.Vol.4.45-57.2004.
[10]Zhang B,Ren J & Huang H.Investigation of the porcine MUC13 gene:isolation,expression,polymorphisms and strong association with susceptibility to E.coli F4ab/ac.[J].Animal Genetics.June.Vol 39(3):258-266,2008. [11]M.Schroyen,E.Cox and N.Buys.Susceptibility of piglets to ETEC is not related to the expression of MUC13 and MUC20.[J]Animal Genetics.April 4,2011.
(编辑:晏兵兵)
中图分类号:S828
文献标识码:B
文章编号:1006-799X(2016)06-0089-04
作者简介:牛晓艳(1984-),女,山西太原人,助理研究员,主要从事海狸色獭兔育种工作。

