藤仓赤霉(Gibberellafujikuroi)分子生物学及发酵工程研究进展
李 超, 杨佳羽, 刘 佳, 朱廷恒
浙江工业大学生物工程学院, 杭州 310004
藤仓赤霉(Gibberellafujikuroi)分子生物学及发酵工程研究进展
李超,杨佳羽,刘佳,朱廷恒*
浙江工业大学生物工程学院, 杭州 310004
摘要:赤霉素是最重要的植物生长调节剂之一,工业化生产是由丝状真菌藤仓赤霉发酵产生。近20年来,随着分子生物学技术的发展,对藤仓赤霉赤霉素生物合成途径中相关基因的分子鉴定和表达调控等研究取得了显著的进展,赤霉素生物合成途径的分子生物学基本研究清楚,使得利用基因工程和代谢工程技术进行赤霉菌改良、提高赤霉素发酵水平成为可能。本文对藤仓赤霉中赤霉素合成机理及其表达调控、关键酶基因功能、外源基因转化系统、发酵技术、利用基因工程技术进行改造等方面的研究进展进行综述。
关键词:藤仓赤霉; 分子机理; 生物合成; 基因表达调控; 发酵
水稻恶苗病原菌藤仓赤霉(Gibberellafujikuroi)在1917年首次被Sawada等从被感染的水稻种子中分离出来,该菌以产生赤霉素(Gibberellins, GAs)而闻名,如GA3、GA4、GA7、GA9等[1]。赤霉素是一种由高等植物、部分真菌和细菌产生的重要的二萜类植物激素。目前,在自然界中已有超过130种赤霉素烷类化合物被发现[2]。其中一些赤霉素可控制植物种子萌发、茎的伸长、开花诱导和果实的生长等过程[3],其中GA3作为藤仓赤霉最主要的赤霉素产物已在农业中广泛应用。而GA4、GA7由于生物活性与GA3不同,并且发酵产量更低,因此其市场价格也更加昂贵[4]。藤仓赤霉除了能合成多种赤霉素,还被发现能产生多种有用的次级代谢产物。Linnemannstons等和Studt等通过研究pks4基因的功能和序列分析证明藤仓赤霉可产生比卡菌素和链红菌素这两种聚酮化合物[5-6]。Bearder等发现一些真菌毒素,如镰刀菌素也是其次级代谢产物[7]。
由于赤霉素在农业上的巨大潜力,对藤仓赤霉的开发与应用进行了大量的研究。近20年来,随着分子生物学技术的发展和应用,对藤仓赤霉赤霉素合成途径的研究取得了突破性的进展。Tudzynski等在1998年通过构建筛选赤霉菌cDNA文库的方法发现了涉及赤霉素合成的基因簇[8]。此后,赤霉素合成途径中所有的相关基因都被克隆出来,这些基因所编码的酶的特性也被逐一鉴定[9]。这不仅为研究赤霉素生物合成途径中复杂的调控机制提供了依据,也为通过基因工程手段改造代谢途径增加产量奠定了基础。
1赤霉素生物合成的分子机理
1.1赤霉素生物合成途径
经过30多年的研究,藤仓赤霉中赤霉素的生物合成途径已经被研究清楚(图1),其中各种中间代谢产物也已阐明[9-10]。赤霉素的生物合成始于羟甲基戊二酰辅酶A,其在3-羟基-3-甲基-戊二酰CoA(HMG-CoA)还原酶的催化生成甲羟戊酸(MVA),甲羟戊酸经异戊二烯基焦磷酸(IDP)、香叶基焦磷酸后(GDP),由法尼基焦磷酸(FDP)合成酶催化成FDP;而后FDP在香叶基香叶基焦磷酸(GGDP)合成酶的作用下生成GGDP。随后,GGDP在一种双功能酶——古巴焦磷酸(CPP)、内根-贝壳杉烯合成酶(CPS/KS)的催化下,经两步反应由古巴焦磷酸(CPP)转化成内根-贝壳杉烯。该步骤被认为是赤霉素合成途径中第一个关键性的限速步骤[11-12]。高度疏水的内根-贝壳杉烯在多功能细胞色素P450-4单氧酶的催化下,经由贝根-贝壳杉烯醇、内根-贝壳杉烯醛转化成内根-贝壳杉烯酸[13]。内根-贝壳杉烯酸在多功能细胞色素P450-1单氧酶的催化下,经由7β-羟基-贝壳杉烯酸、GA12-醛、GA14-醛,转化成GA14[14]。GA14由细胞色素P450-2单氧酶催化转化为GA4[15],而GA4既可在1,2-去饱和酶的作用下转化成GA7,又可在P450-3作用下转化为GA1。最后,GA7在P450-3的作用下转化成GA3[16]。
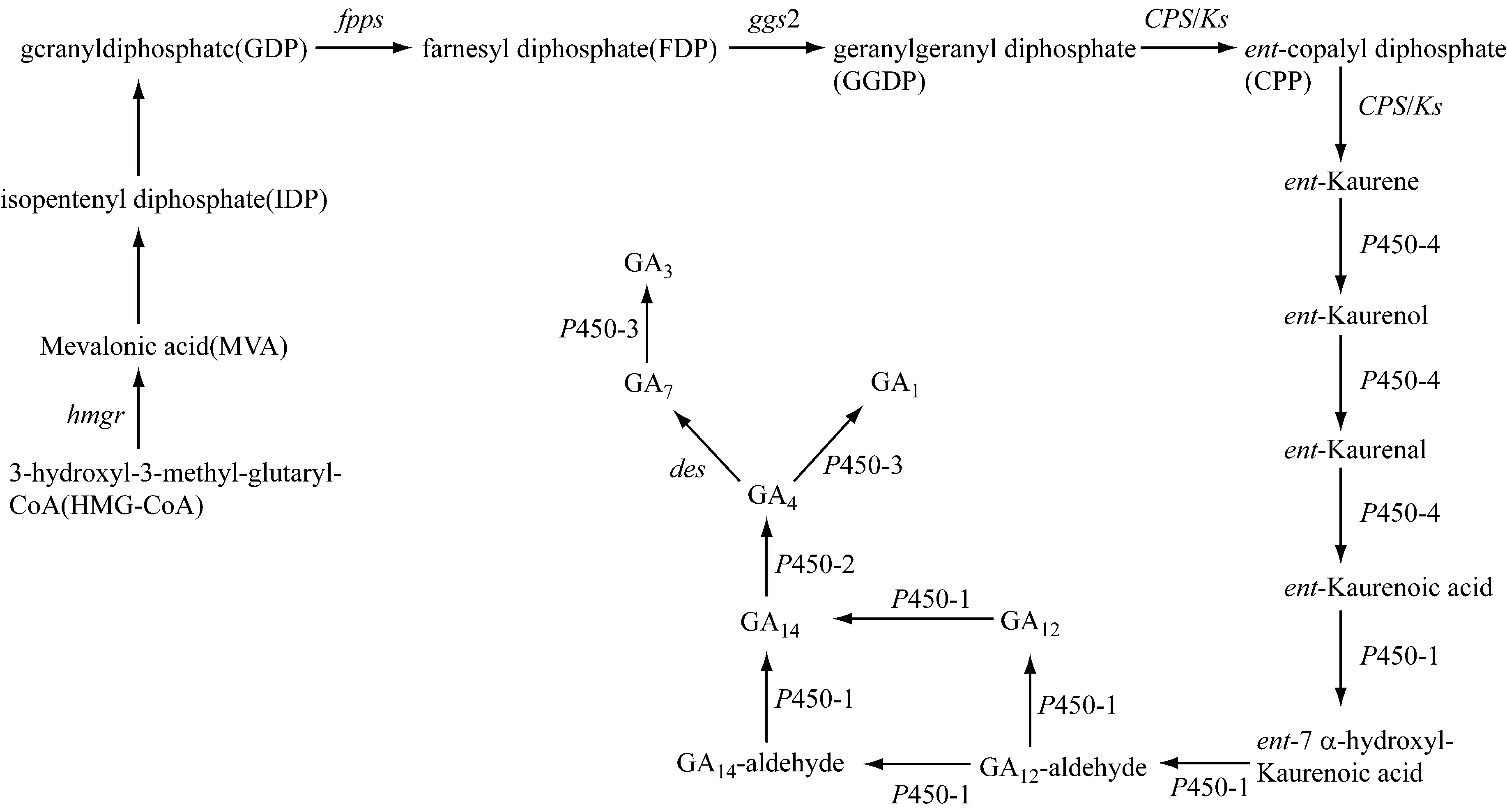
图1 藤仓赤霉中赤霉素合成途径[10]
1.2赤霉素生物合成途径中涉及的基因
赤霉素生物合成途径基因主要分成两部分[9-10]:类异戊二烯途径基因、GAs合成基因。HMG-CoA还原酶基因(hmgr)、FDP合成酶基因(fpps)、香叶基香叶基焦磷酸合成酶基因(ggs2)三个在真菌和植物中高度保守的类异戊二烯途径基因的表达水平并不受光、pH、碳氮源浓度影响[17-19]。相比之下,真菌中控制GA合成部分的7个基因除了ggs2之外,cps/ks、des、p450-4、p450-3、p450-2、p450-1与植物相比同源性却并不高。Tudzynski等利用差异基因筛选、限制酶介导的基因重组、PCR技术克隆了这些基因。同时又利用染色体步移法确定这7个基因紧密连锁,且存在于藤仓赤霉同一条染色体上[20]。
1.3赤霉素合成基因的功能
藤仓赤霉中GAs合成相关的这7个基因组成了一个基因簇,但各自转录方向并不一样(图 2)。Des基因位于该基因簇最左侧含有一个长1 029bp的开放阅读框,编码GA4去饱和酶,催化GA4转化为GA3的反应,此基因的表达受到高浓度氮源的抑制[16]。P450-4位于des右侧,编码多功能细胞色素P450-4单氧酶,该酶催化内根-贝壳杉烯到内根-贝壳杉烯酸的3步氧化反应[13]。P450-1位于P450-4的右侧,编码多功能细胞色素P450-1单氧酶,该酶主要催化内根-贝壳杉烯酸到GA14的4步反应:7β-羟基化、氧化C-6位收缩B环、3β-羟基化、氧化C-7位,还可将赤霉素生物合成途径内的中间产物内根-贝壳杉烯酸和内根-7α-羟基-内壳杉烯酸分别催化成kaurenolides和fujenoic acids[14]。P450-1和P450-4两个基因在基因簇中紧密相连,且共用一个启动子,转录方向相反。P450-2位于P450-1的下游,编码多功能细胞色素P450-2单氧酶,该酶在藤仓赤霉中主要通过催化GA14C-20位氧化将其转化为GA4,同时还涉及少量GA12C-20位氧化将其转化为GA9的反应[15]。Ggs2编码香叶基香叶基焦磷酸(GGDP)合成酶催化法尼基焦磷酸(FDP)转化成GGDP。cps/ks编码一个双功能的酶——古巴焦磷酸(CDP)、内根-贝壳杉烯合成酶(CPS/KS),催化两步环化反应,使GGDP转化成内根-贝壳杉烯。而cps/ks与ggs2两者紧密连锁,共用一个839 bp的启动子,这样可能更利于GGDP合成酶特异性地为CPS/ks提供GGDP[21]。然而在高等植物中,这两步催化反应由两个不同的酶分别催化完成。P450-3位于基因簇最右侧,是藤仓赤霉中赤霉素合成途径最后一个基因,含2个开放式阅读框,该基因编码细胞色素P450-3单氧酶,催化GA7到GA3和GA4到GA1的反应[16]。
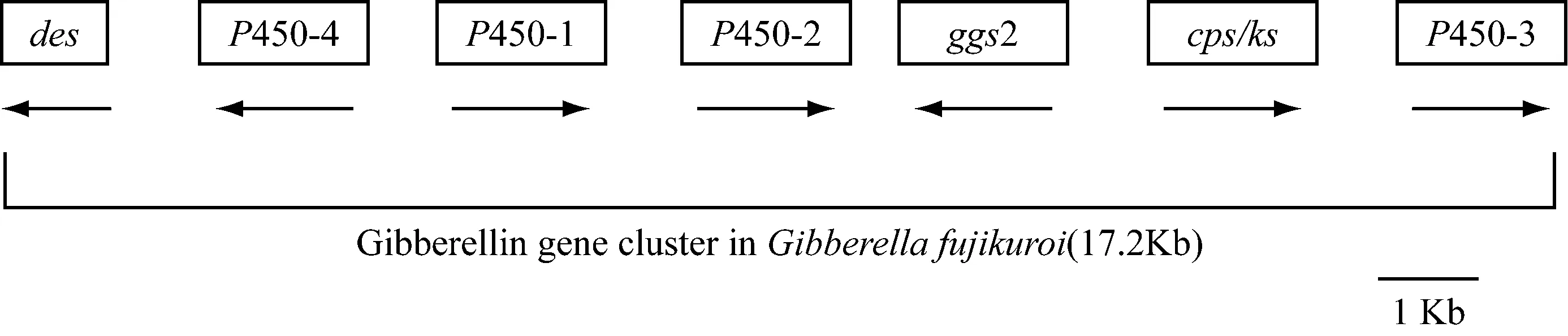
图2 藤仓赤霉中赤霉素生物合成基因簇[10]
2外源基因转化系统
建立一个高效的的藤仓赤霉遗传转化系统,对赤霉菌分子遗传学研究、赤霉素生物合成分子机理研究、基因工程改造都至关重要。自1973年Mishra等[22]首次报道粗糙链孢霉(Neurospora crassa)的转化以来,丝状真菌遗传转化系统的研究发展非常迅速。藤仓赤霉的遗传转化系统也已经建立,包括限制酶介导的整合技术体系(restriction enzyme mediated integration,REMI)[23]和农杆菌介导的转化体系(Agrobacteriumtumefaciensmediated transformation, ATMT)[24]。
2.1载体系统
在REMI技术中,需要制备藤仓赤霉原生质体作为外源基因的受体细胞。原生质体的转化载体主要有两种构建方式,采用细菌质粒构建转化载体和采用融合PCR技术(fusion PCR)构建基因大片段。1991年,Sanchez-Fernandez等[25],用1-甲基-3-硝基-1-亚硝基胍处理野生型藤仓赤霉,得到一株不能在以硝酸盐为唯一氮源的培养基上生长的突变株SG140(niaD11-)。随后,以含完整黑曲霉(Aspergillusniger)硝酸还原酶基因(niaD)的质粒pSTA10为基础,通过营养缺陷互补筛选转化子,构建了第一个藤仓赤霉的转化系统。但其转化效率偏低,仅为1~2个转化子/μg DNA。1992年,Brückner B 等[26]构建了以构巢曲霉(A.nidulans)鸟氨酸氨甲酰转移酶基因argB为筛选标记的载体,虽然筛选效率有所提高,但转化率并没有明显的提高。1996年,Tudzynski 等[27]分离并克隆了藤仓赤霉硝酸还原酶基因(niaD),构建了一个可用于转化niaD基因缺陷菌株niaD57的粘粒载体pGFniaD,转化频率高达200个转化子/μgDNA。
在ATMT体系中,需要构建双源载体,对完整或部分脱壁的细胞为受体进行外源基因转化。1998年,de Groot等[24]建立了赤霉菌的农杆菌介导的转化方法,以农杆菌Ti质粒为基础构建了一个包含潮霉素抗性基因、gpd启动子、trpC终止子的双元载体pUR5750。该方法大大提高了藤仓赤霉外源基因的转化率,可产生300~7 200个转化子/107个原生质体。目前,由于融合PCR技术采用可在不需要内切酶消化和连接酶处理的条件下实现DNA片段的体外链接,避免了繁琐的传统载体构建步骤,已被广泛应用于各种真菌转化载体的构建。Yu J等[28]利用该方法在真菌中成功敲除31个基因,并且顺利地将超过10个基因与一个启动子相融合。
2.2转化方法
在藤仓赤霉工程菌的构建过程中,转化是最关键的步骤之一。1991年,Leslie等采用醋酸锂介导法,成功将外源基因导入藤仓赤霉孢子中,可得到1~2个转化子/μgDNA[29]。但该方法转化效率偏低,且目前赤霉素工业生产用菌株都是经过诱变育种的不产孢的突变菌株。Sanchez-Fernandez等[25]以CaCl2-PEG介导法将含硝酸还原酶基因(niaD)的质粒pSTA10顺利导入藤仓赤霉突变株SG140原生质体中,可得1~2个转化子/μg DNA,该方法此后又逐步得到改进[27]。农杆菌可通过侵染,将Ti质粒中的T-DNA导入丝状真菌孢子或原生质体内,并使这部分DNA随机整合到真菌基因组上。de Groot等[24]利用农杆菌这一性质,构建双元载体成功转化了多种曲霉、木霉等丝状真菌,其转化频率比传统转化方法高600倍。国内,郝丽梅等[30]也报道了农杆菌介导的藤仓赤霉转化方法,但是农杆菌介导的真菌转化以孢子为受体的转化率较高,以菌丝为受体的转化率却较低。2013年,朱廷恒等[31]首次利用电击法转化赤霉菌原生质体,结果证明电压为0.8~1.0 kV时赤霉菌原生质体的转化率最高,可得18个转化子/μgDNA。该方法操作步骤少,过程相对简单,可应用于藤仓赤霉基因工程研究。
3赤霉素生物合成基因的表达调控
丝状真菌次级代谢产物的合成有着极其复杂的调控机制,涉及到如碳/氮源、温度、pH、光照、活性氧、有毒化合物等外界环境因素对真菌细胞的刺激,调控基因的表达,合成途径中相关基因的转录激活和表达翻译等[32]。而外界环境刺激对丝状真菌次级代谢的调控主要通过调控因子来实现。大多数调控因子含有如锌指结构域、α-螺旋-环-α-螺旋、亮氨酸拉链等DNA结合蛋白结构,通过特异性识别并结合靶基因启动子序列调控结构基因的表达[33]。
在藤仓赤霉代谢途径的研究过程中,发现高浓度的氮源(硝酸盐、氨态盐)、葡萄糖会降低赤霉素合成的产量[34],且一些赤霉素合成抑制剂如阳离子络合物、含氮的杂环化合物、类α-酮戊二酸化合物、16,17-二氢-GAs等也能抑制赤霉菌中赤霉素的合成[35]。其中高浓度氮源对赤霉素合成的抑制效果尤为明显[36]。1999年,Tudzynski等[37]首次克隆了藤仓赤霉氮代谢相关基因areA-GF,该基因与粗糙链孢霉基因nit-2、构巢曲霉基因areA等氮代谢调控基因有很高的同源性,并编码一个含有972个氨基酸残基的蛋白质。而这些基因表达蛋白质内含有一个同源性高达98%的锌指DNA结合域。同时,Tudzynski等又通过同源重组和同源互补实验证明,areA-GF的缺失会导致藤仓赤霉无法利用除铵盐和谷氨酸盐外的氮源,且赤霉素产量也大幅降低。2003年,Mihlan等[38]的研究表明,赤霉素虽然是不含氮的二萜类化合物,但areA-GF编码的蛋白质可直接结合在cps/ks、des、p450-4、p450-2、p450-1、ggs2这六个可被氮调节的基因的启动子区域上。因此,当areA-GF缺失将导致这六个基因的表达水平急剧降低。而p450-3是此基因簇中唯一不受氮源调控的基因[16]。在构巢曲霉(A.nidulans)和粗糙链孢霉(N.crassa)中,AREA和NIT2在翻译后直接与nmr表达的基因结合,因此还受到nmr基因的调控[39-40]。Mihlan等通过对藤仓赤霉中nmr的敲除和超表达证明,该基因对氮代谢及赤霉素生物合成的抑制并不剧烈[38]。除此之外,藤仓赤霉谷氨酰胺合成酶基因glnA-GF的敲除导致赤霉素和比卡菌素生物合成的大幅下降,暗示glnA-GF也有可能涉及该菌的氮代谢调控[41]。
在构巢曲霉中,meaB编码bZIP的一个转录因子,该转录因子可影响氮调控基因的表达[42]。在氮源充足的情况下,MeaB通过与nmrA启动子上保守序列结合激活nmrA的表达,从而间接地影响AreA的活性[43]。在赤霉菌中,meaB的转录调控方式与A.nidulans有所不同。meaB由两个转录子组成,meaBL在氮源充足的环境下以依赖AreA的方式起主导作用,而meaBS在氮源受到限制的环境下以同样的方式起主导作用。在氮源受限的条件下,meaB的敲除导致如赤霉素合成基因、比卡菌素合成基因等受氮源调控的基因表达量大幅增加。当meaA和areA被双敲除,GA合成基因不再表达,而比卡菌素合成基因表达量大幅增加。因此,meaB与areA互相合作介导藤仓赤霉的氮代谢调控,但meaB的调控功能并不能代替areA[44]。Studt等[45]证明,cAMP介导的G蛋白信号传导途径对藤仓赤霉次级代谢过程有重要调控作用,其中赤霉素的合成依赖于有火星的腺苷酸环化酶和PKA2,而这种调控并不涉及G蛋白的α亚基。此外,近年来有研究证明,全局调控基因如Velvet、组蛋白甲基转移酶基因LaeA、fSge1有激活赤霉素合成相关基因的作用[44][46]。
4藤仓赤霉基因工程改造
在赤霉素合成的分子生物学基础上,可以通过基因工程手段进行赤霉菌改良,从而调控赤霉素的不同组份、比例、产量等。Tudzynski等[16]对藤仓赤霉赤霉素合成途径末端两个基因des、P450-3进行敲除,以得到只生产GA4或GA7的工程菌株。结果发现,des敲除株不再产生GA3与GA7,而GA1与GA4则大幅增加,两者合成比例约为5∶1。而P450-3敲除株不再生产GA3与GA1,同时GA4与GA7的产量较野生型菌株大幅增加,此结果与藤仓赤霉紫外突变株6314(P450-3 884 bp处带一个点突变)一致,因此该菌株可作为生产GA4+7的工程菌。当des与P450-3两者都被敲除,敲除株只生产GA4,并且其产量比野生型高7~8倍。Wiemann等[47]将藤仓赤霉磷酸泛酰巯基乙酰转移酶编码基因ppt1敲除,导致聚酮化合物合酶(PKSs)和非核糖体缩氨酸合酶(NRPSs)活性丧失,与这两种酶相关的下游次级代谢反应均不再进行,因此代谢流向赤霉素合成途径转移,使GAs产量大幅增加。为了显著提高藤仓赤霉GAs合成效率,2013年Tudzynski等[1]在野生型或6314基础上将合成途径中hmgr、fpps、ggs2、cps/ks等几个关键酶基因进行超表达。结果显示,在同等培养条件下hmgr和fpps的超表达反而导致赤霉素产量下降,这可能是由于上游前体物质的增加激活了途径中的反馈抑制。而将6314菌株中ggs2进行超表达后发现,赤霉素总产量提高135%。由于ggs2与cps/ks共用一个启动子,因此同时将两者进行超表达,结果赤霉素总产量提高近54%。Hmgr表达的酶由两个部分组成,分别为含8个跨膜区域的疏水性N端和一个亲水性的C端,而N端跨膜域被怀疑与反馈抑制相关。因此在6314的基础上敲除hmgr的N端序列,超表达hmgr的C端序列,最后赤霉素总产量相比6314提高了260%。目前,对于藤仓赤霉基因工程改造方面的研究还很少,且在提高赤霉素产量方面的成果也并不显著,因此该方向有待发展。
5藤仓赤霉生产赤霉素的发酵技术
目前利用赤霉菌的工业化大规模发酵,全世界每年可生产超过25吨赤霉素,市场价值高达1亿美元。工业化的赤霉素生产技术以液态发酵为主,也有固态发酵的报道。科学界在提高赤霉素发酵产量和降低生产成本上投入了大量研究[9]。但还存在着产量偏低、成本过高、后期分离难度过大等原因。目前,在发酵方法方面,既对传统的液体发酵技术、固体发酵技术进行改进,也尝试如固定化发酵技术这样的新技术。
5.1液体深层发酵技术
长久以来,工业上一直采用藤仓赤霉的液体深层发酵技术生产赤霉素,但产量通常受到产物反馈抑制、化学分解、菌株退化等原因的限制[48]。1995年Hollman等[49]首次使用持续萃取发酵技术,在发酵过程中持续地将发酵液从发酵罐中泵入一个交叉流微滤装置进行过滤。滤液则被泵入装有65%Genapol和35%水的萃取塔中进行萃取,而滤出的微生物被回收并加入发酵罐重新利用。该方法赤霉素产量为520 mg/L,但从一定程度上降低了产物的反馈抑制。同时,液体深层发酵技术还受到许多外界因素的影响,如pH、温度、光照、通氧、碳氮比等[50]。当pH在3.5~5.0之间,温度25 ℃时,GA3的发酵产量可达到最大;当pH在6~7之间,温度超过32 ℃时,发酵液中GA4、GA7的积累量会增加[51-52]。由于赤霉素的生物合成受到高浓度氮的抑制,所以保持培养基中低浓度的氮源有利于赤霉素产量的提高[53-54]。而高浓度的葡萄糖对赤霉素的合成有明显的抑制作用[55],葡萄糖的抑制机理目前还不清楚。碳源的选择对赤霉素的发酵产量至关重要,有研究表明以麦芽糖、甘露糖、丙三醇、半乳糖等代替葡糖糖作为碳源可增加GA3产量[56]。由于赤霉素的生物合成是在类异戊二烯途径的基础上进行的,因此也有研究表明在培养基中添加植物油可增加GA3的产量[57]。一些如牛乳清粉、糖蜜、甜菜渣等工业废料也可作为碳源,用于赤霉素的发酵生产[58]。有报道证明,以贻贝加工废弃物作为原料液体深层发酵生产赤霉素,产量可达3 g/L[59]。
5.2赤霉素的固态发酵技术
固态发酵技术(SSF)指利用不溶性的原料作为营养源和物理支持物,使微生物在较低水活度的环境下大量繁殖并生产目的产物。工业上生产微生物次级代谢产物,固态发酵技术相较于深层液体发酵技术有着更高的产量,且需要的能量更少,下游处理工艺更简单,大大节省了生产成本[60]。因此,目前固态发酵技术在赤霉素的生产领域也受到了广泛的关注。而赤霉素固态发酵主要受到底物投加方法、环境条件、营养因子、固体支持物种类等因素的影响[50]。1987年,Kumar等[61]通过实验证明玉米麸、麦麸、米糠、木薯粉等廉价原料也可作为固态发酵GA3的原料,且GA3的产量比普通深层液态发酵高1.6倍。1996年,Bandelier等[62]首次在一种无菌小规模反应器(50 L)中,通过分批补料固态发酵的方法以含50%干物质的小麦麸为原料生产赤霉素GA3,测得GA3产量高达3 g/Kg。近几年,有研究发现固态发酵培养基中水活度也是影响因素之一。2005年,Corona等[63]以小麦麸和可溶性淀粉作为有机基质研究不同水活度对赤霉素固态发酵产量的影响,结果发现当水活度为0.99的最适条件下,GA3产量可达(4.5~5.0)g/Kg。一些惰性载体如离子交换树脂,也被尝试着作为藤仓赤霉生长和赤霉素生产的支持物,结果发现其最大生物量达到40 mg/g,赤霉素GA3的产量则可达到0.73 mg/g[64]。麻风树的种子富含油,当油被榨出后剩下的麻风树种子饼也可被作为发酵生产赤霉素的原料,且可大幅度提高产量。有报道指出,以麻风树种子饼作为底物进行液态深层发酵时,保持最佳物理参数,GA3产量最高可达15 g/L;而作为固态发酵底物时,最高产量史无前例地达到了105 g/Kg[65]。
5.3固定化赤霉菌发酵技术
近几年,固定化细胞发酵技术以其独特的优势越来越多地被应用于赤霉素的生产。通过藤仓赤霉菌丝的固定化可以获得较高的细胞密度,并且提高传质速率,有利于保持高生产率;固定化细胞生长消耗的底物更少,且底物补加过程可控性更高从而提高底物利用率[66]。不同的固定化方法对藤仓赤霉的稳定性和赤霉素产量都有不同影响,一些聚合材料如聚氨酯、角叉菜胶、海藻酸盐被报道可用于藤仓赤霉的固定化[50]。Kumar等[67]以海藻酸钠或卡拉胶凝胶珠固定藤仓赤霉菌丝,在一个倒置的圆锥形流态化生物反应器中进行反复分批发酵,结果发现由于培养基的重复使用、固态化细胞不稳定导致GA3的产量反而低于液体发酵20%。有研究证明,将藤仓赤霉细胞附着于被亲水性羟乙基丙烯酸酯和疏水性三甲基丙烯酸酯共聚物覆盖的聚合纤维载体之上,连续发酵12批次的循环超过84天,赤霉素产量稳定地维持在210 mg/L[68]。2000年,Escamilla等[69]以聚半乳糖醛酸钙固定化藤仓赤霉,并优化了培养基中PH、碳氮比、米粉浓度、温度等因素,在一个流态化反应器中进行分批发酵,最终GA3产量高达3.9 g/L。以硅藻土珠子作为材料固定藤仓赤霉细胞,在7L的无菌发酵罐中进行反复分批发酵,赤霉素产量是同样条件下液体发酵的3倍。而发酵过程中添加的棉籽饼粉浓度可对赤霉素产量及操作稳定性产生很大影响,当棉籽饼粉与葡萄糖在培养基中含量达到1∶10时,赤霉素产量达到最大,固定化细胞稳定性达到最高[66]。国内,陈九武[70]等建立了聚乙烯醇水凝胶的固定化方法,并与海藻酸钙凝胶进行了比较,发现该凝胶在机械轻度、使用寿命、贮存稳定性等方面均更优秀。因此,可将该方法应用于各种真菌的固定化发酵。藤仓赤霉固定化发酵与上述液态发酵和固态发酵相比虽然有很多优势,但在大规模发酵生产赤霉素上遭遇了很多问题,如菌种变异、固定化细胞稳定性不高、发酵过程控制困难等。因此,藤仓赤霉的最佳固定化方法和发酵规模的扩大还有待研究。
6结束语
赤霉素作为能有效调控植物生长发育的主要激素,其生物合成无论在植物还是微生物中都受到严格的调控。相比高等植物和可生产赤霉素的细菌,藤仓赤霉的赤霉素生物合成机理已被研究得较为清楚。赤霉素的生物合成途径中的大部分基因的序列、功能特征也通过定点敲除、同源重组、互补等手段被深入地研究。这对在分子水平上系统地认识赤霉素生物合成途径中的酶、调控方式、中间代谢产物、代谢调节机理有极大的帮助,同时也为利用基因工程手段获得高产菌株提供了可能。目前一些与赤霉素生物合成相关的基因表达调控过程已被初步研究。但氮代谢抑制的分子机制并不清楚,是否存在其他调控基因有待进一步地研究。还有途径中涉及调控的各基因之间的作用机制和转录因子识别方式也有待解释清楚。
目前,通过改良传统发酵技术,可在一定程度上提高赤霉素产量。但随着农业上对赤霉素日益增长的需求,通过如菌种选育、培养基优化、发酵技术改良等方法并不能大幅地提高赤霉素产量、简化下游操作。因此,利用基因工程技术对赤霉素生物合成途径进行分子改造,从而得到产量高、赤霉素种类单一的工程菌株势必成为研究赤霉菌的趋势。
参考文献
[1]Albermann S, Linnemannstons P, Tudzynski B. Strategies for strain improvement inFusariumfujikuroi: overexpression and localization of key enzymes of the isoprenoid pathway and their impact on gibberellin biosynthesis. Applied Microbiology and Biotechnology, 2013, 97(7): 2979-2995.
[2]Bomke C, Tudzynski B. Diversity, regulation, and evolution of the gibberellin biosynthetic pathway in fungi compared to plants and bacteria. Phytochemistry, 2009, 70(15-16): 1876-1893.
[3]Hooley R. Gibberellins: perception, transduction and responses. Plant Molecular Biology, 1994, 26(5): 1529-1555.
[4]Lale G, Gadre R. Enhanced production of gibberellin A4 (GA4) by a mutant ofGibberellafujikuroiin wheat gluten medium. Journal of Industrial Microbiology &Biotechnology, 2010, 37(3): 297-306.
[5]Linnemannstons P, Schulte J, Prado MD,etal. The polyketide synthase genepks4 fromGibberellafujikuroiencodes a key enzyme in the biosynthesis of the red pigment bikaverin. Fungal Genetics and Biology, 2002, 37(2): 134-148.
[6]Studt L, Wiemann P, Kleigrewe K,etal.Biosynthesis of Fusarubins Accounts for Pigmentation ofFusariumfujikuroiPerithecia. Applied and Environmental Microbiology, 2012, 78(12): 4468-4480.
[7]Barrero AF, Sanchez JF, Oltra JE,etal. Fusarin-C and 8z-Fusarin-C fromGibberellafujikuroi. Phytochemistry, 1991, 30(7): 2259-2263.
[8]Tudzynski B, Holter K. Gibberellin biosynthetic pathway inGibberellafujikuroi: evidence for a gene cluster. Fungal Genetics and Biology, 1998, 25(3): 157-170.
[9]Tudzynski B. Biosynthesis of gibberellins inGibberellafujikuroi: biomolecular aspects. Applied Microbiology and Biotechnology, 1999, 52(3): 298-310.
[10]Tudzynski B. Gibberellin biosynthesis in fungi: genes, enzymes, evolution, and impact on biotechnology. Applied Microbiology and Biotechnology, 2005, 66(6): 597-611.
[11]Hedden P, Kamiya Y. Gibberellin biosynthesis: Enzymes, genes and their regulation. Annu Rev Plant PhysPlant Mol. Biol., 1997, 48: 431-460.
[12]Sun TP, Kamiya Y. Regulation and cellular localization ofent-kaurene synthesis. Physiol Plantarum, 1997, 101(4): 701-708.
[13]Tudzynski B, Hedden P, Carrera E,etal. The P450-4 gene ofGibberellafujikuroiencodesent-kaurene oxidase in the gibberellin biosynthesis pathway. Applied and Environmental Microbiology, 2001, 67(8): 3514-3522.
[14]Rojas MC, Hedden P, Gaskin P,etal.The P450-1 gene ofGibberellafujikuroiencodes a multifunctional enzyme in gibberellin biosynthesis. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(10): 5838-5843.
[15]Tudzynski B, Rojas MC, Gaskin P,etal. The gibberellin 20-oxidase ofGibberellafujikuroiis a multifunctional monooxygenase. The Journal of Biological Chemistry, 2002, 277(24): 21246-21253.
[16]Tudzynski B, Mihlan M, Rojas MC,etal. Characterization of the final two genes of the gibberellin biosynthesis gene cluster ofGibberellafujikuroi:desandP450-3 encode GA4 desaturase and the 13-hydroxylase, respectively. The Journal of Biological Chemistry, 2003, 278(31): 28635-28643.
[17]Homann V, Mende K, Arntz C,etal. The isoprenoid pathway: Cloning and characterization of fungalFPPSgenes. Current Genetics, 1996, 30(3): 232-239.
[18]Mende K, Homann V, Tudzynski B. The geranylgeranyl diphosphate synthase gene ofGibberellafujikuroi: isolation and expression. Molecular &General Genetics: MGG, 1997, 255(1): 96-105.
[19]Woitek S, Unkles SE, Kinghorn JR,etal. 3-Hydroxy-3-methylglutaryl-CoA reductase gene ofGibberellafujikuroi: Isolation and characterization. Current Genetics, 1997, 31(1): 38-47.
[20]李红民, 贾敬芬, 梅兴国. 赤霉菌分子生物学研究进展. 微生物学报, 2004, 44(4): 552-555.
[21]Domenech CE, Giordano W, Avalos J,etal. Separate compartments for the production of sterols, carotenoids and gibberellins inGibberellafujikuroi. Eur J Biochem, 1996, 239(3): 720-725.
[22]Mishra NC, Tatum EL. Non-Mendelian inheritance of DNA-induced inositol independence inNeurospora. Proceedings of the National Academy of Sciences of the United States of America, 1973, 70(12): 3875-3879.
[23]Linnemannstons P, Voss T, Hedden P,etal. Deletions in the gibberellin biosynthesis gene cluster ofGibberellafujikuroiby restriction enzyme-mediated integration and conventional transformation-mediated mutagenesis. Applied and Environmental Microbiology, 1999, 65(6): 2558-2564.
[24]de Groot MJA.Agrobacteriumtumefaciens-mediated transformation of filamentous fungi. Nat Biotechnol, 1998, 16(11): 1074.
[25]Sanchez-Fernandez R, Unkles SE, Campbell EI,etal. Transformation of the Filamentous FungusGibberellafujikuroiUsing theAspergillusnigerniaDGene Encoding Nitrate Reductase. Molecular & General Genetics, 1991, 225(2): 231-233.
[26]Bruckner B, Unkles SE, Weltring K,etal. Transformation ofGibberellafujikuroi: Effect of theAspergillusnidulansAMA1 Sequence on Frequency and Integration. Current genetics, 1992, 22(4): 313-316.
[27]Tudzynski B, Mende K, Weltring K-M,etal. TheGibberellafujikuroiniaDgene encoding nitrate reductase: isolation, sequence, homologous transformation and electrophoretic karyotype location. Microbiology, 1996, 142(Pt 3): 533-539.
[28]Yu JH, Hamari Z, Han KH,etal. Double-joint PCR: a PCR-based molecular tool for gene manipulations in filamentous fungi. Fungal Genetics and Biology, 2004, 41(11): 973-981.
[29]Leslie JF, Dickman MB. Fate of DNA Encoding Hygromycin Resistance after Meiosis in Transformed Strains ofGibberellafujikuroi(Fusariummoniliforme). Applied and Environmental Microbiology, 1991, 57(5): 1423-1429.
[30]郝丽梅, 龚伟, 梅兴国等. 一种农杆菌介导的赤霉菌CCRC3.572的转化方法. 生物技术通讯, 2006(6): 888-890.
[31]朱廷恒, 易佳炜, 王渭霞等. 赤霉菌原生质体外源基因高效转化方法研究. 浙江工业大学学报, 2013, 41(5): 482-485.
[32]Brakhage AA. Regulation of fungal secondary metabolism. Nat Rev Microbiol, 2013, 11(1): 21-32.
[33]Garvie CW, Wolberger C. Recognition of specific DNA sequences. Mol. Cell, 2001, 8(5): 937-946.
[34]Rodrigues C, Vandenberghe LPD, de Oliveira J,etal. New perspectives of gibberellic acid production: a review. Crit Rev Biotechnol, 2012, 32(3): 263-273.
[35]Rademacher W. Growth retardants: Effects on gibberellin biosynthesis and other metabolic pathways. Annu Rev Plant Phys Plant Mol. Biol., 2000, 51(4): 501-531.
[36]Munoz GA, Agosin E. Glutamine Involvement in Nitrogen Control of Gibberellic Acid Production inGibberellafujikuroi. Applied and Environmental Microbiology, 1993, 59(12): 4317-4322.
[37]Tudzynski B, Homann V, Feng B,etal. Isolation, characterization and disruption of the areA nitrogen regulatory gene ofGibberellafujikuroi. Molecular &General Genetics:MGG, 1999, 261(1): 106-114.
[38]Mihlan M, Homann V, Liu TWD,etal. AREA directly mediates nitrogen regulation of gibberellin biosynthesis inGibberellafujikuroi, but its activity is not affected by NMR. Molecular Microbiology, 2003, 47(4): 975-991.
[39]Andrianopoulos A, Kourambas S, Sharp JA,etal. Characterization of theAspergillusnidulansnmrAgene involved in nitrogen metabolite repression. Journal of Bacteriology, 1998, 180(7): 1973-1977.
[40]Jarai G, Marzluf GA. Analysis of conventional andinvitrogenerated mutants of nmr, the negatively acting nitrogen regulatory gene ofNeurosporacrassa. Molecular & General Genetics: MGG, 1990, 222(2-3): 233-240.
[41]Teichert S, Schonig B, Richter S,etal. Deletion of theGibberellafujikuroiglutamine synthetase gene has significant impact on transcriptional control of primary and secondary metabolism. Molecular Microbiology, 2004, 53(6): 1661-1675.
[42]Polley SD, Caddick MX. Molecular characterisation ofmeaB, a novel gene affecting nitrogen metabolite repression inAspergillusnidulans. FEBS Lett, 1996, 388(2-3): 200-205.
[43]Wong KH, Hynes MJ, Todd RB,etal. Transcriptional control ofnmrAby the bZIP transcription factor MeaB reveals a new level of nitrogen regulation inAspergillusnidulans. Molecular Microbiology, 2007, 66(2): 534-551.
[44]Wagner D, Schmeinck A, Mos M,etal. The bZIP Transcription Factor MeaB Mediates Nitrogen Metabolite Repression at Specific Loci. Eukaryot Cell, 2010, 9(10): 1588-1601.
[45]Studt L, Humpf HU, Tudzynski B. Signaling governed by G proteins and cAMP is crucial for growth, secondary metabolism and sexual development inFusariumfujikuroi. PloS one, 2013, 8(2): e58185.
[46]Michielse CB, Studt L, Janevska S,etal. The global regulator FfSge1 is required for expression of secondary metabolite gene clusters but not for pathogenicity inFusariumfujikuroi. Environ Microbiol, 2015, 17(8): 2690-2708.
[47]Wiemann P, Albermann S, Niehaus EM,etal. The Sfp-Type 4'-Phosphopantetheinyl Transferase Ppt1 ofFusariumfujikuroiControls Development, Secondary Metabolism and Pathogenicity. PloS one, 2012, 7(5): e37519.
[48]O'Donnell K, Cigelnik E, Nirenberg HI. Molecular systematics and phylogeography of theGibberellafujikuroispecies complex. Mycologia, 1998, 90(3): 465-493.
[49]Hollmann D, Switalski J, Geipel S,etal. Extractive Fermentation of GibberellicAcid byGibberellafujikuroi. J Ferment Bioeng, 1995, 79(6): 594-600.
[50]Shukla R, Srivastava AK, Chand S. Bioprocess strategies and recovery processes in gibberellic acid fermentation. Biotechnol Bioproc Eng, 2003, 8(5): 269-278.
[51]Borrow A, Brown S, Jefferys EG,etal. The Effect of Varied Temperature on the Kinetics of Metabolism ofGibberellafujikuroiin Stirred Culture. Canadian Journal of Microbiology, 1964, 10(3): 445-466.
[52]Borrow A, Brown S, Jefferys EG,etal. The Kinetics of Metabolism ofGibberellafujikuroiin Stirred Culture. Canadian Journal of Microbiology, 1964, 10(3): 407-444.
[53]Bruckner B, Blechschmidt D. Nitrogen Regulation of Gibberellin Biosynthesis inGibberellafujikuroi. Applied Microbiology and Biotechnology, 1991, 35(5): 646-650.
[54]Silva EME, Dendooven L, Reynell JAU,etal. Morphological development and gibberellin production by different strains ofGibberellafujikuroiin shake flasks and bioreactor. World J Microb Biot, 1999, 15(6): 753-755.
[55]Bruckner B. Regulation of Gibberellin Formation by the FungusGibberellafujikuroi. Ciba F Symp, 1992, 171(171): 129-143.
[56]Darken MA, Jensen AL, Shu P. Production of gibberellic acid by fermentation. Applied Microbiology, 1959, 7: 301-303.
[57]Gancheva V, Dimova T, Kamenov K,etal. [Biosynthesis of gibberellins. III. Optimization of the nutrient medium for gibberellin biosynthesis by using mathematical methods to plan experiments]. Acta Microbiologica Bulgarica, 1984, 14: 80-84.
[58]Cihangir N, Aksoz N. Production of gibberellic acid byAspergillusnigerusing some food industry wastes. Acta Microbiologica Polonica, 1996, 45(3-4): 291-297.
[59]Pastrana LM, Gonzalez MP, Murado MA. Production of Gibberellic Acid from Mussel Processing Wastes in Submerged Batch Culture. Bioresource Technol, 1993, 45(3): 213-221.
[60]Robinson T, Singh D, Nigam P. Solid-state fermentation: a promising microbial technology for secondary metabolite production. Applied Microbiology and Biotechnology, 2001, 55(3): 284-289.
[61]Kumar PKR, Lonsane BK. Gibberellic acid by solid state fermentation: Consistent and improved yields. Biotechnology and Bioengineering, 1987, 30(2): 267-271.
[62]Bandelier S, Renaud R, Durand A. Production of gibberellic acid by fed-batch solid state fermentation in an aseptic pilot-scale reactor. Process Biochem, 1997, 32(2): 141-145.
[63]Corona A, Saez D, Agosin E. Effect of water activity on gibberellic acid production byGibberellafujikuroiunder solid-state fermentation conditions. Process Biochem, 2005, 40(8): 2655-2658.
[64]Gelmi C, Perez-Correa R, Gonzalez M,etal. Solid substrate cultivation ofGibberellafujikuroion an inert support. Process Biochem, 2000, 35(10): 1227-1233.
[65]Rangaswamy V. Improved Production of Gibberellic Acid byFusariummoniliforme. Journal of Microbiology Research, 2012, 2(3): 51-55.
[66]Kim CJ, Lee SJ, Chang YK,etal. Repeated-batch culture of immobilizedGibberellafujikuroiB9 for gibberellic acid production: An optimization study. Biotechnol Bioproc Engineering, 2006, 11(6): 544-549.
[67]Kumar PKR, Lonsane BK. Immobilized growing cells ofGibberellafujikuroiP-3 for production of gibberellic acid and pigment in batch and semi-continuous cultures. Applied Microbiology & Biotechnology, 1988, 28(6): 537-542.
[68]Lu ZX, Xie ZC, Kumakura M. Production of Gibberellic Acid inGibberellafujikuroiAdhered onto Polymeric Fibrous Carriers. Process Biochem, 1995, 30(7): 661-665.
[69]Escamilla EM, Dendooven L, Magana IP,etal. Optimization of gibberellic acid production by immobilizedGibberellafujikuroimycelium in fluidized bioreactors. J Biotechnol, 2000, 76(2-3): 147-155.
[70]陈九武, 赵学慧,吴思方. 固定化细胞合成酯类载体的研究. 工业微生物, 1997,27(3): 27-31.
Advances in molecular biology and fermentation technology ofGibberellafujikuroi
LI Chao,YANG Jia-yu,LIU Jia,ZHU Ting-heng
Institute of biological engineering, Zhejiang university of technology, Hangzhou 310004
AbstractGibberellins (GAs), produced by fermentation of filamentous fungi Gibberella fujikuroi, is one of the most important plant hormones. With the developments of molecular biology techniques over the past 20 years, remarkable progresses were made in molecular identification and expression regulation of genes involved in gibberellin biosynthesis. Based on these progresses, the genetic engineering and metabolic engineering techniques for improving this fungus and enhancing the GAs fermentation level could be carried out. In this paper, the advances in GAs synthesis mechanism, expression regulation mechanisms, function of key enzyme genes, transformation system, fermentation technology and genetic engineering techniques about Gibberella fujikuroi were summarized.
Key wordsGibberella fujikuroi; molecular mechanisms; biosynthesis; expression regulation; fermentation
doi:10.3969/j.issn.1001-6678.2016.02.009
基金项目:浙江省新苗人才计划(编号2013R403048)。
作者简介:李超(1989~),男,硕士研究生。E-mail:563297356@qq.com。 *通讯作者: 朱廷恒,博士。Tel:86-571-88320741,E-mail:thzhu@zjut.edu.cn。

