稳态和时间分辨荧光光谱法检测pBR322质粒中十字形结构
夏岚 张旭
(1. 南京工业大学生物与制药工程学院,南京 211816;2. 盐城师范学院药学院,盐城 224051;3. 南京大学医学院,南京 210093)
稳态和时间分辨荧光光谱法检测pBR322质粒中十字形结构
夏岚1,2张旭2,3
(1. 南京工业大学生物与制药工程学院,南京 211816;2. 盐城师范学院药学院,盐城 224051;3. 南京大学医学院,南京 210093)
在目的DNA不被酶切成不同片段的情况下,应用光谱方法探测pBR322质粒中十字形结构DNA的形成。吡咯脱氧胞苷(Pyrrolo deoxycytidine,PdC)取代dC掺入到pBR322质粒的特定位点(3 195,3 222或3 281位点)。应用稳态和时间分辨荧光法研究十字形结构DNA的形成。结果显示:(1)稳态荧光特性表明,当PdC掺入到pBR322质粒的3 222位点,PdC-pBR322超螺旋荧光强度大约是松弛型PdC-pBR322的4倍;与此同时,其时间分辨荧光寿命大约比松弛型PdC-pBR322长约0.3 ns。当PdC掺入到3195或3281位点时,稳态荧光光谱和荧光寿命(两位点处约1.42 ns)并没有显著变化。(2)随着盐浓度的增大,荧光寿命略微有变化,但不是很明显。通过检测和分析pBR322质粒的荧光光谱和荧光寿命,表明能够形成在非配对环状部分3222位点含有dC的十字形结构。在一定的盐浓度(0-100 mmol/L)条件下,十字形结构能够保持其稳定性。
十字形结构;pBR322质粒;PdC;稳态和时间分辨荧光光谱法
在某些生理过程或生物技术如细菌的二分裂、病毒的复制、目的基因与运载体的结合等过程中,会发生DNA的复制、转录或翻译,或者RNA的复制或逆转录过程,都遵循碱基的互补配对原则[1]。然而,越来越多的证据表明B型DNA并不是DNA存在的唯一结构形式。基因调控区特殊形式的DNA(非双链结构DNA)在转录调控中起着关键的作用。例如,G-四链体是在转录调控区发现的一种特殊的DNA结构[2]。RNA或者单链DNA片段通过自身回折使互补的碱基配对能够形成发夹结构[3,4]。有研究报道在超螺旋质粒DNA中能够形成另外一种特殊的DNA结构形式,十字形结构,即具有反向重复序列的DNA的单链碱基分别自我互补配对而形成的一种二元对称结构[5,6]。生物体中很多遗传性疾病都与机体内的DNA重复序列有关[7]。强直性肌营养不良、脊髓小脑共济失调3型与发夹结构的形成密切相关[8,9]。在基因调控区的 某些结合位点,小分子或蛋白质与单链D NA具有高亲和力,从而易于结合到单链DNA[10,11]。迄今为止,特殊结构的DNA已成为药物开发中潜在 的重要靶点。
与DNA的发夹结构相似,DNA十字形结构与许多遗传过程密切相关。DNA的十字形突出结构参与了机体的某些转录调控、重组和复制。十字形结构在真核和原核生物基因组中广泛存在[12-14],通常会在启动子、增强子或终止子等基因调控区内形成。有研究报道十字形结构DNA通过与有关蛋白结合在芽殖酵母复制起源中发挥重要作用[15,16]。十字形结构的形成促进局部碱基对解除配对,有助于杆状病毒的转录和复制过程[17]。在牛酪氨酸羟化酶基因启动子的上游区域的十字形结构,可作为激活或阻遏蛋白控制细胞特异性启动子活性的目标位点[18]。在pBR322质粒的“-35”区域的十字形结构能够影响启动子与聚合酶的相互作用[19]。在Alu元件(灵长类动物特有的约300 bp长的重复序列)下游形成的十字形结构可明显抑制T细胞特异性增强子的作用[20]。因此,有必要深入了解十字形结构DNA的形成过程以及进一步揭示它们的生物学作用。
研究DNA十字形结构最常用的方法是使用酶裂解法把目标DNA酶解成碎片,然后通过分析不同DNA片段来判断十字形结 构的位置[20-22]。然而,使用这种方法存在两点不足之处:一是破坏目的基因二级结构;二是十字形结构形成中的某些特异性信息几乎很少能够被检测出来。荧光光谱法是研究核酸结构信息的一种重要方法,同时也有利于研究在体内或者体外溶液中不同浓度条件下它们之间的相互作用。天然的DNA碱基能够吸收200-300 nm区间的紫外辐射,但是激发态寿命很短,只有较弱的内源荧光,荧光量子产量很低。很多种荧光核酸碱基类似物被合成并掺入到DNA中[23,24]。PdC是一种能监测核酸二级结构的荧光碱基类似物,首先,它类似于天然碱基脱氧胞苷而不破坏双链结构的形成,此外,它有很大的斯托克斯位移并且荧光对环境因素的改变很敏感[25]。
本实验在没有改变pBR322质粒原有结构的前提下,用PdC分别代替3 195、3 222和3 281位置上的dC。根据配对的PdC的荧光寿命比非配对的短的理论,用时间分辨荧光光谱方法分别研究3195、3222和3281位置处的时间分辨荧光寿命。
1 材料与方法
1.1 材料
限制性内切酶BbvC I、Nb. BbvC I、Dpn I,deep vent DNA聚合酶,T4连接酶和pBR322 质粒购买于New England Biolabs公司;纯化的大肠杆菌(E.coli)DNA促旋酶和拓扑异构酶I购买于TopoGEN公司;寡核苷酸(引物,PdC寡核苷酸)购买于Midland Certified Reagent Company公司;XL10-gold感受态细胞购买于Stratagene公司。酶和转化细胞的使用严格按照制造商的指示说明进行。2700型PCR扩增仪(GeneAmp PCR System 2700)购买于Applied Biosystems公司。定点突变试剂盒(QuickChange XL Site-Directed Mutagenesis Kit)购买于Stratagene公司。凝胶回收试剂盒(QIAquick Gel Extraction Kit)、质粒提取试剂盒(Plasmid mega kit)均购买于QIAGEN公司。稳态和时间分辨荧光光谱仪购买于美国ISS公司。实验中使用的其他材料见表1。
1.2 方法
1.2.1 聚合酶链反应(PCR) PCR反应体积为50 μL,pBR322质粒的量为100 ng,引物为70 ng,dNTP的浓度为250 μmol/L,DNA聚合酶为5 U 的deep vent。PCR扩增条件为:95℃变性30 s,在50℃退火50 s和72℃延伸8 min,经25次循环后,样品储藏于4℃过夜。用1%的琼脂糖凝胶电泳检测扩增产物。
1.2.2 质粒转化、提取与 纯化 用Dpn I内切酶处理PCR扩增完成后的片段, 然后将突变产物转入XL10-gold超级感受态细胞,转化操作步骤严格按照定点突变试剂盒说明书进行。质粒提取和纯化严格按照质粒提取 和纯化手册操作。纯化后产品用1%琼脂糖凝胶电泳进行分析。突变的质粒DNA由Seqwright公司测序分析。
1.2.3 酶切与纯化 用限制性内切酶Nb. BbvC I酶切双突变的pBR322质粒,处理方法严格按照内切酶的说明书。应用凝胶回收试剂盒,获得纯化的长的酶切DNA片段。
1.2.4 pBR322载体与含PdC的片段连接 将酶切后纯化的DNA片段(pBR322载体)和含有PdC的113碱基序列的DNA片段进行连接。处理如下:将含有PdC的 DNA片段与pBR322载体混合(280∶1),将混合溶液置于75℃水浴中20 min,然后自 然冷却至室温。随后加入5 U T4 DNA连接酶。反应体系于25℃孵育不同时间(10、30、60、90 min)和37℃条件下孵育120 min。最后用含有0.5 μg/mL溴化乙锭的1%琼脂糖凝胶电泳进行分析鉴定。
1.2.5 连接后处理(纯化) 将1.2.4的体系加热到80℃维持20 min从而使T4 DNA连接酶失活。加入1 U的促旋酶置于37℃水浴中孵 育2 h后,应用1%琼脂糖凝胶电泳检测分析。

表1 实验中所用到的DNA序列
1.2.6 稳态和时间分辨荧光光谱分析 选择稳态激发波长为375 nm,发射波长为425 nm测定pBR322质粒的时间分辨荧 光光谱和稳态光谱。室温下,在固定波长为375 nm的激发光作用下,检测样品发射荧光强度在 390 nm和570 nm波长之间的分布情况。并检测在波长为280 nm和380 nm之间的激发光作用下,样品在发射波长为 425 nm处的荧光强度。时间分辨激发光谱用具有激发和发射单色器的K2多频互相关相位调制荧光仪,采用相位调制方式收集。室温下,使用含二甲基POPOP激光燃料的无水乙醇溶液(其荧光寿命在无水乙醇为1.45 ns)作为参考。氙灯为样品激发源。采用单指数拟合,利用ISS187衰减分析软件对相位延迟和调制延迟进行分析。
2 结果
2.1 野生型pBR322和扩增后的pBR322的电泳图的比较
pBR322质粒经PCR扩增后,由1%琼脂糖凝胶电泳结果(图1)可以看出,野生型pBR322质粒与扩增后的pBR322质粒图谱相似,说明 pBR322质粒成功被扩增出来。
2.2 细胞扩增pBR322的电泳图
将突变产物转入XL10-gold超级感受态细胞,质粒提取和纯化后1%琼脂糖凝胶电泳结果(图2)表明,成功获得突变的pBR322质粒。
2.3 双突变型pBR322质粒的酶切鉴定
双突变的质粒因为被酶切了两次,一条含有113个碱基的DNA片段被释放出来,因此,泳道2所显示的DNA的泳动速度比泳道4的DNA稍快(图3)。
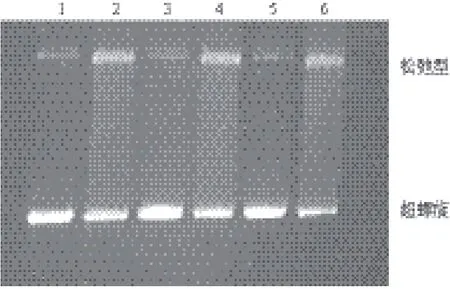
图1 PCR产物凝胶电泳
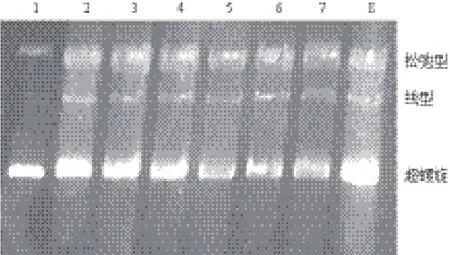
图2 纯化产物凝胶电泳

图3 酶切鉴定凝胶电泳
2.4 连接产物的琼脂糖凝胶电泳图
用溴化乙锭处理后的共价闭合双链DNA环,极易形成正超螺旋结构,导致连接后的超螺旋产物比对照pBR322的泳动速度快(图4)。从泳道7可以看出,当温度升高时,连接产物的产量增多,所以减少了酶切质粒。

图4 不同时间连接产物凝胶电泳
2.5 纯化后的PdC-pBR322连接产物电泳图
超螺旋和松弛型PdC-pBR3222的含量足够用于稳态和时间分辨荧光光谱的测量(图5)。
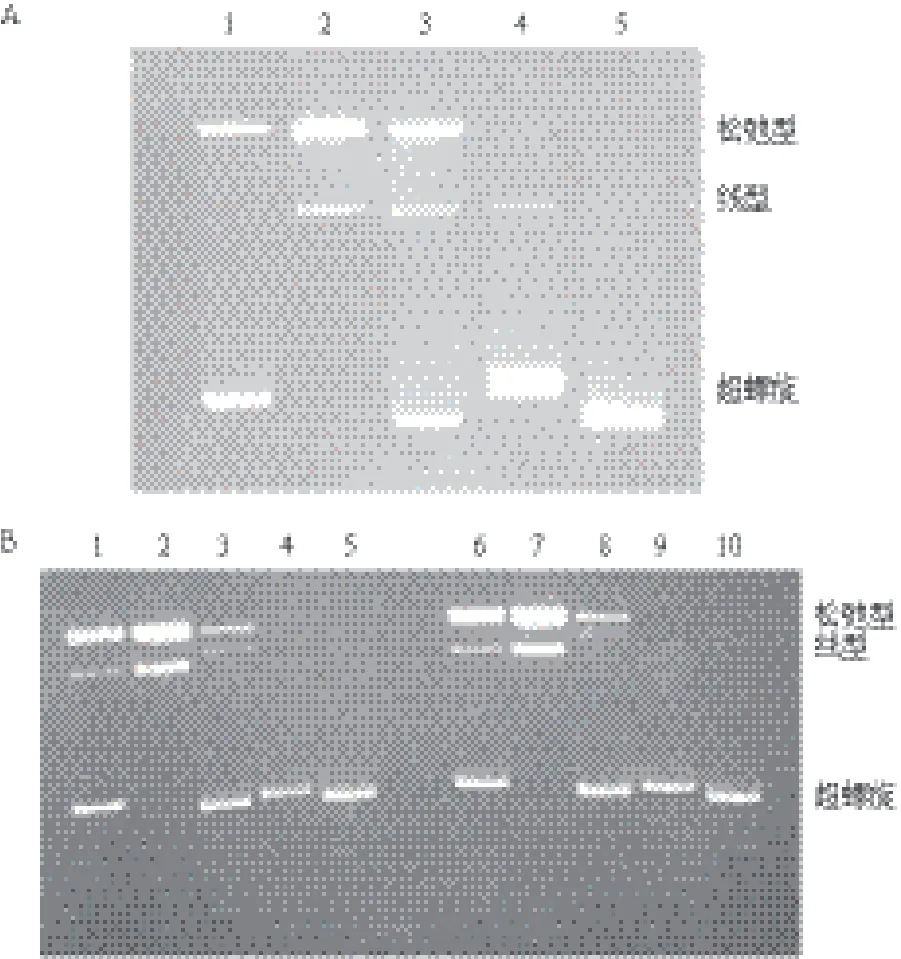
图5 连接产物纯化凝胶电泳
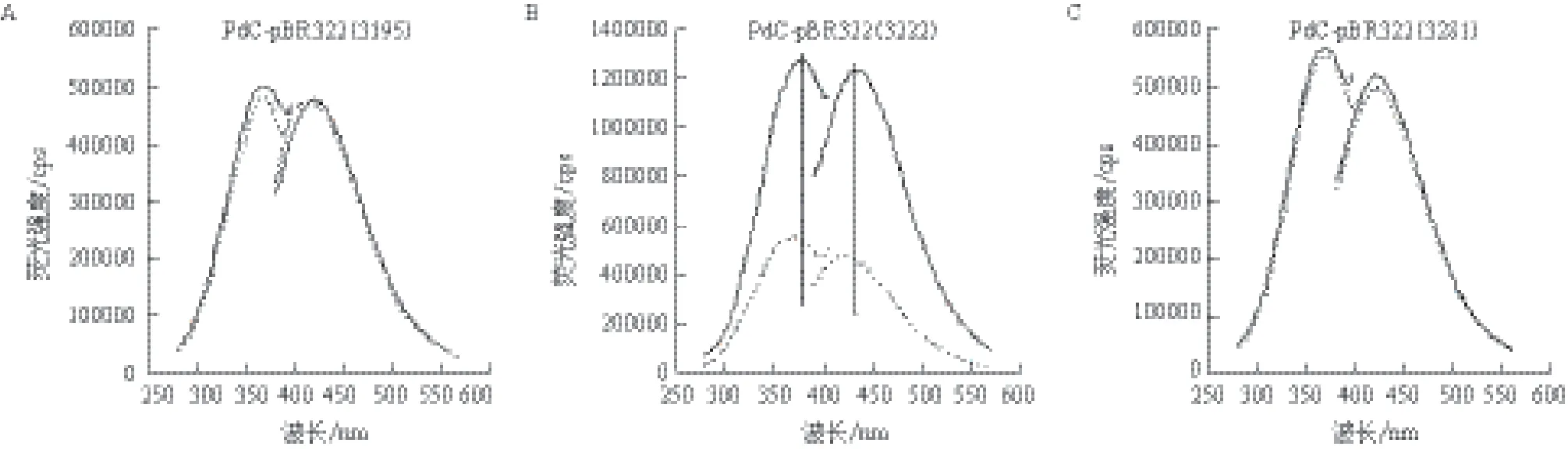
图6 质粒的稳态荧光图谱分析
2.6 稳态荧光分析
稳态荧光分析结果(图6)显示,PdC-pBR322(3195)和PdC-pBR322(3281)的激发和发射光谱没有显著变化。松弛型和超螺旋PdC-pBR322光谱几乎相同。然而,能够观察到松弛型的PdC-pBR322(3222)的激发和发射光谱淬灭,这表明超螺旋PdC-pBR322(3222)中PdC没有配对,十字形环状部分含有PdC。
2.7 时间分辨荧光寿命
荧光寿命检测结果(表2)表明,荧光寿命长短相当接近,除了超螺旋PdC-pBR322(3222)的荧光寿命1.72 ns比松弛型PdC-pBR322(3222)的荧光寿命1.42 ns大约延长了0.3 ns。结果表明,超螺旋PdC-pBR322(3222)中PdC没有配对,这可能意味着在这个位置形成了一个环状结构。
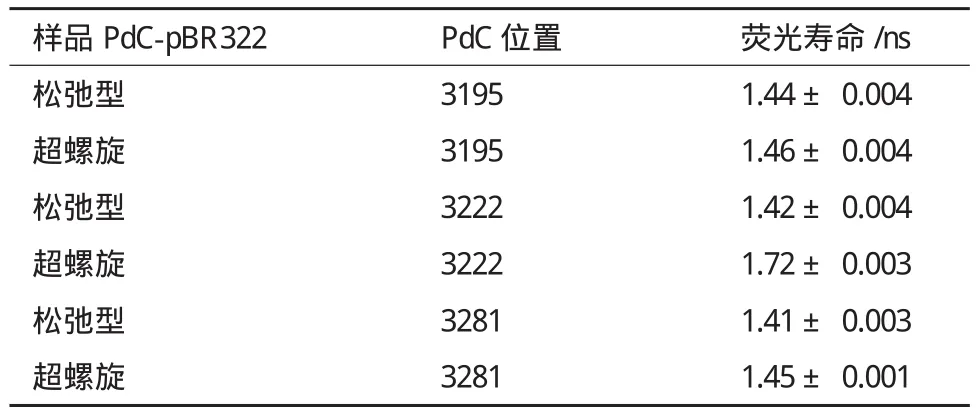
表2 质粒荧光寿命检测结果
2.8 盐浓度对荧光寿命的影响
应用荧光寿命探讨了溶液中离子强度对PdC-pBR322的作用。结果(图7)显示,随着盐浓度的增加,荧光寿命有变化,然而,当盐浓度低于20 mmol/L时,寿命变化相对较大,但仍不显著,表明十字形结构在该实验研究的盐浓度范围内处于稳定状态。

图7 不同盐浓度下的荧光寿命变化
3 讨论
十字形结构DNA与复制,基因表达调控,核小体结构重组等生物过程有关,也与癌症、沃纳氏综合征等疾病的演变与发展有密切联系[26]。很多蛋白质与十字形结构DNA的识别与结合有关。在转录过程中高迁移率族蛋白家族(HMGB)、乳腺癌1号蛋白(BRCA1)等优先与十字形结构DNA结合[27,28]。研究表明,P53在体内更易于结合错配的双链、十字形结构等非典型DNA[29,30]。14-3-3蛋白能够在DNA复制的S期通过与复制起始暂时形成的十字形结合参与真核生物的DNA复制过程[31]。因此,十字形结构DNA的形成过程以及其发挥的生物学作用有待深入研究。
在本实验中我们探讨了如何用PdC取代特定位点的dC掺入pBR322质粒,并用稳态和时间分辨荧光光谱法来探测十字形结构DNA的形成。该实验方法与酶解消化法相比,pBR322质粒不用被酶解成不同DNA片段,因此适用于研究某些完整质粒特性的实验,如超螺旋结构条件下蛋白质和DNA的结合。尽管在连接步骤中的产率很低,但PdC的荧光非常敏感,浓度为1 ng/mL足够应用于荧光光谱测量。
本实验由3种不同位点引入PdC的光谱分析结果分析推测,当DNA片段中只有PdC位点不与G配对时,只能够形成“泡”的形式。唯有PdC位点不与G形成配对,且该DNA具有反向重复序列,其单链DNA在被PdC取代位点相邻的两侧能够互补配对形成一种二元对称结构时,才能够形成十字形结构(图8)。

图8 PdC-pBR322(3222)质粒中形成的十字形结构DNA
该实验中光谱分析检测了十字形结构的形成,除盐浓度对十字形结构的形成影响不明显,其余均与先前文献报道的一致[32]。我们在盐浓度为100 mmol/L NaCl中发现不配对的PdC,而酶解消化法分析在20 mmol/L 以上的NaCl中检测不到这种十字环结构DNA。光谱数据清楚地表明,即使在高盐浓度下仍然存在十字形结构DNA,因此我们推测更高的盐浓度可能影响了酶的功能作用。
4 结论
用PdC取代质粒中指定位置的dC而没用改变质粒的结构,并利用配对的PdC的荧光寿命比非配对的短的理论,用时间分辨荧光光谱分别测量了3 195、3 222和3 281位置的荧光寿命,得到3 222处的PdC未配对。根据3 222处的碱基序列特点(3 222处的碱基序列具有回文结构),推断出质粒在该处具有十字形结构。
[1] 高翼之. DNA双螺旋模型的建立——基因的物质本性[J]. 遗传, 2002, 24(6):691-694.
[2]Qiu J, Liu J, Chen S, et al. Role of hairpin-quadruplex dna secondary structural conversion in the promoter of hnRNP K in gene transcriptional regulation[J]. Org Lett, 2015, 17(18):4584-4587.
[3]Bevilacqua PC, Blose JM. Structures, kinetics, thermodynamics, and biological functions of RNA hairpins[J]. Annu Rev Phys Chem, 2008, 59:79-103.
[4]Chen J, Poddar NK, Tauzin LJ, et al. Single-molecule FRET studies of HIV TAR-DNA hairpin unfolding dynamics[J]. J Phys Chem B, 2014, 118(42):12130-12139.
[5]Coufal J, Jagelská EB, Liao JC, et al. Preferential binding of p53 tumor suppressor to p21 promoter sites that contain inverted repeats capable of forming cruciform structure[J]. Biochem Biophys Res Commun, 2013, 441(1):83-88.
[6]Li D, Lv B, Zhang H, et al. Positive supercoiling affiliated with nucleo some formation repairs non-B DNA structures[J]. Chem Commun(Camb), 2014, 50(73):10641-10644.
[7]赵宏宇, 蔡禄, 赵秀娟, 等. 化学药物对与人遗传病相关的DNA重复序列不稳定性的影响[J]. 生物技术通报, 2010(11):153-156, 161.
[8]García-López A, Llamusí B, Orzáez M, et al. In vivo discovery of a peptide that prevents CUG-RNA hairpin formation and reverses RNA toxicity in myotonic dystrophy models[J ]. Proc Natl Acad Sci USA, 2011, 108(29):11866-11871.
[9]Pushechnikov A, Lee MM, Childs-Disney JL, et al. Rational design of ligands targeting triplet repeating transcripts that cause RNAdominant disease:application to myotonic muscular dystrophy type 1 and spinocerebellar ataxia type 3[J]. J Am Chem Soc, 2009, 131(28):9767-9779.
[10]Wadkins RM, Vladu B, Tung CS, et al. Actinomycin D binds to metastable hairpins in single-stranded DNA[J]. Biochemistry, 199 8, 37(34):11915-11923.
[11]Wadkins RM, Tung CS, et al. The role of the loop in binding of an actinomycin D analog to hairpins formed by single-stranded DNA[J]. Arch Biochem Biophys, 2000, 384(1):199-203.
[12]Jagelská EB, Pivonková H, Fojta M, et al. The potential of the cruciform structure formation as an important factor influencing p53 sequence-specific binding to natural DNA targets[J]. Biochem Biophys Res Commun, 2010, 391(3):1409-1414.
[13]Shlyakhtenko LS, Hsieh P, et al. A cruciform structural transition provides a molecular switch for chromosome structure and dynamics[J]. J Mol Biol, 2000, 296(5):1169-1173.
[14]Brázda V, Laister RC, Jagelská EB, et al. Cruciform structures are a common DNA feature important for regulating biological processes[J]. BMC Mol Biol, 2011, 12:33.
[15]Yahyaoui W, Callejo M, Price GB, et al. Deletion of the cruciform binding domain in CBP/14-3-3 displays reduced origin binding and initiation of DNA replication in budding yeast[J]. BMC Mol Biol, 2007, 8:27.
[16]Zannis-Hadjopoulos M, Yahyaoui W, Callejo M, et al. 14-3-3 cruciform-binding proteins as regulators of eukaryotic DNA replication[J]. Trends B iochem Sci, 2008, 33(1):44-50.
[17]Hilton S, Winstanley D. Identification and functional analysis of the origins of DNA replication in the Cydia pomonella granulovirus genome[J]. J Gen Virol, 2007, 88(Pt 5):1496-1504.
[18]Kim EL, Peng H, Esparza FM, et al. Cruciform-extruding regulatory element controls cell-specific activity of the tyrosine hydroxylase gene promoter[J]. Nucleic Acids Res, 1998, 26(7):1793-1800.
[19]Horwitz MS. Transcription regulation in vitro by an E. coli promoter containing a DNA cruciform in the ‘-35’ region[J]. Nucleic Acids Res, 1989, 17(14):5537-5545.
[20]Hanke JH, Hambor JE, Kavathas P, et al. Repetitive Alu elements form a cruciform structure that regulates the function of the human CD8 alpha T cell-specific enhancer[J]. J Mol Biol, 1995, 246(1):63-73.
[21]Caffarelli E, Leoni L, Sampaolese B, et al. Persistence of cruciform structure and preferential location of nucleosomes on some regions of pBR322 and ColE 1 DNAs[J]. Eur J Biochem, 1986, 156(2):335-342.
[22]Kato M, Hokabe S, Itakura S, et al. Interarm interaction of DNA cruciform forming at a short inverted repeat sequence[ J]. Biophys J, 2003, 85(1):402-408.
[23]张来斌, 任廷琦. 扩环荧光碱基类似物x-腺嘌呤分子基态和激发态性质的理论研究[J]. 物理学报, 2013, 10:353-359.
[24]Bhavsar YP, Reilly SM, Wadkins RM. Evaluation of fluorescent analogs of deoxycytidine for monitoring DNA transitions from duplex to functional structures[J]. J Nucleic Acids, 2011, 2011:986820.
[25]Tinsley RA, Walter NG. Pyrrolo-C as a fluorescent probe for monitoring RNA secondary structure fo rmation[J]. RNA, 2006, 12(3):522-529.
[26]Brázda V, Laister RC, Jagelská EB, et al. Cruciform structures are a common DNA feature important for regulating biological processes[J]. BMC Mol Biol, 2011, 12:33.
[27] Stefanovsky VY, Moss T. The cruciform DNA mobility shift assay:A tool to study proteins that recognize bent DNA[J]. Methods Mol Biol, 2015, 1334:195-203.
[28]Brázda V, Jagelská EB, Liao JC, et al. The central region of BRCA1 binds preferentially to supercoiled DNA[J]. J Biomol Struct Dyn, 2009, 27(1):97-104.
[29]Degtyareva N, Subramanian D, Griffith JD. Analysis of the binding of p53 to DNAs containing mismatched and bulged bases[J]. J Biol Chem, 2001, 276(12):8778-8784.
[30]Jagelska EB, Pivonková H, Fojta M, et al. The potential of the cruciform structure formation as an important factor influencing p53 sequence-specific binding to natural DNA targets[J]. Biochem Biophys Res Commun, 2010, 391(3):1409-1414.
[31]Zannis-Hadjopoulos M, Sibani S, Price GB. Eucaryotic replication origin binding proteins[J]. Front Biosci, 2004, 9:2133-2143.
[32]Kowalski D, Natale DA, Eddy MJ, et al. Stable DNA unwinding, not “breathing, ” accounts for single-strand-specific nuclease hypersensitivity of specific A+T-rich sequences[J]. Proc Natl Acad Sci U S A, 1988, 85(24):9464-9468.
(责任编辑 李楠)
Steady-state and Time-resolved Fluorescence Spectroscopy to Detect the Cruciform Structure in pBR322
XIA Lan1,2ZHANG Xu2,3
(1. College of Biotechnology and Pharmaceutical Engineering,Nanjing Tech University,Nanjing 211816;2. School of Pharmacy,Yancheng Teachers University,Yancheng 224051;3. Medical School of Nanjing University,Nanjing 210093)
The specific aim of this work is to probe the formation process of DNA with cruciform extrusion in a pBR322 plasmid using spectroscopic methods while without digesting DNA into pieces. Pyrrolo deoxycitidine(PdC)was incorporated into pBR322 to substitute for dC at specific sites of 3 195,3 222 or 3 281. Steady-state and time-resolved fluorescence were employed to examine the specific properties of PdC in the cruciform region. Results were as followings:1)Steady-state fluorescent properties indicated that the fluorescence intensity of supercoiled PdC-pBR322 was about 4-fold stronger than that of relaxed PdC-pBR322,when PdC was incorporated into pBR322 at position 3 222;meanwhile,its time-resolved lifetime was about 0.3 ns longer than that of relaxed PdC-pBR3322. When PdC was incorporated at position 3 195 or 3 281,there was no significant change in either steady-state fluorescence spectra or time-resolved lifetime(about 1.42 ns at both positions). 2)Lifetimes changed a little with the increasing of salt concentration,but not significantly. In conclusion,by analyzing fluorescence spectra and lifetimes(both in relaxed state and supercoiled plasmid),a cruciform structure with dC at site 3 222 of impaired loop is formed,and the cruciform is stable within this investigated salt concentration range(0-100 mmol/L).
cruciform extrusion;pBR322 plasmid;PdC;steady-state and time-resolved fluorescence spectroscopy
10.13560/j.cnki.biotech.bull.1985.2016.04.028
2016-02-25
江苏省六大人才高峰资助项目(swyy-030)
夏岚,女,硕士研究生,研究方向:抗瘤大分子的活性研究;E-mail:XLxialan@163.com
张旭,男,副教授,研究方向:抗瘤大分子的活性研究;E-mail:zhangxthunder@sina.com

