适配体在食源性致病菌检测中的应用进展
解沛燕朱龙佼许文涛,2
(1.中国农业大学食品科学与营养工程学院,北京 100083;2.中国农业大学食品科学与营养工程学院 食品安全检测与风险评估实验室,北京 100083)
适配体在食源性致病菌检测中的应用进展
解沛燕1朱龙佼1许文涛1,2
(1.中国农业大学食品科学与营养工程学院,北京 100083;2.中国农业大学食品科学与营养工程学院 食品安全检测与风险评估实验室,北京 100083)
适配体建立在仿生学基础上,是在体外从单链核酸随机序列库中筛选出的对某种靶物质具有高亲和度的寡聚核苷酸(DNA或RNA)片段。相较于食源性致病菌的常规检测手段,适配体具备特异性强、稳定性好及易于修饰等优点,因此在该领域得到了关注和应用。但前人对适配体与靶物质结合的原理认识尚不够深入,对食源性致病菌的适配体筛选及应用总结还存在许多不足。结合食源性致病菌的自身结构特点,介绍了该类物质的适配体筛选特点和检测领域中的最新研究进展,提出了目前存在的问题及发展趋势。
适配体;食源性致病菌;传感器;检测
食源性疾病是全球食品安全问题之首[1]。自1993年至今,食源性疾病的发病率呈现上升态势,每年约数10亿例发生,每天有数百万人感染,仅食物引起的腹泻性疾病每年就导致220万人死亡,WHO将控制食品污染和食源性疾病列为优先重点战略工作领域[2]。一向以食品安全监管严格著称的美国每年发生的食源性疾病达7 600万人次,住院32.5万人次,约5 000人死亡,年均发生食品安全事件350起,经济损失达1 520亿美元[3]。其中,由食源性致病菌导致的食源性疾病占44%。
食源性致病菌是指能够污染食品并通过饮食传播,引起人类疾病的病原菌,通常指的是细菌。它们在食品中生长代谢会引起食品的腐败和变质,有些病原菌会产生特定的有毒物质,直接或间接地导致人患病。常见的细菌类食源性致病菌包括金黄色葡萄球菌、沙门氏菌属、志贺氏菌、致病性大肠杆菌(特别是出血性大肠杆菌O157∶H7)、致病性弧菌(包括:霍乱弧菌、副溶血性弧菌)等[4]。
在全球,每年食源性致病菌(如沙门氏菌、李斯特菌、大肠杆菌O157和弧菌等)的检测量约2.2 4亿个,花费约14亿美元[3]。由卫生部、工业和信息化部、商务部、工商总局、质检总局、粮食局和食品药品监管局联合制订的《2015年国家食品安全风险监测计划》中,对食源性疾病监测以及致病菌的检测制定了全面而详细的监控计划[5]。目前,我国检测食源性致病菌大多采用常规检测方法,存在耗时长、检测范围小等问题,而适配体在该领域的运用弥补了传统手段存在的诸多不足。
1 食源性致病菌的常规检测方法
目前,食源性致病菌的常规检测方法主要有平板分离法、化学分析法、分子生物学方法和免疫学检测法等。
1.1 平板分离法
平板分离法是根据 食源性致病菌的生理生长特性存在差异对其进行鉴定的方法,通常包括増菌、分离、生化分析及血清学鉴定等步骤。该方法原理易懂、操作简便且检测结果稳定性好,我国将其定为食源性致病菌的常规检测手段。然而,平板分离法的前期増菌和分离步骤通常需要1-2 d,且后续生化检测繁琐,整个周期要3-5 d,耗时较长,无法满足对食源性致病菌快速检测的需求。
1.2 化学分析法
化学分析法是根据不同的食源性致病菌在自身组分及代谢产物上存在差异,往往有属于该类菌的标志性化学组成,利用气相色谱或高效液相色谱分析样品中所包含的化学成分,达到检测食源性致病菌的目的。该方法操作灵敏度高,结果可靠,但对样品的前处理要求较为苛刻,并且需要使用昂贵的大型仪器设备,不适合应用于现场的检测。
1.3 分子生物学方法
分子生物学方法是利用不同的食源性致病菌在核酸层面上存在的差异来进行检测分析。电泳技术、分子杂交技术、PCR技术以及DNA序列测序分析技术等搭配使用,可以对样品中食源性致病菌进行定性定量分析。其中,应用最广泛的是PCR技术。该技术检测食源性致病菌的优势在于检测时间短,检测程序简单。但不同种类的PCR技术自身也存在一定的缺陷,例如,普通PCR仅能用于单一致病菌的检测,而通过扩增食源性致病菌中的信使 RNA(mRNA)来检测分析逆转录 PCR只能够检测活细菌细胞。
1.4 免疫学检测法
免疫学检测法是以全菌或菌的某一部分,如菌毛蛋白、脂多糖及细菌毒素等为抗原,利用相应的抗体与其特异性结合,实现对食源性致病菌的检测。该类方法主要以抗体为识别元件,通过酶催化底物反应产生颜色变化,进行定性、定量分析。此法具有良好的特异性和稳定性,是目前我国基层检测单位应用最多的快检方法。由于抗体属于蛋白类物质,通常在动物体内筛选,制备成本较高,并且检测及储存对环境的要求较高,导致使用酶联免疫吸附反应检测食源性致病菌时存在一定的局限性。
适配体的出现,弥补了以往方法检测食源性致病菌时的不足。与平板培养法相比,适配体在检测时能从复杂环境中找到靶物质,检测周期短;与仪器分析法相比,样品的前处理简便;与抗体相比,适配体在体外筛选获得,制备周期短且成本较低。
2 适配体简介
2.1 适配体的起源
1990年,Tuerk 和 Gold 和 Ellington 和 Szostak发现了一类能与蛋白的单链核酸序列,两者的结合特异性及亲和度极高,他们将这类核酸序列命名为适配体(Aptamer)——由拉丁文“aptus”(意为“合适”)及德文“meros”(意为部分、分离)两者结合衍生而来[6]。在此之后,涌现出大量针对不同靶物质的适配体研究,人们通过适配体与其他材料的搭建,发明出各种生物传感器,应用于药物生产、医学检测等领域。
2.2 适配体的特点
适配体是能特异性识别某种靶物质且具有高度亲和力的核酸序列(DNA或RNA),通常含有15-40个碱基,其分子量大致为5-25 kD[6]。追根溯源,适配体是在仿生学的基础上发展应用的,如RNA适配体,其实质是对非编码RNA的一种模仿。与抗体等检测元件相比,适配体具有以下4个显著的特点:(1)筛选周期短 筛选适配体不需要进行动物实验,在体外即可获得目标序列。最常见的适配体筛选技术——指数富集的配基系统进化技术(systematic evolution of ligands by exponential enrichment,SELEX)通常在8-15轮的筛选后得到与靶物质结合亲和性和特异性良好的适配体序列,整个周期在1-2个月左右。而最近诞生的Non-SELEX技术筛选周期更短,与毛细管电泳仪共同使用时可将筛选周期控制在1 d左右。(2)高亲和性和高特异性 适配体是在容量约为1013-1015的核酸文库中进行5-13轮的阳性筛选,最终得到的适配体与靶物质共同孵育时,其解离常数可达 nmol 甚至 pmol 水平。由此可见,适配体与靶物质的结合能力极强。同时,适配体也具有高特异性,以食源性致病菌为例,研究者通常会选择菌上某个特异性组分,如标志性蛋白、致病毒素等作为筛选靶物质,从根本上保证了筛选出的适配体具有特异性,当靶物质是全菌时,研究者会在阳性筛选结束后选择3-5种与该菌结构或致病性相似的菌种进行阴性筛选,使筛选出的适配体具有极高的特异性。(3)适用范围广 由于适配体包含15-40个随机碱基序列,用于筛选适配体的文库容量极大,因此几乎所有的靶物质都能从文库中找到能与其特异性结合的适配体序列。随着研究者的不断探索,适配体已经应用于食品中危害因子检测、医学药物研发、疾病诊断和治疗等众多研究领域。以检测食品中的危害因子为例,适配体在重金属[7-9]、农药残留[10-12]、兽药残留[13,14]以及食源性致病菌[15-28]等的检测中都有突出作用。(4)对检测环境要求低作为核酸类物质,适配体能耐高温、耐酸碱性,并且易于储存。与常规检测方法相比,适配体在使用时对检测环境要求低。再者,适配体具有标记稳定性,可在序列末端标记巯基、生物素等基团,因而广泛用于各类生物传感器的搭建。
3 适配体的筛选
3.1 筛选技术
目前,用于适配体的筛选技术有很多,如亲和层析SELEX、毛细管电泳SELEX(Capillary electrophoresis SELEX)、均衡混合物的非均衡毛细管电泳SELEX(Non-equilibrium capillary electrophoresis of equilibrium mixtures SELEX)、全细胞SELEX(whole cell-SELEX)等方法。根据筛选过程中是否有序列扩增富集,可以将现有的筛选技术分为SELEX筛选和Non-SELEX筛选两大类。
3.1.1 SELEX筛选 大多数的适配体筛选技术属于指数富集的配基系统进化技术(s ystematic evolution of ligands by exponential enrichment,SELEX),主要包括(图1):建立文库、与靶物质孵育、洗脱分离、扩增富集等步骤[29]。
适配体的筛选文库一般由含有15-40个随机碱基的序列组成,容量约为1013-1015,两端为固定序列,便于PCR扩增。在适配体文库与靶物质孵育阶段,研究者会根据靶物质的特点选用合适的结合缓冲液,这也是筛选过程中需要优化的条件之一。
分子量不同的靶物质,其洗脱分离步骤操作差异较大。其中,大分子物质的常用分离方法有:(1)硝酸纤维素膜印迹法[30-35]:可将靶物质固定在硝酸纤维素膜上,随后通过分子印迹分析得到能与靶物质结合的随机单链。“适配体”概念的提出者、SELEX方法的创始者Tuerk等[30]最早应用硝酸纤维素膜筛选得到噬菌体 T4 DNA 聚合酶的适配体,使得该方法在适配体筛选,特别是蛋白类靶物质的筛选中得到广泛应用。Savory[32]在筛选小树肝脏上某种蛋白的适配体时,借助硝酸纤维素膜完成适配体的洗脱分离步骤,该研究建立了复杂机制中靶物质的适配体筛选模型。(2)离心沉淀法[36,37]:能结合上靶物质的随机单链可随靶物质一同被离心沉淀下来,该方法具有耗时短、操作简单等优点,通常用于大分子靶物质,如全菌,适配体的筛选。Hari等[36]在筛选鼠伤寒沙门氏菌适配体时,以全菌作为靶物质,与核酸序列文库室温下共同孵育45 min后,将混合物在1 500×g下离心10 min,去除未与靶物质结合的DNA单链。经过8轮阳性筛选及2轮阴性筛选后,研究者挑选出1条能与鼠伤寒沙门氏菌特异性结合的适配体单链。(3)微孔板筛选技术[38-40]:将靶物质加入微孔板内,经过一段时间孵育后加入核酸文库序列,能与靶物质结合的随机单链可留在微孔板上。该方法利用聚苯乙烯材料特有的吸附作用固定靶物质,稳定性较好,多用于以菌的表面大分子为靶物质的适配体筛选中。Han等[39]筛选金黄色葡萄球菌适配体时,以菌表面的磷壁酸为靶物质,将其包被在96孔板上,加入RNA序列文库后室温下孵育20 min,用缓冲液冲洗后,能与靶物质特异性结合的RNA单链留在微孔板中,最后分离出的适配体单链也与金黄色葡萄球菌结合。
小分子物质的分离方法:(1)磁珠分离法[41-43]:小分子靶物质可以固定在磁珠上,与适配体序列文库共同孵育后在外加磁场的作用下可以实现分离,而后通过加热或加碱等方法使适配体序列与靶物质分开。Mann等[41]首次以分子量很小的乙醇胺(Mr = 61.08)为靶物质,将其固定在磁珠上。加入核酸序列文库后振荡孵育30 min,利用磁性材料的分离富集效应去除未与乙醇胺结合的核酸序列,而后加入含有尿素及EDTA的缓冲液提取结合在靶物质上的核酸序列,进入下一步扩增富集,最终筛选出6条性能良好的适配体。(2)柱层 析分离法[44-49]:将靶物质结合在树脂层析柱上,适配体序列文库与靶物质孵育后,能特异性结合的序列可以留在层析柱上,达到分离的目的。Cruz-Aguado等[44]将靶物质伏马毒素B1固定在凝胶树脂亲和层析柱上,注入含有核酸序列文库的溶液,用结合缓冲液反复冲洗层析柱,成功分离出与伏马毒素B1结合解离常数为100 ± 30 nmol/L的适配体序列。
3.1.2 Non-SELEX筛选 Non-S EL E X 筛 选 技 术 是 相较 于 S E LEX筛选技术提出的。传统的SELEX技术在经过洗脱分离步骤后,要使用PCR等技术富集能与靶物质结合的核酸序列,扩增出的产物将进入下一轮阳性筛选过程,从而提高适配体序列在核酸序列文库中所占有的比例,最终筛选出解离常数低的适配体序列。与SELEX不同的是,Non-SELEX无需进行PCR等核酸序列扩增步骤,经过2-3次分离、分析筛选步骤后直接得到适配体序列(图2)。
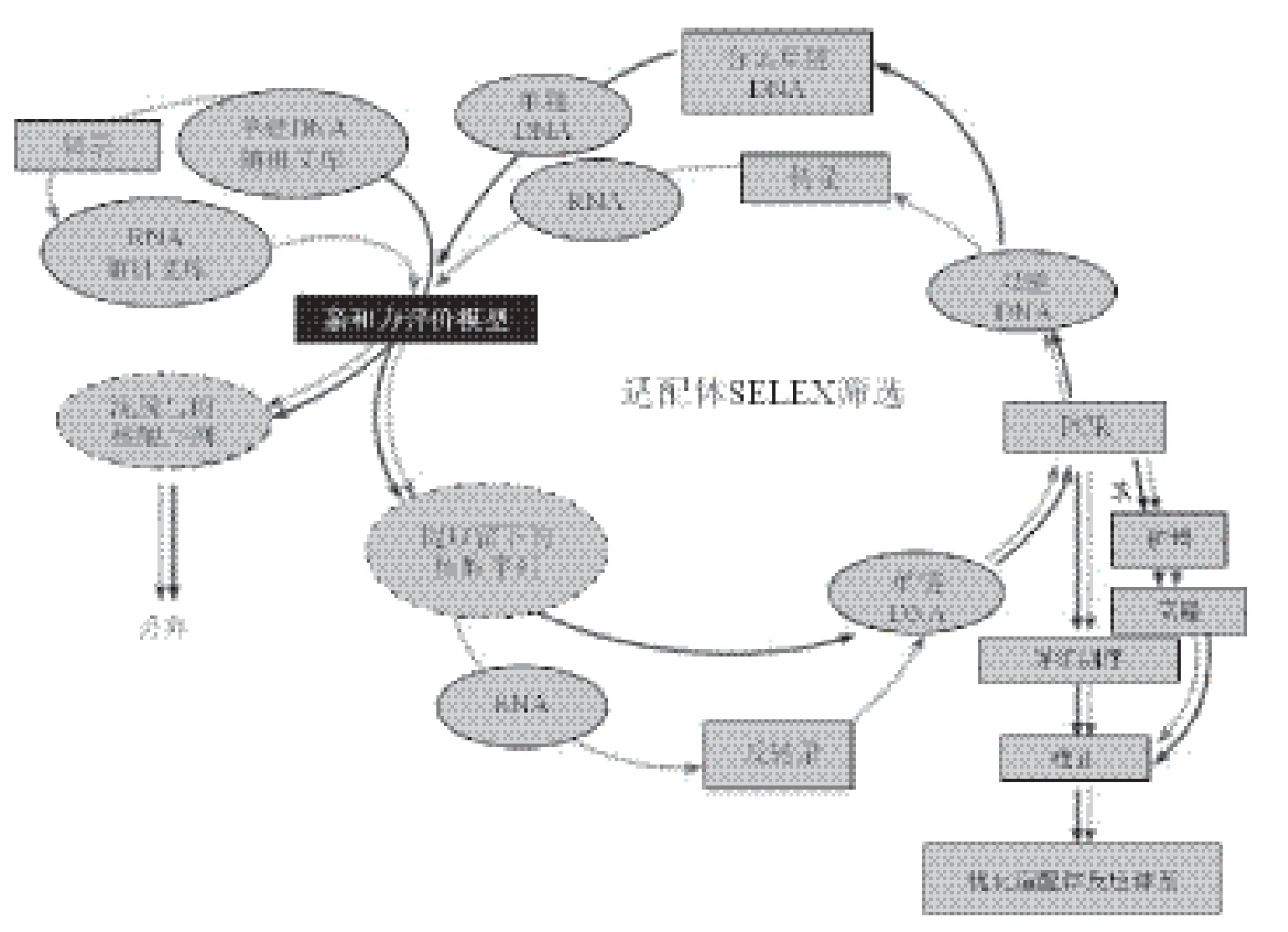
图1 SELEX技术筛选适配体流程图
Berezovski等[50]首次提出Non-SELEX技术的概念,并使用该技术筛选h-RAS蛋白适配体。他们将平衡混合物的非平衡毛细管电泳(non-equilibrium capillary electrophoresis of equilibrium mixtures,NECCM)引入适配体的筛选中,以h-RAS蛋白和适配体文库的平衡混合物为样品,利用h-RAS蛋白和核酸序列两者结合后出现特异性峰形,进而针对这种非线性拟合对反应的解离速率常数进行求解,经过3轮分离、分析后得到能特异性结合h-RAS蛋白的核酸序列。
随后,Non-SELEX技术在适配体筛选中得到了发展和应用[51-54]。Ashley等[51]利用非平衡毛细管电泳,以溶菌酶,胰蛋白酶原,糜蛋白酶原A和肌红蛋白为靶标进行阴性筛选,经过3轮Non-SELEX过程出了能特异性结合牛过氧化氢酶的适配体。Yu等[52]运用数学模型分析了在不同条件,如蛋白浓度、分离效率不同对Non-SELEX筛选过程的影响,为后续研究者的实验设计提供了参考依据。
与筛选周期通常为1-3个月的SELEX筛选技术相比,Non-SELEX筛选技术可在几天甚至几小时内完成适配体筛选全过程,大大缩短了筛选周期。但由于后者需借助毛细管电泳仪进行筛选,筛选靶物质结合核酸序列前后应在电泳迁移率方面有明显变化,因此具有一定的局限性,即Non-SELEX技术大多用于大分子物质的适配体筛选。

图2 SELEX技术与Non-SELEX技术筛选适配体流程对比示意图
3.2 筛选靶物质
食源性致病菌的筛选靶物质可以分为3大类:全细胞、表面组分以及毒素。参照我国《食品安全国家标准——食品中致病菌限量》(GB 29921-2013),本文介绍了国标中规定的食源性致病菌的致病特点,并总结了目前国内外文献中筛选出的食源性致病菌适配体序列(表1)。
以全细胞作为筛选靶物质的研究大多集中在适配体应用于食源性致病菌检测领域的起步阶段,但随着研究的不断深入,致病菌上特异性组分和毒素成为了选择的热门。相较于用整个致病菌为靶物质,后者具有一定的优势:(1)可不做或少做阴性筛选,实验周期得到缩短;(2)以整个细菌作为筛选靶物质会污染操作环境的可能性较大,会带来一定的安全隐患,选择某个组分为靶物质则大大降低了这种风险;(3)以致病菌上的某个特异性部分为靶物质筛选出的适配体可用于整个致病菌的检测,且清楚地了解与致病菌的结合位点,对基于适配体的生物传感器的搭建十 分有利。
3.2.1 全细胞 作为大分子物质,全细胞的表面复杂且结构同源性较高,因此在完成阳性筛选后,通常会选择3-6种与靶致病菌具有相似结构或能导致相似疾病的细菌进行阴性筛选,确保筛选出的适配体能够特异性地识别靶致病菌。Cao等[26]在筛选金黄色葡萄球菌适配体时,将经过11轮阳性筛选后留下的适配体序列与表皮葡萄球菌、嗜热链球菌以及大肠杆菌DH 5共同孵育,反向筛选适配体。
3.2.2 表面组分 筛选适配体的靶物质也可以是致病菌表面的某个特异的功能性部分,如脂多糖、蛋白等。 Li等[17]在筛选肠毒素大肠杆菌K88适配体时,以能吸附宿主肠道黏膜上皮细胞,从而一起宿主肠道疾病的菌毛蛋白为靶物质;Bruno等[18]在筛选大肠杆菌O111∶B4适配体时,以菌膜上具有抗原性的内毒素脂多糖为靶物质。同样地,在筛选原生生物、病毒等的适配体时,大多也会选择使用该类病原微生物上的特异性蛋白作为靶物质,如针对甲、乙、丙型肝炎,已经筛选出以核心蛋白、3C蛋白酶、囊膜糖蛋白E2、NS3螺旋酶为靶标的核酸适配体。
3.2.3 毒素 真菌毒素是真菌在食品或饲料里生长所产生的代谢产物,对人类和动物都有害,而用于适配体筛选的毒素通常指的是真菌毒素,如黄曲霉毒素、赭曲霉毒素及玉米赤霉烯酮等。2008年,Cruz-Aguado和Penner首先通过免疫亲和柱筛选得到了与OTA高亲和识别的DNA适配体,并且成功设计了以OTA适配体为识别模式的荧光偏振检测方法[43],这也是OTA适配体在分析检测上的第一次应用。王文凤等[55]借助磁珠筛选得到了黄曲霉毒素B1和B2的适配体,随后将适配体搭载纳米金颗粒,实现对靶物质的检测。

表1 食源性致病菌适配体汇总表
4 适配体的性能鉴定
研究者通常对筛选出的适配体进行两方面的性能鉴定:适配体与靶物质结合的亲和性以及特异性。
4.1 亲和性
亲和性是指适配体与靶物质结合力的大小,通常用解离常数进行表征。在自然状态下,适配体呈现多种不确定的空间构象,当加入靶物质存在时,适配体会改变自身构象,通过范德华力、氢键、盐桥、静电吸附等作用力嵌入在靶物质表面的结合位点[4],实现两者结合,其解离常数可达nmol 甚至pmol 水平。由此可见,适配体与靶物质的亲和性很高。
4.2 特异性
特异性是适配体与靶物质及非靶物质比较,结合力的强弱是否存在明显差异。就食源性致病菌而言,通常使用与靶物质有相似结构和相同致病性的病原菌或病原菌结构(如鞭毛蛋白等)进行特异性分析。解离常数也是评价适配体特异性的指标,当与靶物质孵育时,Kd值极低,而与其他物质孵育时,Kd值较高,两者相差1-2个数量级,则该适配体序列与靶物质结合的特异性较高。
4.3 亲和性与特异性的关系
适配体与靶物质的亲和性与特异性是必要不充分的关系。核酸序列对靶物质的亲和性要求该序列很容易与靶物质结合;而核酸序列的特异性则要求该序列能且仅能与靶物质结合。通常情况下,筛选得到的适配体能从复杂基质中特异地与靶物质结合,则两者之间相互作用力一定很强。反之则不成立,经过多轮阳性筛选得到的适配体通常与靶物质具有较好的亲和性,但由于两者间的结合位点可能不是靶物质特有的组分,阳性筛选出的适配体序列不一定具有与靶物质较好的特异性,这一特点在以全细胞为靶物质的适配体筛选中尤为突出,因此全细胞筛选一般配合阴性筛选。
5 适配体与靶物质的结合表征
5.1 靶物质的结合位点
研究筛选出的适配体与靶物质的结合位点有助于优化检测体系。段诺等[28]在以副溶血性弧菌为靶物质筛选得到适配体后,使用提取试剂盒提取了副溶血性弧菌表面的外膜蛋白和脂多糖两类组分,比对解离常数和琼脂糖凝胶电泳图,分析了该适配体对膜外两种物质的结合情况,最终得出筛选出的适配体与副溶血性弧菌的结合位点在外膜蛋白上的结论。针对不同靶物质的适配体筛选研究有很多,但对筛选出来的适配体与靶物质,尤其是以菌种上某个特异性组分作为靶物质,结合位点研究的较少。出现这种情况的可能的原因有如下两点:(1)适配体序列与靶物质结合时通常会折叠缠绕改变构象,而这一过程可能会受到靶物质上某些物质的作用,机理复杂;(2)靶物质作为一个复杂的大分子物质,其表面能与核酸序列结合的位点较多,探究两者结合的具体作用位点必须将靶物质进行切割分离,难度较大。
5.2 适配体的功能片段
研究者在探究适配体序列上能与靶物质结合的功能片段时,通常会根据适配体序列的结构特点,找到保守序列,即筛选出的核酸序列中共有的片段,再根据茎环、支撑臂等结构将其分解为若干部分,探讨各部分与靶物质的结合情况,从而确定该适配体序列的功能片段。
Lee等[15]在筛选大肠杆菌O157∶H7的RNA适配体时,选取与靶物质结合率最高的适配体序列,将其分解为3个部分,而后用3个序列片段及全序列分别与靶物质进行孵育,用Kd值评价两者结合的亲和性。结果表明,保守序列部分与靶物质结合的亲和性最佳,此结果能很好地反映适配体与靶物质结合的有效片段,后续研究者在使用适配体检测大肠杆菌O157∶H7时可直接合成功能序列,既能提高检测效率,又可以节约成本。
6 适配体检测应用
6.1 菌种的样品前处理
在检测之前,需要对样品中的菌种进行前处理,即选择合适的培养基扩增菌种使其达到对数期,通过离心去除杂质,加入筛选适配体时用到的结合液重悬,使菌种在数量和质量上都处于良好的待检状态[56]。
以菌上某个特异性组分,如膜外蛋白、脂多糖等作为待检物质时,可在菌种扩增完成后使用对应组分的提取试剂盒进行提取[28]。
6.2 适配体光学传感器
基于适配体的光学传感器是利用适配体检测食源性致病菌中应用最广泛的模型,其优势在于光学元件种类繁多且适配体易于修饰,两者结合还可以搭建可视化颜色改变的生物传感器,这对现场检测来说至关重要。常见的有比色传感器[16,56,57]、化学发光传感器[58-61]、荧光传感器[62-66]、表面等离子共振传感器[67-69]以及表面增强拉曼光谱传感器[70-72]。各种传感器模型间是相辅相成的,研究者会同时引入多种材料,如纳米材料与荧光材料并用等,从而提高反应体系的灵敏度,降低检测限。
6.2.1 比色传感器 适配体搭载可发生颜色变化的新型纳米材料,构成比色传感器。新性纳米材料在溶液中处于分散状态,当靶物质出现时,适配体与之结合,从而拉近粒子间的距离产生色差。研究者以颜色变化为信号,对靶物质进行定性定量检测。
Wu 等[16]利用聚二乙炔(polydiacetylene,PDA)纳米球在不同状态下会产生颜色变化来检测大肠杆菌。10,12-二十五碳二炔酸在紫外灯下照射可形成PDA纳米球,经过氨基修饰的大肠杆菌O157: H7脂多糖特异性结合的适配体可以与羧基化的PDA形成肽键,从而将两者结合在一起。PDA纳米球在分散状态下呈现蓝色,修饰完适配体后仍旧保持分散状态,不发生颜色变化。当大肠杆菌O157:H7存在时,适配体会迅速改变自身构象与之结合,从而导致PDA纳米球出现聚集,呈现出红色(图3)。当大肠杆菌O157:H7存在时,该体系可产生肉眼可见的颜色变化,具有较大的优势,比色反应可在2 h内完成,借助紫外可见分光光度计可实现定量分析。但由于反应体系中使用的材料单一,方法的灵敏度较差,其检测区间为104-108CFU/mL。

图3 PDA搭载适配体检测大肠杆菌O157:H7的原理示意图[16]
DNA酶也是常见的搭建比色传感器的元件。DNA酶具有生物催化作用,而血红素与富含G碱基的适配体结合后形成的超分子复合物具有类似过氧化物酶活性,可以催化过H2O2化ABTS的反应,产物APTS+是一种有色物质,可利用其作为传感信号,搭载适配体序列组成比色传感器。
Teller等[57]运用DNA酶比色发光传感器实现了对AMP和溶菌酶的检测。研究者设计了两条DNA杂交链,其中一条为“识别序列”,同时包括靶物质适配体和血红素适配体,另一条为“阻碍序列”,与“识别序列”中的两段适配体序列分别有9个碱基互补。当靶物质存在时,“识别序列”中与其对应的适配体序列会结合上去,从而打开双链,另一端的血红素适配体形成G4结构,催化H2O2化 ABTS的显色反应(图4)。该方法原理简单易懂,操作步骤较少,建立了一种可以推广的DNA酶比色传感器模型。但其方法中需要设计两条 含有茎环结构的DNA杂交链,因此对适配体序列长度的有一定要求。
6.2.2 化学发光传感器 化学发光是物质在进行化学反应过程中伴随的一种光辐射现象。因其具有灵敏度高、检测区间宽及装置简单等优点,化学发光被广泛用于蛋白、毒素等分子的检测。在适配体序列上标记化学发光因子[58,59],或引入DNA酶催化鲁米诺与H2O2反应[60,61],是目前最常见的化学发光传感器体系。
Mun等[61]首次以适配体代替酶联抗体,用竞争法检测黄曲霉毒素B1。研究者在黄曲霉毒素B1适配体的末端连接了类辣根 过氧酶的DNA序列,当待检样品中存在 黄曲霉 毒 素 B1时,竞争性地与适配体结合,固定在 9 6 孔板上的黄曲霉毒素B1-卵蛋白失去DNA序列,在 孔 中加入H2O2和鲁米诺后不再出现化学发光(图 5)。该 方法用于玉米样品中黄曲霉毒素B1的检测,其检测限为0.11 ng/mL。与酶联抗体相比,适配体竞争法大大降低了检测限。
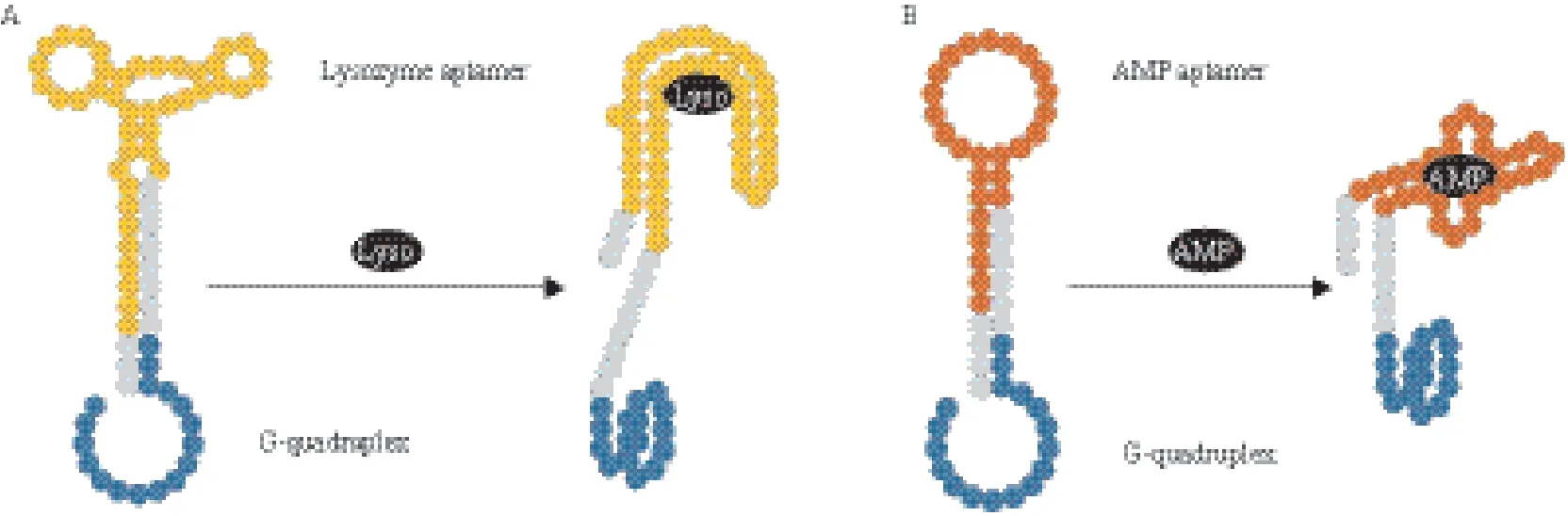
图4 DNA酶比色传感器检测AMP、溶菌酶示意图[57]

图5 DNA酶化学发光传感器检测黄曲霉毒素B1示意图[61]
6.2.3 荧光传感器 荧光适配体生物传感器主要是用荧光基团标记核酸适配体,基于目标分子与适配体作用后产生的荧光偏振或荧光强度的改变来检测食源性致病菌[62-66];或者将荧光基团或淬灭基团分别标记在核酸适配体的两端,加入靶物质后引起适配体自身构象改变,从而产生荧光信号上的差异[57]。
段颖芬等[62]搭建检测鼠伤寒沙门氏菌的荧光传感器时,以氧化石墨烯为平台,将荧光染料标记的鼠伤寒沙门氏菌适配体作为分子识别探针,加入纳米材料氧化石墨烯后,荧光探针会吸附在其表面导致荧光淬灭。当鼠伤寒沙门氏菌存在时,适配体与靶物质结合使荧光信号再度恢复(图6)。根据荧光信号恢复的强弱可以实现对鼠伤寒沙门氏菌的定量检测。该方法的淬灭效率高、背景值低,其检测限为102CFU/mL,在检测食源性致病菌的荧光传感器中属于检测限较低的体系。由此可见,纳米材料的引入大大提高了反应的灵敏性。该方法的淬灭效率高、背景值低,其检测限为102CFU/mL,在检测食源性致病菌的荧光传感器中属于检测限较低的体系,由此可见纳米材料的引入大大提高了反应的灵敏性。

图6 氧化石墨烯搭载荧光标记适配体检测鼠伤寒沙门氏菌的原理示意图[62]
6.2.4 表面等离子共振传感器 表面等离子共振(surface plasmon resonance,SPR)是一种物理现象,当入射光以临界角入射到两种不同折射率的介质界面(比如玻璃表面的金或银镀层)时,可引起金属自由电子的共振,由于共振致使电子吸收了光能量,从而使反射光在一定角度内大大减弱。将适配体修饰在等离子共振装置表面,靶物质的出现会导致等离子共振装置的折射率发生位移。
Tombelli 等[68]使用SPR适配体传感器来检测艾滋病病毒HIV-1Tat蛋白。使用生物素链霉亲和素将适配体固定在芯片表面,流动注射不同浓度的HIV-1Tat蛋白时,检测折射角的信号变化,检出限为0.12 mg/L。Lei等[69]在构建SPR传感器检测沙门氏菌时,在链霉亲和素的适配体末端加入与沙门氏菌侵袭蛋白A基因互补的核酸序列,将其作为信号放大体系引入SPR传感器中(图7),该方法的检测限可达到60 CFU /mL。SPR传感器不需任何标记物,可用于生物分子的无损检测。
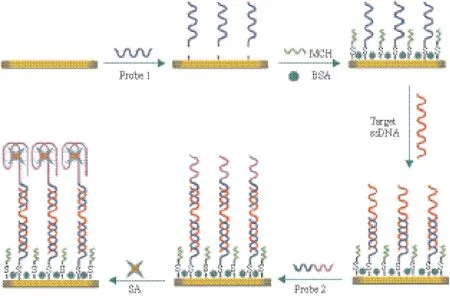
图7 SPR传感器检测鼠伤寒沙门氏菌的原理示意图[69]
6.2.5 表面增强拉曼光谱传感器 拉曼光谱是光子与分子相互碰撞发生方向和能量改变,出现散射光频率变化的联合散射光谱。拉曼散射效应是一个非常弱的过程,研究者将分子吸附在粗糙表面,如金属和纳米粒子,形成表面增强拉曼光谱(surface enhanced raman spectroscopy,SERS),可用于分子水平的检测[70-72]。
Zhang等[70]利用SERS传感器同时检测鼠伤寒沙门氏菌和金黄色葡萄球菌。研究者将特异性识别两种菌的适配体固定在Fe3O4磁性纳米粒子上,形成捕获探针。拉曼分子巯基乙酸(MBA)与鼠伤寒沙门氏菌适配体、拉曼分子5,5'-二硫代二(2-硝基苯甲酸)(DNTB)与金黄色葡萄球菌适配体分别修饰在金纳米粒子表面,形成信号探针(图8)。当反应体系存在两种菌时,捕获探针中的适配体可特异性地与靶物质结合,加入信号探针形成夹心结构。两种拉曼分子可形成不同的表面增强拉曼光谱,实现对细菌的检测和鉴定。该方法对鼠伤寒沙门氏菌的检测限为15 CFU/mL,金黄色葡萄球菌的检测限为30 CFU/mL,具有简单快捷、灵敏度高等优点,引入多种拉曼分子,可实现对多种致病菌的同时检测。
6.3 适配体电化学传感器
基于适配体的电化学传感器起步较晚,但由于优势明显,在近几年得到了迅速发展,常见的有电极修饰传感器[73-75]及纳米管传感器[76-80]。
6.3.1 电极修饰传感器 电极修饰传感器由固定了适配体的电极和电化学活性传感元件构成。在加入待检样品后,适配体与靶物质结合,导致电极表面结构改变,通过检测电流、电阻等的变化定性定量检测靶物质。Luo等[74]将大肠杆菌O111适配体与捕获探针相连,固定在金电极表面,当大肠杆菌O111存在时,适配体脱离捕获探针,与靶物质结合。识别探针与裸露的捕获探针连接留在金电极表面,加入识别元件链霉抗碱性磷酸酶后引起电势改变(图9)。该方法可用于牛奶中大肠杆菌O111的检测,其检测限为305 CFU/mL,3.5 h可完成检测分析。
6.3.2 纳米管传感器 纳米管电化学传感器与电极修饰传感器原理相似,是将适配体修饰在纳米管内壁上,通电后记录初始电势,当靶物质通过纳米管时会引起电势信号的改变。常用于电化学检测的纳米管有硅纳米孔[76]及单壁碳纳米管[77-80]。Gustavo等[77]利用单壁碳纳米管同时检测大肠杆菌O157∶H7及单增李斯特菌两种菌(图10)。研究者将大肠杆菌O157∶H7及单增李斯特菌的适配体分别修饰在单壁碳纳米管壁内部,当两种菌或其中一种存在于样品中时,与其对应适配体与靶物质结合,通电的单壁碳纳米管会产生电势变化,实现对两种菌的实时定量检测。该方法的检测区间为2-103CFU/ mL,灵敏度较高。但受到单壁碳纳米管内径大小的限制,其检测上限较低,即当样品中的靶物质浓度较高时,可能会出现假阴性的试验结果。
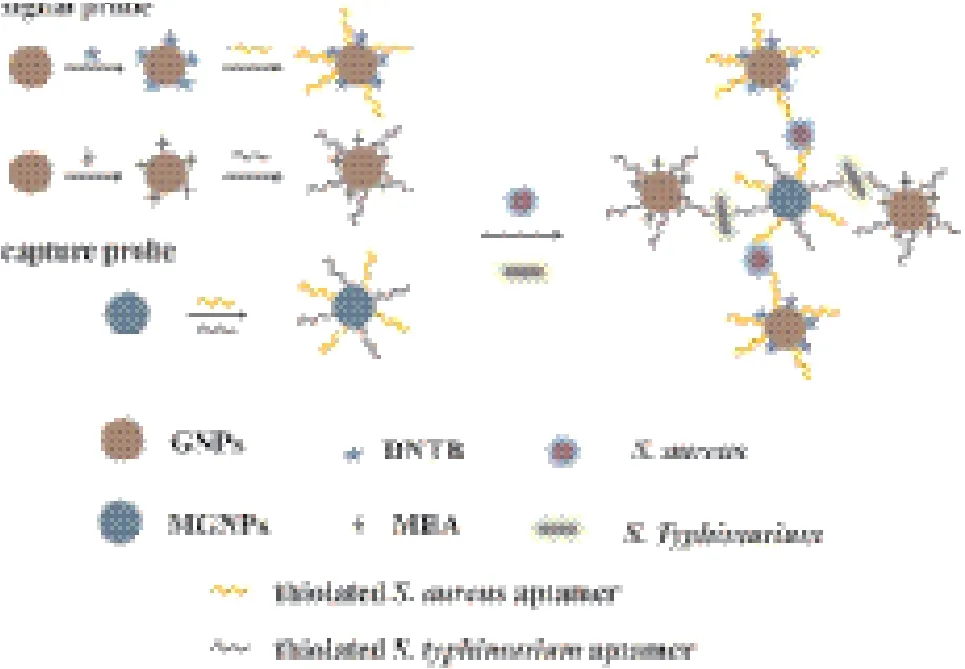
图8 SERS传感器检测鼠伤寒沙门氏菌的原理示意图[70]

图9 电极修饰传感器检测大肠杆菌O111原理示意图[74]

图10 单壁碳纳米管传感器检测大肠杆菌O157:H7原理示意图[77]
6.4 适配体压电晶体传感器
压电晶体是非中心对称晶体,在机械力作用下可产生形变,使带电质点发生相对位移,从而在晶体表面出现正、负束缚电荷,极轴两端产生的电势差可作为检测信号。其中,石英晶体微天平(quartz crystal mierobalance,QCM)是最常见的压电晶体传感[68,58]。Ozalp等[81]将特异性识别致病菌的适配体修饰在QCM传感器上,当样品中出现靶物质时,两者的结合可导致QCM传感器发生形变,出现电势差(图11)。研究者利用这一原理检测了牛奶中沙门氏菌的含量,方法的检测限为100 CFU/ mL。该方法装置简单,每个晶体能重复使用多次,可用于实际样品的批量检测。

图11 石英晶体微天平传感器检测细菌原理示意图[81]
7 结语
适配体对靶物质可特异性识别且结合力很强,同时适配体还具有分子小、易合成、易修饰、相对稳定等优势,作为一类分子识别元件易于搭载各种传感元件组建不同类型的生物传感器,满足当下对食品中可能存在的食源性致病菌的现场检测要求。
但是,目前适配体的发展还存在着一些局限性:(1)针对不同食源性致病菌,尤其是真菌类物质,适配体种类有限;(2)缺乏对适配体与靶物质结合的位点系统研究;(3)同时检测多种食源性致病菌的生物传感器模型较少。
针对目前存在的不足,适配体检测食源性致病菌可以从以下3个方面进行深入研究:(1)筛选更多种类的食源性致病菌适配体;(2)借助生物信息学手段,精准定位适配体在靶物质上的结合位点;(3)建立能检测食源性致病菌的高通量、高特异性、高灵敏度的适配体传感器。此外,适配体与磁珠等结合可以抓取复杂机制中的靶物质,也为实际样品中食源性致病菌的分离提取提供了新的思路。
[1]徐小英. 我国食物(食品)质量安全存在的主要问题及应对措施[J]. 首都师范大学学报:自然科学版, 2013, 34(6):76-85.
[2]刘士敬. 食源性疾病的流行病学[J]. 中国社区医师, 2008, 24(4):9-10.
[3]Tim, Lawruk. 食源性致病菌的检测现状与突破[J]. 食品安全导刊, 2015(z1):44-45.
[4]GB29921-2013《食品安全国家标准——食品中致病菌限量》[S]
[5]李宁, 杨大进, 郭云昌, 等. 我国食品安全风险监测制度与落实现状分析[J]. 中国食品学报, 2011, 11(3):5-8.
[6]Banerjee J, Nilsen-Hamilton M. Aptamers:multifunctional molecules for biomedical research[J]. Journal of Molecular Medicine, 2013, 91(12):1333-1342.
[7]Chen B, Wang Z, Hu D, et al. Determination of nanomolar levels of mercury(II)by exploiting the silver stain enhancement of the aggregation of aptamer-functionalized gold nanoparticles[J]. analytical letters, 2014, 47(5):795-806.
[8]Wang X, Guo XG. Ultrasensitive Pb2+detection based on fluorescence resonance energy transfer(FRET)between quantum dots and gold nanoparticles. [J]. Analyst, 2009, 134(7):1348-1354.
[9]Wu S, Duan N, Shi Z, et al. Dual fluorescence resonance energy transfer assay between tunable upconversion nanoparticles and controlled gold nanoparticles for the simultaneous detection of Pb2+and Hg2+[J]. Talanta, 2014, 128:327-336.
[10]Wang L. Selection of DNA aptamers that bind to fourorganophosphorus pesticides[J]. Biotechnology Letters, 2012, 34(5):869-874.
[11]Jiang He, Yuan Liu, Mingtao Fan, et al. Isolation and identification of the DNA aptamer target to acetamiprid[J]. J Agric Food Chem, 2011, 59(5):1582-1586.
[12]Kang BK, Kim JH, Kim S, et al. Aptamer-modified anodized aluminum oxide-based capacitive sensor for the detection of bisphenol A[J]. Applied Physics Letters, 2011, 98(7):073703.
[13]倪姮佳. 恩诺沙星和磺胺二甲嘧啶核酸适配体的筛选及化学发光检测方法的研究[D]. 北京:中国农业大学, 2014.
[14]Liu L. Development of an immunochromatographic strip test for rapid detection of ciprofloxacin in milk samples[J]. Sensors, 2014, 14(9):16785-16798.
[15]Lee YJ, Han SR, Maeng JS, et al. In vitro selection of Escherichia coli O157∶H7-specific RNA aptamer[J]. Biochemical and Biophysical Research Communications, 2012, 417(1):414-420.
[16]Wu W, Zhang J, Zheng M, et al. An aptamer-based biosensor for colorimetric detection of Escherichia coli O157∶H7[J]. PLoS One, 2012, 7(11):e48999.
[17]Li H, Ding X, Peng Z, et al. Aptamer selection for the detection of Escherichia coli K88[J]. Canadian Journal of Microbiology, 2011, 57(6):453-459.
[18]Bruno JG, Carrillo MP, Phillips T. In vitro antibacterial effects of antilipopolysaccharide DNA aptamer-C1qrs complexes[J]. Folia Microbiologica, 2008, 53(4):295-302.
[19]Kim SE. Harnessing aptamers for electrochemical detection of endotoxin[J]. Analytical Biochemistry, 2012, 424(1):12-20.
[20]Bruno JG. A novel screening method for competitive FRET-aptamers applied to E. coli assay development[J]. Mccarthy, 2010, 20(6):1211-1223.
[21]Savory N, Nzakizwanayo J, Abe K, et al. Selection of DNA aptamers against uropathogenic Escherichia coli NSM59 by quantitative PCR controlled Cell-SELEX. [J]. J Microbiol Methods, 2014, 104:94-100.
[22]Janagama JH, HP Dwivedi. Selection, characterization, and application of DNA aptamers for the capture and detection of Salmonella enterica serovars[J]. Mol Cell Probes, 2009, 23(1):20-28.
[23]Pan Q, Zhang XL, Wu HY, et al. Aptamers that preferentially bind type IVB pili and inhibit human monocytic-cell invasion by Salmonella enterica serovar typhi[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(10):4052-4060.
[24]Liu GQ, Yu XF, Xue F, et al. Screening and preliminary application of a DNA aptamer for rapid detection of Salmonella O8[J]. Microchimica Acta, 2012, 178(1-2):237-244.
[25]Hyeon JY, Chon JW, Choi IS, et al. Development of RNA aptamers for detection of Salmonella enteritidis[J]. Journal of Microbiological Methods, 2012, 89(1):79-82.
[26]Cao X, Li S, Chen L, et al. Combining use of a panel of ssDNA aptamers in the detection of Staphylococcus aureus[J]. Nucleic Acids Research, 2009, 37(14):4621-4628.
[27]Suh SH, Dwivedi HP, Choi SJ, et al. Selection and characterization of DNA aptamers specific for Listeria species[J]. Analytical Biochemistry, 2014, 459(18):39-45.
[28]Duan N, Wu S, Chen X, et al. Selection and identification of a DNA aptamer targeted to Vibrio parahemolyticus[J]. J Agric Food Chem, 2012, 60(16):4034-4038.
[29]Kärkkäinen RM, Drasbek MR, McDowall I, et al. Aptamers for safety and quality assurance in the food industry:detection of pathogens[J]. International Journal of Food Science and Technology, 2011, 46(3):445-454.
[30]Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment:RNA ligands to bacteriophage T4 DNA polymerase[J]. Science, 1990, 249(4968):505-510.
[31]Ogihara K, Savory N, Abe K, et al. DNA aptamers against the Cry j 2 allergen of Japanese cedar pollen for biosensing applications[J]. Biosen Bioelectron, 2015, 63:159-165.
[32]Savory N, Goto S, Yoshida W, et al. Two-dimensional electrophoresis-based selection of aptamers against an unidentified protein in a tissue sample[J]. Analytical Letters, 2013, 46(18):2954-2963.
[33]Xi Z, Huang R, Li Z, et al. Selection of HBsAg-specific DNA aptamers based on carboxylated magnetic nanoparticles and their application in the rapid and simple detection of hepatitis B virus infection[J]. ACS Appl Mater & Interfaces, 2015, 21:11215-23.
[34]Ashley J, Ji K, Li SF. Selection of cholesterol esterase aptamers using a dual-partitioning approach[J]. Electrophoresis, 2015, 36(20):2616-2621.
[35]Hijiri H, Koji S, Kazunori I. Selection of DNA aptamers against VEGF165 using a protein competitor and the aptamer blotting method[J]. Biotechnol Lett, 2008, 30:829-834.
[36] Dwivedi HP, Smiley RD, Jaykus LA. Selection of DNA aptamers for capture and detection of Salmonella typhimurium using a wholecell SELEX approach in conjunction with cell sorting[J]. Appl Microbiol Biotechnol, 2013, 97(8):3677-3686.
[37] Duan N, Ding X, He L, et al. Selection, identification and application of a DNA aptamer against Listeria monocytogenes[J]. Food Control, 2013, 33(1):239-243.
[38]Liu XM, Zhang DJ, Cao GJ, et al. RNA aptamers specific for bovine thrombin[J]. Mol Recognit, 2003, 16:23-27.
[39]Han SR, Lee SW. In vitro selection of RNA aptamer specific to Staphylococcus aureus[J]. Ann Microbiol, 2014, 2:883-885.
[40]Huge BJ, Flaherty RJ, Dada OO, et al. Capillary electrophoresis coupled with automated fraction collection[J]. Talanta, 2014, 130:288-293.
[41]Mann D, Reinemann C, Stoltenburg R, et al. In vitro selection of DNA aptamers binding ethanolamine[J]. Biochemical and Biophysical Research Communications, 2005, 338:1928-1934.
[42]Huang Y, Chen X, Xia Y, et al. Selection, identification and application of a DNA aptamer against Staphylococcus aureus enterotoxin A[J]. Analytical Methods, 2014, 6(3):690-697.
[43]Stoltenburg R, Reinemann C, Strehlitz B. FluMag-SELEX as an advantageous method for DNA aptamer selection[J]. Analytical and Bioanalytical Chemistry, 2005, 383(1):83-91.
[44]Cruz-Aguado JA, Penner G. Determination of ochratoxin a with a DNA aptamer[J]. J Agric Food Chem, 2008, 22:10456-10461.
[45]McKeague M, Bradley CR, Girolamo AD, et al. Screening and initial binding assessment of fumonisin B1 aptamers[J]. International Journal of Molecular Sciences, 2010, 11(12):4864-4881.
[46]Liu M, Kagahara T, Abe H, et al. In vitro selection of RNA aptamer to hemin[C]//Nucleic Acids Symposium Series. Oxford University Press, 2008, 52(1):513-514.
[47]Liu M, Kagahara T, Abe H, et al. Direct in vitro selection of heminbinding DNA aptamer with peroxidase activity[J]. Bulletin of the Chemical Society of Japan, 2009, 82(1):99-104.
[48]Vianini E, Palumbo M, Gatto B. In vitro selection of DNA aptamers that bind L-tyrosinamide[J]. Bioorganic & Medicinal Chemistry, 2001, 9(10):2543-2548.
[49]Zhang H, Hamasaki A, Toshiro E, et al. Automated in vitro selection to obtain functional oligonucleotides[C]//Nucleic acids symposium series. Oxford University Press, 2000, 44(1):219-220.
[50]Berezovski M, Musheev M, Drabovich A, et al. Non-SELEX Selection of Aptamers[J]. Journal of the American Chemical Society Jacs, 2006, 128(3):1410-1411.
[51]Ashley J, Ji K, Li SF. Selection of bovine catalase aptamers using non SELEX[J]. Electrophoresis, 2012, 33(17):2783-2789.
[52]Yu X, Yu Y. A mathematical analysis of the selective enrichment of NECEEM-based non-SELEX[J]. Applied Biochemistry and Biotechnology, 2014, 173(8):2019-2027.
[53]Tok J, Lai J, Leung T, et al. Selection of aptamers for signal transduction proteins by capillary electrophoresis[J]. Electrophoresis, 2010, 31(12):2055-2062.
[54]Yufa R, Krylova SM, Bruce C, et al. Emulsion PCR significantly improves nonequilibrium capillary electrophoresis of equilibrium mixtures-based aptamer selection:allowing for efficient and rapid selection of aptamer to unmodified ABH2 protein[J]. Analytical Chemistry, 2014, 87(2):1411-1419.
[55]王文凤. 真菌毒素寡核苷酸适配体的筛选与应用[D].无锡:江南大学, 2012.
[56]Wu H, Li M, Wang Y, et al. Aptasensors for rapid detection of Escherichia coli O157∶H7 and Salmonella typhimurium[J]. Nanoscale Research Letters, 2012, 7(47):765-781.
[57]Teller C, Shimron S, Willner I, et al. Aptamer-DNAzyme hairpins for amplified biosensing[J]. Analytical Chemistry, 2009, 81(21):9114-9119.
[58]Cao ZJ, Peng QW, Qiu X, et al. Highly sensitive chemiluminescence technology for protein detection using aptamer-based rolling circle amplification platform[J]. Journal of Pharmaceutical Analysis, 2011, 1(3):159-165.
[59]Song Y, Yang X, Li Z, et al. Label-free chemiluminescent ATP aptasensor based on graphene oxide and an instantaneous derivatization of guanine bases[J]. Biosensors and Bioelectronics, 2014, 51:232-237.
[60] Shim WB, Mun H, Joung HA, et al. Chemiluminescence competitive aptamer assay for the detection of aflatoxin B1 in corn samples[J]. Food Control, 2014, 36(1):30-35.
[61]Mun H, Jo EJ, Li T, et al. Homogeneous assay of target moleculesbased on chemiluminescence resonance energy transfer(CRET)using DNAzyme-linked aptamers[J]. Biosensors and Bioelectronics, 2014, 58:308-313.
[62] 段颖芬. 鼠伤寒沙门氏菌适配体的筛选及应用研究[D]. 长沙:湖南师范大学, 2014.
[63]Kellenberger CA, Wilson SC, Sales-Lee J, et al. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP[J]. Journal of the American Chemical Society, 2013, 135(13):4906-4909.
[64]Duan N, Wu S, Dai S, et al. Simultaneous detection of pathogenic bacteria using an aptamer based biosensor and dual fluorescence resonance energy transfer from quantum dots to carbon nanoparticles[J]. Microchimica Acta, 2014, 182(5-6):917-923.
[65]Kim LH, Yu HW, Kim YH, et al. Potential of fluorophore labeled aptamers for Pseudomonas aeruginosa detection in drinking water[J]. Journal of the Korean Society for Applied Biological Chemistry, 2013, 56(2):165-171.
[66]Le L, Dong X, Xinkai P, et al. Aptamer biosensor for Streptococcus hemolyticus detection based on fluorescence quenching by gold nanoparticles[J]. African Journal of Microbiology Research, 2012, 6(45):7230-7236.
[67]Di Primo C, Dausse E, Toulmé JJ. Surface plasmon resonance investigation of RNA aptamer-RNA ligand interactions[M]// Therapeutic Oligonucleotides. Humana Press, 2011:279-300.
[68]Tombelli S, Minunni M, Luzi E, et al. Aptamer-based biosensors for the detection of HIV-1 Tat protein[J]. Bioelectrochemistry, 2005, 67(2):135-141.
[69]Lei P, Tang H, Ding S, et al. Determination of the invA gene of Salmonella using surface plasmon resonance along with streptavidin aptamer amplification[J]. Microchimica Acta, 2015, 182(1-2):289-296.
[70]Zhang H, Ma X, Liu Y, et al. Gold nanoparticles enhanced SERS aptasensor for the simultaneous detection of Salmonella typhimurium and Staphylococcus aureus[J]. Biosensors and Bioelectronics, 2015, 74:872-877.
[71]Negri P, Kage A, Nitsche A, et al. Detection of viral nucleoprotein binding to anti-influenza aptamers via SERS[J]. Chemical Communications, 2011, 47(30):8635-8637.
[72]Ravindranath SP, Wang Y, Irudayaraj J. SERS driven crossplatform based multiplex pathogen detection[J]. Sensors and Actuators B:Chemical, 2011, 152(2):183-190.
[73]Cheng AKH, Sen D, Yu HZ. Design and testing of aptamer-based electrochemical biosensors for proteins and small molecules[J]. Bioelectrochemistry, 2009, 77(1):1-12.
[74]Luo C, Lei Y, Yan L, et al. A rapid and sensitive aptamer-based electrochemical biosensor for direct detection of Escherichia coli O111[J]. Electroanalysis, 2012, 24(5):1186-1191.
[75]Labib M, Zamay AS, Kolovskaya OS, et al. Aptamer-based viability impedimetric sensor for bacteria[J]. Analytical Chemistry, 2012, 84(21):8966-8969.
[76]Urmann K, Walter JG, Scheper T, et al. Label-free optical biosensors based on aptamer-functionalized porous silicon scaffolds[J]. Analytical Chemistry, 2015, 87(3):1999-2006.
[77]Zelada-Guillén GA, Riu J, Düzgün A, Rius FX.Immediate detection of living bacteria at ultralow concentrations using a carbon nanotube based potentiometric aptasensor[J]. Angew Chem Int Ed Engl, 2009, 48(40):7334-7337.
[78] Zelada-Guillén GA, Sebastián-Avila JL, Blondeau P, et al. Label-free detection of Staphylococcus aureus in skin using realtime potentiometric biosensors based on carbon nanotubes and aptamers[J]. Biosens Bioelectron, 2012, 1:226-232.
[79]So HM, Park DW, Jeon EK, et al. Detection and titer estimation of Escherichia coli using aptamer-functionalized single-walled carbonnanotube field-effect transistors[J]. Small, 2008, 2:197-201.
[80]Zelada-Guillén GA, Blondeau P, Rius FX, et al. Carbon nanotubebased aptasensors for the rapid and ultrasensitive detection of bacteria[J]. Methods, 2013, 63(3):233-238.
[81]Ozalp VC, Bayramoglu G, Erdem Z, et al. Pathogen detection in complex samples by quartz crystal microbalance sensor coupled to aptamer functionalized core-shell type magnetic separation[J]. Analytica Chimica Acta, 2015, 853:533-540.
(责任编辑 狄艳红)
Application Progress of Aptamers in the Detection of Food-borne Pathogenic Bacteria
XIE Pei-yan1ZHU Long-jiao1XU Wen-tao1,2
(1. College of Food Science and Nutritional Engineering,China Agricultural University,Beijing 100083;2. Laboratory of Food Safety Detection and Risk Assessment,College of Food Science and Nutritional Engineering,China Agricultural University,Beijing 100083)
Aptamers are artificial DNA/RNA oligonucleotides binding target ligands with high affinities,and they are selected from the libraries of single-strand nucleic acid random sequence in vitro by SELEX. Compared with the traditional methods,application of aptamers has the obvious advantages of high specificity,chemical stability and simplicity of modification in the detection of food-borne pathogenic bacteria,thus it draws attentions and applications in this field. However,the combination principle of aptamers to the target has not been understood clearly yet,and summaries on the selection of aptamers against food-borne pathogenic bacteria and its application are still deficient. Combined with the characteristics of food-borne pathogenic bacteria,this article describes the selection characteristics of aptamers against them,and the latest progress in the detection field. Finally,the existing problems and development trend are proposed.
aptamer;food-borne pathogens;biosensor;detection
10.13560/j.cnki.biotech.bull.1985.2016.04.006
2015-07-06
国家高技术研究发展计划(“863”计划,2012AA101606),北京市科技新星计划(XX2014B069)
解沛燕,女,硕士,研究方向:食品工程;E-mail:495366514@qq.com
许文涛,男,副教授,博士生导师,食品安全分子检测及分子毒理、转基因生物安全;E-mail:xuwentao1111@sina.com

