应用甲基化基因检测技术分析前列腺癌中HIC1的甲基化状态
单孟林,丁 滔,郑江花,程增辉,郭 颖,夏前林.复旦大学附属公共卫生临床中心医学检验科,上海 0508;.上海交通大学附属第六人民医院南院泌尿外科,上海 0033
应用甲基化基因检测技术分析前列腺癌中HIC1的甲基化状态
单孟林1,丁 滔2,郑江花1,程增辉1,郭 颖1,夏前林1
1.复旦大学附属公共卫生临床中心医学检验科,上海 201508;2.上海交通大学附属第六人民医院南院泌尿外科,上海 200233
[摘要]背景与目的:癌高甲基化基因1(hypermethylated in cancer 1,HIC1)因表观遗传甲基化修饰导致其在多种癌细胞和组织中表达沉默,可能与肿瘤的发生、发展有关。而这一现象在前列腺癌研究中的报道甚少。该研究采用甲基化基因检测技术对前列腺癌中HIC1启动子进行甲基化状态分析。方法:采用甲基化特异性聚合酶链式反应(methylation-specific polymerase chain reaction,MSP)和亚硫酸盐测序PCR(bisulfate sequencing PCR,BSP)技术,对前列腺癌细胞PC3、C4-2B和正常前列腺上皮细胞PrEC,以及前列腺癌组织标本和正常前列腺穿刺标本,进行HIC1启动子甲基化分析;并使用去DNA甲基化酶5-Aza-CdR处理PC3、C4-2B和PrEC,经反转录PCR(reverse transcription-PCR,RT-PCR)和蛋白[质]印迹法(Western blot)分析HIC1在基因水平和蛋白质水平上的变化。结果:MSP分析结果发现,在PC3、C4-2B和PrEC中HIC1甲基化率分别为78.23%、72.15%和10.63%,差异有统计学意义(P<0.05)。对36例人前列腺癌实体瘤和36例人前列腺正常穿刺组织进行BSP测序,统计显示,甲基化率分别是80.30%和31.56%。5-Aza-CdR处理后的PC3、C4-2B和PrEC经RT-PCR和Western blot分析发现,与未处理对照相比,HIC1转录水平和蛋白水平表达均显著上调。结论:在前列腺癌中,抑癌基因HIC1启动子呈高度甲基化状态并出现表达沉默或下调,可作为前列腺癌的早期预测指标和潜在的治疗靶点。
[关键词]甲基化特异性PCR;亚硫酸盐测序PCR;前列腺癌;癌高甲基化基因1;甲基化
Correspondence to:ZHENG JianghuaE-mail:zhengjianghua2015@163.com
近年来,我国前列腺癌的发病率逐年上升,已跃居男性泌尿生殖系肿瘤的第3位[1],但前列腺癌侵袭转移的作用机制尚不清楚[2-3]。癌高甲基化基因1 (hypermethylated in cancer 1,HIC1)是一种抑癌基因[4]。目前研究表明,在多数肿瘤中,HIC1经常高甲基化,提高了甲基化转移酶的活性,以至在癌细胞和癌组织中出现HIC1表达沉默[5-6],这可能与肿瘤的进展有关。然而,在前列腺癌中,HIC1的高甲基化情况及是否与前列腺癌进展有关,目前报道甚少。因此有必要探讨HIC1在前列腺癌不同进展时期的甲基化状态。
本研究采用甲基化特异性聚合酶链式反应(methylation-specific polymerase chain reaction,MSP)和亚硫酸盐测序PCR(bisulfate sequencing PCR,BSP)技术,对前列腺癌细胞和正常前列腺上皮细胞,以及前列腺癌组织标本和正常前列腺穿刺标本进行HIC1启动子甲基化分析,以探讨HIC1甲基化水平对前列腺癌发生、发展的影响。
1 材料和方法
1.1标本及临床病理资料
前列腺癌组织标本来自2011年4月—2014年5月在汕头大学医学院附属粤北人民医院泌尿外科住院的36例患者,年龄56~85岁。术前均无药物、外科去势及放射治疗史,经手术或穿刺活检,所有标本经组织病理学检查证实为前列腺腺癌。按照Gleason评分、前列腺特异性抗原(prostate-specific antigen,PSA)水平、2002年AJCC的TNM分期系统,所有患者均随访至2014 年3月。同时选取36例正常前列腺穿刺组织作为对照组,年龄55~89岁,术前无长期药物治疗史,血清PSA检测均为阴性。所有组织样本于离体30 min内以0.9%NaCl溶液冲洗表面污物血渍后,于肿瘤边缘细胞活力较好处切取肿瘤组织,剔除可能附着的血管、神经及结缔组织等迅速置于液氮中冻存,并转存于-80 ℃冰箱备用。
1.2细胞复苏及培养
人前列腺癌细胞系PC3、C4-2B、DU145和LnCap购自美国ATCC公司,保存于液氮中,复苏后在含10%胎牛血清的RPMI1640培养基(美国GIBCO公司)中传代培养,培养箱条件为CO2体积分数为5%、37 ℃恒温。人前列腺正常上皮细胞PrEC购自武汉原生原代生物医药科技有限公司,需用专用培养基,其中加有特制的细胞因子和10%FBS。取对数生长期细胞进行实验。
1.3基因组DNA提取及亚硫酸盐处理
采用基因组DNA小量抽提试剂盒(离心柱式,购自碧云天生物技术研究所)抽提前列腺癌细胞与组织的基因组DNA,并进行纯度鉴定,-20 ℃保存备用。按照EZ DNA Methylation Gold Kit(ZYMO研究试剂公司)说明书操作,使抽提到的DNA中未发生甲基化的胞嘧啶通过亚硫酸氢盐处理后全部转化为尿嘧啶,而甲基化的胞嘧啶不变。
1.4HIC1启动子区MSP和BSP分析和引物设计
从NCBI数据库中选取HIC1(NM-006497.3,GeneID:3090)转录起始点上游-1 000 bp的核苷酸序列输入分析软件。使用的在线分析软件为Methprimer(http://www.urogene.org/methprimer/ indexl.html),可以预测被分析序列内的CpG岛,完成MSP、甲基化非特异性PCR(unmethylationspecific PCR,USP)和BSP技术的引物设计。
1.5HIC1启动子区MSP和BSP反应
以修饰后的DNA作为模板,按照热启动Taq酶试剂盒ZymoTaqTMPreMix进行PCR扩增。反应体系:ZymoTaqTMPreMix 25.0 μL,0.3~1.0 μmol/L的上下游引物(图1),<200 ng/50 μL的模板DNA,加ddH2O至50 μL。PCR反应条件:95 ℃预变性10 min;95 ℃变性30 s,65 ℃退火40 s,72 ℃延伸1 min,36个循环;72 ℃终延伸7 min。取PCR产物5 μL,经1%琼脂糖凝胶电泳分离。

图1 HIC1启动子CpG岛的情况Fig. 1 HIC1 promoter region CpG islands
1.6目的基因测序
在紫外灯下从琼脂糖凝胶中切下含待回收DNA的凝胶,经QIAquick Gel Extract Kit(购自德国QIAGEN公司)纯化后用A Cloning Kit(购自美国Invitrogen公司)连接到PCR2.1载体,并将连接产物转化DH5α感受态细菌(购自上海申能博彩生物技术公司)。挑取选择性培养基平板上白色的单菌落10~20个进行PCR鉴定,以电泳片段的大小区分目的基因,把含有目的基因的菌落接种于含有氨苄西林的LB中,37 ℃、250 r/min摇菌过夜,取1 mL菌液送上海生物工程公司测序,利用在线甲基化分析网站QOMA(http://quma.cdb. riken.jp/top/index.html)分析测序结果。
1.7HIC1表达的反转录PCR(reverse transcription-PCR,RT-PCR)检测
分别提取5-Aza-Cd R处理后的实验组和对照组的P C 3、C 4 - 2 B和P r E C细胞的总R N A,采用c D N A合成反转录系统(购自美国P r o m e g a公司)将其反转录成cDNA,再进行RT-PCR检测。引物(正义链):5'-GAGAGCTTCGGTGACAACCTGTA-3',引物(反义链):5'-TTCTTCCCGCAGATGGTGCA-3',反应体系:cDNA 1 μL,10×Buffer 2.5 μL,dNTP (10 mmol/L) 0.5 μL,Taq 酶0.25 μL,无菌水5 μL,引物各2.5 μL,最后加DEPC水到25 μL。取PCR产物5 μL,经1%琼脂糖凝胶电泳鉴定为单一条带,产物大小符合。PCR所用引物采用Primer5.0在线软件设计,基因序列通过NCBI网站获得。所有引物均通过BLAST比对验证其正确性,产物经测序证实,以GAPDH作内参。
1.8HIC1蛋白表达的蛋白[质]印迹法(Western blot)检测
将5-Aza-CdR处理后的实验组和对照组的PC3、C4-2B和PrEC细胞按单层贴壁细胞总蛋白提取方法抽提细胞的蛋白质,经Western blot检测HIC1蛋白的表达。一抗为Anti-HIC1抗体(H8539,购自美国Sigma公司),按照说明书用5%脱脂奶粉TBST以1∶5 000比例稀释后使用。
1.9统计学处理
实验结果用SPSS 15.0软件进行统计分析,所有数据均以±s表示,P<0.05为差异有统计学意义。
2 结果
2.1前列腺癌细胞中HIC1启动子的分析
使用在线分析软件M e t h p r i m e r预测H I C 1(N M - 0 0 6 4 9 7 . 3,G e n e I D:3 0 9 0)转录起始点上游- 1 0 0 0 b p区域内存在3个主要的C p G岛,G C百分比>50.0%,每个岛均>100 bp,Obs/Exp>0.60(图1);并且获得HIC1启动子CpG岛2(-624~-495 bp)区域的MSP、USP和BSP引物共5对,选取其中1对行MSP、USP和BSP分析(表1)。

表1 HIC1启动子CpG岛2(-624~-495 bp)区域检测引物Tab. 1 Primers used for MSP and BSP in HIC1 promoter region CpG island 2
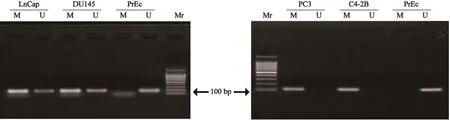
图2 MSP产物琼脂糖凝胶电泳结果Fig. 2 MSP reaction electrophoregram
2.2HIC1启动子区MSP和BSP分析结果
MSP分析出现的大小为119 bp的琼脂糖电泳条带为阳性PCR产物,发现在人前列腺癌细胞株PC3、C4-2B中,HIC1启动子呈高甲基化,在DU145、LnCap中,HIC1启动子呈部分甲基化,而在人前列腺正常上皮细胞PrEC中,HIC1的启动子几乎没有甲基化(图2)。统计分析显示,PC3、C4-2B和PrEC中HIC1甲基化率分别为78.23%、72.15%和10.63%,前列腺癌细胞PC3 和C4-2B分别与PrEC比较,差异有统计学意义(P<0.05)。BSP分析出现的大小为129 bp的琼脂糖电泳条带为阳性PCR产物,结果显示,在人前列腺癌细胞系PC3中HIC1呈高甲基化,此结果与MSP结果相符(图3)。

图3 BSP产物琼脂糖凝胶电泳结果Fig. 3 BSP reaction electrophoregram
2.3目的基因测序结果
进一步针对HIC1启动子区CpG岛2的-624~-495 bp区域,对人前列腺癌细胞PC3、C4-2B和人前列腺正常上皮细胞PrEC进行BSP测序(图4)。另外,对36例前列腺癌实体瘤和36例前列腺正常穿刺组织进行BSP测序,其中分别检测到29例和11例HIC1启动子发生高甲基化(表2),统计显示甲基化比例分别是80.30%和31.56%,差异有统计学意义(P<0.05)。

图4 人前列腺癌细胞系和人前列腺正常细胞系中HIC1启动子BSP分析的测序峰图Fig. 4 The sequencing results of HIC1 promoter region CpG island 2 purple represent methylated cytosine

表2 人前列腺癌实体瘤的甲基化水平和临床资料Tab. 2 Clinical information and body tumor methylation status of prostate cancer patients
2.4RT-PCR和Western blot检测结果
使用去DNA甲基化酶试剂5-Aza-CdR处理前列腺癌细胞株PC3、C4-2B和前列腺正常细胞株PrEc,经RT-PCR(图5A)和Western blot(图5B)检测发现,与未处理对照相比,HIC1转录水平和蛋白的表达均显著上调。
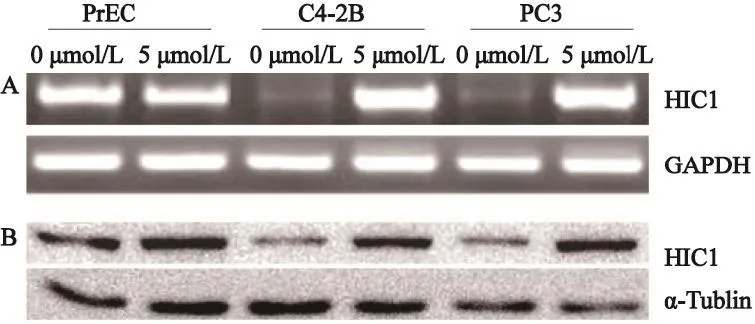
图5 5-Aza-CdR处理前列腺癌细胞株和PrEc细胞中HIC1表达变化结果Fig. 5 The efects of 5-Aza-CdR on HIC1 expression
3 讨论
表观遗传学在肿瘤的形成过程中具有重要作用,是指在不改变基因组序列的前提下,通过DNA和组蛋白的修饰等来调控基因表达,其中以DNA甲基化最为常见[7-8]。在人类表观遗传学研究中,最常见的就是CpG二核苷酸中胞嘧啶的甲基化修饰。在正常细胞中,位于抑癌基因启动子区的CpG岛处于低水平或未甲基化状态,此时抑癌基因处于正常的开放状态,抑癌基因不断表达抑制肿瘤的发生。而在肿瘤细胞中,该区域的CpG岛被高度甲基化,染色质构象发生改变,抑癌基因的表达被关闭,从而导致细胞进入细胞周期,凋亡丧失,DNA修复缺陷,血管生成及细胞黏附功能缺失等,最终导致肿瘤发生[9]。由于CpG岛的局部高度甲基化要早于细胞恶性增生,早在肿瘤临床确诊之前就可检测出特异基因的甲基化异常的现象,故其甲基化的检测对肿瘤的筛查和风险评估、早期诊断、分期分型、预后判断及治疗监测具有重要的意义[10-11]。DNA甲基化研究的检测方法很多,MSP和BCP是2种候选基因甲基化分析方法。
HIC1是定位在CpG岛的一种抑癌基因,位于人类第17号染色体短臂上(17p13.3)。有研究表明,HIC1转录起始于P0、P1和P2这3个独立的启动子,其中P1和P2富含GC序列[12]。HIC1启动子CpG岛2包含有11个CpG位点[13],其甲基化是一个渐进的过程,每一个胞嘧啶碱基都是逐渐被甲基化的。Gonzalgo等[14]认为CpG岛甲基化达到一定比例(>60%)时才足以完全关闭基因表达,而低比例的甲基化只能降低其表达。本研究对HIC1启动子CpG岛2(-624~-495 bp)区域的11个CpG位点行MSP、USP和BSP分析,结果显示,在前列腺癌细胞系和患者组织中,抑癌基因HIC1启动子的高甲基化水平均明显高于正常对照(P<0.05)。经DNA甲基化酶试剂5-Aza-CdR处理的PC3、C4-2B和PrEC细胞与未处理对照相比,HIC1转录和蛋白表达均显著上调,而前列腺正常细胞株PrEC中,HIC1转录和蛋白表达均没有变化。这表明5-Aza-CdR作用于高甲基化前列腺癌细胞,使其发生去甲基化,恢复HIC1表达;而对低甲基化的前列腺正常上皮细胞,5-Aza-CdR处理没有影响其表达。这说明可能是前列腺癌细胞中HIC1高甲基化引起其表达沉默。
前列腺癌是一种老年性疾病,而且早期难以发现,用现有的诊断方法确诊时,患者大多已经发生了骨和肺部的转移,延误了治疗时机。Kilinc等[15]发现在Gleason评分高的前列腺癌组织中抑癌基因HIC1、SFRP2和DAPK1呈高度甲基化。我们在前列腺癌细胞株和前列腺癌组织标本中得到的结果与这些报道一致。认为在前列腺癌细胞株和前列腺癌组织中,HIC1启动子是高甲基化的,并且呈现表达缺失或下调。本研究结果提示,对前列腺癌的早期诊断可以从检测HIC1的甲基化水平入手。Chen等[16]发现杂合敲除基因HIC1(+/-)小鼠能诱发多种肿瘤发生,提示HIC1很可能参与肿瘤的发生、发展。HIC1启动子高度甲基化如何调节前列腺癌的发生、发展和侵袭转移,将是未来需要深入研究的方向。
[参 考 文 献]
[1] CHIANG A C, MASSAGUÉ J. Molecular basis of metastasis [J]. N Engl J Med, 2008, 359(26):2814-2823.
[2] ENGSTROM C A. Hot flashes in prostate cancer:state of the science [J]. Am J Mens Health, 2008, 2(2):122-132.
[3] FITZPATRICK J M, SCHULMAN C, ZLOTTA A R, et al. Prostate cancer:a serious disease suitable for prevention [J]. BJU Int, 2009, 103(7):864-870.
[4] JENAL M, BRITSCHGI C, FEY M F, et al. Inactivation of the hypermethylated in cancer 1 tumour suppressor--not just a question of promoter hypermethylation?[J]. Swiss Med Wkly, 2010, 140:w13106.
[5] SAITO K, SAKURAI S, SANO T, et al. Aberrant methylation status of known methylation-sensitive CpG islands in gastrointestinal stromal tumors without any correlation to the state of c-kit and PDGFRA gene mutations and their malignancy[J]. Cancer Sci, 2008, 99(2):253-259.
[6] DUMONT N, WILSON M B, CRAWFORD Y G, et al. Sustained induction of epithelial to mesenchymal transition activates DNA methylation of genes silenced in basallike breast cancers[J]. Proc Natl Acad Sci U S A, 2008, 105(39):14867-14872.
[7] CARMONA F J, ESTELLER M. Epigenomics of human colon cancer[J]. Mutat Res, 2010, 693(1-2):53-60.
[8] JONES P A, BAYLIN S B. The epigenomics of cancer[J]. Cell, 2007, 128(4):683-692.
[9] ESTELLER M. Cancer epigenomics:DNA methylomes and histone-modification maps[J]. Nat Rev Genet, 2007, 8(4):286-298.
[10] RAKYAN V K, HILDMANN T, NOVIK K L, et al. DNA methylation profiling of the human major histocompatibility complex:a pilot study for the human epigenome project[J]. PLoS Biol, 2004, 2(12):e405.
[11] FINAK G, BERTOS N, PEPIN F, et al. Stromal gene expression predicts clinical outcome in breast cancer[J]. Nat Med, 2008, 14(5):518-527.
[12] ZHENG J, XIONG D, SUN X, et al. Signification of hypermethylated in cancer 1 (HIC1) as tumor suppressor gene in tumor progression[J]. Cancer Microenviron, 2012, 5(3):285-293.
[13] ZHENG J, WANG J, SUN X, et al. HIC1 modulates prostate cancer progression by epigenetic modification[J]. Clin Cancer Res, 2013, 19(6):1400-1410.
[14] GONZALGO M L, HAYASHIDA T, BENDER C M, et al. The role of DNA methylation in expression of the p19/p16 locus in human bladder cancer cell lines[J]. Cancer Res, 1998, 58(6):1245-1252.
[15] KILINC D, OZDEMIR O, OZDEMIR S, et al. Alterations in promoter methylation status of tumor suppressor HIC1, SFRP2, and DAPK1 genes in prostate carcinomas[J]. DNA Cell Biol, 2012, 31(5):826-832.
[16] CHEN W Y, ZENG X, CARTER M G, et al. Heterozygous disruption of Hic1 predisposes mice to a gender-dependent spectrum of malignant tumors[J]. Nat Genet, 2003, 33(2):197-202.
Detection of HIC1 promoter methylation in prostate cancer using MSP and BSP methods
SHAN Menglin1,DING Tao2,ZHENG Jianghua1,CHENG Zenghui1,GUO Ying1,XIA Qianlin1(1.Department of Laboratory Medicine,Shanghai Public Health Clinical Center,Fudan University,Shanghai 201508,China;2.Department of Urology,the Sixth People's Hospital South Campus,Shanghai Jiao Tong University,Shanghai 200233,China)
[Key words]Methylation specific PCR;Bisulfate sequencing PCR;Prostate cancer;Hypermethylated in cancer 1;Methylation
[Abstract]Background and purpose:Hypermethylated in cancer 1 (HIC1) is silenced in multiple cancer cells and tissues by DNA methylation of epigenetic modification, which may modulate the initiation and progression of tumors. However, there are few reports about this phenomenon in prostate cancer. This study aimed to investigate the status of HIC1 promoter methylation in prostate cancer using methylation methods. Methods:Methylation-specific polymerase chain reaction (MSP) and bisulfate sequencing PCR (BSP) were used to detect the methylation status of HIC1 promoter in prostate cancer cell lines PC3 and C4-2B, prostate normal cell line PrEC, primary Chinese PCa tissues and the respective healthy control cases. HIC1 expression level was respectively determined by reverse transcription-PCR (RT-PCR) and Western blot assays in PC3, C4-2B and PrEC cells treated with 5-Aza-CdR. Results:We found that the percentages of HIC1 promoter methylation were 78.23%, 72.15% and 10.63% in PC3, C4-2B and PrEC cells by MSP analyses. Moreover, the levels of methylated HIC1 promoter in 36 primary Chinese PCa tissues compared with the respective healthy control cases were 80.30% vs 31.56%. Expressions of HIC1 mRNA and protein level were restored in PC3 and C4-2B cells after5-Aza-CdR treatment. Conclusion:These findings demonstrate that HIC1 promoter region is hypermethylated in prostate cancer, which results in silence or downregulation of HIC1. The status of HIC1 methylation can be a valuable marker in the early stage of prostate cancer and a potential therapeutic target.
DOI:10.3969/j.issn.1007-3969.2016.04.002
中图分类号:R737.25
文献标志码:A
文章编号:1007-3639(2016)04-0290-07
基金项目:上海市自然科学基金面上项目(13ZR1435000);上海市科委2014年“科技创新行动计划”实验动物研究领域科技支撑重点项目(14140901400);国家自然科学基金面上项目(81372318);上海市教委科研创新项目(13YZ049);上海市卫生局科研课题项目(20124309)。
通信作者:郑江花 E-mail:zhengjianghua2015@163.com
收稿日期:(2015-07-09修回日期:2015-09-01)

