芸芥果胶酸裂解酶基因的克隆及序列分析
方 彦,杨 刚,孙万仓,武军艳,刘自刚,曾秀存,李学才,董 云
(1.甘肃农业大学 研究测试中心,兰州 730070;2.甘肃省油菜工程技术研究中心,兰州 730070;3.甘肃省农业科学院 作物研究所,兰州 730070)
芸芥果胶酸裂解酶基因的克隆及序列分析
方彦1,2,杨刚2,孙万仓2,武军艳2,刘自刚2,曾秀存2,李学才2,董云3
(1.甘肃农业大学 研究测试中心,兰州730070;2.甘肃省油菜工程技术研究中心,兰州730070;3.甘肃省农业科学院 作物研究所,兰州730070)
摘要采用RACE 技术从芸芥自交亲和系花药中克隆出果胶酸裂解酶(Pectate lyases,PL)基因,全长1 657 bp,其完整开放阅读框(Open Read Frame, ORF)为1 371 bp,编码456个氨基酸,分子质量51.179 ku,等电点9.42。序列比对结果表明,该蛋白与其他植物的PL蛋白具有较高的同源性,因此命名为EsPL。进化树分析表明,芸芥EsPL基因与十字花科芸薹属植物甘蓝型油菜、白菜型油菜的PL基因亲缘关系较近。实时荧光定量PCR结果显示,EsPL在芸芥开花后自交亲和系花药中的表达量显著高于自交不亲和系花药中表达量,该基因可能在芸芥自交亲和性状调控过程中发挥重要作用。
关键词芸芥;EsPL基因;RACE;序列分析
芸芥(ErucasativeMill) 又名芝麻菜,属十字花科芝麻菜属,茎叶可作饲料、种子可榨油,是干旱半干旱地区的重要油料作物和节水作物。芸芥野生性强,对干旱具有优异的抵御能力,是十字花科植物抗旱育种的重要资源[1-3],其以孢子体自交不亲和的方式进行繁殖,这种繁殖方式不利于芸芥产量潜力的发挥[4]。已有研究[5-7]报道,芸芥中存在自交亲和突变体。植物的自交亲和性利于自身有益基因的保留和累加,增强抗逆性,提高产量;并且利于选育优良自交系、种质保纯,增加品种的稳定性,延长种子的使用寿命[8-9]。因此,研究已发现的芸芥自交亲和基因资源遗传机制以及克隆控制自交亲和性的特异基因,在生物学和育种等方面具有重要意义。
自交亲和性产生于自交不亲和性,植物自交亲和机制的研究是在对自交不亲和机制深入理解的基础上建立起来的。植物孢子体自交不亲和的研究主要集中在芸薹属植物,前人对芸薹属植物自交不亲和研究发现,其自交不亲和性受S位点复等位基因控制[10-11]。Richards等[12]、Sampson[13]和Fang等[14]曾分别对Brassicacampestrisssp.oleifera、raphanusraphanistrum和SinapisalbaS等位基因的分布和遗传做过深入研究。现已分离到3类与S位点连锁的基因,分别编码位于雌蕊中的SRK(Slocus receptor kinase, S位点受体酶)和SLG(S-locus glycoprotein, S位点糖蛋白),位于雄蕊中的SCR(S-locus cysteinrich protein,S位点富半胱氨酸蛋白)[15-18]。自交不亲和的实质是雌蕊细胞对同源花粉认识或拒绝的分子过程[19],涉及雌蕊和花粉相互识别的问题。自交不亲和植物自交亲和性的产生不仅与S位点连锁基因发生片段缺失有关[20-22],同时还受其他因子的调控[23]。在花粉和柱头相互作用过程中,花粉萌发后,柱头组织就与花粉管进行信号交流,一些负责降解果胶质的酶具有破坏细胞壁结构、帮助花粉管伸长的作用[24-25],这些都会影响到自交亲和的产生。 以往对芸芥亲和性的研究和探讨主要集中在生理生化[5]、遗传分析[6]和蛋白质差异表达[26]等方面,而对芸芥亲和性相关基因的分离和表达调控途径等方面的研究较少,目前分离和鉴定与自交亲和相关基因的工作中,除陈姣荣等[7]报道木葡聚糖内糖基转移酶/水解酶基因与芸芥自交亲和性的调控相关外,未见其他相关报道。为获得芸芥自交亲和相关基因的表达情况,前期的研究[27]已通过差异显示分析技术获得果胶酸裂解酶基因的EST片段。本文拟在此基础上,采用快速扩增cDNA末端(RACE)的方法,从自交亲和的芸芥花药中克隆EsPL基因全长cDNA。同时,利用实时荧光定量方法研究其在芸芥自交亲和系(Self-compatibility, SC)和自交不亲和系(Self-incompatibility, SI)开花前后柱头、花药的表达模式,并进行序列分析,以期为芸芥自交亲和分子机制的研究提供理论基础。
1材料与方法
1.1试验材料
1.1.1植物材料以芸芥自交不亲和系(SI)和突变体自交亲和系(SC)为试验材料,大田统一种植、管理,芸芥现蕾时套袋,开花前花蕾长约5 mm时剥蕾,分别取SI和SC的花药和柱头;开花后在花蕾开放当天分别取SI和SC的花药和柱头,所采样品在液氮中速冻, -80 ℃冰箱保存,备用。
1.1.2酶、试剂盒和主要化学试剂5′RACE System for Rapid Amplification of cDNA Ends,Version 2.0试剂盒购自Invitrogen公司;SMARTerTMRACE cDNA Amplification Kit试剂盒购自Clontech公司;KOD FX Neo为TOYOBO公司出品的高保真DNA聚合酶;各种限制性内切酶购自NEB公司;普通的化学药品均为国产分析纯。
1.2总RNA提取及cDNA第一链合成
按照RNA提取试剂盒说明书提取芸芥SI和SC开花前后花药和柱头的总RNA,电泳检测完整性,紫外分光光度计测定纯度, -80 ℃冰箱保存,备用。
RACE cDNA合成:根据Clontech 公司的SMARTerTMRACE cDNA Amplification Kit的操作说明书,以芸芥SC开花后花药总RNA 为模板,利用试剂盒提供的逆转录引物,在逆转录酶作用下合成3′-RACE Ready cDNA第一链;采用Invitrogen公司的5′ RACE System for Rapid Amplification of cDNA Ends,Version 2.0试剂盒操作说明书,以芸芥SC开花后花药总RNA 为模板,使用SUPERSCRIPT II 反转录酶和引物GSP1进行5′-RACE Ready cDNA 第一链的合成,并使用RNase Mix对合成的cDNA进行去RNA处理。
1.3RACE
1.3.1引物设计已知芸芥SC开花后花药的EsPL基因部分序列为甘肃省油菜工程技术研究中心实验室差异显示分析获得[27],利用Primer 5.0软件根据已知的部分序列设计5′RACE特异巢式引物和3′RACE引物(表1)。
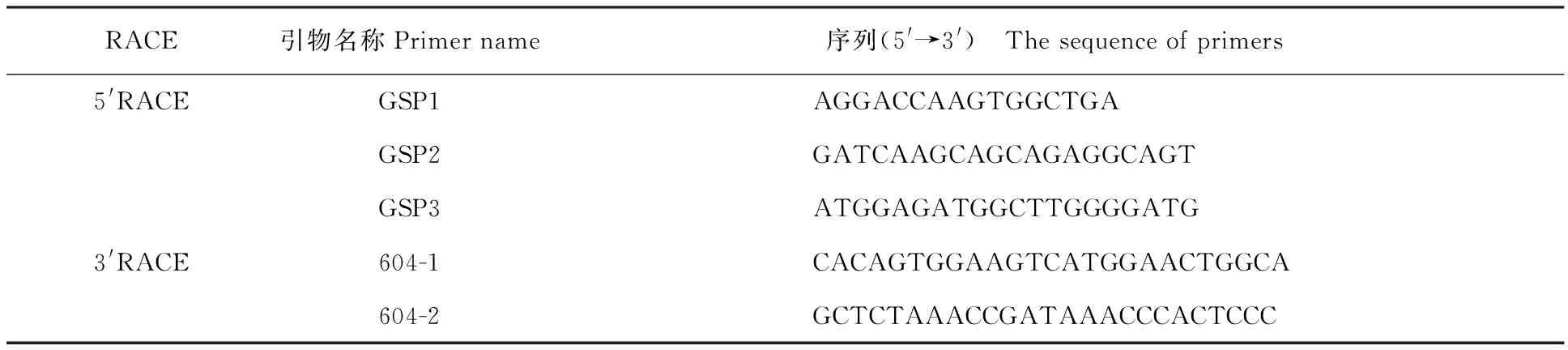
表1 5′RACE和 3′RACE引物序列
1.3.2EsPL基因 5′端序列的获得5′RACE 根据Invitrogen 公司的5′RACE System for Rapid Amplification of cDNA Ends,Version 2.0试剂盒步骤操作,以5′-RACE Ready cDNA为模板,用引物GSP2和试剂盒附带的桥连铆钉引物AAP对已经加dC尾的cDNA进行1扩PCR,以1扩PCR产物为模板,用引物GSP3和试剂盒附带的桥连通用扩增引物AUAP进行2扩PCR,将第2轮PCR产物电泳,并将目的条带切胶回收,连接pMD18T载体,转化大肠杆菌DH5α,筛选阳性克隆进行测序。
1.3.3EsPL基因3′端序列的获得3′RACE 根据Clontech 公司的SMARTerTMRACE cDNA Amplification Kit试剂盒步骤进行,以3′-RACE Ready cDNA 为模板,用引物3′604-1和引物UPM进行1扩PCR,以1扩PCR 产物为模板,用引物3′604-2和引物UPM进行2扩PCR,将第2轮PCR产物电泳,并将目的条带切胶回收,连接pMD18T载体,转化大肠杆菌DH5α,筛选阳性克隆测序。
1.3.4全长cDNA序列的拼接、编码区(CDS)的克隆与序列分析使用DNAMAN软件将DDRT-PCR分离的差异片段、3′RACE和5′RACE拼接,分析获得目的基因的全长cDNA序列。根据RACE扩增获得的EsPL基因序列全长设计上游引物P487F ( 5′- CGCGGATCCATGGCGGTTGCTTATTCG-3′) 和下游引物P487R ( 5′- GCTCTAGAGCAGGACTTGCCTGGTTTAC-3′) , 以芸芥SC开花后花药组织RNA反转录后的cDNA为模板, 进行EsPL基因CDS序列扩增。反应条件为:98 ℃预变性5 min; 98 ℃10 s,60 ℃30 s,68 ℃1.5 min,共30个循环;68 ℃延伸5 min。将全长cDNA序列及其推导的氨基酸序列登录到NCBI网站,用BLAST程序进行核苷酸和氨基酸序列的生物信息学分析。
1.4芸芥EsPL基因编码蛋白质功能分析
利用DNAMAN软件进行序列分析、拼接;NCBI在线氨基酸序列同源性比对;联网(httP://www.nebi.nlm.nih.gov/gorf/gorf.html)寻找开放阅读框;利用ProtParam、TMHMM、Signalp和SOPMA软件进行理化性质、蛋白质跨膜区段预测、信号肽预测和蛋白质二级结构预测;利用NCBI在线CDD进行结构域预测。
依据获得的EsPL基因的cDNA 全长序列,分别设计正向(CAACGCGATTGATAAATGCTGG)和反向(ATGCGTCGGTGACTACGTAGAT)特异引物,Actin基因(正向:TCACCAGAGTCGAGCACA; 反向: TTCTTACCCTCAAGTATCCG)作为内参基因,用实时荧光定量分析果胶酸裂解酶在芸芥SI和SC开花前后花药和柱头中的表达模式。试验操作和计算方法同文献[27]。
2结果与分析
2.1芸芥EsPL基因的RACE分析
由于EsPL基因的部分序列已经获得,本研究只进行5′-RACE和3′-RACE的PCR 扩增。5′RACE 扩增得到一个大小为1 318 bp的片段(图1)。将此条带切胶回收纯化后与pMD18T连接,转化后对阳性克隆测序。测序结果经核苷酸序列比对发现,与差异显示扩增得到的芸芥EsPL基因部分片段的重叠部分的核苷酸序列完全相同,并且该序列与白菜型油菜和甘蓝型油菜等的果胶酸裂解酶基因均具有较高同源性,由此可初步判定其为芸芥EsPL基因的一个片段。同时,在起始密码子ATG前有103 bp的5′非翻译区序列((5′-untranslated region,5′UTR)。
3′RACE 扩增得到一个大小约300 bp的片段(图2)。将此条带切胶回收纯化后与pMD18T连接,转化后对阳性克隆测序。测序结果经核苷酸序列比对发现,与差异显示扩增得到的芸芥EsPL基因部分片段的重叠部分的核苷酸序列完全相同,由此可初步判定其为芸芥EsPL基因的一个片段。同时,在终止密码子TAG 后有183 bp 的3′非翻译区序列(3′-untranslated region,3′UTR),包括68 bp的polyA序列。

1.巢式PCR扩增EsPL基因的5′末端结果Result of 5′RACE Nest PCR;M.DL2000 Marker
图1巢式PCR扩增EsPL基因的5′末端结果
Fig.1Nest PCR analysis for theEsPL5′RACE
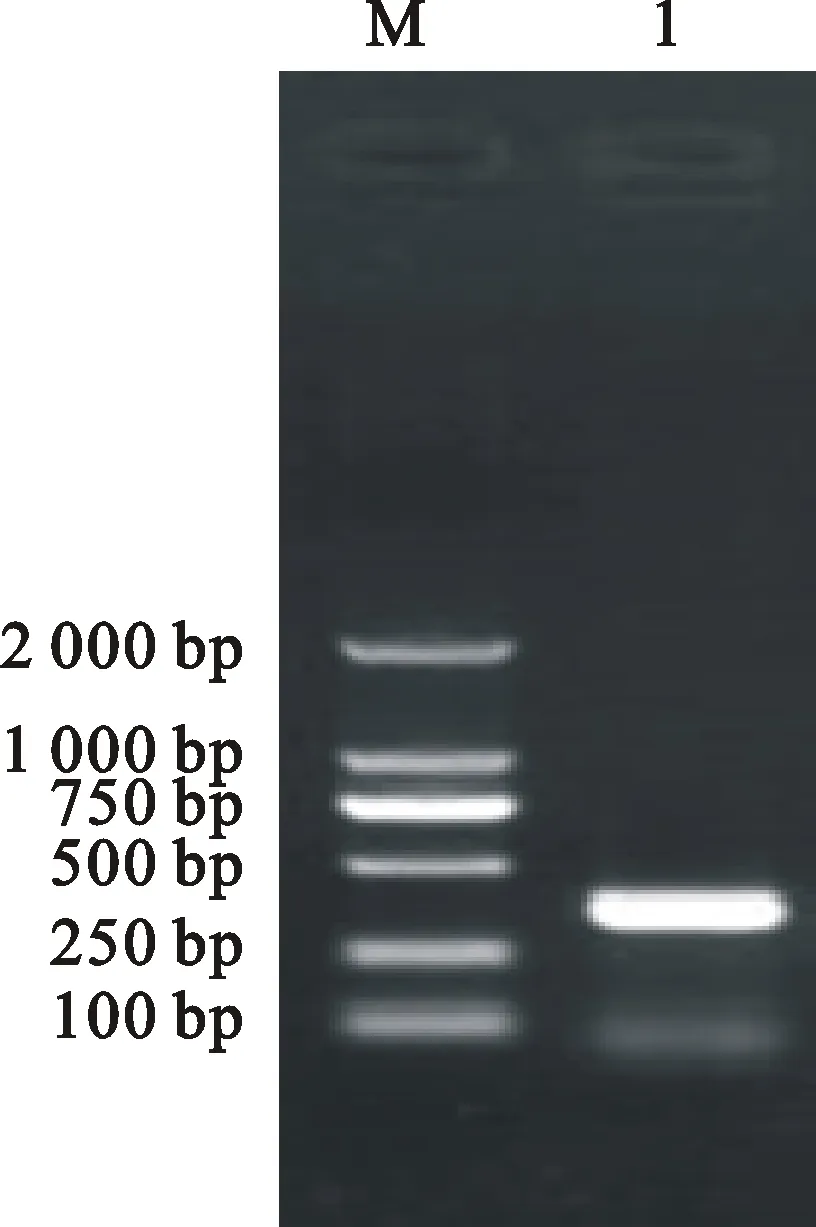
1.巢式PCR扩增EsPL基因的3′末端结果Result of 3′RACE Nest PCR; M.DL2000 Marker
图2巢式PCR扩增EsPL基因的3′末端结果
Fig.2Nest PCR analysis for theEsPL3′RACE
2.2全长cDNA序列的拼接与编码区(CDS)片段的获得
将DDRT-PCR分离得到的中间片段、5′RACE和3′RACE扩增得到的序列进行分析拼接后,得到一条1 657 bp的序列,包含1个长1 371 bp的完整开放阅读框(图3),起始密码子位于104位,终止密码子在1 474位,编码456个氨基酸,5′非编码区长103 bp,3′非编码区长183 bp,在3′非编码区存在l个mRNA的加尾信号AATAAA。

方框为起始密码子ATGATG is indicated in box; *为终止密码子TGATGA is marked by sterisk
图3EsPL基因的核酸序列及推导氨基酸序列
Fig.3Nucleotide sequence and deduced amino a
cid sequence ofEsPLgene
为进一步确证芸芥EsPL基因拼接正确与否,根据拼接所得果胶酸裂解酶全长序列设计引物PCR扩增基因的编码区,序列全长约1 380 bp(图4),回收目的片段,与pMD18-T载体连接、转化后,对质粒pMD18-T-EsPL进行PCR和双酶切鉴定,结果表明,质粒PCR条带为1 529 bp,酶切条带约为1 378 bp,对质粒pMD18-T-EsPL测序验证,序列长1 380 bp,与NCBIgenbank中同源基因及其RACE全长序列比对结果一致,将该序列命名为EsPL基因,并提交 NCBI 数据库,登录号为KP064392。

1~2.EsPL基因全长cDNA 序列扩增产物Product of the complete cDNA sequence ofEsPL;M.DL2000 Marker
图4EsPL基因全长cDNA电泳检测
Fig.4Complete cDNA ofEsPLgene
2.3芸芥EsPL基因预测蛋白的生物信息学分析
EsPL基因编码蛋白相对分子质量为51.179 ku,理论等电点为9.42,芸芥EsPL基因编码蛋白由20种氨基酸组成,其中以Gly所占比例最高,为9.0%;含有55个碱性氨基酸(K、R),39个酸性氨基酸(D、E),144 个疏水氨基酸(A、F、I、L、V、W),133个极性氨基酸(C、N、Q、S、T、Y),131个带电荷的氨基酸(C、D、E、H、K、R、Y);预测不稳定系数为31,是一个稳定蛋白(稳定系数<40时稳定);脂肪系数为75.26%,总平均疏水指数(grand average of hydropathicity,GRAVY)为-0.406,预测该蛋白属于亲水性蛋白。
疏水性分析表明(图5),EsPL编码的蛋白质是亲水性蛋白。信号肽预测表明,芸芥EsPL基因编码的蛋白存在信号肽,信号肽剪切位点位于第28与第29位氨基酸残基之间,是分泌蛋白。用TMHMM在线软件预测EsPL蛋白跨膜曲线可以更直观的反映跨膜蛋白情况,发现N端有一个跨膜蛋白(图6)。EsPL蛋白含有约24.56%的α-螺旋(α-helix),29.61%的延伸链(extended strand ),13.16%的β-转角(beta turn)和32.68%的无规则卷曲(random coil)(图7)。
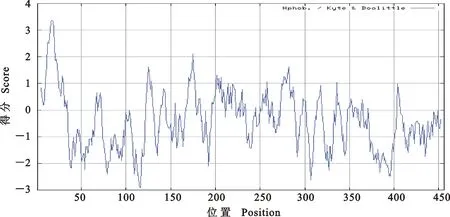
图5 芸芥EsPL蛋白的亲水性与疏水性预测
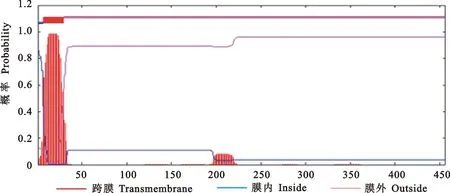
图6 芸芥EsPL蛋白跨膜区段的预测分析
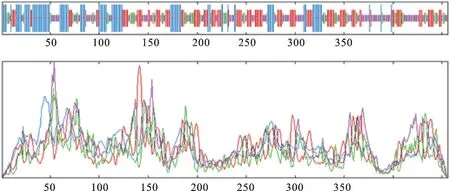
蓝色.α-螺旋Blue.α-helix; 紫色.无规则卷曲Purple.Random coil;红色.延伸链Red.Extended strand;绿色.β-转角Green.β-turn
图7 芸芥EsPL蛋白二级结构分析
Fig.7Predicted second structure of EsPL protein
对芸芥EsPL基因编码蛋白的保守结构域功能分析发现,该蛋白含有2个PL家族特有的保守结构域,分别为N端第29~84位氨基酸之间的Pec-lyase-N 结构域和C端第177~371位氨基酸之间的Amb-all结构域,其中Pec-lyase-N 结构域属于果胶酸裂解酶N家族,Amb-all结构域属于果胶酸裂解酶C家族,该家族酶具有一个右手β-螺旋结构(图8)。
2.4芸芥EsPL基因编码蛋白的同源性及进化树分析
从GenBank数据库中选择7个其他十字花科植物的PL基因序列,用DNAMAN软件进行氨基酸序列同源性分析,结果显示,芸芥EsPL基因与其他植物的PL基因具有较高的同源性。芸芥EsPL与甘蓝型油菜(CDY09356.1 )的同源性为90.0% ;与白菜型油菜(XP_009129128.1)同源性为90% ;与甘蓝型油菜(CDY41679.1)同源性为90%;与拟南芥(NP_178375.1)同源性为82%;与琴叶拟南芥(XP_002875142.1)同源性81%(图9)。构建的进化树(图10)进一步证实芸芥EsPL基因编码蛋白与十字花科芸薹属植物甘蓝型油菜(CDY09356.1 )、白菜型油菜(XP_009129128.1)的PL蛋白亲缘关系更近。
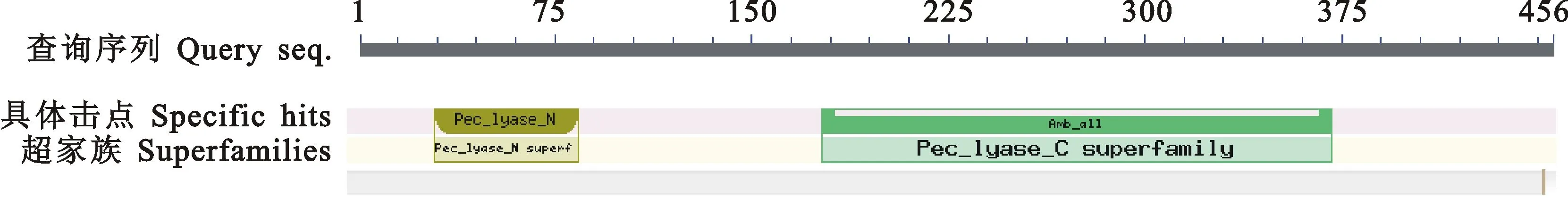
图8 芸芥EsPL蛋白质结构域预测

1.芸芥Erucasativa;2.甘蓝型油菜Brassicanapus(CDY09356.1);3.白菜型油菜Brassicarapa(XP_009129128.1);4.甘蓝型油菜Brassicanapus(CDY41679.1);5.拟南芥Arabidopsisthaliana(4NP_178375.1);6.琴叶拟南芥Arabidopsislyratasubsp.lyrata(XP_002875142.1);7.白菜型油菜Brassicarapa(XP_009101245.1);8.拟南芥Arabidopsisthaliana(NP_172894.1)
图9EsPL与同源蛋白序列多重比对
Fig.9The multiple sequence alignment of EsPL with homologous proteins
2.5果胶酸裂解酶基因在芸芥SI和SC开花前后的表达分析
以芸芥的SI开花前花药、SC开花前花药、SI开花后花药、SC开花后花药、SI开花前柱头、SC开花前柱头、SI开花后柱头和SC开花后柱头为分析对象,Actin作为内参基因,对上述不同组织部位中EsPL基因的表达进行分析,结果显示:EsPL基因在SC开花后花药中的表达量最高(图11),其次为SC开花后柱头中和SC开花前花药中。开花前与开花后SC花药中EsPL基因表达量均显著高于SI花药中表达量,为SI开花前花药中的26.18倍、SI开花后花药中的62.82倍。SC开花后柱头中PL基因表达量是SI开花后柱头中的5.68倍。

图10 十字花科植物PL氨基酸序列的系统进化树
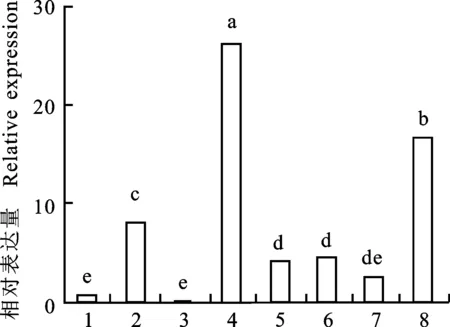
1.SI开花前花药Anther of SI pre-bloom;2.SC开花前花药Anther of SC pre-bloom;3.SI开花后花药Anther of SI after flowering;4.SC开花后花药Anther of SC after flowering;5.SI开花前柱头Stigma of SI pre-bloom;6.SC开花前柱头Stigma of SC pre-bloom;7.SI开花后柱头Stigma of SI after flowering;8.SC开花后柱头 Stigma of SC after flowering
柱状图上的不同字母代表差异达到显著水平(P<0.05) Histograms capped with different letters indicate significant difference atP<0.05
图11果胶酸裂解酶基因在芸芥花药和柱头的差异表达
Fig.11Differential expression of pectate
lyase gene in anther and stigma
3讨 论
果胶酸裂解酶是一种能降解植物细胞壁、导致植物组织软化的解聚酶[28]。它主要通过β-转移和消除机制等作用于果胶物质。果胶主要存在于植物的初生细胞壁中,参与植物花粉发育的多个过程。对果胶酸裂解酶的相关功能研究发现,在植物花粉管萌发和延伸过程中,花粉壁中的果胶酸裂解酶具有能够促进花粉管萌发、伸长,破坏花柱转导组织细胞壁、降解柱头表面的角质膜,使花粉管顺利到达胚囊[29-30]的作用。这主要通过果胶酸裂解酶降解花粉内壁果胶后促进植物花粉萌发和花粉管伸长。另外,果胶酸裂解酶也可能通过其他途径促进花粉管伸长,这一过程主要是通过降解传递系统的果胶物,利用降解产物作为花粉管形成的物质基础,不断促进其持续伸长。有研究[31-32]表明,适宜浓度的外源微生物果胶酶对番茄花粉管伸长具有显著的促进作用 。蒋晶晶[33]采用同源克隆的方法获得白菜类果胶酸裂解酶基因 BcPLL9,对 BcPLL9的表达进行反义RNA抑制后,花粉管生长无力且长度变短,到达胚珠的花粉管比例降低,植株结籽数明显减少。
孢子体型自交不亲和性在细胞学上表现为自花的花粉不能在自己的柱头上正常萌发,或萌发后在柱头乳突细胞上缠绕而无法进入柱头,花粉和雌蕊之间表现为相互抑制的作用[34]。对芸薹属植物亲和研究发现,花粉和柱头的识别要经历接触、花粉水化、花粉壁渗出蛋白质、花粉活化萌发和角质被穿透花粉管伸入等5个阶段。在此过程中,最关键的是柱头表膜接受到花粉壁蛋白信号后的识别过程,把关的是柱头角质层。如果是亲和花粉,释放出的壁蛋白中含有角质酶,经过表膜活化后,可分解表膜下的角质层,使花粉管伸入柱头[35]。芸芥属于孢子体型自交不亲和植物,自交授粉后花粉萌发数较少,柱头乳突细胞内有胼胝质沉积反应,且只有少量花粉管能穿入柱头[36]。本研究中果胶酸裂解酶是在芸芥SC开花后花药中分离出的差异表达基因,荧光定量分析发现,它在芸芥开花后SC花药中的表达量显著高于SI花药中表达量,因此它可能是主要负责降解果胶质的酶,通过破坏细胞壁结构促进花粉管伸长,同时降解花粉壁胼胝质的沉积,促进花粉从柱头吸水,这样花粉内壁就会被柱头上的蛋白质溶解,并开始萌发使花粉管进入柱头,芸芥表现为自交亲和。
4结 论
本研究通过RACE 方法从芸芥自交亲和系开花后花药中克隆得到EsPL基因全长cDNA。该基因具有完整的开放阅读框,编码456 个氨基酸,其编码的蛋白质5′端具有信号肽和跨膜区;实时荧光定量分析表明,EsPL在芸芥开花后SC花药中的表达量显著高于SI花药中表达量,初步说明EsPL基因在芸芥SI和SC性状调控过程中发挥重要作用。
参考文献Reference:
[1]官春云,李方球,李 栒,等.芸芥(ErucasativaMill.)对菌核病的抗性研究[J].中国农业科学,2004,37(8):1138-1143.
GUAN CH Y,LI F Q,LI X,etal.Resistance of rocketsalad(ErucasativaMill.) to seem rot(Sclerotiniasclerotiorum)[J].ScientiaAgriculturaSinica,2004,37(8):1138-1143(in Chinese with English Abstract).
[2]孙万仓,官春云,张金文,等.中国芸芥遗传多样性RAPD标记分析[J].中国农业科学,2003,36(11):1248-1253.
SUN W C,GUAN CH Y,ZHANG J W,etal.Genetic diversity analysia of Yunjie(ErucasativaMill.) in China by RAPD marker[J].ScientiaAgriculturaSinica,2003,36(11):1248-1253(in Chinese with English Abstract).
[3]孙万仓,官春云,孟亚雄,等.芸芥(ErucasativaMill.)与芸薹属(BrassicaL.)3个油用种的远缘杂交[J].作物学报,2005,31(1):36-42.
SUN W C,GUAN CH Y,MENG Y X,etal.Intergeneric crosses betweenErucasativaMill andBrassicaspecies[J].ActaAgronomicaSinica,2005,31(1):36-42(in Chinese with English Abstract).
[4]NEWBIGIN E D,MARCY K,UYENOYAMA.The evolutionary dynamics of self-incompatibility systems[J].TrendsinGenetics,2005,9(21):500-505.
[5]王保成,孙万仓,范惠玲,等.芸芥自交亲和系与自交不亲和系SOD、POD和CAT酶活性[J].中国油料作物学报,2006,28(2):162- 165.
WANG B CH,SUN W C,FAN H L ,etal.Studies on SOD,POD and CAT activity between self- compatible and self- incompatible lines inErucasativaMill[J].ChineseJournalofOilCropSciences,2006,28(2):162-165(in Chinese with English Abstract).
[6]范惠玲.芸芥自交亲和性相关基因mRNA差异显示分析[D].兰州:甘肃农业大学,2007.
FAN H L.Analysis of genes related to self-compatibility inErucasativaMill by mRNA differential display technique[D].Lanzhou:Gansu Agricultural University,2007 (in Chinese with English Abstract).
[7]陈姣荣,方 彦,孙万仓,等.芸芥木葡聚糖内糖基转移酶/水解酶基因 EsXTH1的cDNA克隆和生物信息学分析[J].中国油料作物学报,2013,35(2):131-136.
CHEN J R,FANG Y,SUN W C,etal.Cloning and bioinformatics of xyloglucan endotrans glycosylase and hydrolase EsXTH1 gene inErucasativaMill [J].ChineseJournalofOilCropSciences,2013,35(2):131-136(in Chinese with English Abstract).
[8]方智远,刘玉梅,杨丽梅,等.甘蓝制种技术上的一项重要变革[J].中国蔬菜,2004(5):33.
FANG ZH Y,LIU Y M,YANG L M,etal.An important change of plant species technology of cabbage[J].ChinaVegetables,2004(5):33(in Chinese).
[9]YAMANE H,USHIJIMA K,SASSA H,etal.The use of the shaplotype-specific F-box protein gene,SFB,as a molecular marker for s-haplotypes and self- compatibility in Japanese apricot(Prunusmume)[J].TheoreticalandAppliedGenetics,2003,107(8):1357-1361.
[10]BATEMAN A J.Self-incompatibility systems in angiosperms III[J].CruciferaeHeredity,1955,9:53-68.
[11]NASRALLAH J,NASRALLAH M E.Pollen-stigma signaling in the sporophytic self-incompatibility response[J].PlantCell,1993,5(10):1325-1335.
[12]RICHARDS R A,THURLING N.The genetics of self-incompatibility inBrassicacampestrisL.ssp.oleiferaMetzg.I.characteristics of S-locus.control of self-incompatibitlity[J].Genetica,1973,44(3):428-438.
[13]SAMPSON D R.Frequency and distribution of self-incompatibility alleles inRaphanusraphanistrum[J].Genetica,1967,56(2):241-251.
[14]FANG Q Z,BI F C.Self-(in)compatibility inheritance and allele-specific marker development in yellow mustard (Sinapisalba) [J].MolecularBreeding,2014,33(1):187-196.
[15]NASRALLAH J B,YU S M,NASRALLAH M E.Self-incompatibility genes of brassica oleracea:expression,isolation,and structure[J].ProceedingsoftheNationalAcademyofSciences,1988,85(15):5551-5555.
[16]SCHOPFER C R,NASRALLAH M E,NASRALLAH J B.The male determinant of self-incompatibility in brassica[J].Science,1999,286(5445):1697-1700.
[17]SUZUKI G,KAI N,HIROSE T,etal.Genomic organization of the S locus:identification and characterization of genes in SLG/SRK region of S(9) haplotype ofBrassicacampestris(syn.rapa)[J].Genetics,1999,153(1):391-400.
[18]WATANABE M,HATAKEYAMA K,TAKADA Y,etal.Molecular aspects of self-incompatibility inBrassicaspecies[J].PlantandCellPhysiology,2001,42(6):560-565.
[19]华志明.植物自交不亲和分子机理研究的一些进展[J].植物生理学通讯,1999,35(1):77-82.
HUA ZH M.Recent advances in molecular mechanisms of self-incompatibility in higher plants[J].ScienceCommunicationofPlantPhysiology,1999,35(1):77-82(in Chinese with English abstract).
[20]GORING D R,GLAVIN T L,SCHAFER U.An s receptor kinase gene in self-compatible brassica napus has a 1-bp deletion[J].PlantCell,1993,5(5):531-539.
[21]何余堂,马朝芝,马 勇,等.PCR步行法克隆油菜自交不亲和基因[J].中国油料作物学报,2004,26(1):1-5.
HE Y T,MA CH ZH,MA Y,etal.Cloning of S-locus gene by PCR walking inBrassicacampestrisL[J].ChineseJournalofOilCropSciences,2004,26(1):1-5(in Chinese with English Abstract).
[22]NASRALLAH J B,RUNDLE S J,NASRALLAH M E.Genetic evidence for the requirement of the brassica S-locus receptor kinase gene in the self-incompatibility response[J].PlantJournal,1994,5(3):373-384.
[23]赵永斌,朱利泉,王小佳.植物孢子体自交不亲和信号转导功能分子研究进展[J].生物技术通讯,2004,15(2):176-178.
ZHAO Y B,ZHU L Q,WANG X J.The functional molecules of SSI signal transduction in plants[J].LettersinBiotechnology,2004,15(2):176-178(in Chinese with English Abstract).
[24]SUEN D F,WU S S,CHANG H C,etal.Cell wall reactive proteins in the coat and wall of maize pollen:potential role in pollen tube growth on the stigma and through the style[J].JournalofBiologicalChemistry,2003,278(44):43672-43681.
[25]SUEN D F,HUANG A H C.Maize pollen coat xylanase facilitates pollen tube penetration into silk during sexual reproduction [J].JournalofBiologicalChemistry,2007,282(1):625-636.
[26]王保成.芸芥自交亲和系与自交不亲和系生理生化特性研究[D].兰州:甘肃农业大学,2006.
WANG B CH.Studies on physiological and biochemical characteristics between self-compatible and self-incompatible lines inErucasativaMill[D].Lanzhou:Gansu Agricultural University,2007(in Chinese with English Abstract).
[27]方彦,孙万仓,武军艳,等.芸芥自交亲和相关基因的差异显示及表达分析[J].中国油料作物学报,2014,36(5):580-585.
FANG Y,SUN W C,WU J Y,etal.Differential display and expression analysis of self-compatibility associated gene inErucasativaMill[J].ChineseJournalofOilCropSciences,2014,36(5):580-585(in Chinese with English Abstract).
[28]CARPITA N C,GIBEAUT D M.Structural models of primary cell walls in flowering plants:consistency of molecular structure with the physical properties of the walls during growth[J].PlantJournal,1993,3(1):1-30.
[29]TANIGUCHI Y,ONO A,SAWATANI M,etal.Cry j I ,a major allergen of Japanese cedar pollen,has pectate lyase enzyme activity[J].Allergy,1995,50(1):90-93.
[30]WU Y Z,QIU X,DU S,etal.PO149,a new member of pollen pectate lyase-like gene family from alfalfa[J].PlantMolecularBiology,1996,32(6):1037-1042.
[31]ROGERS H J,HARVEY A,LONSDALE D M.Isolation and characterization of a tobacco gene with homology to pectate lyase which is specifically expressed during microsporogenesis[J].PlantMolecularBiology,1992,20(3):493-502.
[32]PARRE E,GEITMANN A.Pectin and role of the physical properties of the wall in pollen tube growth ofSolanumchacoense[J].Planta,2005,220(4):582-592.
[33]蒋晶晶.白菜花粉发育和授粉受精相关基因的表达分析与功能验证[D].杭州:浙江大学,2012.
JIANG J J.Expression and functional characterization of genes during pollen development and fertilization inBrassicacampestrisssp.chinensis[D].Hangzhou:Zhejiang University,2012 (in Chinese with English Abstract).
[34]刘后利,王兆木.实用油菜栽培学[M].上海:上海科学技术出版社,1987.
LIU H L,WANG ZH M.Culture of Rapeseed[M].Shanghai:Shanghai Science and Technology Press,1987(in Chinese).
[35]马静芳,安彩泰.植物界中的不亲和性[M].兰州:甘肃科学技术出版社,2007.
MA J F,AN C T.Incompatibility of Plants[M].Lanzhou:Gansu Science and Technology Press,2007(in Chinese).
[36]武军艳,孙万仓.利用花粉-柱头的相互作用鉴定5个十字花科油用植物的自交亲和性[J].西北农业学报,2006,15(5):61-64.
WU J Y,SUN W C.Observation on interaction of pollen-pistil on cruciferae rapeseeds[J].ActaAgriculturaeBoreali-occidentalisSinica,2006,15(5):61-64(in Chinese with English abstract).
Received 2015-06-29Returned2015-07-22
Foundation itemThe National Natural Science Foundation of China (No.30960199,No.31260334); China Agriculture Research System (No.CARS-13); Gansu Provincial Engineering Research Center of Rapeseed (No.1306NTGA022); Gansu Provincial Natural Science Foundation((No.1308RJZA205).
First authorFANG Yan,female,Ph.D,associate research fellow.Research area:plant genetics and breeding.E-mail:ffyv@163.com
(责任编辑:顾玉兰Responsible editor:GU Yulan)
Cloning,Sequence Analysis ofEsPLGene fromErucasativeMill
FANG Yan1,2,YANG Gang2, SUN Wancang2, WU Junyan2,LIU Zigang2, ZENG Xiucun2, LI Xuecai2and DONG Yun3
(1.Instrumental Research and Analysis Center of Gansu Agricultural University, Lanzhou730070,China;2.Rape Engineering and Technology Research Center of Gansu,Lanzhou730070,China;3.Crop Institute of Gansu Agricultural Academy, Lanzhou730070,China)
AbstractThe full-length cDNA of EsPL was obtained by the technology of rapid amplification of cDNA ends (RACE).The full-length cDNA was 1 657 bp, containing a 1 371 bp opening reading frame(ORF).The deduced protein was 456 amino acids with molecular mass 51.179 ku and isoelectric point 9.42.The sequence aligment demonstrated that EsPL was high identity with other PL proteins from other species.So it was named the EsPL.Phylogram tree showes that there were closest evolutionary relationship of Brassica napus, Brassica rapa and Eruca sative Mill gene.Analysis by real-time PCR indicated that EsPL gene expression levels in SC anther is significant higher than SI anther after flowering,which suggested that EsPL played an important role in SI and SC characteristics of regulation of Eruca sative Mill.
Key wordsEruca sative Mill; EsPL gene; RACE;Sequence analysis
Corresponding authorSUN Wancang, male,Ph.D,professor.Research area:rapeseed breeding and cruciferae germplasm resources research.E-mail:18293121851@163.com
中图分类号Q781
文献标志码A
文章编号1004-1389(2016)03-0386-10
通信作者:孙万仓,男,博士,教授,研究方向为油菜育种及十字花科种质资源。E-mail:18293121851@163.com
基金项目:国家自然科学基金(30960199,31260334);国家现代农业产业技术体系建设专项(CARS-13);甘肃省油菜工程技术研究中心(1306NTGA022);甘肃省自然科学基金(1308RJZA205)。
收稿日期:2015-06-29修回日期:2015-07-22
网络出版日期:2016-03-06
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20160306.1610.018.html
第一作者:方彦,女,博士,副研究员,研究方向为作物遗传育种。E-mail:ffyv@163.com

