PRRSV经典株和变异株RT-nPCR鉴别诊断方法的建立
朱小甫,吴旭锦
(咸阳职业技术学院 畜牧兽医研究所 动物疫病分子生物学诊断实验室,陕西 咸阳 712000)
PRRSV经典株和变异株RT-nPCR鉴别诊断方法的建立
朱小甫,吴旭锦
(咸阳职业技术学院 畜牧兽医研究所 动物疫病分子生物学诊断实验室,陕西 咸阳 712000)
[摘要]【目的】 建立一种能够区分猪繁殖与呼吸综合征病毒(PRRSV)经典株和变异株的RT-nPCR诊断方法,能够从病料组织、血清或精液中直接进行目的基因检测,达到快速诊断的目的。【方法】 根据GenBank上发表的猪繁殖与呼吸综合征病毒基因序列,设计并合成了2对引物,通过改变不同的反应体系和反应条件,建立PRRSV经典株和变异株的RT-nPCR诊断方法,并通过灵敏性试验、特异性试验、病料组织和血清或精液样品检测,验证建立RT-nPCR方法的适用性。【结果】 建立了PRRSV经典株和变异株的RT-nPCR诊断方法,该方法检测的cDNA含量极限为1.3×10-7pg/μL;且仅能从PRRSV毒株中扩增到目的条带,从CH-1R经典株扩增出649 bp条带,从SXXY/2013变异株扩增出562 bp条带,电泳后能清晰区别,而从其他参考毒株中均不能扩增出目的条带。从48份疑似病料组织和血清中能直接检测出31份阳性结果。【结论】 建立了一种灵敏度高、特异性好、可直接从病料组织和血清中快速鉴别PRRSV经典株和变异株的RT-nPCR方法。
[关键词]猪繁殖与呼吸综合征;经典株;变异株;诊断方法
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)又称蓝耳病,是由猪繁殖与呼吸综合征病毒 (Porcine reproductive and respiratory syndrome virus,PRRSV)引起的一种猪群重大疫病[1-2]。1987年,本病首次在美国北卡罗来纳州爆发,随后迅速扩散到世界各地[3]。1996年,我国郭宝清首次从北京地区发病的猪群中分离出PRRSV,从而证实了本病在我国的存在[4]。2006年,首先在我国南方爆发的“猪无名高热”疫情中,农业部最终调查结果表明,猪繁殖与呼吸障碍综合征变异株是该疫病的元凶[5]。2008年,我国农业部规定高致病性猪蓝耳病为一类动物疫病,经典蓝耳病为二类动物疫病,本病成为严重威胁养猪业的重大疫病之一。
PRRSV属于尼多病毒目、动脉炎病毒科、动脉炎病毒属的成员,根据病毒的抗原性质、基因组及致病性的差异,PRRSV分为欧洲型(LV株为代表株)和美洲型(ATCC-VR2332株为代表株)。PRRSV基因组为不分节段的单链正股RNA,大小约为15 kb[6]。目前诊断蓝耳病的技术手段主要有病毒分离鉴定、病毒中和试验、荧光抗体技术、酶联免疫吸附试验和RT-PCR技术[7]。大多数诊断方法耗时较长,不能满足临床快速诊断的需要,RT-PCR技术是目前诊断方法中最为灵敏和快速的技术,但目前建立的大多数方法不能直接区分经典毒株和变异株。由于我国猪群经典蓝耳毒株和变异株共存,急需开发快速、准确的鉴别诊断技术。序列比较分析表明,与经典毒株相比,变异株在NSP2基因上连续缺失87个核苷酸[8]。针对这一特点,本研究旨在建立一种能够直观区分经典蓝耳毒株和变异株的RT-nPCR诊断方法,以期提供一种快速、准确的诊断手段,为临床防控猪蓝耳病提供科学依据。
1材料与方法
1.1材料
1.1.1病毒蓝耳病毒变异株是由本实验室从旬邑县某猪场采集病料,用Marc-145细胞传代分离获得,测定了NSP2基因序列,命名为SXXY/2013株。参考毒株中,蓝耳经典毒株CH-1R、猪瘟病毒疫苗C株、伪狂犬Bartha-K毒株疫苗,均为普莱柯生物工程股份有限公司生产;圆环病毒2型(PCV-2)、细小病毒(PPV)、流行性腹泻病毒(PEDV)、传染性胃肠炎病毒(TGEV),均为本实验室分离保存。
1.1.2病料本实验室采集或猪场送检病料组织(肺脏、淋巴结、脾脏、肝脏和肾脏)、血清或精液,分别来自咸阳、渭南、西安和宝鸡等地区不同规模猪场,共计48份,组织病料研磨处理后,12 000 r/min离心10 min,收集上清液-70 ℃保存备用。
1.1.3试剂TRIzol Reagent、DNAzol Reagent为Invitrogen公司生产;AMV反转录酶(10 U/μL)、RNA酶抑制剂(40 U/μL)、DEPC处理水、rTaq酶(5 U/μL)、dNTP(各成分均为10 mmol/L)等,均购自生工生物工程(上海)有限公司。DL 2000 Marker为宝生物工程(大连)有限公司产品。其他试剂均为国产分析纯。
1.1.4引物设计与合成根据GenBank上公开的PRRSV CH-1a株(登录号:AY032626)、HUN4株(登录号:EF635006)、JXA1株(登录号:EF112445)和VR2332株(登录号:AY150564)等全基因组序列,设计并合成了2对引物,NSP2-1F:5′- CGATCTGGTACCCGTCAA-3′(2 272~2 289,为相对于CH-1a株基因组位置,下同),NSP2-1R:5′-CAATGGTCATGGCACC-3′(3 564~3 681),NSP2-2F:5′-GAATGTCGCAGCTCACT-3′(2 726~2 744),NSP2-2R:5′-GCGTCGCATGGTACCTA-3′(3 357~3 374)。预期扩增片段长度经典毒株为649 bp,变异株为562 bp。引物由生工生物工程(上海)有限公司合成,均用DEPC处理水稀释到20 μmol/L。
1.2方法
1.2.1PRRSV RNA的提取和第一链cDNA的合成取SXXY/2013株细胞培养液250 μL,按照TRIzol Reagent试剂说明提取总RNA,倒置离心管自然干燥。在核酸干燥过程中,按照以下体系配制反转录反应液: DEPC处理水10.5 μL,上游引物NSP2-1R 1.0 μL,dNTP 4.0 μL,5×AMV Buffer 4.0 μL,AMV 0.25 μL,RNA酶抑制剂 0.25 μL,总体积20.0 μL。核酸干燥后用配制好的反转录反应液充分溶解核酸,置42 ℃水浴反转录90 min,取出后在微量紫外分光光度计上测定cDNA的含量。
1.2.2PRRSV RT-nPCR检测方法的建立取已知浓度的cDNA溶液为模板,摸索扩增条件。第1次扩增反应体系中10×PCR Buffer 2.5 μL,cDNA 2.0 μL, dNTP 1.0 μL,引物NSP2-1F和NSP2-1R各0.5 μL,改变rTaqDNA聚合酶用量(0.25~1.0 μL)和Mg2+用量(0.5~3.0 μL),用超纯水补足总体积25.0 μL。反应条件设定为:95 ℃预变性5 min; 94 ℃变性50 s,退火温度由52~58 ℃按1 ℃递增设定退火1 min,72 ℃延伸 1 min,共进行35个循环;最后72 ℃延伸10 min。第2次扩增时,取2.0 μL第1次扩增产物作为模板,其他成分同第1次扩增进行设定摸索,只是引物更换为NSP2-2F、NSP2-2R。反应条件设定为:95 ℃预变性5 min; 94 ℃变性 40 s,退火温度由52~58 ℃按1 ℃递增设定退火45 s,72 ℃ 延伸45 s,共35个循环;最后72 ℃延伸10 min。
1.2.3PRRSV RT-nPCR方法灵敏性试验将cDNA溶液做10倍梯度稀释至10-10倍,分别以各稀释后cDNA溶液作为模板进行PCR扩增。按照摸索出的最优反应体系与条件进行RT-nPCR反应,取5.0 μL第2次扩增产物,用1.5%琼脂糖凝胶电泳,凝胶成像系统中照相。
1.2.4PRRSV RT-nPCR方法特异性试验提取PRRSV SXXY/2013株、PRRSV CH-1R株及CSFV C株、流行性腹泻病毒(PEDV)、传染性胃肠炎病毒(TGEV)等RNA病毒的核酸并反转录获得cDNA;按照DNAzol Reagent试剂说明提取伪狂犬Bartha-K株、圆环病毒2型(PCV-2)、细小病毒(PPV)等DNA病毒DNA,用NSP2-1F/NSP2-1R、NSP2-2F/NSP2-2R引物对分别进行第1次和第2次扩增,PCR产物用1.5%琼脂糖凝胶电泳,成像系统中观察照相。
1.2.5病料中PRRSV的检测用以上建立的检测方法对收集的48份组织病料进行检测,验证建立的RT-nPCR方法的实用性。
2结果与分析
2.1PRRSV RT-nPCR检测方法的建立
通过改变反应体系中rTaqDNA聚合酶用量和Mg2+用量,以及反应条件中的退火温度,优化反应体系和反应条件,最终确定的最优反应体系和反应条件为:第1次扩增cDNA 2.0 μL,超纯水16.0 μL,10×PCR Buffer 2.5 μL,Mg2+2.0 μL, dNTP 1.0 μL,引物NSP2-1F和NSP2-1R各0.5 μL,rTaqDNA聚合酶0.5 μL,总体积25.0 μL。反应条件为:95 ℃预变性5 min;94 ℃ 50 s,55 ℃ 1 min,72 ℃ 1 min,共进行35个循环;最后72 ℃延伸10 min。第2次扩增反应体系:取2.0 μL第1次扩增产物作为模板,体系其余组分同第1次扩增,引物为NSP2-2F/NSP2-2R。反应条件为:95 ℃预变性5 min;94 ℃ 40 s,58 ℃ 45 s,72 ℃ 45 s,共35个循环;最后72 ℃延伸10 min。
2.2PRRSV RT-nPCR检测方法的灵敏性
经测定,反转录第一链cDNA含量为130 pg/μL,将其10倍梯度稀释后进行套式扩增。从图1可见,所建立的检测方法能够扩增出可见目的条带的最大稀释度为10-9,即检测的cDNA含量极限为1.3×10-7pg/μL,表明此方法灵敏度较高,可完全满足PRRSV临床检测的需要。

图 1 PRRSV NSP2基因 RT-nPCR检测方法灵敏度试验结果
2.3PRRSV RT-nPCR检测方法的特异性
用所建立的方法对PRRSV SXXY/2013株、CH-1R株及CSFV C株、流行性腹泻病毒(PEDV)、传染性胃肠炎病毒(TGEV)、伪狂犬Bartha-K株、圆环病毒2型(PCV-2)、细小病毒(PPV)等进行扩增,结果只有PRRSV SXXY/2013株扩增出了562 bp的目的条带,PRRSV CH-1R株扩增出了649 bp的目的条带,与预期条带长度一致,其他6种毒株未扩增出条带(图2),提示所建立的检测方法特异性好。

图 2 PRRSV NSP2基因RT-nPCR检测方法特异性试验结果
2.4PRRSV RT-nPCR方法对临床样品的检测结果
用建立的RT-nPCR方法检测陕西省部分地区采集的48份疑似PRRS的猪肺脏、淋巴结、脾脏、肝脏、肾脏等组织以及血清、精液等样品,结果有31份样品为阳性,阳性率为64.6%,部分病料检测结果见图3。由图3可知,2~4号血清样品为蓝耳病毒变异株阳性,7号精液样品为蓝耳病毒变异株阳性,9、10、12、13号组织样品为蓝耳病毒变异株阳性,而15号组织样品为蓝耳病毒经典株阳性。
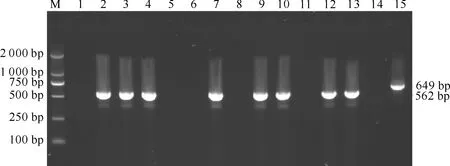
图 3 部分疑似PRRS病料中PRRSV NSP2基因检测结果
3讨论
猪繁殖与呼吸综合征是近年严重危害我国养猪业的重大传染病之一,其传播迅速,发病率和死亡率均较高,造成了严重的经济损失[9]。由于目前对PRRS尚无有效的治疗药物和办法,因此建立快速有效的诊断方法是控制PRRS的重要前提。由于PRRS与多种猪繁殖障碍性疾病临诊症状相似,临床诊断无法确诊。血清学技术中目前应用最广的是ELISA技术[7],但由于临床应用的PRRS疫苗无论是灭活苗还是弱毒苗均为非基因缺失苗,无法用ELISA技术区分疫苗免疫抗体和野毒感染抗体,不能用于诊断。
在我国《高致病性猪蓝耳病防治技术规范》中规定,要确诊高致病性猪蓝耳病必须满足病原学指标,即高致病性猪蓝耳病病毒分离鉴定阳性或高致病性猪蓝耳病病毒反转录聚合酶链式反应(RT-PCR)检测阳性。因此,在PRRS的诊断中最为可靠的方法是病毒分离鉴定和RT-PCR诊断。病毒分离鉴定的优点是准确可靠,并能获得宝贵的病毒毒株,但缺点是需要进行细胞培养传代,费用高、耗时长,不能满足临床快速诊断的需要。随着分子生物学的兴起,RT-PCR技术得到迅速发展,这一技术已广泛应用于PRRSV的快速诊断。周丹等[10]针对PRRSV普通株和变异株NSP2基因设计合成了3条引物,建立了一步鉴别诊断PRRSV普通株与变异株的RT-PCR检测方法,通过不同长度的RT-PCR扩增产物将二者区分开来,该方法最低能检测到1.5 pg的核酸含量。李和平等[11]建立了检测PRRSV的套式RT-PCR,该方法具有极高的敏感性,将106.5TCID50的PRRSV细胞毒稀释10 000倍后,依然能扩增出相应的目的片段,并且具有较好的重复性和特异性。Reicks等[12]发现,采用套式RT-PCR从接种感染24 h后的血清中能检测出PRRSV。但是,以上学者建立的检测方法中,李和平和Reicks 建立的套式RT-PCR不能区分经典毒株和变异株,周丹建立的方法能够区分但灵敏度稍低。本研究在分析PRRSV经典毒株和变异株基因差异的基础上,针对NSP2基因设计了2对引物,扩增片段包含了变异株缺失的87个核苷酸,通过改变反应体系和反应条件等参数进行优化组合,建立了鉴别诊断PRRSV经典株和变异株的RT-nPCR方法,结果表明,本方法检测cDNA极限质量浓度为1.3×10-7pg/μL,远优于周丹等建立的一步法RT-PCR检测方法,提示套式PCR方法灵敏度确实高于普通PCR。特异性试验表明,本方法能从变异株扩增出562 bp特异性片段,从经典株扩增出649 bp特异性片段,但从其他常见猪病毒中均不能扩增特异性条带,提示建立的方法特异性高,且通过电泳图能直观区分经典株和变异株,解决了李和平等建立的套式检测方法不能区分PRRSV经典毒株和变异株的问题,对临床病例快速鉴别诊断提供了有力的技术手段,具有较高的应用价值。
应用建立的鉴别诊断方法,对收集的48份临床样品进行检测,发现31份样品为阳性,阳性率为64.6%,提示陕西咸阳地区不同规模的猪场均存在PRRSV感染且感染率高,而且检测证实PRRSV变异株和经典株在猪群中均有流行,以变异株感染为主,在PRRS防控上要引起重视,这一结果与国内不同学者的研究结果[13-15]一致。在检测的样品中,包括组织样、血清和精液,本方法从这3种样品中均成功扩增出特异性目的条带,提示对PRRSV的检测样品选择比较宽泛,尤其是血清来源较易,不需剖检采集组织,为流行病学调查提供了便利手段。精液中检测出PRRSV的存在,提示公猪精液带毒并可能通过人工授精传播,需要引起高度重视。
[参考文献]
[1]Kim W I,Kim J J,Cha S H,et al.Significance of genetic variation of PRRSV ORFS in virus neutralization and molecular determinants corresponding to cross neutralization among PRRS viruses [J].Veterinary Microbiology,2013,162(1):10-22.
[2]Choi E J,Lee C H,Hyun B H,et al.A survey of porcine reproductive and respiratory syndrome among wild boar populations in Korea [J].J Vet Sci,2012,13(4):377-383.
[3]Keffaber K K.Reproductive failure of unknown etiology [J].Am Assoc Swine Pract Newsl,1989(1):1-9 .
[4]郭宝清,陈章水,刘文兴,等.从疑似PRRS流产胎儿分离PRR-SV的研究 [J].中国畜禽传染病,1996(2):1-4.
Guo B Q,Chen Z S,Liu W X,et al.Isolation and identification of porcine reproductory and resbiratory syndrome (PRRS)virus from aborted fetuses suspected of PRRS [J].China Animal Infectious Diseases,1996(2):1-4.(in Chinese)
[5]Zhou I,Yang H C.Porcine reproductive and respiratory syndrome in China [J].Virus Res,2010,154:31-37.
[6]Han J,Rutherford M S,Faaberg K S.The porcine reproductive and respiratory syndrome virus nsp2 cysteine protease domain possesses both trans and cis-cleavage activities [J].J Virol,2009(18):9449-9463.
[7]程亮亮,占松鹤,朱良强.猪繁殖与呼吸综合征诊断技术研究进展 [J].动物医学进展,2013,34(3):113-117.
Cheng L L,Zhan S H,Zhu L Q.The research progress of diagnostic techniques in porcine reproductive and respiratory syndrome [J].Progress in Veterinary Medicine,2013,34(3):113-117.(in Chinese)
[8]卢晓艳,周永辉,李伟娟,等.猪繁殖与呼吸综合征病毒河南分离株NSP2和GP5基因遗传变异分析 [J].华北农学报,2012,27(2):100-104.
Lu X Y,Zhou Y H,Li W J,et al.Analysis ofNSP2 andGP5 gene variation of porcine reproductive and respiratory virus strain isolated in Henan province [J].Acta Agriculturae Boreali-Sinica,2012,27(2):100-104.(in Chinese)
[9]童光志,周艳君,郝晓芳,等.高致病性猪繁殖与呼吸综合征病毒的分离鉴定及其分子流行病学分析 [J].中国预防兽医学报,2007(5):323-326.
Tong G Z,Zhou Y J,Hao X F,et al.Identification and molecular epidemioloqy of the very virulent porcine reproductive and respiratory syndrome viruses emerged in China [J].Chinese Journal of Preventive Veterinary Medicine,2007(5):323-326.(in Chinese)
[10]周丹,郭万柱,漆信桥,等.PRRSV普通株与变异株鉴别诊断RT-PCR方法的建立与临床应用 [J].中国兽医学报,2012(3):367-370.
Zhou D, Guo W Z, Qi X Q, et al. Establishment of differential diagnostic method for normal and mutant PRRSV strains by RT-PCR [J].Chin J Vet Sci,2012(3):367-370.(in Chinese)
[11]李和平,吴发兴,李晓成,等.猪繁殖与呼吸综合征的套式PCR方法建立及应用 [J].华北农学报,2009,24(1):115-118.
Li H P,Wu F X,Li X C,et al.Establishment and application of the PRRSV nest PCR methods [J].Acta Agriculturae Boreali-Sinica,2009,24(1):115-118.(in Chinese)
[12]Reicks D L,Munoz-Zanzi C,Mengeling W,et al.Detection of porcine reproductive and respiratory syndrome virus in semen and serum of boars during the first six days after inoculation [J].J Swine Health Product,2006,14(1):35.
[13]安春霞,闫若潜,吴志明,等.高致病性与低致病性猪繁殖与呼吸综合征病毒二重RT-PCR检测方法的建立及应用 [J].中国畜牧兽医,2013,40(5):66-70.
An C X,Yan R Q,Wu Z M,et al.Establishment and application of duplex RT-PCR assay for detection of highly and lowly pathogenic porcine reproductive and respiratory syndrome virus [J].China Animal Husbandry and Veterinary,2013,40(5):66-70.(in Chinese)
[14]邹敏,吴发兴,王福军,等.2007-2009年部分猪群PRRSVORF5和NSP2基因的克隆与序列分析 [J].西北农林科技大学学报:自然科学版,2010,38(10):7-14.
Zou M,Wu F X,Wang F J, et al.Cloning and sequence analysis ofORF5 andNSP2 genes of porcine reproductive and respiratory syndrome virus from some pig farms in 2007-2009 [J].Journal of Northwest A&F University:Nat Sci Ed,2010,38(10):7-14.(in Chinese)
[15]张冲,武小椿,王波,等.野猪源与家猪源PRRSV陕西分离株全基因测序与遗传进化分析 [J].西北农林科技大学学报:自然科学版,2011,39(2):9-16.
Zhang C,Wu X C,Wang B,et al.Genomic sequencing and phylogenetic analysis of PRRSV isolated from domestic pig and wild boar in Shaanxi Province [J].Journal of Northwest A&F University:Nat Sci Ed,2011,39(2):9-16.(in Chinese)
Establishment of a RT-nPCR diagnostic method for PRRSV classical and variant strains
ZHU Xiao-fu,WU Xu-jin
(AnimalEpidemicDiseaseDiagnosticLaboratoryofMolecularBiology,InstituteofAnimalHusbandryandVeterinaryMedicine,XianyangVocationalTechnicalCollege,Xianyang,Shaanxi712000,China)
Abstract:【Objective】 This study established a RT-nPCR diagnostic method to distinguish classical and variants strains of porcine reproductive and respiratory virus (PRRSV) quickly from tissue and serum.【Method】 Based on PRRSV gene sequences in GenBank,two pairs of primers were designed and synthesized and a RT-nPCR diagnostic method was established to distinguish classical and variants strains of PRRSV through sensitive test,specificity test,and detection of tissue and serum samples.【Result】 The established method had the detection limit of 1.3×10-7pg/μL and it only amplified target band from PRRSV.A total of 649 bp bands were amplified from classic strain CH-1R and 562 bp bands were amplified from variant strain SXXY/2013.These bands were clearly distinguished after electrophoresis,while no bands were amplified from other reference strains.31 out of the 48 suspected tissues and serum were detected positive.【Conclusion】 A highly sensitive and specific RT-nPCR method was established and it can directly differentiate PRRSV classic strains and variant strains from diseased tissues and serum.
Key words:porcine reproductive and respiratory syndrome;classical strains;variant strain;diagnostic methods
[文章编号]1671-9387(2016)01-0025-06
[中图分类号]S852.65+1
[文献标志码]A
[作者简介]朱小甫(1977-),男,陕西眉县人,执业兽医师,硕士,主要从事动物疫病分子病原学与免疫学研究。
[基金项目]咸阳市科学技术研究发展计划项目(2014K02-21);咸阳职业技术学院重点科研项目(2013KYA01)
[收稿日期]2014-04-25
DOI:网络出版时间:2015-12-0214:2510.13207/j.cnki.jnwafu.2016.01.005
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20151202.1425.010.html
E-mail:zhuxiaofu2004@aliyun.com

