不同分集方法对牛肉嫩度高光谱检测模型的比较
朱荣光,段宏伟,王龙,姚雪东,许程剑
1(石河子大学 机械电气工程学院,新疆 石河子,832003) 2(石河子大学 食品学院,新疆 石河子,832003)
不同分集方法对牛肉嫩度高光谱检测模型的比较
朱荣光1*,段宏伟1,王龙1,姚雪东1,许程剑2
1(石河子大学 机械电气工程学院,新疆 石河子,832003) 2(石河子大学 食品学院,新疆 石河子,832003)
摘要选用牛肉嫩度作为研究对象,开展了4种不同样品集划分方法的选取对其高光谱模型的影响研究。首先选取了70个具有代表性的牛肉样品并提取其肌肉感兴趣区域(ROIs)的光谱,比较分析了浓度梯度法(C-G)、随机法(R-S)、Kennard-Stone(K-S)和光谱-理化值共生矩阵法(SPXY)获取的校正集建立的牛肉嫩度PCR和PLSR模型效果。结果表明:在PCR和PLSR中,SPXY均为最适的样品分集方法,并且4种样品集划分方法下的PLSR模型效果均较优。最优模型SPXY-PLSR校正集的相关系数(Rcal)和均方根误差(RMSEC)分别为0.94和0.48,预测集的相关系数(Rp)和均方根误差(RMSEP)分别为0.93和0.63。研究表明SPXY方法结合高光谱PLSR模型能够实现牛肉嫩度的快速无损检测。
关键词高光谱图像(HSI);牛肉嫩度;分集方法;SPXY
牛肉及其制品是我国主要的肉类消费品[1],嫩度常被列为肉品品质优劣的重要检测指标[2],直接影响着肉的商品价值以及消费者的满意度,因此有必要基于嫩度对牛肉品质进行分级评价。在进行牛肉嫩度检测时,传统感官评定结果误差较大,理化检测又对肉品有损坏,操作过程繁琐。高光谱图像技术作为一种有效的检测方法,已在牛肉质量检测中有相关的研究[3-9],为快速无损准确检测牛肉嫩度提供了可能。
在利用高光谱图像技术进行无损检测时,模型效果验证方法一般有内部验证、外部验证和独立验证,而分集方法的选择对模型验证结果有一定的影响。国内报道[10-13]相关的分集方法主要有:浓度梯度法(C-G)、随机法(R-S)、kennard-stone(K-S)、光谱-理化值共生矩阵法(SPXY)。其中C-G将样本按照化学测量值x由小至大排序,从中按序选取一定数量的样本进行建模,R-S选取样本时完全是随意无规律的或者只遵循简单的规律[14],K-S只选取光谱值y差异大的样本作为模型校正集。而SPXY[15]是基于K-S法发展而来的,在计算样本间距离时将光谱信息x和化学测量值y同时考虑在内,并且样本在x和y空间具有相同的权重。当样品集划分时,其化学值信息或光谱信息所占据的权重不同,高光谱模型效果也不尽相同,因此可以通过比较分析不同牛肉嫩度的高光谱检测模型结果以选取最为合理的样品分集方法。
为快速准确检测牛肉嫩度,首先选取了70个具有代表性的牛肉样本并提取样品肌肉部分感兴趣区域(ROIs)的光谱。采用C-G、R-S、K-S和SPXY分别获取对应的样品校正集和预测集并对其进行了光谱预处理,对比分析了4种分集方法下牛肉嫩度的PCR和PLSR模型效果。根据模型效果确定最合理的分集方法和最优的建模方法。
1材料与方法
1.1牛肉试验样本制备
试验样本选择牛肉品种为新疆褐牛,购置于石河子西部牧业零售店。根据实验要求,取样过程中严格遵照中华人民共和国《肉与肉制品的取样标准GB/T 9695—2008》的规定进行。取8只牛后腿部距离股骨上端止点5 cm位置的肌肉块,用无菌刀切割成基本尺寸为6 cm×4 cm×3cm的小块,保证所切取的样品厚度大体一致。取样后采用保鲜袋密封包装,冷藏于4 ℃恒温箱中,保存1~15 d。整个过程中保证操作规范、取样工具清洁。
1.2牛肉高光谱图像采集系统
高光谱图像采集系统如图1所示。主要包括:光谱仪(ImSpector V10E-QE, Finland),150 W光纤卤素灯(SCHOTT DCR Ⅲ),线阵CCD摄像机,脉冲输送装置(Zolix, SC300-1A, Beijing),暗箱,图像采集卡和计算机等。整套系统置于自制的暗箱之中,以消除外界干扰对于图像采集的影响。
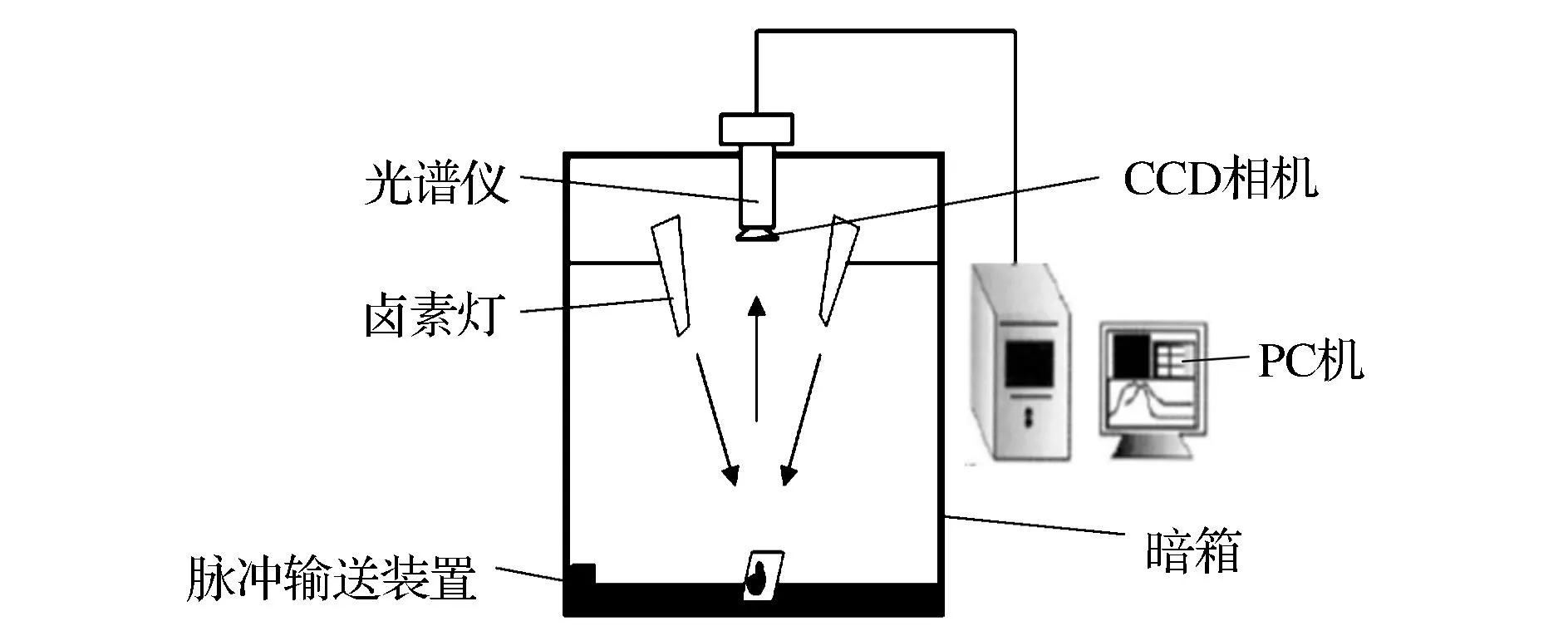
图1 高光谱图像采集系统Fig.1 Hyperspectral imaging acquisition system
为准确获取样品高清图像,在进行牛肉的高光谱漫反射检测时,需要对高光谱仪器进行参数设置,曝光时间设为16 ms,物距为33.5 cm,图像采集速度为1.35 mm/s,样本表面光照强度为2 910 lx。为去除高光谱图像系统工作过程中暗电流等噪声,图像采集前需进行黑白校正[16],公式为R=(I-B)/(W-B),R为校正后图像,I为原始图像,W和B分别为全白、黑校正图像。
1.3牛肉嫩度的剪切力测定
牛肉样品嫩度剪切力的测定步骤严格遵循我国农业部行业标准《NY/T1180—2006肉嫩度的测定剪切力测定法》进行。测量前,使用水浴将肉中心温度控制在70~72 ℃,保证剪切试验条件的统一。用C-LM3B型数显式肌肉嫩度仪配备的取样器进行取样,观察测定结果并得到最大剪切力值,每个样品记录5组数据,取5组数据的平均值作为该样品的嫩度值。
1.4数据处理
高光谱图像数据处理用 ENVI 4.7和TQ 8.01软件。对70个具有代表性的牛肉样本进行肌肉部分感兴趣区域(ROIs)光谱的提取,分别采用C-G、R-S、K-S和SPXY获取对应的校正集和预测集,比较了4种不同分集方法的牛肉嫩度PLSR和PCR模型效果。确定最合理的分集方法及对应的模型。模型效果评价标准[17]包括建模效果和预测效果,若RMSEC越小,表明模型的建模效果越好,RMSEP越小,表明模型的预测效果越好。Rcal和Rp越大,预测值与实际测量值的相关性越好,且RMSEC和RMSEP差值越小,表明模型稳健性较好。
2结果与讨论
2.1牛肉嫩度结果统计分析
嫩度根据剪切力值可分为“老”、“嫩”,传统评价方法[18]中剪切力值≤6 kg的样本为嫩牛肉,剪切力值>6 kg的样本为老牛肉。试验采集的“嫩”牛肉样品数量为49个,“老”牛肉样品数量为21个,其剪切力统计结果如表1所示。包括校正集样品数、预测集样品数、样品最大值、最小值、平均值和标准偏差,牛肉嫩度以均值为中心,大致呈高中低正态分布。

表1 牛肉样品剪切力统计结果
2.2牛肉样品的光谱提取
首先采用波段运算减法和掩膜去除图像背景及阴影部分,其次采用波段运算加法、二值化和掩膜去除亮点、脂肪和结缔组织,从而提取高光谱图像中纯肌肉部分作为感兴趣区域(ROIs),并对该区域进行全波段400~1 000 nm光谱数据的提取。由于所采集到的光谱数据存在尖峰等噪声,因此选取480~860 nm光谱作为高光谱图像所采集到的牛肉样品代表性原始光谱。由图2可知,在596、760 nm附近有2处明显的吸收峰。牛肉在储藏过程中,其肉质的老、嫩与肉品的含水量以及蛋白质的分解等密切相关,且760 nm附近为水的吸收峰,596 nm附近为氧合血红蛋白的吸收峰[19],表明所提取的光谱信息与牛肉嫩度具有相关性。

图2 牛肉样品的代表性原始光谱Fig.2 Representative raw spectra of beef samples
2.3不同预处理方法的选择
由于所采集到的光谱数据存在较多的噪声,为了提高后续建模精度,需选用合适的预处理方法对原始光谱数据进行预处理。常用的光谱预处理方法有:多元散射校正(MSC)、变量标准化(SNV)、1阶导数(1D)、2阶导数(2D)、Savitzky-Golay(S-G)平滑和中心化处理(mean-center)等。表2为SPXY获取的样品集光谱经不同预处理后建立的PLSR模型结果,通过比较Rcal、Rp、RMSEC 和 RMSEP,发现采用2阶导数进行光谱预处理时,其模型效果明显提高,但当将2阶导数与MSC相结合时的模型效果又有所下降,因此选取的最优预处理方法为2D、S-G平滑(5点)和 mean-center相结合的方法。

表2 最优预处理方法的选取
2.4不同分集方法下牛肉嫩度模型建立与评价
选用的样品分集方法分别为浓度梯度法(C-G)、随机法(R-S)、kennard-stone(K-S)和光谱-理化值共生矩阵法(SPXY),样本划分后建立的PCR和PLSR模型结果如表3所示。

表3 4种分集方法的PCR和PLSR模型结果
当采用C-G、R-S、K-S和SPXY 4种分集方法,其分别获取的牛肉嫩度样品校正集建立的PCR模型,在选取的PCs分别为10、12、13和14时,对应预测集的RMSEP均远大于与该4种分集方法相对应的PLSR模型结果,由此得出,在4种分集方法中,PLSR的模型效果均优于PCR模型。
当建模方法为PCR时,若样本划分方法为R-S,其最优模型的RMSEP为0.90,大于C-G的模型结果。此时C-G的最优模型选取的PCs为10,对应的RMSEP为0.88,大于K-S的模型结果,其成因可能是C-G模型的PCs较少导致的模型欠拟合。该情况下K-S的模型最优PCs为13,RMSEP为0.84,亦远大于SPXY的建模结果。可见,PCR模型中,样品分集方法的优次顺序为SPXY>K-S>C-G>R-S。
当建模方法为PLSR时,R-S方法获取的样品集建立的最佳模型在选取的LVs为4时,其RMSEP为0.89,远大于C-G的建模结果,其原因可能是R-S方法自身的随机性导致的模型效果较差。当前C-G获取的样品集所建立PLSR最优模型的RMSEP为0.75,大于K-S方法下的模型结果,二者模型效果相近。此时K-S的模型选取了5个LVs,对应的RMSEP为0.73,远大于SPXY的模型结果。可知,PLSR模型中,SPXY分集方法最为合适,其顺序依次为SPXY>K-S>C-G>R-S。
因此在PCR和PLSR2种建模方法中,SPXY均为最适样品分集方法,并且与SPXY对应的PLSR模型效果最优。原因可能是SPXY将光谱信息和化学测量值信息考虑在内时,同时选取了差异大的样本作为模型校正集,确保了所选校正集样本能够均匀覆盖整个样本集试验区域,提高了模型预测结果。最优模型校正集的Rcal和RMSEC分别为0.94和0.48,预测集的Rp和RMSEP分别为0.93和0.63,如图3(a)、图3(b)所示。

图3 SPXY对应的PLSR模型效果Fig.3 PLSR model effects corresponding to SPXY
3结论
(1)对于PCR和PLSR 2种建模方法,最合理的样品分集方法均为SPXY,其优次顺序均为SPXY>K-S>C-G>R-S。分析其原因可能在于样品集选取时,光谱信息和化学测量值信息均需考虑在内,并且光谱信息所占据的权重稍高于化学测量值。
(2)对于C-G、R-S、K-S和SPXY 4种分集方法,其PLSR模型效果均较优,并且与SPXY相对应的PLSR模型效果最优,其校正集的相关系数(Rcal)和均方根误差分别为0.94和0.48,预测集的相关系数(Rp)和均方根误差分别为0.93和0.63。
参考文献
[1]叶强,贾彩荷. 湖岭牛肉干加工技术研究[J]. 肉类研究,2009,23(9): 34-36.
[2]周光宏, 李春保, 徐幸莲. 肉类食用品质评价方法研究进展[J]. 中国科技论文在线, 2007, 2(2): 75-82.
[3]宋育霖,彭彦昆,郭辉,等. 光学扩散特征的生鲜肉细菌总数的无损检测方法[J]. 光谱学与光谱分析, 2014, 34(3): 741-745.
[4]吴建虎,彭彦昆,陈菁菁,等. 基于高光谱散射特征的牛肉品质参数的预测研究[J]. 光谱学与光谱分析,2010, 30(7): 1 815-1 819.
[5]朱荣光,姚雪东,高广娣,等. 不同储存时间和取样部位牛肉颜色的高光谱图像检测[J]. 农业机械学报,2013, 44(7): 165-169.
[6]赵杰文,翟剑妹,刘木华,等. 牛肉嫩度的近红外光谱法检测技术研究[J]. 光谱学与光谱分析, 2006, 26(4): 640-642.
[7]PENG Y, ZHANG J, WANG W, et al. Potential prediction of the microbial spoilage of beef using spatially resolved hyperspectral scattering profiles[J]. Journal of food engineering, 2011, 102(2): 163-169.
[8]WU J,PENG Y, LI Y, et al. Prediction of beef quality attributes using VIS/NIR hyperspectral scattering imaging technique[J]. Journal of Food Engineering, 2012, 109(2): 267-273.
[9]ElMASRY G, SUN D W, ALLEN P. Non-destructive determination of water-holding capacity in fresh beef by using NIR hyperspectral imaging[J]. Food Research International, 2011, 44(9): 2 624-2 633.
[10]刘伟,赵众,袁洪福,等. 光谱多元分析校正集和验证集样本分布优选方法研究[J]. 光谱学与光谱分析, 2014, 34(4): 947-951.
[11]展晓日,朱向荣,史新元,等. SPXY样本划分法及蒙特卡罗交叉验证结合近红外光谱用于橘叶中橙皮苷的含量测定[J]. 光谱学与光谱分析,2009, 29(4): 964-968.
[12]詹雪艳,赵娜,林兆洲,等. 校正集选择方法对于积雪草总苷中积雪草苷NIR定量模型的影响[J]. 光谱学与光谱分析, 2014, 34(12): 3 267-3 272.
[13]朱向荣, 单杨, 李高阳, 等. 近红外光谱法快速测定液态奶中蛋白质和脂肪含量[J]. 食品科学, 2011, 32(12): 191-195.
[14]李运锋,袁景淇,薛耀锋. 发酵过程中神经网络训练样本的选取[J]. 化工自动化及仪表, 2004, 31(6): 21-24.[15]GALVAO R K H, ARAUJO M C U, JOSE G E, et al. A method for calibration and validation subset partitioning[J]. Talanta, 2005, 67(4): 736-740.
[16]WANG W, LI C,TOLLNER E W, et al. Journal of Food Engineering, 2012, 109(1): 38-48.
[17]KAMRUZZAMANA M, ELMASRY G, SUN D W, et al. Prediction of some quality attributes of lamb meat using near-infrared hyperspectral imaging and multivariate analysis[J]. Anal Chim Acta, 2012, 54(714): 57-67.
[18]PARK B, CHEN Y R,HRUSCHKA W R, et al. Near-infrared reflectance analysis for predicting beef longissimus tenderness[J]. Journal of Animal Science-Menasha Then Albany Then Champaign Illinois, 1998, 76: 2 115-2 120.
[19]浦瑞良,宫鹏. 高光谱遥感及其应用[M].北京:高等教育出版社,2000.
Comparative research on hyperspectral detection model of beef tenderness with different sample set partitioning methods
ZHU Rong-guang1*, DUAN Hong-wei1,WANG Long1, YAO Xue-dong1, XU Cheng-jian2
1(College of Mechanical and Electrical Engineering,Shihezi University, Shihezi 832003, China) 2(2.Food College,Shihezi University, Shihezi 832003, China)
ABSTRACTSelection of sample set partitioning methods has an influence on the prediction results of hyperspectral quantitative analysis model. In this paper, beef tenderness was chosen as the research object, and study on the effects of 4 different sample set partitioning methods on hyperspectral model was carried out. Initially, 70 representative beef samples were adopted and spectra of muscle regions of interest (ROIs) were extracted. Subsequently, PCR and PLSR model effects of beef tenderness were separately compared and analyzed, whose models were established by the calibration set separately obtained from concentration gradient method (C-G), Random Sampling (R-S), Kennard stone (K-S) and sample set partitioning based on joint X-Y distance (SPXY). The results indicated SPXY was the best sample set partitioning method in PCR and PLSR models, the PLSR model effects under 4 sample set partitioning methods were all better than that of PCR. And SPXY-PLSR model effect was optimal, which correlation coefficient(Rcal) and root mean square error(RMSEC) of calibration set were 0.94 and 0.48 respectively, correlation coefficient(Rp) and root mean square error(RMSEP) of prediction set were 0.93 and 0.63 respectively. The research shows that the SPXY method combined with hyperspectral PLSR model can realize the rapid and nondestructive detection of beef tenderness.
Key wordshyperspectral; beef tenderness; sample set partitioning methods; SPXY
收稿日期:2015-08-14,改回日期:2015-11-05
基金项目:国家自然科学基金资助(No.31460418);高等学校博士学科点专项科研基金(No.20136518120004)资助
DOI:10.13995/j.cnki.11-1802/ts.201604034
第一作者:博士,副教授(本文通讯作者,E-mail: rgzh_jd@163.com)。

