南方型紫花苜蓿根系盐胁迫应答转录因子鉴定与分析
马 进,郑 钢,裴翠明,张振亚(浙江农林大学风景园林与建筑学院,浙江临安311300)
南方型紫花苜蓿根系盐胁迫应答转录因子鉴定与分析
马进,郑钢,裴翠明,张振亚
(浙江农林大学风景园林与建筑学院,浙江临安311300)
摘要:转录因子可以调节众多下游基因的表达,在植物抗逆境中起重要的调节作用。为了解析转录因子在南方型紫花苜蓿适应盐胁迫环境的分子机制,以南方型紫花苜蓿Medicago sativa‘Millennium’为材料,以正常培养(WT_ck1)和氯化钠(盐)胁迫(WT_N1)条件下的2个样品根系进行转录组分析,鉴定紫花苜蓿根系盐胁迫应答转录因子基因。同时,随机挑选4个转录因子差异表达基因进行实时荧光定量qRT-PCR(3次重复),验证转录组测序技术(RNA-Seq)结果的可靠性。结果表明:紫花苜蓿根系在250 mmol·L(-1)氯化钠胁迫下72 h,共检测到31 907个基因表达量发生了改变,表达量差异达到2倍以上的基因共2 758个。其中,隶属于38个转录因子家族199个转录因子在盐胁迫下差异表达,上调表达104个,下调表达95个。在各转录因子家族中,盐胁迫应答基因数量最多的是MYB基因家族,其后分别是AP2-EREBP,bHLH,WRKY,NAC和GRAS基因家族,这暗示了紫花苜蓿根系对盐胁迫响应可能是多种转录因子家族共同参与的应答过程。qRT-PCR分析表明:4个随机选择的基因在胁迫前后的表达特点与表达谱测序结果一致。此外,MsERF-2b,MsbHLH,MsbZIP,MsGRAS,MsNAC,MsMGT-3a和MsWRKY等转录因子被选为与盐胁迫应答相关的候选转录因子。该研究结果为阐明植物对盐胁迫的应答机制提供了新的线索。图3表3参36
关键词:植物育种学;南方型紫花苜蓿;转录因子;根转录;盐胁迫
Key words: plant breeding; southern type alfalfa; transcription factor; root transcriptome; salt stress
土壤盐化是全球面临的最严峻的环境问题之一[1]。植物盐胁迫实际上是一种综合胁迫,包括生理性干旱胁迫、渗透胁迫以及离子毒害胁迫,涉及到信号传递、多基因协同控制、蛋白质翻译与表达等一系列复杂的分子调控网络。发掘和鉴定与植物盐耐性相关重要基因,有助于了解植物盐分子机制,为培育耐盐植物新品种提供理论依据。紫花苜蓿Medicago sativa主要分布在温带地区,除了作为牧草外,也是一种很好的水土保持绿化植物。近年来,国外高秋眠级紫花苜蓿品种引进,率先解决了在南方的种植难题,但紫花苜蓿耐盐能力有限,限制了在南方盐碱地大面积种植。因此,发掘参与其盐逆境响应中耐盐功能基因,并分析其耐盐分子机制,有助于利用基因工程技术创制适合南方盐碱地种植紫花苜蓿新品种。植物受盐胁迫后,许多基因的表达量会发生明显的变化,其中部分差异表达基因在耐盐应激调控过程中发挥着重要作用。LUO等[2]利用逆转录聚合酶链式反应(RT-PCR)方法,从紫花苜蓿中分离到受胁迫诱导表达的解旋酶基因MH1,该基因可提高植物活性氧(ROS)的清除及渗透调节能力。MA等[3]从紫花苜蓿盐胁迫中成功克隆1个MsGME基因,该基因对抗坏血酸合成起着重要的作用。郭鹏等[4]从紫花苜蓿盐胁迫中成功克隆出1个类受体蛋白激酶MsSIK1基因,过量表达该基因可提高拟南芥Arabidopsis thaliana的抗盐性。李明娜等[5]从紫花苜蓿成功克隆MsHB2基因,该基因可能通过ABA信号途径参与紫花苜蓿盐胁迫应激调控。蒺藜苜蓿Medicago truncatula作为豆科Leguminosae模式植物,与苜蓿亲缘关系很近,与四倍体苜蓿基因组具有很高的同源性。ZAHAF等[6]对蒺藜苜蓿盐胁迫下进行全基因组分析,为开展紫花苜蓿耐盐分子机制研究提供了重要信息,但紫花苜蓿为同源四倍体、异花授粉植物,其基因组非常复杂,仍然难以揭示紫花苜蓿耐盐的分子机制。转录因子是转录调控中的核心功能蛋白。植物胁迫反应过程中,众多的转录因子承上启下,调控一系列基因的表达,在植物逆境应答网络过程中发挥着不可或缺的关键性作用。鉴于转录因子在转录调控中的重要意义,在逆境胁迫应答中,转录因子及其下游调控靶基因的识别,已成为后基因组时代研究的热点之一。目前,采用高通量技术从转录组水平上鉴定和分析紫花苜蓿盐胁迫应答转录因子研究较少,本研究利用转录组测序技术(RNA-Seq),对盐胁迫处理前后的南方型紫花苜蓿幼苗根系进行转录组测序,鉴定根系响应盐胁迫应答转录因子及表达特性,以期更深入地理解南方型紫花苜蓿对盐胁迫响应的分子机制,为进一步鉴定和克隆其重要的耐盐基因,提高南方型紫花苜蓿耐盐性状奠定基础。
1 材料与方法
1.1材料及处理
选择耐盐性相对较强的高秋眠级南方型紫花苜蓿Medicago sativa‘Millennium’为材料[7],将种子先用体积分数为75%乙醇消毒10 min,然后用灭菌水清洗3次,灭菌水中浸泡2 h于20℃生长箱内发芽。7 d后,挑选生长一致小苗转移至塑料盆装满基质[V(细沙)∶V(珍珠岩)=3∶1],每天浇霍格兰营养液。30 d后加入250 mmol·L-1氯化钠至营养液中处理,选取胁迫处理0 h和72 h的紫花苜蓿地下部根系组织,混合样于液氮中保存备用。
1.2RNA的提取及检测
利用TRNzol Reagent试剂盒分别提取处理与对照组幼根RNA,提取过程按试剂盒说明进行。生物学重复3次·处理-1的RNA样品混合为50 μL。所提取的RNA经电泳检测合格后送至深圳华大基因科技有限公司进一步确认质量后,将同一样本所提取的幼根RNA进行高通量测序。
1.3Illumina测序
将盐处理组与对照组总核糖核酸(RNA)由华大基因科技有限公司完成文库构建与测序,将构建好的文库进行其质量和产量检测。对质量检测合格后的文库采用IlluminaHiSeqTM 2000进行测序。将经碱基识别(base calling)测序产生的原始图像转化为原始序列数据(raw data),继而转换为有效数据(clean data)。采用短reads比对软件SOAPaligner/SOAP2将有效数据分别与参考基因组蒺藜苜蓿进行比对。
1.4差异基因的选择
最后,大学的教学模式、课程要求、体系难度都远远不同于高中生已经非常熟悉的状况。而大学一年级,可能与学生在大学的整个基调是息息相关的。
测序后使用RPKM(reads per kilo bases per million reads)法计算基因表达量,根据基因表达量(RP-KM值)计算其在处理与对照中的差异表达倍数。差异表达倍数大于2倍以上(含2倍)的基因认定为差异表达基因。
1.5荧光定量RT-PCR
以分别提取的处理组与对照组根系RNA为材料,采用TaKaRa公司的PrimeScript RT reagent Kit With cDNA Eraser试剂盒反转录合成cDNA,作为实时荧光聚合酶链式反应(PCR)反应的模板。根据测序的基因测序结果随机挑取4个盐胁迫转录因子基因,用Primer Premier 5.0软件设计荧光定量PCR引物(表1)。以苜蓿GAPDH(GenBank登录号为MTR_4g131180)为内参,引物为5′-GTGGTGCCAAGAAGGTTGTTAT-3′和5′-CTGGGAATGATGTTGAAGGAAG-3′。各处理均做3次重复计算基因的相对表达量。
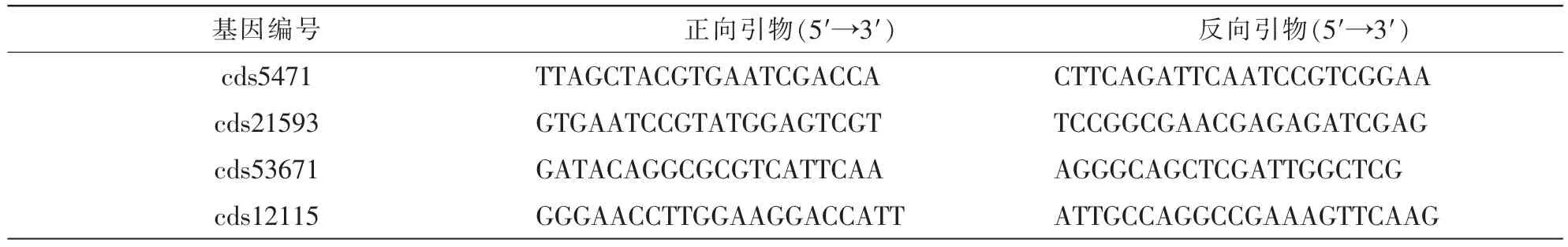
表1 验证DGE数据准确性的qRT-PCR引物Table 1 Primers to detect the accuracy of DGE date by qRT-PCR
2 结果与分析
2.1测序质量分析
对测得的reads分析表明:对照样本(WT_ck1)和盐胁迫处理样本(WT_N1)raw reads去杂后获得的clean reads比例接近,说明cDNA文库构建和测序质量较高。由于受到遗传背景的差异和注释信息量的限制,对照样本clean reads中近52.25%可被参考基因组注释,47.75%的clean reads未能注释到对应的基因。盐胁迫处理样本(WT_N1)样本的reads注释情况同对照样本基本上相近(表2)。

表2 DGE样品测序数据统计结果Table 2 Statistics of upgraded version of the DEG
2.2紫花苜蓿根系盐胁迫应答转录因子分析
将盐处理组与对照组总RNA,由华大基因科技有限公司完成文库构建与测序。以差异检验发现错误率FDR(false discovery rate,FDR)值≤0.001且差异倍数不低于2倍为差异表达基因(DEGs, differentially expressed genes)的选择标准,紫花苜蓿根系在250 mmol·L-1氯化钠胁迫下72 h,共检测到31 907个基因表达量发生了改变,对照样本(WT_ck1)和盐胁迫处理样本(WT_N1)之间有2 758个基因表现为差异表达,其中1 338个DEGs在盐胁迫响应中表达上调,1 420个DEGs表达下调。此外,南方型紫花苜蓿根系盐响应下,共有隶属于38转录因子家族199个转录因子差异表达,占总差异表达基因的7.72%,其中上调表达104个,下调表达95个。
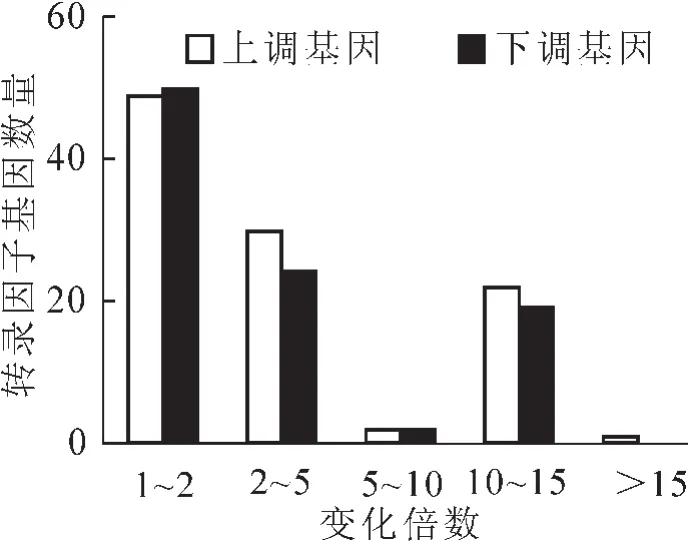
图1 紫花苜蓿根响应盐胁迫转录因子差异基因倍数变化Figure 1 Fold change of differentially expressed genes of transcription factor in the root of alfalfa under control and salt stress
从图2可以看出:紫花苜蓿根系响应盐胁迫下有38个转录因子家族的差异表达。MYB转录因子家族上调和下调数量最多。Alfin-like,C2C2-Dof,C2H2和SBP转录因子家族基因全部上调,DBP,HB和Tify转录因子家族基因全部下调。其他的转录因子家族基因如AP2-EREBP,bHLH,NAC,MADS,Trihelix 和WRKY等转录家族成员基因既有转录水平上调的,也有转录水平下调的,这也表明了转录家族基因在盐响应中调控方式的复杂性。

图2 紫花苜蓿根响应盐胁迫转录因子家族分布Figure 2 Distribution of transcription factor family in the root of alfalfa under control and salt stress
2.3紫花苜蓿根系响应盐胁迫诱导表达重要转录因子基因
转录因子在逆境胁迫下,可以激活或抑制下游基因的转录表达。目前已经证实bZIP,WRKY,AP2,NAC,bHLH,DERB,MYB和C2H2等转录因子家族的成员参与了植物对盐胁迫的应答反应。根据BLASTP结果对表达DEGs进行功能注释,发现多个盐胁迫标志性转录因子基因显著上调或下调表达(表3)。上调的基因如乙烯响应因子MsERF-RAP2(gi|357464931)和MsERF-2b(gi| 357442393),bHLH转录因子MsbHLH93(gi|357466829),NAC转录因子MsNAC(gi|657395215,gi| 657400593),WRKY转录因子MsWRKY(gi|657385964),MYB转录因子MsMYB(gi|657378081,gi| 357476493)及Trihelix转录因子MsMGT-3a(gi|657378410)等。下调的基因如DREB转录因子(gi| 357481745),bHLH转录因子MsbHLH(gi|657389916),C3H转录因子(gi|357446695),GRAS转录因子MsGRAS(gi|357454173,gi|657390120和gi|657400125),MYB转录因子MsMYB(gi|657404698,gi| 657387760)及Trihelix转录因子MsTFIIIB(gi|657378081)。
2.4差异表达基因的qRT-PCR荧光定量验证
为了验证测序差异表达基因数据的可靠性,随机挑选4个差异表达基因,其中2个上调基因cds5471和cds21593,2个下调基因cds53671和cds12115,以苜蓿GAPDH(GenBank登录号为MTR_4g131180)为内参,进行qRT-PCR验证。结果表明:虽然4个基因在盐胁迫处理后发生不同程度的表达(图3),但胁迫诱导表达的变化趋势基本一致,说明Illumina测序获得的转录组数据的可靠性。

图3 DEGs qRT-PCR验证Figure 3 Validation of DEGS data by qRT-PCR

表3 紫花苜蓿根系响应盐胁迫诱导表达重要转录因子Table 3 Salt-stress induced key transcription factors in the root of alfalfa
3 讨论
在干旱、盐碱等逆境胁迫下,植物可通过改变基因的表达,调控不同代谢和信号转导途径,从转录和翻译等不同水平上作出响应,而转录因子可在逆境胁迫下,激活或抑制下游基因的转录表达。目前,已经证实bZIP,WRKY,AP2/EREBP,C2H2,bHLH,DERB,MYB和NAC等转录因子家族的成员参与了植物对盐胁迫的应答反应[8]。本研究结果表明:紫花苜蓿根系在250 mmol·L-1氯化钠胁迫72 h时后,检测到表达量差异达到2倍以上的基因2 758个,在差异表达基因中,包含着38转录家族共199个转录因子基因差异表达,其中上调转录因子104个,下调转录因子95个。这也暗示着紫花苜蓿根系盐胁迫应答各类转录因子调控方式的复杂性。
AP2/EREBP是一个大的转录因子家族,包含EREBP和AP2等2个亚族[9]。研究证实,AP2/EREBP转录家族基因与植物逆境响应有关[10]。本研究发现,转录因子多数属于ERFs转录因子家族,ERFs类转录因子可激活抗氧化基因的表达,来减轻盐胁迫造成的氧化损伤[11]。如过表达GmERF7和GmERF6可提高大豆Glycine max抗盐性和抗旱性。但也有研究报道:过表达SlERF3可降低番茄Solanum lycopersicum对盐的耐受性[12]。本研究发现:28个AP2/EREBP基因参与紫花苜蓿根系响应盐胁迫应答过程,其中15个表达上调,13个表达下调。AP2/EREBP基因在盐胁迫响应下呈现上调和下调现象,表明其参与盐胁迫响应的调控方式不同。
NAC转录家族因子在植物发育、逆境应答和衰老和次生壁合成等众多的生物学过程中发挥着重要作用[13-14]。研究证实,NAC家族转录因子在逆境胁迫应答中存在着正负两种调控机制。过表达SNAC1能提高水稻Oryza sativa干旱、高盐的耐受性,且可以同OsPP18的启动子结合共同调控其表达[15];过表达AtNAC2可降低拟南芥转基因的耐盐性[16]。本研究发现,9个NAC家族转录因子参与盐胁迫应答过程,其中8个表达上调,1个表达下调。在9个NAC家族转录因子成员中,其中gi|657395215和gi| 657400593上调10倍以上,这可能是紫花苜蓿根部参与盐胁迫应答重要调节因子。
作为植物特有的WRKY家族转录调控因子,在植物抗逆过程中扮演着重要的角色。蒺藜苜蓿中已鉴定出28个成员[17]。现已证实WRKY家族转录因子在逆境胁迫应答中存在着正负2种调控机制。过表达GhWRKY17可提高棉花Gossypium hirsutum对干旱、高盐的耐受性[18],但过表达OsWRKY45-1可降低水稻对高盐的耐受性[19]。本研究发现:13个WRKY转录因子基因参与紫花苜蓿根系相应盐胁迫应答过程,其中10个表达下调,3个表达上调,这也暗示着WRKY转录因子参与植物的盐胁迫响应机制方式的复杂性。
碱性亮氨酸拉链(basic leucine zipper,bZIP)转录因子广泛分布于真核生物中,并参与调控植物对干旱及盐胁迫的抗性。现已证实bZIP家族转录因子在逆境胁迫应答中存在着正负2种调控机制。过表达OsbZIP23可明显提高转基因水稻植株对干旱、高盐的耐受性[20]。GmbZIP44,GmbZIP62和GmbZIP78可作为负调控因子参与大豆调控对盐胁迫响应[21]。李燕等[22]成功将紫花苜蓿MsZIP基因转到烟草Nicotiana tabacum中,为后期研究该基因在耐盐中的作用提供了材料。本研究检测到2个bZIP转录因子参与紫花苜蓿根系盐胁迫应答过程,其中1个bZIP转录因子基因下调明显(gi|657385763,差异倍数为-13.13),推测这个bZIP转录因子可能作为负调控因子参与紫花苜蓿盐胁迫应答过程。
C2H2型锌指蛋白植物中最重要的转录调节因子家族之一。如拟南芥有176个C2H2型锌指蛋白,水稻有189个C2H2型锌指蛋白成员。研究表明:C2H2型转录家族因子参与植物生长发育和非生物胁迫应答的多种细胞生化过程[23]。CHEN等[24]研究发现ZPF179在水稻体内对盐胁迫应答中发挥重要作用。本研究发现,1个C2H2转录因子基因上调(gi|657403499,差异倍数为2.33),推测这个C2H2转录因子可能参与紫花苜蓿盐胁迫应答过程。bHLH转录因子广泛存在于植物中,在应对非生物胁迫应答中起到重要的调控作用。目前已在蒺藜苜蓿中发现100多个bHLH转录家族成员[25]。研究发现:蒺藜苜蓿对盐胁迫逆境同bHLH转录因子有关系[26]。过表达OrbHLH001和bHLH92分别可增加水稻和拟南芥对高盐胁迫的耐受性[27-28]。本研究发现,17个bHLH转录因子参与紫花苜蓿根系响应盐胁迫应答过程,其中12个表达上调,5个表达下调。在差异表达bHLH转录因子中,gi|657395215和gi|657400593上调达到11倍以上,推测这2个bHLH转录因子可能是紫花苜蓿盐胁迫应答中重要潜在转录因子基因。
作为植物特有的GRAS蛋白家族转录调控因子,已在拟南芥和水稻基因组中分别发现GRAS家族的基因成员33个和57个[29]。研究表明:GRAS家族的基因成员在植物抗逆性中发挥了关键作用[30-31]。本研究中,发现9个GRAS家族基因成员,其中3个表达上调,6个表达下调,表明其该类基因可能同紫花苜蓿根系的盐胁迫响应逆境调节有关。作为转录因子家族Trihelix一个小家族,已经在拟南芥发现30个,水稻有31个。已有证据表明,小家族Trihelix转录因子在逆境胁迫响应中起着重要调控作用[32-34]。本研究也发现6个Trihelix转录因子差异表达。热激转录因子(HSF)对提高植物逆境耐受力方面起着重要调控作用[35-36]。本研究发现4个HSF转录因子全部上调表达,暗示了HSF家族的转录因子同盐胁迫应答可能有一定的关联性。
在复杂的植物逆境胁迫应答网络中,转录调控基因可以通过调控一系列与逆境相关的功能基因的表达,从而明显提高植物对逆境的抵抗能力。本研究利用RNA-Seq技术鉴定出199个转录因子参与南方型紫花苜蓿根系盐胁迫应答过程,但大多数转录因子在盐胁迫应答中发挥功能及其控制下游靶基因调控机制仍知之甚少,还需要进一步深入的研究,从而更深入揭示紫花苜蓿对盐胁迫的调控机制。
4 参考文献
[1]左照江,张汝民,高岩.盐胁迫下植物细胞离子流变化的研究进展[J].浙江农林大学学报,2014,31(5):805 - 811.ZUO Zhaojiang,ZHANG Rumin,GAO Yan.Advances in plant cell ion flux with salt stress: a review[J].J ZhejiangA & F Univ, 2014, 31(5): 805 - 811.
[2]LUO Yan, LIU Yubo, DONG Yuxiu, et al.Expression of a putative alfalfa helicase increases tolerance to abiotic stress in Arabidopsis by enhancing the capacities for ROS scavenging and osmotic adjustment[J].J Plant Physiol, 2009, 166(4): 385 - 394.
[3]MA Lichao, WANG Yanrong, LIU Wenxian, et al.Overexpression of an alfalfa GDP-mannose 3, 5-epimerase gene enhances acid, drought and salt tolerance in transgenic Arabidopsis by increasing ascorbate accumulation[J].Biotechnol Let, 2014, 36(11): 2331 - 2341.
[4]郭鹏,邢鑫,张万筠,等.紫花苜蓿盐诱导类受体蛋白激酶基因MsSIK1的克隆及功能分析[J].中国农业科学,2014,47(23):4573 - 4581.GUO Peng,XING Xing,ZHANG Wanjun,et al.Cloning and function analysis of a salt-stress-induced receptor like protein kinase gene MsSIK1 from alfalfa[J].Sci Agric Sin,2014,47(23): 4573 - 4581.
[5]李明娜,龙瑞才,杨青川,等.紫花苜蓿盐诱导HD-Zip类转录因子MsHB2的克隆及功能分析[J].中国农业科学,2014,47(4):622 - 632.LI Mingna,LONG Ruicai,YANG Qingchuan,et al.Cloning and function analysis of a salt-stress-induced HD-Zip transcription factor MsHB2 from alfalfa[J].Sci Agric Sin, 2014,47(4): 622 - 632.
[6]ZAHAF O, BLANCHET S, de ZÉLICOURT A, et al.Comparative transcriptomic analysis of salt adaptation in roots of contrasting Medicago truncateula genotypes[J].Mol Plant, 2012,5(5): 1068 - 1081.
[7]马进,郑钢,蔡建国.南方型紫花苜蓿基因型在组织培养条件下对NaCl的耐性[J].浙江农业学报,2011,23 (4):782 - 786.MA Jin, ZHENG Gang, CAI Jianguo.NaCl tolerance of different genotypes of southern type of Medicago sativa under tissue culture[J].Acta Agric Zhejiang, 2011,23(4): 782 - 786.
[8]MILLER G A D, SUZUKI N, CIFTCI-YILMAZ S, et al.Reactive oxygen species homeostasis and signalling during drought and salinity stresses[J].Plant Cell Environ, 2010, 33(4): 453 - 467.
[9]王庆灵,刘文鑫,赵嘉平.山海关杨PdERF-18转录因子的表达特征分析[J].浙江农林大学学报,2014,31 (5):716 - 723.WANG Qingling,LIU Wenxin,ZHAO Jiaping.Expression patterns for a PdERF-18 response to different stresses in Populus deltoides‘Shanhaiguan’[J].J Zhejiang A & F Univ, 2014,31(5): 716 - 723.
[10]HUSSAIN S S,KAYANI M A,AMJAD M.Transcription factors as tools to engineer enhanced drought tolerance in plants[J].Biotechnol Prog, 2011, 27(2): 297 - 306.
[11]ZHANG Zhijin,WANG Juan,ZHANG Rongxue,et al.The ethylene response factor AtERF98 enhances tolerance to salt through the transcriptional activation of ascorbic acid synthesis in Arabidopsis[J].Plant J, 2012, 71: 273 -287.
[12]PAN I C, LI C W, SU R C, et al.Ectopic expression of an EAR motif deletion mutant of SlERF3 enhances tolerance to salt stress and Ralstonia solanacearum in tomato[J].Planta,2010,232(5): 1075 - 1086.
[13]PURANIK S, SAHU P P, SRIVASTAVA P S,et al.NAC proteins: regulation and role in stress tolerance[J].Trends Plant Sci, 2012, 17(6): 369 - 381.
[14]NAKASHIMA K, TAKASAKI H,MIZOI J,et al.NAC transcription factors in plant abiotic stress responses[J].Biochim Biophys Acta, 2012, 1819(2): 97 - 103.
[15]YOU Jun, ZONG Wei, HU Honghong, et al.A stress-responsive nac1-regulated protein phosphatase gene rice protein phosphatase18 modulates drought and oxidative stress tolerance through abscisic acid-independent reactive oxygen species scavenging in rice[J].Plant Physiol,2014,166(4): 2100 - 2114.
[16]BALAZADEH S, SIDDIQUI H, ALLU A D, et al.A gene regulatory network controlled by the NAC transcription factor ANAC092/AtNAC2/ORE1 during salt-promoted senescence[J].Plant J, 2010, 62(2): 250 - 264.
[17]江腾,林勇祥,刘雪,等.苜蓿全基因组WRKY转录因子基因的分析[J].草业学报, 2011, 20(3): 211 - 218.JIANG Teng,LIN Yongxiang,LIU Xue,et al.Genome-wide analysis of the WRKY transcription factor family in Medicago truncatula[J].Acta Pratacultuae Sin, 2011, 20(3): 211 - 218.
[18]YAN Huiru, JIA Haihong, CHEN Xiaobo, et al.The cotton WRKY transcription factor GhWRKY17 functions in drought and salt stress in transgenic Nicotiana benthamiana through ABA signalling and the modulation of reactiveoxygen species production[J].Plant Cell Physiol, 2014,55(12): 2060 - 2076.
[19]TAO Zeng , KOU Yanjun , LIU Hongbo, et al.OsWRKY45 alleles play different roles in abscisic acid signalling and salt stress tolerance but similar roles in drought and cold tolerance in rice[J].J Exp Bot, 2011, 62(14): 4863 - 4874.
[20]XIANG Yong, TANG Ning, DU Hao, et al.Characterization of OsbZIP23 as a key player of the basic leucine zipper transcription factor family for conferring abscisic acid sensitivity and salinity and drought tolerance in rice[J].Plant Physiol, 2008, 148(4): 1938 - 1952.
[21]LIAO Yong, ZOU Hongfeng, WANG Huiwen, et al.Soybean GmMYB76, GmMYB92 and GmMYB177 genes confer stress tolerance in transgenic Arabidopsis plants[J].Cell Res, 2008, 18(10): 1047 - 1060.
[22]李燕,康俊梅,张铁军,等.紫花苜蓿MsZIP基因超表达载体的构建及烟草转化[J].草地学报,2012,20 (4): 735 - 740.LI Yan,KANG Junmei, ZHANG Tiejun, et al.Construction and transformation of super-expressing plasmid of MsZIP gene from Medicago sativa L.[J].Acta Agrest Sin, 2012, 20(4): 735 - 740.
[23]ZHANG Hong, LAN Ni, LIU Yanpei, et al.The C2H2-type zinc finger protein ZFP182 is involved in abscisic acidinduced antioxidant defense in rice[J].J Integr Plant Biol,2012,54(7): 500 - 510.
[24]SUN Shunjing, GUO Shuqiao, YANG Xia, et al.Functional analysis of a novel Cys2/His2-type zinc finger protein involved in salt tolerance in rice[J].J Exp Bot,2010,61(10): 2807 - 2818.
[25]HE Ji, BENEDITO V A, WANG Mingyi, et al.The Medicago truncatula gene expression atlas web server[J].BMC Bioinform, 2009, 10(30): 441 - 449.
[26]ZAHAF O, BLANCHET S, ZÉLICOURT A, et al.Comparative transcriptomic analysis of salt adaptation in roots of contrasting Medicago truncatula genotypes[J].Mol Plant, 2012,5(5): 1068 - 1081.
[27]CHEN Yuan, LI Fei, MA Yan, et al.Overexpression of OrbHLH001, a putative helix-loop-helix transcription factor, causes increased expression of AKT1 and maintains ionic balance under salt stress in rice[J].J Plant Physiol,2013, 170(1): 93 - 100.
[28]JIANG Yuanqing, YANG Bo, DEYHOLOS M K.Functional characterization of the Arabidopsis bHLH92 transcription factor in abiotic stress[J].Mol Genet Genom,2009,282(5): 503 - 516.
[29]SIBYLLE H, OLDROYD G E D.GRAS-domain transcription factors that regulate plant development[J].Plant Signal Behav, 2009, 4(8): 698 - 700.
[30]HOU Xingliang, LEE L Y C, XIA Kuaifei, et al.DELLAs modulate jasmonate signaling via competitive binding to JAZs[J].Dev Cell, 2010, 19(6): 884 - 894.
[31]SUN Xiaolin, JONES W T, RIKKERINK E H, et al.GRAS proteins: the versatile roles of intrinsically disordered proteins in plant signaling[J].Biochem J, 2012, 442(1): 1 - 12.
[32]XIE Zongming, ZOU Hongfeng, LEI Gang, et al.Soybean trihelix transcription factors GmGT-2A and GmGT-2B improve plant tolerance to abiotic stresses in transgenic Arabidopsis[J].PLoS One, 2009, 4(9): e6898.
[33]FANG Yujie, XIE Kabin, HOU Xin, et al.Systematic analysis of GT factor family of rice reveals a novel subfamily involved in stress responses[J].Mol Genet Genomics, 2010, 283(2): 157 - 169.
[34]WANG Xiaohong,LI Qingtian,CHEN Haowei,et al.Trihelix transcription factor GT-4 mediates salt tolerance via interaction with TEM2 in Arabidopsis[J].BMC Plant Biol, 2014, 14(1): 1 - 14.
[35]CHARNG Y Y,LIU H C,LIU N Y, et al.A heat-inducible transcription factor, HsfA2, is required for extension of acquired thermotolerance in Arabidopsis[J].Plant Physiol, 2007, 143(1): 251 - 262.
[36]AYAKO N, YUKINORI Y, ERIKO Y, et al.Arabidopsis heat shock transcription factor A2 as a key regulator in response to several types of environmental stress[J].Plant J, 2006, 48(4): 535 - 547.
Identification and characterization of salt-responsive transcription factors in roots of southern type alfalfa
MA Jin, ZHENG Gang, PEI Cuiming, ZHANG Zhenya
(School of Landscape Architecture, Zhejiang A & F University, Lin’an 311300, Zhejiang, China)
Abstract:Transcription factors(TFs), which can regulate downstream gene expression, play an important role in plant stress responses.In order to investigate the molecular mechanism of salt tolerance, the TFs of southern type alfalfa, in this study with 250 mmol·L(-1)NaCl stress, Illumina RNA-sequencing was performed to evaluate the expression spectrum of transcription factors in roots of the southern alfalfa cultivar“Millennium”.Then to verify the expression of four randomly selected genes, Quantitative Reverse-Transcriptase Polymerase Chain Reaction(qRT-PCR)(three repeated)was used.Results showed 31 907 differentially expressed genes, 2 758 of which showed a difference of over two fold.Among these genes, 199 transcription factors belonging to 38 TF families were up-regulated and 95 were down-regulated.Genes from the MYB family were observed most, followed by AP2-EREBP, bHLH, WRKY, NAC, and GRAS.The qRT-PCR assay of four randomly selected genes confirmed the results of RNA-Seq analysis.In addition, candidate genes such as MsERF-2b,MsbHLH, MsbZIP, MsC2H2, MsGRAS, MsNAC, MsMGT-3a, and MsWRKY that may be involved in salt stress responses were identified.This study indicated that multiple TF families were involved in salt stress responses in the root of southern alfalfa types, and it provided new information for further study of the mechanism of a plant’s response to salt stress.[Ch, 3 fig.3 tab.36 ref.]
作者简介:马进,副教授,博士,从事植物逆境生理研究。E-mail: majinzjl@163.com
基金项目:国家自然科学基金资助项目(31272494);浙江省自然科学基金资助项目(LY16C170003)
收稿日期:2015-04-25;修回日期:2015-07-19
doi:10.11833/j.issn.2095-0756.2016.02.003
中图分类号:S722.3
文献标志码:A
文章编号:2095-0756(2016)02-0201-08

