特异长链非编码RNA在肝细胞癌中的表达和验证
厉新妍 方 亮 黄淑琳
(1广州市第一人民医院,广东 广州 510180;2香港大学理学院,中国 香港)
·论著·
特异长链非编码RNA在肝细胞癌中的表达和验证
厉新妍1*方 亮1黄淑琳2
(1广州市第一人民医院,广东 广州 510180;2香港大学理学院,中国 香港)
目的:分析肝细胞癌组织与配对癌旁组织中表达差异的长链非编码RNA(lncRNA),探讨可能与肝细胞癌发生发展有关的特异lncRNA。 方法:采用lncRNA芯片技术,检测5例肝细胞癌和其配对癌旁正常组织中的lncRNA和mRNA,筛选出差异表达的lncRNA和mRNA。运用分层聚类分析,GO分析和pathway分析等生物信息学技术对其进行分类。最后从中选择10个lncRNA用RT-PCR进行定量验证。 结果:有1 359个lncRNA表达水平出现显著性差异,差异倍数>2倍。629个显著上调,占46.2%,其中125个表达5倍以上;730个显著下调,占53.7%,其中110个表达5倍以上。聚类分析结果:① 基因间 lncRNA有11 706条,其中与已知编码基因相距<300 kb的lncRNA共有352条。② 增强子型 lncRNA 共有1 802条,其中与已知编码基因相距<300 kb的lncRNA共有42条。③ HOX 基因簇共有101条。GO分析将mRNA分成分子功能、生物过程和细胞组分3类。Pathway分析结果:差异表达的mRNA基因所参与的细胞生命活动调控通路共有61条,上调表达的mRNA参与12条通路,下调表达的mRNA参与49条通路。参与细胞周期和化学致癌作用的表达水平明显改变。RT-PCR结果:8条lncRNA表达趋势与基因芯片结果一致。对癌组和癌旁组进行检验,有6条有显著性差异(P<0.05)。 结论:与癌旁组织比较,lncRNA肝细胞癌组织中的表达水平明显改变,可能与肝细胞癌的发生发展有关。
肝细胞癌;长链非编码RNA;长链非编码RNA芯片
肝细胞癌目前在全世界恶性肿瘤中发病率排第4位,死亡率第2位[1]。其具有侵袭性强、转移发生率高的特点,病死率高、预后差,5年生存率低于10%[2]。研究肝癌的发生发展的病理生理机制非常重要,早期发现肝癌,有利于提高肝癌的有效治疗和整体预后。
长链非编码RNA(long non-coding RNA,lncRNA)是新发现的一类转录本大于200个核酸单位的非编码RNA分子。lncRNA 与人类肿瘤的发生发展关系密切[3-4]。GWAS(genome-wide association study,全基因组关联分析)证明, lncRNA 在表观遗传学、转录水平调控和转录后调控等多个层面参与了肿瘤的发生发展过程[5]。越来越多的研究证据表明,许多异常表达的lncRNA在肝细胞癌的发生发展过程中起着重要的作用[6]。近年来,运用基因芯片技术, lnc RNAs 参与肝癌的发生机制的研究也有了很大进展。本研究运用lncRNA基因芯片,检测肝细胞癌及配对癌旁组织中lncRNA的表达谱及特点,以期探索肝细胞癌新的早期诊断标记物。
1 材料与方法
1.1 实验对象
收集2012年1月至2014年1月在广州市第一人民医院肝胆外科住院,确诊为肝细胞癌行肝切除术治疗的10例患者活组织标本,其中肝细胞癌组织5例、其配对癌旁组织5例,取标本前均得到患者本人及家属同意。癌旁组织均取自于肿瘤边缘的2 cm外的相对正常组织并经病理检测确认无肿瘤组织。患者中男 4例,女1例;年龄35~70岁,中位年龄54岁。肿瘤分化程度:高分化2例、中分化3例;术后经病理学检查证实为肝细胞癌(T1N0M0-T3N0M0),术前没有接受放疗、化疗、栓塞、射频等治疗;未合并其他良、恶性病变。
1.2 芯片选择
所用基因芯片为Arraystar人类LncRNA芯片V3.0芯片。此芯片能够检测30 586个LncRNAs和26 109个蛋白质编码转录本。
1.3 样品总RNA 的提取及质量检测
于5对肝细胞癌组织和其配对癌旁组织中,每一例分别取50~100 mg 液氮内保存的肝细胞癌组织或癌旁组织,匀浆,再抽提 RNA。 使用Agilent Nanodrop ND-1000分光光度计检测样品在260、280、230 nm吸光度值,检测RNA是否降解并测定RNA的浓度。RNA完整性通过标准变性凝胶电泳进行评估。
1.4 RNA标记和芯片杂交
从总RNA中移除rRNA后,得到mRNA(mRNA-ONLY(tm) Eukaryotic mRNA Isolation Kit,Epicentre)。使用随机引物方法将每个样品放大并转录成带荧光的cRNA。使用RNeasy Mini Kit (Qiagen)纯化标记的cRNAs,并用NanoDrop ND-1 000检测浓度和活性。芯片杂交。洗涤、固定并扫描杂交芯片[Agilent DNA Microarray Scanner(part number G2505C)]。
1.5 图像采集
使用Agilent Feature Extraction软件(v11.0.1.1) 获得芯片图,并读值,得到原始数据。使用Gene Spring GX v12.1软件(Agilent Technologies)对原始数据进行Quantile标准化和随后的数据处理。原始数据标准化后经过筛选高质量探针(某探针在9个样品中至少有1个被标记为Present或Marginal)进行进一步分析。两组样品间具有统计学意义的差异表达LncRNA或差异表达mRNA通过P-value/FDR筛选。两个样品间差异表达LncRNAs或差异表达mRNAs通过Fold Change筛选。
1.6 生物信息学分析
聚类分析:一般来说,同一类样品能通过聚类出现在同一个簇(cluster)中,聚在同一个簇的样品可能具有相似的生物学性质。利用聚类分析方法分析所有lncRNA,按照lncRNA表达水平将其分类。通过寻找定位并分析lncRNA邻近的编码基因,观察 lncRNA的表达水平与其邻近编码基因mRNA表达水平是否相关,寻找lncRNA潜在的靶基因。
1.7 实时定量 PCR
从2倍以上差异表达的 LncRNA 中选取 10条在5例肝细胞癌组织及配对癌旁正常组织中进 RT-PCR验证。其中5条在肝癌组织中上调表达,5条在肝癌组织中下调表达。分别以序列名表达基因,简写为X1-X5代表上调表达基因,X6-X10代表下调表达基因。各基因及其引物如表1所示。
1.7.1 RT-PCR所用试剂2×PCR master mix(Arraystar,货号:AS-MR-OO6-25)。引物设计软件:Primer 5.0ViiA 7 Real-time PCR System (Applied Biosystems)。RNA抑制酶:Epicentre (货号:SRI6310K)。 SuperScript Ⅲ Reverse Transcriptase: Invitrogen (货号:18080-044)。5xRT缓冲液:Invitrogen(反转录酶自带产品)。2.5MmdNTP 混合液:HYTest Ltd (货号:7DN1)。Primer:由上海康成公司提供序列,英俊公司合成。
1.7.2 RT-PCR条件 95 ℃,10 min;40个PCR循环(95 ℃,10 s;60 ℃,60 s)。每个样品3管重复,每次都将待测样品及内参同时进行qRT-PCR。为了建立PCR产物的熔解曲线,扩增反应结束后,按(95 ℃,10 s;60 ℃,60 s;95 ℃,15 s);并从60 ℃缓慢加热到99 ℃(仪器自动进行-Ramp Rate为0.05 ℃/s)。
使用管家基因GAPDH(不同样品间表达量基本恒定)作为内参,以样品待测基因的值除以此样品内参的值,最终得到的比值为样品的待测基因相对含量。
1.8 统计学分析
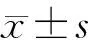
2 结 果
2.1 肝细胞癌和其配对癌旁组织基因芯片原始图片
肝细胞癌和其配对癌旁组织基因芯片原始图片见图1~5。
2.2 肝细胞癌组和配对癌旁组织差异表达LncRNA的表达谱分析
通过表达谱芯片检测,与癌旁组织比较,肝细胞癌组织中有1 359个LncRNA表达水平出现显著性差异,差异倍数>2倍。629个显著上调,占46.2%,其中125个表达5倍以上;730个显著下调,占53.7%,其中110个表达5倍以上。见表2~4。

表1 各基因对应引物序列表

图1第一对;图2第二对;图3第三对;图4第四对;图5第五对
图1-5 肝细胞癌和其配对癌旁组织基因芯片原始图片

表2 差异表达lncRNA芯片结果(个,%)
2.3 火山图及散点图
火山图中所有红点代表肝细胞癌和癌旁组织中差异表达的LncRNA(P<0.05,fold change>2.0)。其后行RT-PCR实验验证的10个lncRNA(ENST00000412092、ENST00000550805、AA151944、ENST00000448255、ENST00000329536、ENST00000504368、ENST00000579251、TCONS_00016357、ENST00000508147、TCONS_00010747)均在图中具体标注,见图6。
散点图可直观观察差异表达的LncRNA在肝细胞癌组和癌旁组织组之间的相关性。X和Y轴的值都是标准化处理后的信号值。绿色线为折叠倍率线,折叠倍率≥2。差异表达的LncRNA分别在肝细胞癌组和癌旁组表达水平变量呈正相关,见图7。

表3 肝细胞癌和癌旁组织上调表达差异5倍以上的部分lncRNA信息

表4 肝细胞癌和癌旁组织下调表达差异5倍以上的部分lncRNA信息

876543210-log10(pvalue)log2(FoldChange)CancervsPara-505ENST00000504368ENST00000579251TCONS_00016357ENST00000508147TCONS_00010747ENST00000329536AA151944ENST00000448255ENST00000550805ENST00000412092
图6 肝细胞癌和癌旁组织中LncRNA变化火山图

18161412108644681012141618CancerParaX-Axis[Para]X-Axis[Cancer]
图7 肝细胞癌和癌旁组织中LncRNA变化的散点图
2.4 聚类分析结果
采用聚类分析方法分析所有lncRNA,按照其特征性表达水平,可分成3类:基因间lncRNA(即LinclncRNA),增强子型lncRNA(即Enhancer lncRNA),HOX基因簇。通过定位并分析lncRNA邻近的编码基因,发现lncRNA的表达水平与其邻近的mRNA表达有关。
①基因间lncRNA有 11 706条,其中与已知编码基因相距<300 kb的lncRNA共有352条。② 增强子型lncRNA共有1 802条,其中与已知编码基因相距<300 kb的lncRNA共有42条。③ HOX 基因簇共有101条。将聚类分析的结果用热图(Heat Map)表示,见图8。

[4ca,Cancer][8ca,Cancer][6ca,Cancer][5ca,Cancer][9ca,Cancer][4para,Para][8para,Para][6para,Para][5para,Para][9para,Para]samplesgroups
注:纵列为样本,横行为lncRNA;红色为上调表达lncRNA,绿色为下调表达lncRNA
图8 差异表达lncRNA的聚类分析热图
2.5 基因本体论分析和生物学途径分析
在5对样本中可检测到23 335 mRNA。通过对5对样本的数据进行分析,与癌旁非肿瘤肝组织相比,肝细胞癌组织中有518个mRNA的表达水平显著上调,954个mRNA表达水平显著下调(差异倍数>2,P<0.05)。对差异显著表达的mRNA进行了基因本体论(gene ontology ,GO)分析和生物学途径分析(pathway analysis)。
2.5.1 GO分析 将mRNA从以下3类对mRNA进行属性归属和富集分析:生物学过程类(biological process,BP)、细胞组分类(cellular component,CC)、分子功能类(molecular function,MF)。表达上调和下调的3类mRNA的构成比见图9,10。
2.5.2 生物学途径分析 根据最新版本的KEGG数据库,对肝细胞癌和癌旁组织中差异表达上调和下调的mRNA进行了生物学途径分析。其中差异表达的mRNA基因所参与的细胞生命活动调控通路,上调表达的mRNA参与12条通路,下调表达的mRNA参与49条通路。在上调表达的细胞通路中,细胞周期信号通路富集度最高。在下调表达的细胞通路中,化学致癌作用富集度最高。见图11。
2.6 对差异表达的lncRNA进行RT-PCR验证结果
编号为X1-X10的lncRNA中,在肝细胞癌组织和癌旁组织中均有表达。其中X1、X2、X3、X4在肝细胞癌组织中为上调表达;X6、X8、X9、X10在肝细胞癌组织中下调表达,与芯片结果一致。
肝细胞癌和癌旁组织比较,上调基因中X2、X3、X4差异有统计学意义(P<0.05);下调基因中X6、X9、X10差异有统计学意义(P<0.05)。见表5。

CellcyclephaseCellcycleprocessMphasemitoticcellcyclecellcycleMphaseofmitoticcellcyclemitosisnucleardivisionorganellefissionorganelleorganization10152025303505EnrichmentScore[-log10(Pvalue)]SlgGOtermsofDEgene-BP1015202505EnrichmentScore[-log10(Pvalue)]02468EnrichmentScore[-log10(Pvalue)]ABCchromosomechromosomalpartchromosomecentromericregionnucleuscondensedchromosomespindleintracellularnon-membrane-boundedorganellenon-membrane-boundedorganellecondensedchromosomekinetochorecondensedchromosomecentromericregionproteinbindingbindingDNAbindingnucleicacidbingdingpyrophosphataseactivitynucleoside-triphosphataseactivityhydrolaseactivity,actingonacidanhydrides…microtubulemotoractivityhydrolaseactivity,actingonacidanhydrideskinasebingdingSigGOtermsofDEgene-MFSlgGOtermsofDEgene-CC

注:A图为BP、B图为CC、C图MF

注:A图为BP、B图为CC、C图MF
图11 表达上调(A)和下调(B)的mRNA富集度较高的10条信号通路

表5 肝细胞癌和癌旁组织中差异表达lncRNA的RT-PCR验证结果(n=5)
3 讨 论
肝细胞癌是常见的恶性肿瘤之一,其发生发展过程复杂,涉及多种基因和环境因素的协同作用,经过多个病理及分子阶段改变,实现由正常肝细胞演变为癌细胞[7]。
lncRNA曾被称为基因组中的"暗物质"而未被重视[8-9]。但近年来,越来越多的研究报道发现,lncRNA某些特定区段在各种肿瘤细胞中广泛表达,其与正常细胞的原癌基因有关,能抑制细胞凋亡。若其异常表达,则会导致肿瘤发生[10-11]。多研究表明,lncRNA与肝细胞癌存在密切关系[12-14]。
本研究中,我们运用美国Arraystar公司提供的lncRNA V3.0基因芯片,其可同时检测mRNA的表达,并包含了当今所有权威数据库的lncRNA,对肝细胞癌的lncRNA表达谱进行了深入全面的分析。选择5对肝细胞癌和配对癌旁组织检测其差异表达lncRNA,通过聚类分析,基因本体论和生物学途径分析,对其中差异显著表达的lncRNA和mRNA进行分类,预测lncRNA的分子功能,最后利用qRT-PCR验证实验结果。
通过实验,我们确认在肝细胞癌组织中,有629个lncRNA上调表达, 730个lncRNA下调表达。这些lncARNA中的绝大部分尚未深入研究,我们预测其可能参与肝细胞癌发生发展的多个环节,为我们研究肝细胞癌提供了新的思路。
为了更好的分析差异显著的LncRNA,我们运用了多种生物信息学方法进行分析。聚类分析可以获得样本间lncRNA和mRNA的表达模式的差异,根据基因芯片所测mRNA表达水平及变化,对邻近的差异表达的lncRNA进行分类,我们可以推测,LncRNA邻近的编码基因可能是其潜在的靶基因。GO分析显示肝细胞癌组和癌旁组织对比有差异表达的mRNA与新陈代谢过程、细胞周期、染色体片段等方面相关。pathway显示其中差异表达的mRNA基因所参与的细胞生命活动调控通路,上调表达的mRNA参与12条通路,下调表达的mRNA参与49条通路。在上调表达的细胞通路中,细胞周期信号通路富集度最高。在下调表达的细胞通路中,化学致癌作用富集度最高。说明这条信号通路可能在肝细胞癌的发生发展过程中起重要作用。
为了验证芯片分析的结果,我们选择了10个lncRNA通过RT-PCR方法进行验证。结果显示,与其在非肿瘤组织上的表达水平相比,ENST00000550805、AA151944、 ENST00000448255在肝细胞癌组织中的表达水平显著升高;ENST00000504368、ENST00000508147、TCONS_00010747在肝细胞癌组织中的表达水平显著降低。到目前为止对这6种lncRNA的功能特征及在人类疾病中的作用还非常缺乏了解。但是,基于其在肝细胞癌中的显著高或低表达,它们极有可能与肝细胞癌的发生或发展有密切的关联,其潜在价值值得未来进一步研究去探讨。
本研究通过基因芯片技术,成功筛选并验证肝细胞癌和癌旁组织之间差异表达的lncRNA。进一步通过与mRNA表达谱结合的聚类分析,基因本体论及生物学途径分析,预测这些差异表达的lncRNA与相关编码蛋白基因间的功能联系,为进一步研究肝细胞癌新的诊断指标和治疗靶点提供思路。
[1] Raza A,Sood GK. Hepatocellular carcinoma review: current treatment,and evidence-based medicine[J]. World J Gastroenterol,2014,20: 4115-4127.
[2] 周 俭,肖永胜.肝癌的转化医学研究——从基础到临床[J]. 中国普通外科杂志,2016,25(1):1-5.
[3] Zhou J,Xiao YS.Translational medical research of hepatoce-llular carcinoma: from bench to bedside[J].Chinese Journal of General Surgery,2016,25(1):1-5.
[4] Bartel DP. Micro RNAs: target recognition and regula-toryfunctions [J].Gell,2009,136(2):215 233.
[5] Krol J,Loedige I,Filipowicz W. The widespreadregulation of microRNA biogenesis,function and decay[J].NatRev Genet,2010,11 (9):597-610.
[6] Ponting CP,Oliver PL,Reik W. Evolution and functions of long noncoding RNAs[J]. Cell,2009,136 (4) : 629-641.
[7] 刘 冰,陆骊工.肝细胞癌相关基因的研究进展[J].中华生物医学工程杂志,2013,19(6):494-497.
[8] Forner A,Lovet JM,Bruix J. Hepatocellularcarcinoma [J].Lancet,2012,379 (9822):1245-1255.
[9] Pang RW,John JW,Johnson PJ,et al. Biology of hepatocellular carcinoma [J].Ann SurgOncol,2008,15 (4):962-971.
[10] Ponting CP,Oliver PL,Reik W. Evolution and functions of long noncoding RNAs[J]. Cell,2009,136(4):629-641.
[11] Guttcnan M,Amit I,Garter M,et al.Chromatin signature reseals over a thousand highly conserved largenon coding RNA. In mammals[J] Nature,2009,458(7235):223-227.
[12] Wilusz JE,Sunwoo H,Spector DL. Long noncoding RNAs:functional surprises from the RNA,world [J]. Genes&development,2009,23(13)1:1494-1504.
[13] Yang Z,Zhou L,Wu LM,et al. Overexpression of long non-coding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation[J]. Ann SurgOncol,2011,18(5) : 1243-1250.
[14] Lai MC,Yang Z,Zhou L,et al. Long non-coding RNAMALAT-1 overexpression predictstumor recurrenceof hepatocellular carcinoma after liver transplantation[J]. Med Oncol,2012,29(3) : 1810-1816.
[15] Braconi C,Kogure T,Valeri N,et al. MicroRNA-29 can regulate expression of the long non-coding RNA gene MEG3 in hepatocellular cancer[J]. Oncogene,2011,30(47):4750-4756.
(本文编辑:张辉)
Expression and verification of specific long non-coding RNA in hepatocellular carcinoma
LiXinyan1*,FangLiang1,HuangShulin2
(1GuangzhouFirstMunicipalPeople′sHospital,Guangzhou510180;2FacultyofScience,UniversityofHongKong,HongKong,China)
*Correspondingauthor:Email:lixinyan1005@126.com
Objective:To analyze the differentially expressed of long non-coding RNA (lncRNA) in hepatocellular carcinoma (HCC) tissues and matched adjacent normal tissues,and to investigate the specific lncRNA that may be involved in the occurrence and development of HCC. Methods: lncRNA and mRNA were determined in 5 cases of HCC and the matched adjacent normal tissues using lncRNA chip technique,and the differentially expressed lncRNA and mRNA were screened out,and classified by bioinformatics techniques of hierarchical clustering analysis,GO analysis and pathway analysis. Finally,10 lncRNAs were selected for quantitative validation by RT-PCR. Results: There were significant differences in the expression levels of 1 359 lncRNAs,with a fold change > 2-fold. A total of 629 lncRNAs were significantly up-regulated,accounting for46.2%,of which 125 were more than 5-fold; 730 were down-regulated,accounting for 53.7%,110 of which were more than 5-fold. Cluster analysis: ① there were 11 706 lncRNAs between genes,among which 352 lncRNAs were<300 kb from the known coding genes. ② There were 1 802 enhancer-type lncRNAs,of which there were 42 lncRNAs with<300 kb distance from the known coding genes. ③ There were 101 HOX gene clusters. GO analysis divided the mRNA into molecular function,biological process and cellular component. Pathway analysis: There were 61 pathways in the differentially expressed mRNA genes involved regulation of cell life activity. Up-regulated mRNA was involved in 12 pathways,and down-regulated mRNA was involved in 49 pathways. The expression levels of the lncRNAs involved in the cell cycle and chemical carcinogenesis were significantly changed. RT-PCR: The expression trends of 8 lncRNAs were consistent with the gene chip. There were 6 significant differences between the HCC group and the matched adjacent normal group (P<0.05). Conclusion: The expression level of lncRNA in HCC is significantly changed compared with that in adjacent normal tissues,which may be related to the occurrence and development of HCC.
hepatocellular carcinoma; long non-coding RNA; long non-coding RNA chip
10.3969/j.issn.2095-9664.2016.05.01
广东省科技计划项目(2012B031800010)
*通讯作者:Email:lixinyan1005@126.com
R735.7
A
2095-9664(2016)05-0001-08
2016-08-15)

