甜椒核心种质资源比较构建研究
刘子记,曹振木,朱 婕,申龙斌(.中国热带农业科学院热带作物品种资源研究所/农业部华南作物基因资源与种质创制重点开放实验室,海南 儋州 57737;.海南大学园艺园林学院,海口 5708)
甜椒核心种质资源比较构建研究
刘子记1,曹振木1,朱婕2,申龙斌1
(1.中国热带农业科学院热带作物品种资源研究所/农业部华南作物基因资源与种质创制重点开放实验室,海南儋州571737;2.海南大学园艺园林学院,海口570228)
摘要:构建核心种质可大幅提高种质资源利用效率。以410份甜椒种质资源为材料,基于8个性状表型数据,采用混合线性模型分析方法无偏地预测基因型值,利用马氏距离计算种质间遗传距离,分别采用两种聚类方法(最短距离法和类平均法)和两种取样方法(随机取样法和偏离度取样法),按照25%抽样比率构建甜椒核心种质库。采用均值、方差、极差和变异系数4个指标评价不同取样和聚类构建核心种质库水平。结果表明,最短距离法能极显著增加性状方差和变异系数,明显优于类平均法;偏离度取样法优于随机取样法;基于马氏距离、最短距离法和偏离度取样方法获取的102份甜椒核心种质资源能代表原群体遗传多样性。该研究可为甜椒种质资源有利基因发掘和新品种选育奠定基础。
关键词:甜椒;基因型值;聚类方法;取样方法;核心种质
刘子记,曹振木,朱婕,等.甜椒核心种质资源比较构建研究[J].东北农业大学学报, 2016, 47(1): 21-29.
Liu Ziji, Cao Zhenmu, Zhu Jie, et al. Comparative study on the construction of sweet pepper core collection[J]. Journal of Northeast Agricultural University, 2016, 47(1): 21-29. (in Chinese with English abstract)
辣椒(Capsicum spp.,2n=2x=24)在植物学分类上属茄科(Solanaceae)辣椒属(Capsicum)1年或多年生草本植物[1],原产于中南美洲[2],于明代传入中国。辣椒属包括5个栽培种,分别为1年生辣椒(Capsicum annum)、灌木状辣椒(Capsicum frutes⁃cens)、中国辣椒(Capsicum chinense)、下垂辣椒(Capsicum baccatum)和茸毛辣椒(Capsicum pubes⁃cens)[3]。甜椒(Capsicum annuum L. var. grossum Sendt.)属1年生辣椒,果实不辣而略带甜味,以果实特有色泽和营养成分成为世界性蔬菜作物[4-5]。大量研究表明,甜椒果实含有丰富维生素A[6]、维生素C、类胡萝卜素[7]及钙、铁等矿物质,可保护生物有机体免受氧化伤害[8-9],预防坏血病并提高机体免疫力。
种质资源蕴含极其丰富的遗传变异和性状的有利基因,不仅是农业生产和育种工作物质基础,更是生物学研究重要材料[10]。常规育种、远缘杂交、倍性育种、辐射育种和遗传工程等均离不开种质资源。种质资源数量和质量,及其遗传特性研究的深度与广度是决定育种效果的重要条件。植物育种的重大突破,均与重要种质资源发现与利用有关。随种质资源不断收集和积累,种质资源库管理费用不断提高,且筛选特异种质材料难度加大。辣椒作为世界性蔬菜作物,具有丰富的遗传多样性[11],但大多数种质资源未得到有效利用[12]。Frankel和Brown于1984年最早提出构建核心种质理念[13]。对种质资源深入评价与鉴定,并建立核心种质,有利于种质库管理,可促进种质资源深入利用,大幅提高种质资源利用效率。近年来,核心种质研究蓬勃发展,先后对菜豆[14]、玉米[15]、水稻[16-17]、花生[18]、大豆[19]、橄榄[20]、短柄草[21]等多种作物构建核心种质库。园艺作物核心种质研究起步较晚,甜椒核心种质研究鲜有报道。
以最少数量遗传资源包含最大限度遗传多样性,是构建核心种质关键问题。作物性状表型受基因型和环境条件控制,准确度量不同遗传材料间遗传差异是有效构建核心种质前提。本研究采用混合线性模型预测性状的基因型效应值,比较不同取样和聚类方法优劣,有效构建甜椒核心种质,以期为甜椒种质资源高效利用和新品种选育提供理论依据。
1 材料与方法
1.1供试种质材料和基因型值预测
供试甜椒种质资源共410份,其中12份来自山东,13份来自河北,26份来自湖南,30份来自山西,22份来自甘肃,20份来自新疆,24份来自江苏,15份来自福建,35份来自广东,34份来自云南,38份来自海南,18份来自广西,15份来自美国,11份来自英国,18份来自墨西哥,10份来自法国,11份来自泰国,5份来自日本,22份来自巴西,8份来自格林纳达,10份来自河南,13份来自四川(见表1)。不同种质间株高、叶片、果实等性状存在显著差异。
将410份甜椒种质资源按田间行列编号顺序种植,以一定间隔穿插对照种质,连续三年试验,按照文献[22]调查株高、株幅、首花节位、果纵径、果横径、果柄长、果肉厚和单果重。采用朱军提出的混合线性统计分析模型,利用调整无偏预测法无偏预测性状基因型效应值[23]。
1.2遗传距离计算与聚类分析
采用马氏距离基于基因型预测值计算不同种质间遗传距离,假设共有n份种质资源,采用m个性状聚类。第i个种质与第j个种质基因型效应向量分别为giT= (gi1, gi2,…gim);gjT= (gj1, gj2,…gjm),则第i个种质与第j个种质间马氏距离计算公式为Dij2=(gi-gj)TVG-1(gi-gj)[24]。基于甜椒种质间遗传距离,分别采用最短距离法和不加权类平均法聚类分析[25]。
1.3抽样和核心种质遗传变异评价
分别采用随机取样[26]、偏离度取样[27]和25%抽样比率构建甜椒核心种质库,本研究采用均值、方差、极差和变异系数4个指标评价核心资源库构建水平,分别利用F测验和t测验分析方差和均值差异性。
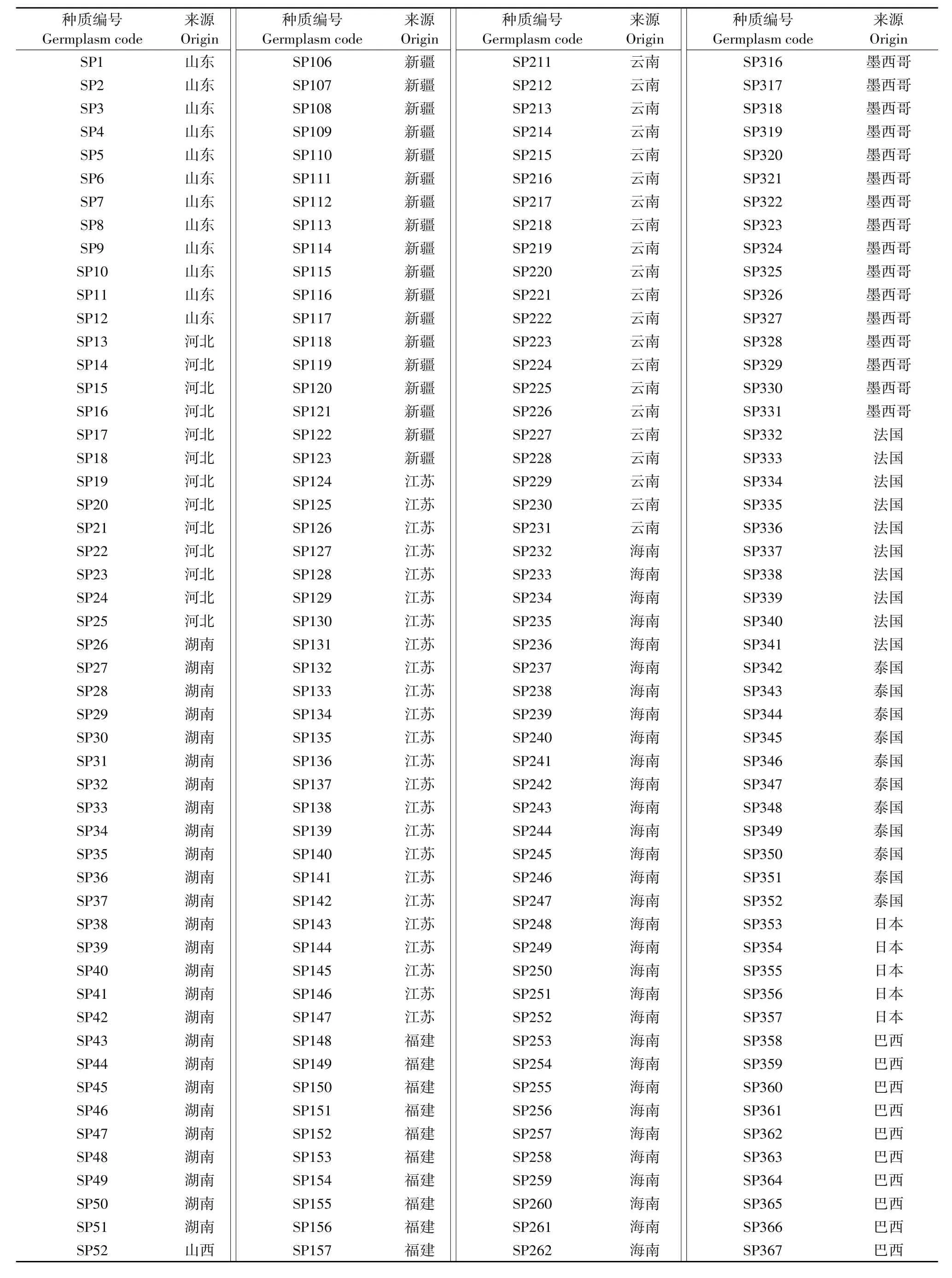
表1甜椒种质资源来源Table 1 Origin of sweet pepper germplasm
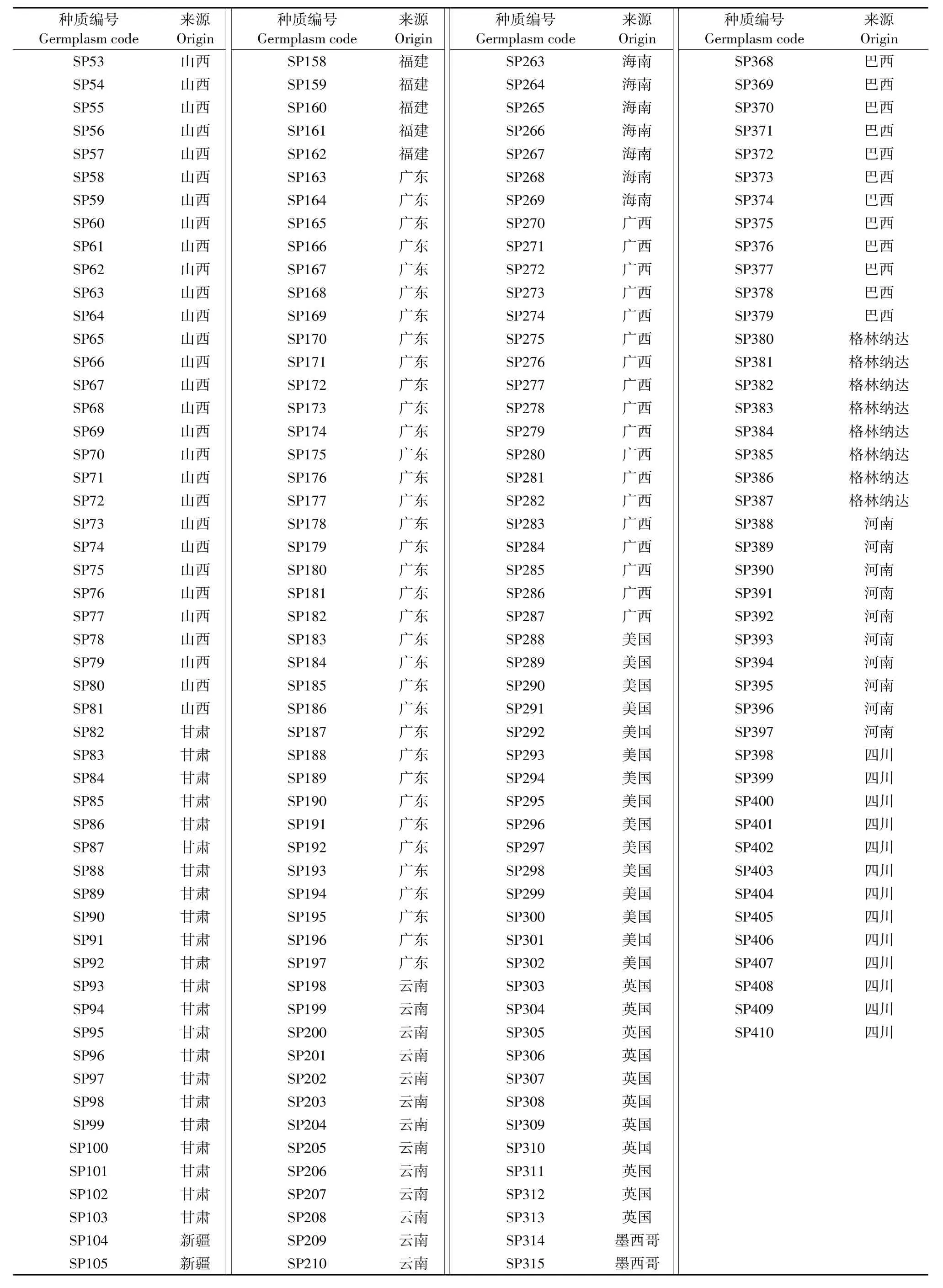
续表
2结果与分析
2.1比较两种聚类方法构建甜椒核心种质
根据性状基因型值,采用马氏距离、偏离度取样法和25%抽样比率,分别基于两种聚类方法(最短距离法和类平均法)构建核心种质。结果表明,利用最短距离法和类平均法构建核心种质均值与原群体无显著差异。与原群体相比,8个性状方差均得到不同程度提高。利用最短距离法构建核心种质,所有8个性状方差与原群体差异达极显著水平。采用类平均法构建核心种质,5个性状(株幅、果纵径、果横径、果肉厚、单果重)方差与原群体差异达极显著水平,果柄长方差与原群体无显著差异。最短距离法构建的核心种质6个性状保存原群体极差,类平均法构建的核心种质5个性状保存原群体极差。最短距离法和类平均法均能提高性状变异系数,采用最短距离法构建核心种质变异系数均大于类平均法(见表2)。综上结果,采用最短距离法聚类分析构建的甜椒核心种质具有相对较大遗传变异,优于类平均法。
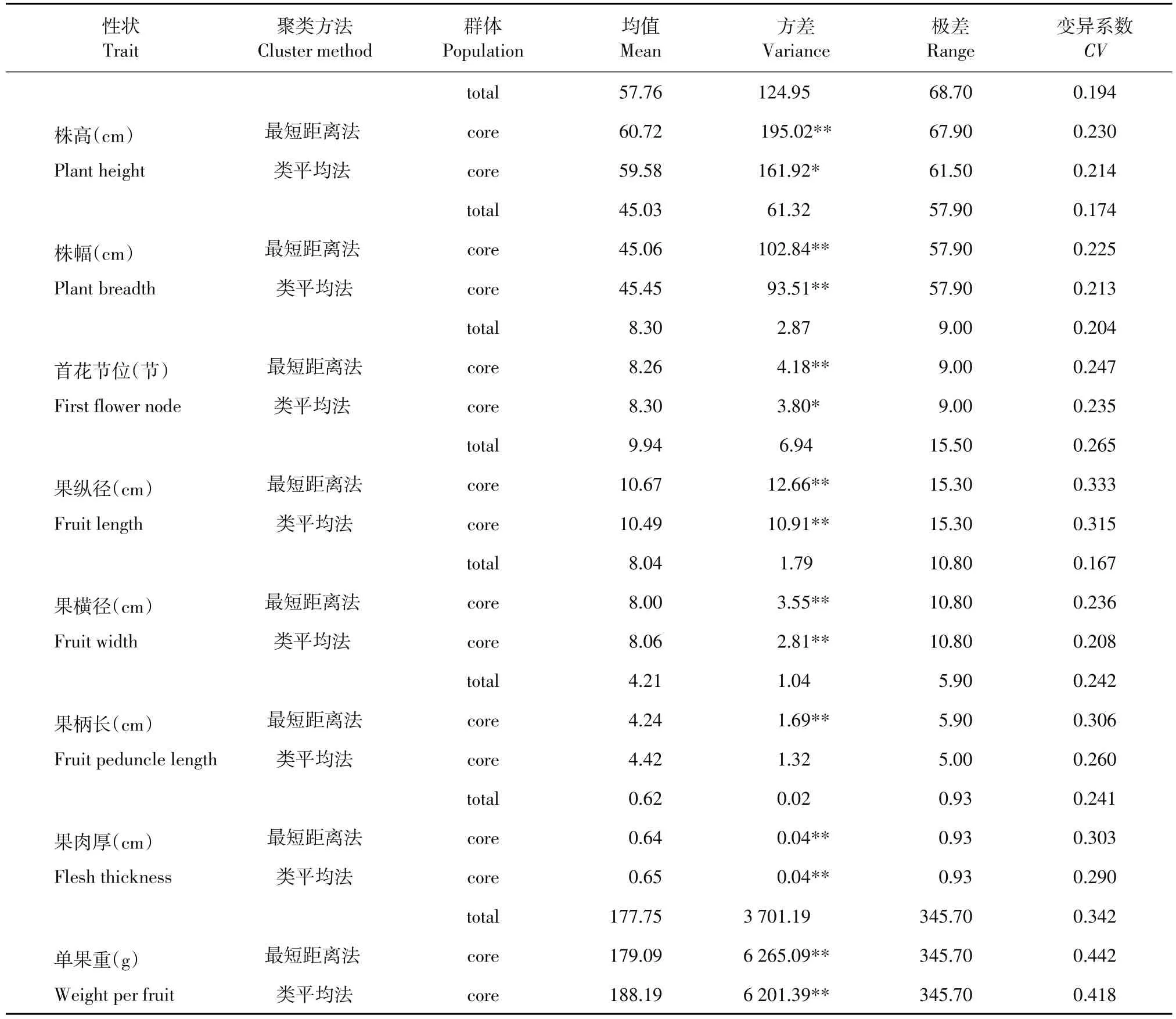
表2两种聚类方法构建甜椒核心种质与原群体间遗传变异比较Table 2 Comparison of genetic variation between initial collection and core collections constructed based on two kinds of cluster strategies in sweet pepper
2.2比较两种取样方法构建的甜椒核心种质
根据性状基因型值,采用马氏距离、非加权类平均法和25%抽样比率,分别基于两种取样方法(随机取样和偏离度取样)构建核心种质。结果表明,利用随机取样和偏离度取样法构建核心种质均值与原群体无显著差异。利用偏离度取样法构建核心种质,8个性状方差均大于随机取样法构建核心种质和原群体,其中5个性状(株幅、果纵径、果横径、果肉厚、单果重)方差与原群体差异达极显著水平,2个性状(株高、首花节位)方差与原群体差异达显著水平。采用随机取样法构建核心种质仅首花节位方差与原群体差异达显著水平,其余性状方差与原群体无显著差异。采用偏离度取样法构建核心种质5个性状极差与原群体一致,采用随机取样法构建核心种质3个性状极差与原群体一致。偏离度取样法和随机取样法构建核心种质8个性状变异系数均高于原群体,采用偏离度取样法构建核心种质,7个性状变异系数高于随机取样法,仅果柄长变异系数略低于随机取样法(见表3)。综合以上分析结果表明,采用偏离度取样法构建甜椒核心种质具有相对较大的遗传变异,优于随机取样法。
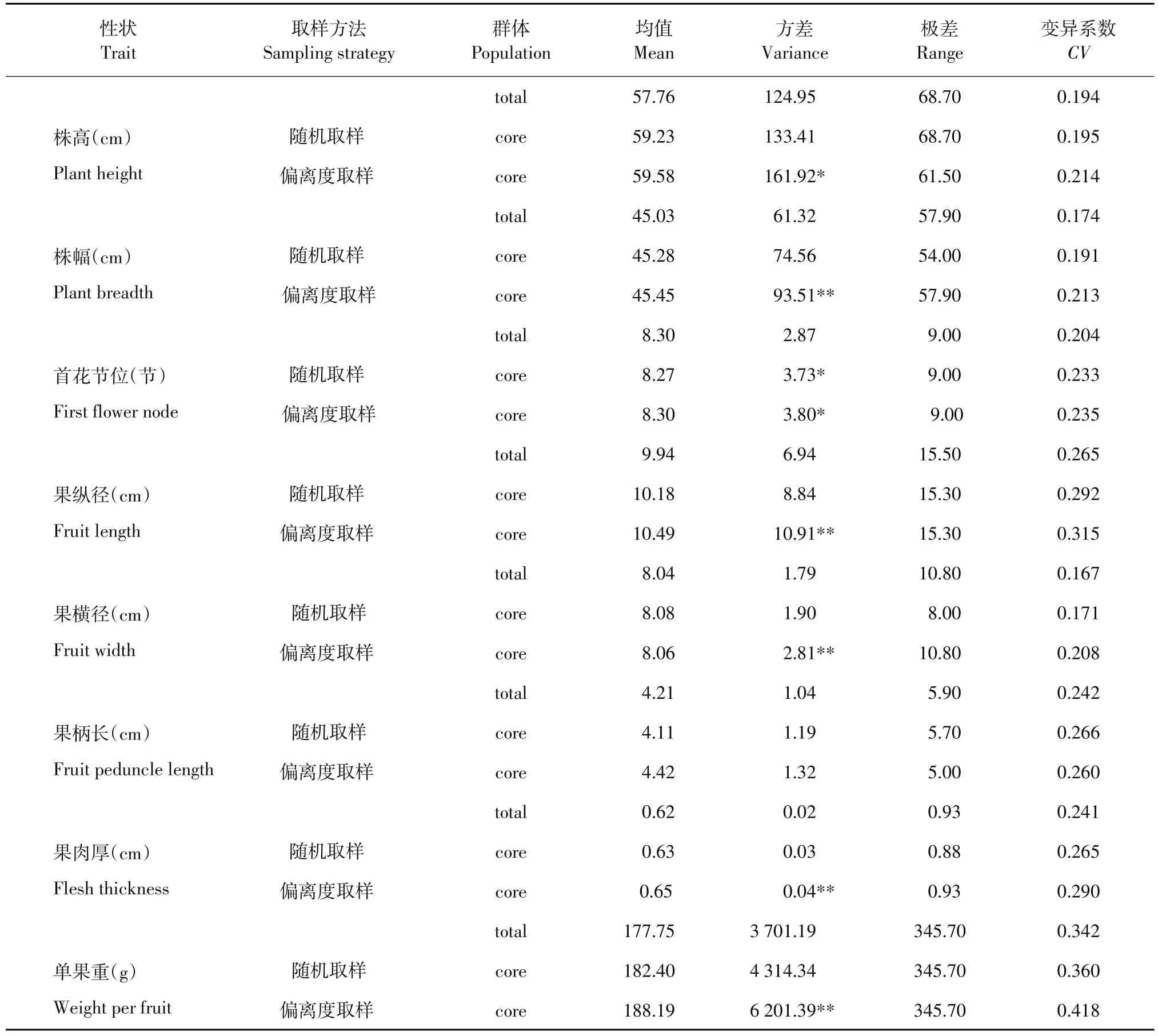
表3两种取样方法构建的甜椒核心种质与原群体间遗传变异比较Table 3 Comparison of genetic variation between initial collection and core collections constructed based on two kinds of sampling strategies in sweet pepper
2.3甜椒核心种质构建
采用马氏距离、最短距离法聚类、偏离度取样法和25%抽样比率,构建甜椒核心种质。核心种质均值与原群体无显著差异。所有8个性状方差均极显著大于原群体。核心种质6个性状保存原群体极差,2个性状极差略低于原群体。核心种质所有8个性状变异系数均高于原群体(见表4)。综上分析,获取的102份甜椒核心资源能代表原群体遗传多样性。甜椒核心种质编号为:SP10、SP16、SP17、SP26、SP30、SP31、SP32、SP36、SP37、SP40、SP50、SP55、SP56、SP62、SP68、SP74、SP75、SP76、SP79、SP81、SP87、SP92、SP93、SP100、SP104、SP106、SP107、SP108、SP114、 SP118、SP119、SP121、SP123、SP131、SP142、SP143、SP145、SP152、SP155、SP158、SP160、SP165、SP177、SP178、SP180、SP182、SP196、SP205、SP206、SP208、SP214、SP219、SP225、SP237、SP241、SP242、SP243、SP257、SP259、SP261、SP262、SP263、SP265、SP266、SP270、SP275、SP276、SP279、SP285、SP301、SP312、SP314、SP315、SP318、SP319、SP322、SP324、SP327、SP329、SP332、SP333、SP336、SP339、SP341、SP346、SP354、SP355、SP359、SP361、SP364、SP365、SP368、SP373、SP376、SP381、SP382、SP383、SP386、SP391、SP393、SP399、SP404。
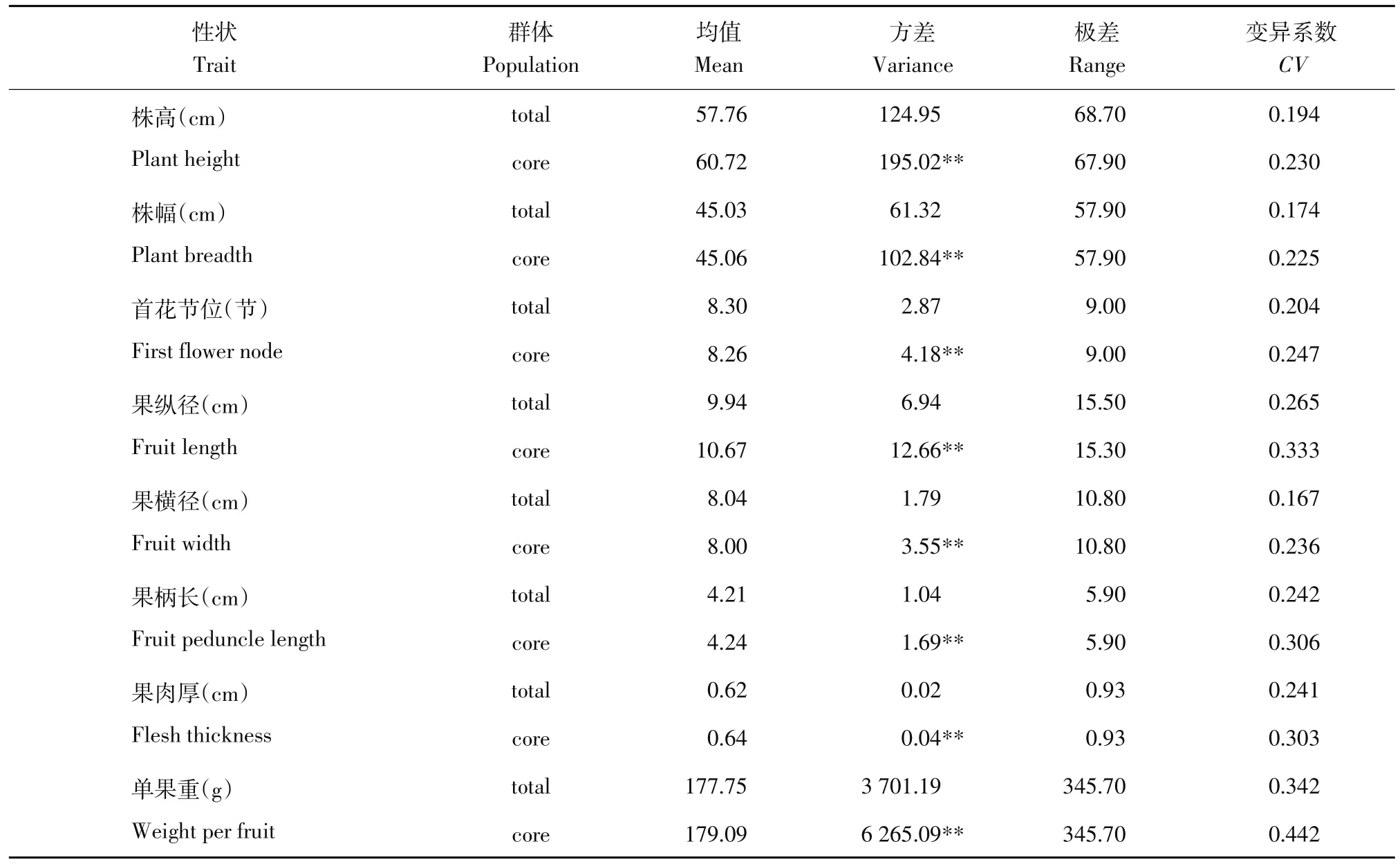
表4甜椒核心种质与原群体间遗传变异比较Table 4 Comparison of genetic variation between initial collection and core collections in sweet pepper
3讨论与结论
种质资源对高产、优质、抗逆新品种选育具有重要意义[28-29]。核心种质构建可提高种质资源利用效率和种质库管理水平。核心种质由种质库中一部分材料组成,以最少数量遗传资源包含最大限度遗传多样性[30],同时又能反映整个种质资源群体遗传结构,是评价和利用种质资源切入点。核心库中入选的遗传材料间均存在生态上或遗传上的距离,覆盖整个种质库内遗传变异[31]。农艺性状多数为数量性状,与环境存在互作,基于农艺性状的遗传分类不能准确度量基因型间遗传差异。所得遗传分类不能真实反映种质资源固有遗传结构[32]。采用合理统计模型及统计分析方法预测基因型值,可有效排除试验误差、环境效应、基因型与环境互作效应。本研究采用混合线性模型无偏预测性状基因型值,利用基因型值的聚类分析,结果更具可靠性。
聚类分析作为一种重要多变量分析工具,常应用于种质资源遗传多样性分析[33]。为确保构建核心库尽可能多地保存原有种质资源遗传变异,首先要对原种质群体遗传分类,明确不同材料间遗传关系。本研究结果表明,采用最短距离法的聚类分析构建甜椒核心种质,所有性状方差和变异系数均大于类平均法,种质间具有相对较大遗传变异,优于类平均法。
完成种质群体遗传分类后,需要根据分类结果采用一定抽样比率和策略对各类群抽样,抽样策略直接关系核心库优劣。胡晋等[26]和徐海明等[27]分别提出多次聚类随机取样和偏离度取样方法。本文以甜椒种质资源为研究对象,比较随机取样和偏离度取样策略构建核心种质库水平。偏离度取样法构建核心种质方差均大于随机取样法,7个性状变异系数高于随机取样法,核心种质大部分保存原群体极差,优于随机取样法。因此核心种质是否有效保存原有种质群体遗传变异,可利用各性状均值、方差、极差、变异系数等参数评价;并且核心种质各性状均值与极差应与原群体无显著差异,方差和变异系数大于原群体[34]。
[参考文献]
[ 1 ] Kim S, Park M, Yeom S I, et al. Genome sequence of the hot pep⁃per provides insights into the evolution of pungency in Capsicum species [J]. Nat Genet, 2014, 46(3): 270-278.
[ 2 ] Barchi L, Bonnet J, Boudet C, et al. A high-resolution, intraspecif⁃ic linkage map of pepper (Capsicum annuum L. ) and selection of reduced recombinant inbred line subsets for fast mapping[J]. Ge⁃nome, 2007, 50(1): 51-60.
[ 3 ]孟金贵,张卿哲,王硕,等.涮辣与辣椒属5个栽培种亲缘关系的研究[J].园艺学报, 2012, 39(8): 1589-1595.
[ 4 ] Kim H J, Nahm S H, Lee H R, et al. BAC-derived markers con⁃verted from RFLP linked to Phytophthora capsici resistance in pepper (Capsicum annuum L. ) [J]. Theor Appl Genet, 2008, 118 (1): 15-27.
[ 5 ] Chen C M, Hao X F, Chen G J, et al. Characterization of a new male sterility-related gene Camf1 in Capsicum annum L. [J]. Mol Biol Rep, 2012, 39(1): 737-744.
[ 6 ] Mejia L A, Hudson E, de Mejia E G, et al. Carotenoid content and vitamin-A activity of some common cultivars of Mexican peppers (Capsicum annuum) as determined by HPLC [J]. J Food Sci, 1998, 53(5): 1448-1451.
[ 7 ] Ha S H, Kim J B, Park J S, et al. A comparison of the carotenoid accumulation in Capsicum varieties that show different ripening colours: Deletion of the capsanthin-capsorubin synthase gene is not a prerequisite for the formation of a yellow pepper [J]. J Exp Bot, 2007, 58(12): 3135-3144.
[ 8 ] Marín A, Ferreres F, Tomás-Barberán F A, et al. Characterization and quantitation of antioxidant constituents of sweet pepper (Cap⁃sicum annuum L.)[J]. J Agric Food Chem, 2004, 52(12): 3861-3869.
[ 9 ] Sun T, Xu Z, Wu C T, et al. Antioxidant activities of different col⁃ored sweet bell peppers (Capsicum annuum L.) [J]. J Food Sci, 2007, 72(2): 98-102.
[10]朱岩芳,祝水金,李永平,等. ISSR分子标记技术在植物种质资源研究中的应用[J].种子, 2010, 29(2): 55-59.
[11] Lefebvre V, Pflieger S, Thabuis A, et al. Towards the saturation of the pepper linkage map by alignment of three intraspecific maps including known-function genes[J]. Genome, 2002, 45(5): 839-854.
[12] Rao G U, Ben Chaim A, Borovsky Y, et al. Mapping of yield-relat⁃ed QTLs in pepper in an interspecific cross of Capsicum annuum and C. frutescens[J]. Theor Appl Genet, 2003, 106(8): 1457-1466.
[13] Frankel O H, Brown A H D. Current plant genetic resources-a critical appraisal. Genetics: New Frontiers Vol. IV [M]. London: Oxford and IBH Publishing, 1984.
[14] Blair M W, Díaz L M, Buendía H F, et al. Genetic diversity, seed size associations and population structure of a core collection of common beans (Phaseolus vulgaris L. ) [J]. Theor Appl Genet, 2009, 119(6): 955-972.
[15] Coimbra R R, Miranda G V, Cruz C D, et al. Development of a Brazilian maize core collection [J]. Genet Mol Biol, 2009, 32(3): 538-545.
[16] Li X, Yan W, Agrama H, et al. Genotypic and phenotypic charac⁃terization of genetic differentiation and diversity in the USDA rice mini-core collection[J]. Genetica, 2010, 138(11-12): 1221-1230.
[17] Zhang H, Zhang D, Wang M, et al. A core collection and mini core collection of Oryza sativa L. in China [J]. Theor Appl Genet,2011, 122(1): 49-61.
[18] Wang M L, Sukumaran S, Barkley N A, et al. Population structure and marker-trait association analysis of the US peanut (Arachis hypogaea L. ) mini-core collection [J]. Theor Appl Genet, 2011, 123(8): 1307-1317.
[19] Kaga A, Shimizu T, Watanabe S, et al. Evaluation of soybean germplasm conserved in NIAS genebank and development of mini core collections [J]. Breed Sci, 2012, 61(5): 566-592.
[20] EI Bakkali A, Haouane H, Moukhli A, et al. Construction of core collections suitable for association mapping to optimize use of Mediterranean olive (Olea europaea L.) genetic resources[J]. PLoS One, 2013, 8(5): e61265.
[21] Tyler L, Fangel J U, Fagerström A D, et al. Selection and pheno⁃typic characterization of a core collection of Brachypodium dis⁃tachyon inbred lines [J]. BMC Plant Biol, 2014, 14: 25.
[22]李锡香,张宝玺,沈镝,等.辣椒种质资源描述规范和数据标准[M].北京:中国农业出版社, 2006.
[23]朱军.作物杂种后代基因型值和杂种优势的预测方法[J].生物数学学报, 1993, 8(1): 32-44.
[24] Mahalanobis P C. On the generalized distance in statistics[J]. Proc Natl Inst Sci India, 1936, 2(1): 49-55.
[25]裴鑫德.多元统计分析及其应用[M].北京:北京农业大学出版社, 1991.
[26]胡晋,徐海明,朱军.基因型值多次聚类法构建作物种质资源核心库[J].生物数学学报, 2000, 15(1): 103-109.
[27]徐海明,胡晋,朱军.构建作物种质资源核心库的一种有效抽样方法[J].作物学报, 2000, 26(2): 157-162.
[28]王志强,郭松,刘声锋,等.西瓜种质资源果实主要数量性状的主成分分析[J].东北农业大学学报, 2014, 45(3): 59-64.
[29]刘守伟,刘淑芹,周新刚,等.分蘖洋葱种质资源的农艺学性状分析[J].东北农业大学学报, 2014, 45(12): 49-57.
[30] Brown A H D. Core collection: A practical approach to genetic re⁃sources management [J]. Genome, 1989, 31: 818-824.
[31] IBPGR. Annual report 1990[R]. Rome: International Board for Plant Genetic Resources, 1991.
[32] Tanksley S D, Mc Couch S R. Seed banks and molecular maps: unlocking genetic potential from the wild [J]. Science, 1997, 277 (5329): 1063-1066.
[33] Peeters J P, Martinelli J A. Hierarchical cluster analysis as a tool to manage variation in germplasm collections[J]. Theor Appl Gen⁃et, 1989, 78(1): 42-48.
[34] Diwan N, McIntosh M S, Bauchan G R. Methods of developing a core collection of annual Medicago species[J]. Theor Appl Genet, 1995, 90(6): 755-761.
Comparative study on the construction of sweet pepper core collection
LIU Ziji, CAO Zhenmu, ZHU Jie, SHEN Longbin(1. Tropical Crops Genetic Resources Institute, Chinese Academy of Tropical Agricultural Sciences/Key Laboratory of Crop Gene Resources and Germplasm Enhancement in Southern China, Ministry of Agriculture, Danzhou Hainan 571737, China; 2. School of Horticulture and Landscape Architecture, Hainan University, Haikou 570228, China)
Abstract:Core collection construction could greatly improve the utilization efficiency of genetic resources. Taking 410 sweet pepper germplasm as tested materials, genotypic values of eight traits were unbiasedly predicted by using linear mixed model analysis method. Genetic distances among accessions were calculated using Mahalanobis distance based on genotypic values. The core collections of sweet pepper were constructed with 25% sampling ratio by using two cluster methods (single linkage and UPGMA) and two sampling strategies (random sampling and deviation sampling), respectively. The core collections derived from different sampling strategies and cluster methods were evaluated with the mean, variance, range and coefficient of variation. The results showed that the single linkage cluster method could significantly increase the variances and coefficients of variation of eight traits, obviously better than UPGMA; the deviation sampling was better than the random sampling. Total 102 core accessions of sweet pepper, selected based on Mahalanobis distance, single linkage cluster and deviation sampling, could represent thebook=47,ebook=27genetic diversity of the initial collection. This study laid the foundation for the identification of favorable genes and breeding of new varieties in sweet pepper.
Key words:sweet pepper; genotype value; cluster method; sampling strategy; core collection
作者简介:刘子记(1982-),男,副研究员,博士,研究方向为蔬菜分子生物学及遗传育种。E-mail:liuziji1982@163.com
基金项目:中国热带农业科学院院本级基本科研业务费专项资金(1630032015003)
收稿日期:2015-07-15
中图分类号:S641.3
文献标志码:A
文章编号:1005-9369(2016)01-0021-09

