概率主成分分析联合支持向量机的前列腺SELDI-TOF质谱数据分析方法研究
李肃义,嵇梦颖,徐 壮,王跃洋,申博文,熊文激
(1.吉林大学 仪器科学与电气工程学院,吉林 长春 130061;2.吉林大学 化学学院,吉林 长春 130012;
3.吉林大学第一医院,吉林 长春 130021)
概率主成分分析联合支持向量机的前列腺SELDI-TOF质谱数据分析方法研究
李肃义1,嵇梦颖1,徐壮1,王跃洋1,申博文2,熊文激3*
(1.吉林大学仪器科学与电气工程学院,吉林长春130061;2.吉林大学化学学院,吉林长春130012;
3.吉林大学第一医院,吉林长春130021)
摘要:基于前列腺癌检测中获取的表面增强激光解吸/离子化飞行时间质谱 (SELDI-TOF-MS)数据,提出一种概率主成分分析(PPCA)联合支持向量机(SVM)的分类方法。对临床322例血清样本的质谱数据进行特征提取,以随机选取训练样本集(225例)构造SVM判别模型,对剩余样本集(97例)进行测试。采用均方根误差、识别率与预测率指标,将所构造的PPCA-SVM模型分别与偏最小二乘(Partial least squares,PLS)和PCA-SVM模型进行比较,发现PLS模型的识别率和预测率分别为90.92%和76.38%,PCA-SVM模型分别为99.23%和 84.63%,而PPCA-SVM模型分别为99.01%和90.41%。因此SELDI-TOF-MS技术结合PPCA-SVM在样品分类中具有准确、重复性好等优点,为前列腺癌早期诊断提供了一种新方法。
关键词:前列腺癌;概率主成分分析;支持向量机;SELDI-TOF-MS
前列腺癌是威胁男性生命和健康的重大疾病,在全世界范围内具有较高的发病率与死亡率[1]。早期的及时、准确诊断是前列腺癌控制及治疗的关键。近年来,SELDI-TOF-MS技术通过探察生命复杂体内蛋白质分子,分析癌症细胞相对于正常细胞中蛋白质丰度表达的差异,筛查出与癌症相关的生物标记物,为实现癌症的早期检测提供了新的技术和平台[2-6]。然而利用SELDI-TOF-MS技术产生的质谱数据海量、信息冗余,尤其患者多时,海量数据会严重影响对患病程度的判断。因此,如何有效提取与疾病相关的特征参数,准确建立特征参数与患病程度之间的映射关系是SELDI-TOF-MS技术检测早期癌症的关键问题。
Lamberto等[7]提出了基于PCA的食用油甘油三酯质谱分析方法,作为常见的提取特征方法,降低了模型多参数计算量和分析问题的复杂性;徐琨等[8]利用偏最小二乘法(PLS)建立了健康者与乙肝患者的血清蛋白质图谱数据分类模型,但当数据类别增多时,质谱数据和样本类别之间非线性因素会随之变大,从而影响PLS的预测效果,产生较大的检测误差[9];Miller等[10],余小兰等[11],曹素梅等[12]分别提出基于人工神经网络的质谱数据分类方法,但人工神经网络的学习目标为训练误差最小化,导致所建模型的泛化能力较低;为了改善人工神经网络分析质谱数据存在的不足,Marchiori等[13]利用支持向量机SVM构建质谱数据分析判别模型,预测率较人工神经网络得到显著提高;张玉玺等[14]通过比较K最邻近法、助推法、分类回归树与支持向量机几种方法构建的质谱数据分析模型,进一步证明了SVM的预测结果更优;Suarez 等[15],Lokhov等[16],王春艳等[17]分别将PCA联合SVM方法用于质谱和光谱数据分析,取得了较好效果。但由于PCA基于重建方差最小投影原理,具有缺少概率模型结构和缺失高阶统计量信息的不足[18]。概率主成分分析(PPCA)则将传统PCA中丢弃的非主成分因子以噪声方差估计的形式对因子载荷矩阵进行约束[19],然后通过最大期望算法估计参数而得到最佳概率模型,因此PPCA可以更有效地从高维数据中寻找到主成分方向,取得较PCA更优的特征提取效果。
综上分析,本文提出了一种PPCA联合SVM的前列腺SELDI-TOF-MS数据分类方法。通过PPCA技术挖掘与提取前列腺癌、前列腺炎患者以及健康者的特征参数;再通过SVM方法建立预测模型;并且利用识别率与预测率指标,将该方法与传统的PLS、PCA-SVM方法进行比较,以验证PPCA联合SVM分类方法的有效性。
1实验部分
实验部分包括实验数据的获取及预处理、PPCA-SVM分类方法的建立与方法评估。程序编写基于MATLAB2013a及台湾大学林智仁等开发的LIBSVM工具箱[20]。
1.1实验数据
FDA-NCI Clinical Proteomics Program Website是由美国国立卫生研究院NIH联合美国食品和药物管理局FDA共同创建的临床蛋白质研究资源网站[21],可提供利用h1蛋白质芯片(Ciphergen Biosystems,Inc.,Palo Alto,CA,USA)结合Ciphergen PBS1 SELDI-TOF质谱技术分析血清样本获取的前列腺癌数据。该数据集共包含322例血清样本的质谱数据,其中包括63例健康者、190例前列腺炎患者与69例前列腺癌患者(43例晚期癌症患者,26例早期癌症患者);每组数据中包含15 154个蛋白质表达丰度值,图1为从SELDI-TOF-MS中任选的3类代表性蛋白质表达丰度图谱。
322个样本随机分为训练样本集(70%)和测试样本集(30%),每组包含健康者、前列腺炎患者和前列腺癌患者,表1为每组中的具体样本数。

表1 训练组与测试组中包含的具体样本数
从图1可看出,原始图谱中含有基线漂移、随机噪声和高频化学噪声,并且有效信号大多集中在1 000≤m/z≤10 000区间。本实验根据质谱噪声与有效信号的主要特征,通过适当的预处理,降低各种非目标因素对质谱的影响,净化图谱信息,避免噪声与基线漂移等引起的特征参数计算误差。预处理的具体步骤如下:信号截取:由图1可看到,m/z<1 000主要被高频化学噪声所污染,m/z>10 000特征的差异不显著,因此,截取1 000≤m/z≤10 000之间的数据进行后续分析。基线校正:通过做递归直方图估计基线,窗口长度为200,对质谱信号进行基线校正。谱线平滑:利用局部加权回归散点平滑法对质谱信号中引入的电子噪声与随机噪声进行平滑滤波。
1.2PPCA-SVM分类方法
1.2.1数据集前列腺癌检测质谱样本集{sn,n=1,2,……,N},其中N为322,代表样本个数。每个样本的输入向量为xn,p,其中p代表维数,由预处理后的7 327维的质谱表达丰度值组成;输出向量{Yn=-1,0,1}对应分类结果,1代表前列腺癌患者,0代表前列腺炎患者,-1代表健康者。随机选取225(70%)例质谱数据作为训练集,97(30%)例质谱数据作为测试集。
1.2.2特征提取预处理后的SELDI-TOF-MS数据具有高维特性,采用降维技术进行特征提取不仅可以简化模型结构,还可提高训练与检测速度。本文利用PPCA对训练集样本预处理后的质谱数据进行降维与特征提取。主要实现步骤如下:
利用高斯噪声ε~N(0,σ2I)描述特征空间的非主成分因子,建立隐变量模型映射d维质谱数据s与其q维特征矩阵x之间的关系:s=Wx+μ+ε,其中W为d×q维因子载荷矩阵,μ为s的非零均值。
在以上隐变量模型下,建立特征矩阵条件下质谱数据的概率分布,依据贝叶斯概率公式,推导出特征矩阵关于质谱数据的后验概率密度分布。
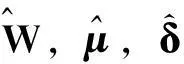
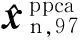
1.2.3SVM建模近年来SVM在临床医学的疾病诊断、预测以及筛查等方面得到广泛应用[22]。
核函数选取及模型参数估计:SVM核函数选用较稳定的RBF核函数,在训练前给定正则化参数和核函数参数的范围,即c∈[-10,10],g∈[-10,10],使c和g在这个范围内遍历取值,每个子集验证1次,对每一个组合参数均进行交叉验证求取SVM模型参数。
模型训练及测试:随机选取225例样本建立PPCA-SVM训练模型,利用剩余的样本作为测试集对模型进行检验,重复进行10次实验。
1.3模型性能评价
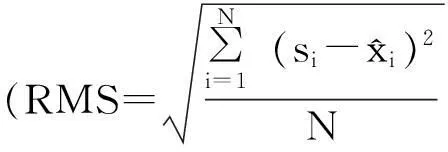
为了评价模型的分类效果,利用随机选取的训练样本集与测试样本集,通过比较识别率与预测率,对PLS模型、PCA-SVM与PPCA-SVM3种模型进行了分类准确率的评估,同时,为验证模型的稳定性,避免实验的随机性,将此过程重复10次,最后通过平均识别率与预测率来评价模型性能。
2结果与讨论
2.1预处理结果
原始图谱中含有基线漂移、随机噪声和高频化学噪声,通过有效信号截取,基线校正与谱线平滑等预处理降低各种噪声对有效信号的影响,避免后续的特征参数计算误差。任意选取某一正常血清样本的原始质谱数据(如图2A),预处理后的谱线如图2B所示。由图可知,预处理后的信号较为有效地校正了原始信号中的基线漂移(8 500≤m/z≤10 000区间较为明显),抑制了部分电子噪声与随机噪声(1 000≤m/z≤4 000区间)。
2.2质谱数据特征提取结果
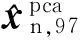
2.3模型对比结果
通过交叉验证法求得PPCA-SVM模型参数,然后对PLS,PCA-SVM与PPCA-SVM 3种模型的识别率及预测率进行评估。表2分别给出了PLS,PCA-SVM,PPCA-SVM模型的10次平均识别率与预测率,PLS模型的平均值分别为90.92%和76.38%,PCA-SVM模型的平均值分别为99.23%和84.63%,PPCA-SVM模型的平均值分别为99.01%和90.41%。
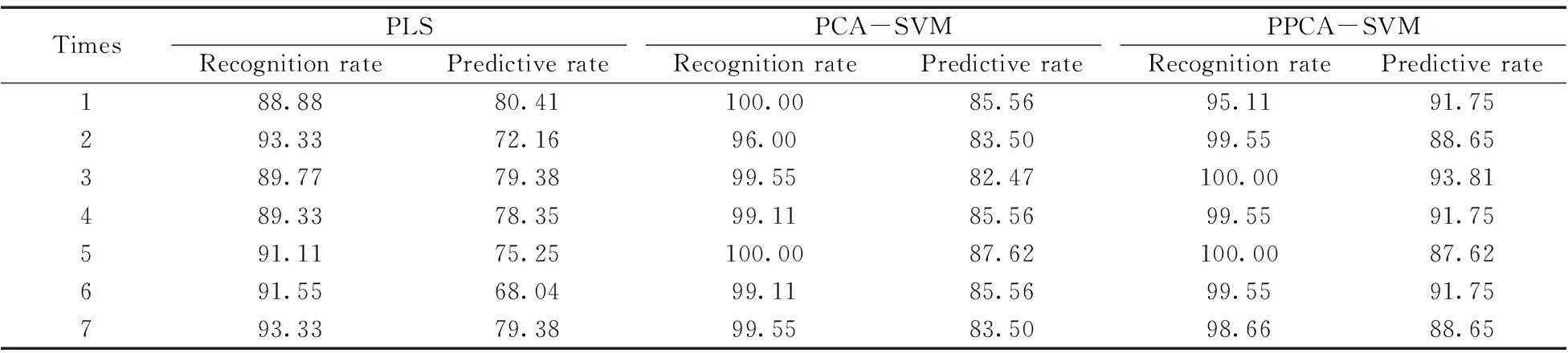
表2 PLS,PCA-SVM,PPCA-SVM模型的平均识别率与预测率
(续表2)
PLS模型集主成分分析与多元线性回归为一体,但其训练目标为经验风险最小化,主要适用于小样本事件,而SVM模型的训练目标则为结构风险最小化。因此,SVM的预测效果更优,其中以PPCA-SVM模型的预测效果最优。
3结论
研究结果表明,3种模型在利用训练集时,识别率均高于90%,但在利用测试集时,PPCA-SVM模型的预测效果最好,其原因主要是由于:①传统PLS模型的学习目标为经验风险最小化,而SVM模型的学习目标则为结构风险最小化,即同时考虑了经验风险与置信误差,因此SVM模型的泛化能力较好,预测效果最优。②PCA与PPCA虽然降低了数据的维度,但PPCA通过最佳概率模型可以更为有效地从高维数据中寻找到主成分方向,更加有效地提取特征参数,因此PPCA-SVM模型的性能优于PCA-SVM模型。基于以上分析与实验验证,本文提出的PPCA-SVM模型优于传统的PLS与PCA-SVM检测模型,具有预测准确率高与稳定性好等特点,为其应用于临床前列腺癌的早期诊断奠定了理论与实验基础。
参考文献:
[1]American Cancer Society.Cancer Facts and Figures 2015[M/OL].Atlanta:American Cancer Society,2015.
[2]CHO W C S.Chin.J.Biotechnol.(曹志成.生物工程学报),2006,22(6):872-876.
[3]Xu C M,Zhang W,Zhang J Y,Liu H,Sun H C,Ma H B,Zhu Y P,Xie H W.Prog.Biochem.Biophys.(徐长明,张伟,张纪阳,刘辉,孙汉昌,马海滨,朱云平,谢红卫.生物化学与生物物理进展),2011,38(6):506-518.
[4]Petricoin E F,Ardekani A M,Hitt B A,Levine P J,Fusaro V A,Steinberg S M,Mills G B,Simone C,Fishman D A,Kohn E C,Liotta L A.Obstetrical&GynecologicalSurvey,2002,57(6):572-577.
[5]Cazares L H,Adam B L,Ward M D,Nasim S,Schellhammer P F,Semmes O J.Clin.CancerRes.,2002,8(8):2541-2552.
[6]Conrads T P,Fusaro V A,Ross S,Johann D,Rajapakse V,Hitt B A,Steinberg S M,Kohn E C,Fishman D A,Whitely G,Barrett J C,Liotta L A,Petricoin E F,Veenstra T D.Endocrine-relatedCancer,2004,11(2):163-178.
[7]Lamberto M,Saitta M.J.Am.OilChem.Soc.,1995,72(8):867-871.
[8]Xu K,Zhu E Y,Yang P Y,Liu Y K.Chin.J.Anal.Chem.(徐琨,朱尔一,杨芃原,刘银坤.分析化学),2009,37(2):211-215.
[9]Bai Y K,Meng X J,Ding D,Shen X G.Spectrosc.SpectralAnal.(白英奎,孟宪江,丁东,申铉国.光谱学与光谱分析),2005,25(3):381-383.
[10]Miller J H,Schrom B T,Kangas L J.MethodsMol.Biol.,2015,1260:89-100.
[11]Yu X L,Yao Y Z.Mod.Med.J.(余小兰,姚永忠.现代医学),2012,40(2):241-244.
[12]Cao S M,Gou X,Chen F J,Yang A K,Chen W K,Li N W.Chin.J.Cancer(曹素梅,郭翔,陈福进,杨安奎,陈文宽,李宁炜.癌症杂志),2007,26 (7):767-770.
[13]Marchiori E,Jimenez C R,West-Nielsen M,Heegaard N H.LectureNotesinComputerScience,2006,3907:79-90.
[14]Zhang Y X,Xiong Q,Yang G,Li M L.Chin.J.Anal.Chem.(张玉玺,熊庆,杨刚,李梦龙.分析化学),2007,35(10):1449-1454.
[15]Suarez E,Hien P N,Israel P O,Lee K J,Kim S B,Jaroslaw K,Kevin A S.Anal.Chim.Acta,2011,706(1):157-163.
[16]Lokhov P G,Kharybin O N,Archakov A I.Int.J.MassSpectrom.,2012,309:200-205.
[17]Wang C Y,Shi X F,Li W D,Ren W W,Zhang J L.J.Instrum.Anal.( 王春艳,史晓凤,李文东,任伟伟,张金亮.分析测试学报),2014,33(3):289-294.
[18]Jolliffe I T.PrincipalComponentAnalysis.Second Edition:Springer Series in Statistics.New York:Springer,2002:1-27.
[19]Tipping M E,Bishop C M.J.RoyalStat.Soc.B,1999,61(3):611-622.
[20]Chang C C,Lin C J.ACMTrans.Intell.Syst.Technol.,2011,2(27):1-27.
[21]http://home.ccr.cancer.gov/ncifdaproteomics/.
[22]Suykens J A K,Vandewalle J.NeuralProcess.Lett.,1999,9(3):293-300.
A SELDI-TOF-MS Data Classification Method for Prostate Based on Probabilistic Principal Components Analysis and Support Vector MachineLI Su-yi1,JI Meng-ying1,XU Zhuang1,WANG Yue-yang1,SHEN Bo-wen2,XIONG Wen-ji3*
(1.College of Electrical Engineering and Instrumentation,Jilin University,Changchun130061,China;2.College
of Chemistry,Jilin University,Changchun130012,China;3.The First Clinical Hospital of Jilin University,
Changchun130021,China)
Abstract:A method combined probabilistic principal components analysis(PPCA) with support vector machine(SVM) was presented for analyzing SELDI-TOF-MS data generated from clinical proteomic study.Using PPCA for feature extraction on 322 MS data set,225 MS data set were randomly selected as learning set for establish SVM model,and the remaining 97 data set were selected as a testing set for prediction and verification.Root mean square error,recognition rate and predictive rate were used to evaluate the model′s classification performance,respectively.To verify the PPCA-SVM model′s classification performance further,the proposed model with partial least squares (PLS) model and PCA-SVM model were compared.The results showed that the recognition rates for PLS,PCA-SVM and PPCA-SVM were 90.92%, 99.23%and 99.01%,respectively,the predictive rates for PLS,PCA-SVM and PPCA-SVM were 76.38%,84.63% and 90.41%,respetively.Experimental results showed that proposed PPCA-SVM model was an accurate and repeatable method for automatically detecting prostate cancer.The method provides a new approach for early diagnosis of prostate cancer in clinic.
Key words:prostate cancer;probabilistic principal components analysis;support vector machines;SELDI-TOF-MS
中图分类号:O657.63;Q461
文献标识码:A
文章编号:1004-4957(2016)01-0091-05
doi:10.3969/j.issn.1004-4957.2016.01.015
通讯作者:*熊文激,博士,教授,研究方向:医学信号处理与肿瘤早期检测技术,Tel:0431-88502382,E-mail:450331530@qq.com
基金项目:国家自然科学基金(201101071);吉林省自然科学基金(20140101063JC)
收稿日期:2015-07-06;修回日期:2015-08-04

