长期移居高原的健康汉族人HIF-2α基因型分布变化及意义
余震坤,常荣,韩永建,陈单单,张晓菲
(1青海省人民医院,西宁810007;2青海大学医学院)
长期移居高原的健康汉族人HIF-2α基因型分布变化及意义
余震坤1,2,常荣1,韩永建2,陈单单2,张晓菲2
(1青海省人民医院,西宁810007;2青海大学医学院)
目的 探讨长期移居高原的健康汉族人低氧诱导因子2α(HIF-2α)基因型分布变化及其意义。方法选择39例移居高原(平均海拔约3 300米)且居住20年以上的健康汉族志愿者(青海汉族组),采用Sanger基因测序法检测其HIF-2α基因14个位点(rs1867785、rs2881324、rs11125068、rs11675441、rs10193827、rs13419896、rs4953360、rs4953361、rs7571879、rs7589621、rs7571218、rs7598371、rs7594912、rs1562453)的基因型,分别与45例北京健康汉族人(北京汉族组,来源于人类基因组单体型图计划数据库)、47例青海健康藏族人(青海藏族组,来源于以往文献)的基因型进行比较。结果青海汉族组和北京汉族组HIF-2α基因14个位点的基因型分布比较差异均无统计学意义(P均>0.05);与青海藏族组比较,青海汉族组和北京汉族组HIF-2α基因9个位点(rs11125068、rs11675441、rs10193827、rs13419896、rs4953360、rs4953361、rs7571879、rs7589621、rs7598371)的基因型分布比较差异均有统计学意义(P均<0.05)。结论长期移居高原的健康汉族人与北京健康汉族人的HIF-2α基因型分布具有一致性,其对高原低氧环境的适应能力与HIF-2α基因型分布无关。
低氧诱导因子2α;基因分布;高原健康汉族;人类基因组单体型图计划数据库
青藏高原是世界最高的高原,世居在这里的藏族人及夏尔巴族人遗传基因分布差异使其对低氧环境产生适应性,也是环境对种群的适应性选择[1]。低氧诱导因子2α(HIF-2α,又称EPAS1)基因为低氧适应的主要遗传基因之一,其多态性与低氧适应密切相关。虽然汉族等其他平原族群移居至高原时容易继发高原红细胞增多症、高原心脏病等慢性高原性疾病(简称慢性高原病),但仍有部分汉族人移居于高原数十年未发生慢性高原病,其是否与HIF-2α基因型分布有关鲜见报道。为此,我们进行了如下研究。
1 资料与方法
1.1 临床资料 选择2015年3~7月青海省人民医院医疗队支援青海省刚察县(平均海拔3 300米)期间招募的39例健康汉族志愿者(青海汉族组),男28例、女11例,年龄(53.13±9.38)岁,BMI 25.93±3.21;收缩压(125.10±12.63)mmHg,舒张压(81.41±5.92)mmHg,肺动脉收缩压(22.47±3.67)mmHg;血红蛋白(152.2±5.6)g/L,血氧饱和度91.79%±3.62%。纳入标准:①符合高原健康人群诊断标准[2];②均由平原移居高原,且在高原居住20年以上;③无异族通婚史;④相互间无血缘关系;⑤无慢性呼吸及心脏疾病病史。采用《第六届国际高原医学和低氧生理学术大会颁布的慢性高原病青海诊断标准》[3],排除合并慢性高原病者。本研究通过青海省人民医院伦理委员会审核,受试对象均知情同意。
于人类基因组单体型图计划(HapMap)数据库(ftp://ftp.ncbi.nlm.nih.gov/hapmap/)[4]中随机选择北京健康汉族人45例(北京汉族组),男22例、女23例,年龄(41.23±11.12)岁。参照柯金坤等[5]的文献,选择青海健康藏族人47例(青海藏族组)。记录北京汉族组和青海藏族组HIF-2α基因14个位点(rs1867785、rs2881324、rs11125068、rs11675441、rs10193827、rs13419896、rs4953360、rs4953361、rs7571879、rs7589621、rs7571218、rs7598371、rs7594912、rs1562453)基因型分布情况。
1.2 青海汉族组HIF-2α基因14个位点基因型检测 采集青海汉族组晨起空腹外周静脉血3 mL,EDTA试管抗凝,4 000 r/min离心10 min,分离中间白细胞层。按照全血DNA试剂盒(批号158389,德国Qiagen公司)标准步骤提取基因组DNA,采用琼脂糖凝胶电泳对DNA进行质检,条带清晰且无拖尾为合格。合格样品送北京博奥晶典生物技术有限公司,检测HIF-2α基因14个位点的基因型。
1.3 统计学方法 采用SPSS19.0统计软件。基因型遗传代表性采用Hardy-Weinberg平衡定律检验;各组基因型频率比较采用χ2检验。P<0.05为差异有统计学意义。
2 结果
青海汉族组、北京汉族组、青海藏族组HIF-2α基因14个位点的基因型经Hardy-Weinberg平衡检验显示均具有遗传代表性(P均>0.05)。青海汉族组和北京汉族组HIF-2α基因14个位点的基因型分布比较差异均无统计学意义(P均>0.05);与青海藏族组比较,青海汉族组和北京汉族组HIF-2α基因9个位点(rs11125068、rs11675441、rs10193827、rs13419896、rs4953360、rs4953361、rs7571879、rs7589621、rs7598371)的基因型分布比较差异均有统计学意义(P均<0.05)。三组HIF-2α基因14个位点基因型分布情况见表1。
3 讨论
HIF-1由Semenza等[6]于1992年首次发现,具有促进红细胞生成素(EPO)合成的作用。HIF-1是由α亚基和β亚基组成的异二聚体,分别为氧调节亚单位与结构亚单位,在低氧环境中共同维持其稳定性,主要作用是促进血管形成以及儿茶酚胺和EPO的生成。α亚基目前有三种同源体[7,8],即HIF-1α、HIF-2α和HIF-3α,其中HIF-1α、HIF-2α的结构及功能具有相似性。青藏高原为低氧环境,机体内的HIF-2α容易被过度激活,导致红细胞增多、组织血管生成紊乱等病理改变,从而引发慢性高原病。基因多态性使得藏族人群能够适应高原低氧环境,不会因低氧环境而发生前述病理改变,是环境对其族群的正选择。
HapMap是由日本、英国、加拿大、中国、尼日利亚和美国的科学家及资助机构合作的国际项目,旨在确定人类遗传基因的相似性和差异性,并绘制出揭示人类遗传多态信息的“单体型图”。HapMap标签单核苷酸分布(SNPs) 可作为科研的参考数据,科研人员将目的基因信息与HapMap数据库进行比对,可能发现与人类健康、疾病和环境因素等相关的个体差异基因。该项目共取样270例正常个体,其中45例为北京健康汉族人。国外多项研究均认为HapMap为实用的关联分析工具,并广泛应用于不同族群基因分布的对比研究[9~11]。基因型的检测结果稳定,只要检测方法正确,一般不会因时间或人为因素的影响而导致结果发生变化,因此本研究直接选择既往柯金坤等[5]研究中47例青海健康藏族人的HIF-2α基因检测结果作为对照。本研究结果显示,青海汉族组和北京汉族组HIF-2α基因14个位点的基因型分布比较差异均无统计学意义,与陈志军等[12]研究结果一致,说明不同海拔的健康汉族人HIF-2α基因型分布差异具有一致性。青海藏族和青海汉族组HIF-2α基因9个位点(rs11125068、rs11675441、rs10193827、rs13419896、rs4953360、rs4953361、rs7571879、rs7589621、rs7598371)的基因型分布比较差异均有统计学意义,说明高原健康汉族能耐受低氧环境可能与HIF-2α的基因多态性无关。
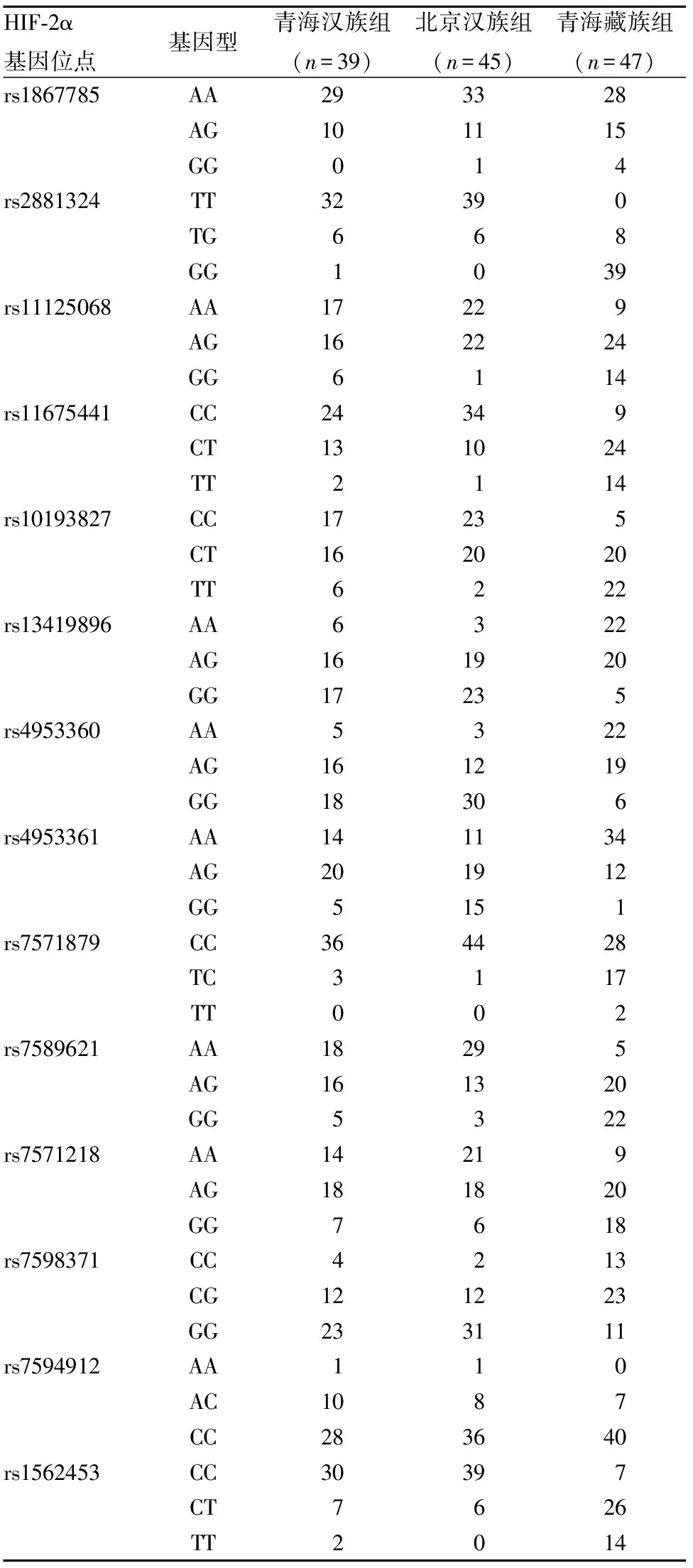
表1 三组HIF-2α基因14个位点基因型分布情况
此外,与低氧密切相关的基因还有EGLNl、PPARA、CYPl7AI、CYP2E1、HOMX2和EDNRA等[13],长期移居高原的健康汉族人对低氧环境的适应是否与上述基因相关还需进一步研究。
[1] Emilia HS, Xin J, Zhuoma B, et al. Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA[J]. Nature, 2014,512(7513):194-197.
[2] 格日力.高原医学[M].北京:北京大学医学出版社,2015:43-67.[3] 国际高原医学会慢性高原病专家小组.第六届国际高原医学和低氧生理学术大会颁布慢性高原病青海诊断标准[J].青海医学院学报,2005,26(1):3-5.
[4] Frazer KA, Ballinger DG, Cox DR, et al. A second generation human haplotype map of over 3.1 million SNPs[J]. Nature, 2007,7164(449):851-861.
[5] 柯金坤,姚宇峰,刘舒媛,等.不同海拔高度低氧环境差异对EPAS1基因多态性的影响[J].中华医学遗传学杂志,2011,28(5):583-588.
[6] Semenza GL, Wang GL. A nuclear factor induced by hypoxia via de novo protein synthesis binds to the human erythropoietin gene enhancer at a site required for transcriptional activation[J]. Mol Cell Biol, 1992,12(12):5447-5454.
[7] Wu D, Potluri N, Lu J, et al. Structural integration in hypoxia-inducible factors[J]. Nature, 2015,7565(524):303-308.
[8] Semenza GL. Hypoxia-inducible factors in physiology and medicine[J]. Cell, 2012,148(3):399-408.
[9] Takeuchi F, Serizawa M, Kato N. HapMap coverage for SNPs in the Japanese population[J]. J Hum Genet, 2008,53(1):96-99.
[10] Lundmark PE, Liljedahl U, Boomsma DI, et al. Evaluation of HapMap data in six populations of European descent[J]. Eur J Hum Genet, 2008,16(9):1142-1150.
[11] Joubert BR, North KE, Wang Y, et al. Comparison of genome-wide variation between Malawians and African ancestry HapMap populations[J]. J Hum Genet, 2010,55(6):366-374.
[12] 陈志军,邱勇,王斌,等.江苏汉族人群基因多态性与HapMap数据库的比较[J].江苏医药,2009,35(4):410-414.
[13] Simonson TS, Yang Y, Huff CD, et al. Genetic evidence for high-altitude adaptation in Tibet[J]. Science, 2010,5987(329):72-75.
国家自然科学基金资助项目(81360301);青海省应用基础研究计划资助项目(2013-Z-743)。
常荣(E-mail: qhschangrong@126.com)
10.3969/j.issn.1002-266X.2016.48.026
R394.3
B
1002-266X(2016)48-0077-03
2016-06-22)

