碳青霉烯类抗生素耐药肺炎克雷伯菌耐药基因分布和分子分型
杨 梦,王 鹏,徐晓倩,熊长辉,刘晓青,袁 辉,潘欢弘
碳青霉烯类抗生素耐药肺炎克雷伯菌耐药基因分布和分子分型
杨 梦,王 鹏,徐晓倩,熊长辉,刘晓青,袁 辉,潘欢弘
目的 探讨碳青霉烯类耐药肺炎克雷伯菌的耐药基因分布及分子分型特征。方法收集38株临床分离的肺炎克雷伯菌,采用肉汤稀释法药敏实验、改良Hodge试验、PCR电泳、脉冲场凝胶电泳分型及多位点序列分型测定其耐药性、耐药基因型及分子型别。结果对碳青霉烯类药物亚胺培南、美罗培南、厄他培南耐药率分别为18.42%(7/38)、28.95%(11/38)、34.21%(13/38),其中有7株(18.42%)同时耐3种药物。对阿米卡星、左氧氟沙星、四环素、呋喃妥因、甲氨苄啶/磺胺甲恶唑、庆大霉素耐药率为23.68%~44.74%,阿莫西林/克拉维酸、头孢唑啉、头孢他啶、头孢曲松、头孢吡肟、氨曲南、环丙沙星耐药率均大于52.63%,氨苄西林、哌拉西林耐药率100%。有11 株肺炎克雷伯菌碳青霉烯酶表型检测为阳性,占28.95%(11/38),其中5株携带blaKPC基因,均为KPC-2型;4株携带blaNDM基因,均为NDM-1型。9株携带blaKPC或blaNDM耐药基因的菌株同源性分析显示有8种不同PFGE带型,5种MLST 型别(ST型)。5种ST型为ST11、ST15、ST395、ST1031、ST1412,其中以ST11为主。结论肺炎克雷伯菌耐药及多重耐药现象严重,肺炎克雷伯菌耐碳青霉烯类药物的主要的原因是携带KPC-2或者NDM-1基因,菌株基因型多态性大,提示病例之间不存在关联性。
肺炎克雷伯菌;碳青霉烯酶;耐药基因;分子分型
肺炎克雷伯菌(Klebsiella pneumoniae)是一种重要的机会致病菌,常导致肺炎、支气管炎、泌尿道和创伤感染等医院感染。由于高耐药性而被广泛关注。碳青霉烯类抗生素是抗菌谱广、抗菌活性最强的一类β-内酰胺酶抗生素,因其具有对β-内酰胺酶稳定以及毒性低等特点,已成为治疗严重细菌感染最主要的抗菌药物之一。但是,随着碳青霉烯类抗生素在临床上使用的增加,碳青霉烯类抗生素的耐药菌株也不断增加,特别是耐碳青霉烯类抗生素的肺炎克雷伯菌快速增加。耐药菌株中表达碳青霉烯酶[1-2],主要是KPC酶和NDM金属酶。本研究对肺炎克雷伯菌临床分离株进行菌种鉴定和药敏试验,筛选出对碳青霉烯类抗生素耐药的肺炎克雷伯菌,并通过脉冲场凝胶电泳(pulsed-field gel electrophoresis typing,PFGE)和多位点序列分型(multilocus sequence typing,MLST)方法进行分子分型研究。
1 材料与方法
1.1 菌株来源 试验菌株为江西省临床实验室分离的肺炎克雷伯菌共38株,分离标本来源为痰液22株、尿液8株、血液4株、引流液2株、伤口分泌物2株,见表1。耐药表型实验和耐药基因检测的质控菌株为大肠埃希菌ATCC 25922、肺炎克雷伯菌ATCC700603,肺炎克雷伯菌ATCCBAA-1705,肺炎克雷伯菌ATCCBAA-1706;PFGE分子量标准参考菌株沙门菌H9812。
1.2 主要仪器 VITEK 2 Compact全自动微生物分析仪 (法国Bio-Mrieux 公司) ;PCR 扩增仪PTC200、脉冲肠凝胶电泳仪CHEF-DRIII system、凝胶成像系统Gel Doc2000(美国Bio-Rad);电泳仪(北京六一厂);恒温培养箱(日本TOKYO RIKAKAI EYELA);微量高速离心机(德国eppdoff)。
1.3 主要试剂 VITEK 2 Compact 全自动微生物分析仪鉴定卡及GN 14药敏卡(法国Bio-Mrieux 公司) ,碳青烯酶类药敏板条(上海星佰有限公司),TaqDNA聚合酶、dNTPs 、DL2000 Marker、限制性内切酶XbaⅠ、蛋白酶K、TBE(大连TaKaRa公司),PCR引物合成(上海生工生物公司),Gold View核酸染料(上海赛百盛公司),琼脂糖(GENE 公司),核酸提取试剂盒(德国QIAGEN公司)。
1.4 菌株复核及抗菌药物敏感性试验 菌株复苏后经VITEK 2 Compact 分析仪鉴定。先用VITEK 2 Compact 全自动微生物分析仪GN14药敏卡进行药物敏感性试验,再用稀释法对碳青烯酶类药物进行分析,判读及结果解释依照2014年美国临床和实验室标准化协会(CLSI) 文件进行。
表1 实验菌株来源
Tab.1 Information of strains

菌株编号StrainID标本Specimen菌株编号StrainID标本Specimenkp6sputumkp37sputumkp7sputumkp38woundsecretionkp8urinekp42bloodkp9urinekp44drainagekp10sputumkp45sputumkp11urinekp49sputumkp12sputumkp52sputumkp16urinekp62urinekp22sputumkp66sputumkp24urinekp70sputumkp25bloodkp71sputumkp26woundsecretionkp72sputumkp27sputumkp76sputumkp28sputumkp77urinekp31sputumkp78urinekp32sputumkp79sputumkp33sputumkp96drainagekp34bloodkp99bloodkp36sputumkp105sputum
1.5 碳青霉烯酶检测(改良Hodge 试验) 将0.5麦氏浊度单位的大肠埃希菌ATCC25922菌液稀释10倍后均匀涂布在M-H平板上,中间贴美罗培南纸片(10 μg /片) ,使用1 μL接种环挑取3个过夜生长实验菌株菌落垂直于药敏纸片从纸片边缘开始划线,长度为20~25 mm,35 ℃孵育16~20 h,美罗培南抑菌圈处出现凹进现象者为改良Hodge试验阳性,提示该菌株产碳青霉烯酶。阳性质控菌为肺炎克雷伯菌ATCC BAA-1705,阴性质控菌为肺炎克雷伯菌ATCCBAA-1706。
1.6 DNA提取 挑取一定量的新鲜培养物悬浊于100 μL纯水,沸水浴30 min后,10 000 r/min离心5 min,吸取上清液即为DNA模板,-20 ℃以下保存。1.7 基因检测与分析 以聚合酶链反应(PCR)法检测耐药基因blaKPC、blaNDM基因,引物由上海生工合成,引物序列和扩增条件详见表2,所有反应体系为25 μL,含有正向引物(F)和反向引物(R)各1 μL、DNA 模板2 μL,10×Buffer 2.5 μL、2.5 mmol/L dNTPs 2 μL、ddH2O 16 μL,Taq酶0.5 μL。反应过程各项参数为:94 ℃预变性5 min;94 ℃ 1 min,退火1 min,(退火温度各基因略有不同),72 ℃ 1min, 30 个循环,72 ℃延伸7 min。PCR 扩增产物经Gold View核酸染料的1.5%琼脂糖电泳。用凝胶成像系统观察结果并拍照。对携带blaKPC、blaNDM基因的菌株采用PCR方法扩增基因全长及测序,所测得序列与GenBank已发表的blaKPC-2、blaNDM-1基因序列比对。
表2 基因引物序列、产物大小及退火温度
Tab.2 Primers product size and annealing temperature used in this study

引物名称Primername引物序列(5'→3')Primersequences产物大小(bp)Productsize退火温度(℃)AnnealingtemperatureblaKPC⁃FATGTCACTGTATCGCCGTCT88256blaKPC⁃FTTTTCAGAGCCTTACTGCCCNDM⁃1⁃UF:TCGCATAAAACGCCTCTG100755NDM⁃1⁃LR:GAAACTGTCGCACCTCAT
1.8 PFGE聚类性分析 参照PulseNet China标准实验方案,实验菌株和沙门菌H9812均选用XbaI酶切。PFGE电泳系统为Bio-Rad公司CHEF DRIII型,电泳电压6 V,夹角120°,脉冲条件为6~36 s,18.5 h。获得的电泳图像以BioNumerics6.6数据库软件进行分型分析。
1.9 MLST分析 对实验菌株的7个管家基因(rpoB、gapA、mdh、pgi、phoE、infB、tonB) 参照http://www.pasteur.fr/recherche/genopole/PF8/mlst/primersKpneumoniae. html 公布的通用引物及退火温度进行PCR扩增并测序,将序列与数据库中储存的序列比对,确定其等位基因谱型及菌株序列型(ST型) 利用官方提供的工具生成Minimum Spanning Trees。
2 结 果
2.1 抗生素敏感性试验结果 根据美国临床和实验室标准化协会(CLSI)推荐的解释标准。 我们分别用Vitek GN14药敏板条和上海星佰公司的碳青霉烯类板条开展了抗生素敏感性试验。
38株肺炎克雷伯菌用GN14板条的药敏结果,对碳青霉烯类药物厄他培南、亚胺培南、美洛培南耐药率为23.68%(9/38),(菌株号分别为KP11、KP22、KP24、KP25、KP28、KP31、KP33、KP36、KP49),阿米卡星、左氧氟沙星、四环素、呋喃妥因、甲氨苄啶/磺胺甲恶唑、庆大霉素耐药率为23.68%~44.74%,阿莫西林/克拉维酸、头孢唑啉、头孢他啶、头孢曲松、头孢吡肟、氨曲南、环丙沙星耐药率均大于52.63%,氨苄西林、哌拉西林耐药率100%,有9株菌同时对3种碳青霉烯类药物耐药。
38株肺炎克雷伯菌用上海星佰公司的碳青霉烯类板条进行试验,其结果为对碳青霉烯类药物亚胺培南、美罗培南、厄他培南耐药率分别为18.42%(7/38)、28.95%(11/38)、 34.21%(13/38),其中有7株(18.42%)同时耐3种药物(菌株号分别为KP11、KP12、KP22、KP24、KP25、KP26、KP28、KP31、KP33、KP36、KP42、KP44、KP49)见表3。
表3 38株肺炎克雷伯菌菌株的药敏结果
Tab.3 Antibiotic sensitivity results of 38 K. pneumonia strains

抗菌素名称antibiotics检测菌株数No.ofstrainstested敏感菌株数No.ofsensitivestrains中介菌株数No.ofintermediarystrains耐药菌株数No.ofresistancestrains耐药率%Resistantrate亚胺培南Imipenem38274718.42美罗培南Meropenem382701128.95厄他培南Ertapenem382501334.21
2.2 碳青霉烯酶检测结果 经改良 Hodge 试验检测,38株肺炎克雷伯菌中有11 株产碳青霉烯酶,占28.95%,分别为KP7、KP11、KP22、KP24、KP25、KP28、KP33、KP36、KP45、KP49、KP105。
2.3 耐药基因PCR检测及测序结果 采用blaKPC、blaNDM基因特异引物进行PCR扩增及产物测序,确定菌株是否携带blaKPC或blaNDM基因及其型别。38株肺炎克雷伯菌经PCR扩增检测出5株携带blaKPC基因(KP25、KP28、KP33、KP45、KP49、),4株携带blaNDM基因(KP11、KP22、KP24、KP105),KPC或NDM基因阳性的,通过测序和BLAST比对,与KPC-2或NDM-1基因完全一致。
2.4 PFGE 结果分析 对携带blaKPC-2、blaNDM-1耐药基因的9株菌株进行PFGE分型分析,经XbaⅠ内切酶酶切后进行PFGE分型,其中1株菌(KP45)发生DNA降解未获得图谱,其余8株PFGE图谱条带清晰。将图谱录入BioNumerics软件进行分析和构建聚类树(图1)。结果显示,8株菌株中PFGE带型各不相同,相似系数在56%~71%之间。
2.5 MLST分析 通过MLST分型,9株菌分为5种MLST 型别(ST型)。其中携带blaKPC-2基因的5株菌分为2种ST型,其中4株为ST11型,1株为ST15型;4株携带blaNDM-1基因的菌株分为4种不同的ST型,分别为ST15、ST395、ST1031、ST1412。见表4、图2。

图1 8株肺炎克雷伯菌PFGE结果聚类图Fig.1 Cluster tree of eight K.pneumonia strains based on PFGE patterns
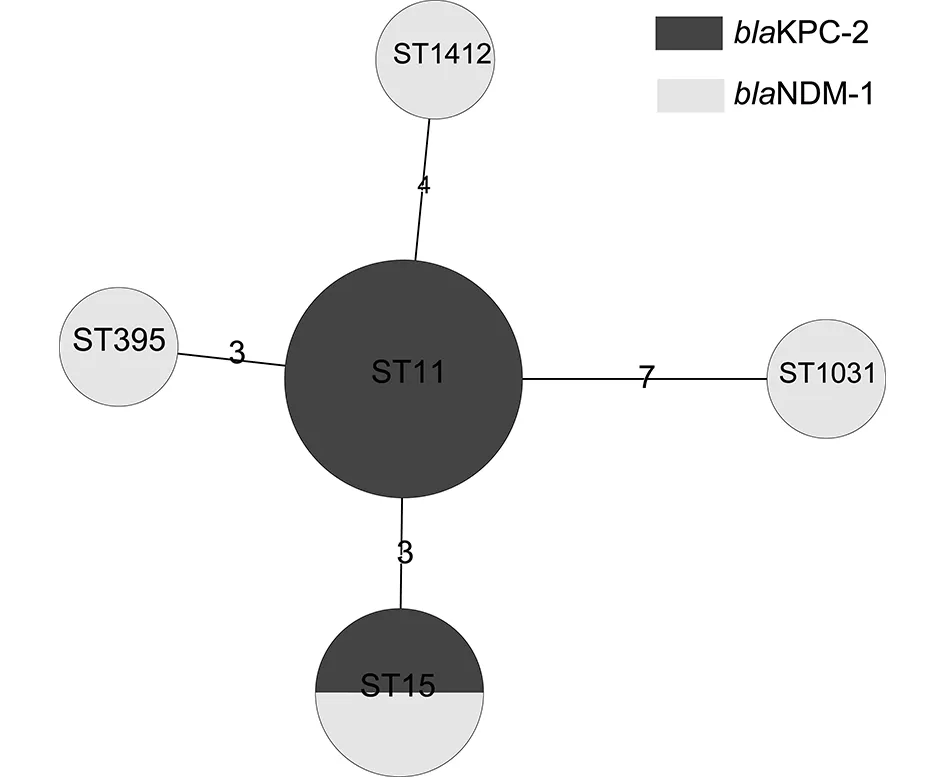
图2 9株肺炎克雷伯菌MLST分型最小生成树Fig.2 Minimum spanning tree of nine K.pneumonia strains based on MLST
表4 MLST等位基因和耐药基因结果
Tab.4 Results of MLST and distribution of antibiotic resistance genes

实验菌株StrainID耐药基因AntibioticresistancegenesMLST等位基因谱MLSTprofileST型MLSTtypeJX2014KP11NDM⁃13⁃1⁃2⁃4⁃1⁃1⁃4ST⁃395JX2014KP22NDM⁃11⁃1⁃1⁃1⁃1⁃1⁃1ST⁃15JX2014KP24NDM⁃12⁃5⁃1⁃1⁃4⁃1⁃18ST⁃1412JX2014KP25KPC⁃21⁃1⁃1⁃1⁃1⁃1⁃1ST⁃15JX2014KP28KPC⁃23⁃3⁃1⁃1⁃1⁃1⁃4ST⁃11JX2014KP33KPC⁃23⁃3⁃1⁃1⁃1⁃1⁃4ST⁃11JX2014KP45KPC⁃23⁃3⁃1⁃1⁃1⁃1⁃4ST⁃11JX2014KP49KPC⁃23⁃3⁃1⁃1⁃1⁃1⁃4ST⁃11JX2014KP105NDM⁃118⁃22⁃18⁃23⁃134⁃13⁃51ST⁃1031
3 讨 论
肺炎克雷伯菌是最常见的医院感染菌,可引起下呼吸道、泌尿生殖道、血流、伤口及颅内等感染。随着碳青霉烯类抗生素这类药物在临床上的广泛应用,碳青霉烯类耐药的肺炎克雷伯菌菌株逐渐增多并出现播散的现象,给临床的治疗带来了极大的困难。我们从临床病例标本中分离出的38株肺炎克雷伯菌,经改良Hodge试验结果显示,11株菌产碳青霉烯酶,可见产碳青霉烯酶是造成肺炎克雷伯菌对碳青霉烯酶类抗菌药物敏感性下降的一个重要原因[3]。
碳青霉烯酶依据Ambler分类有A、B和D 3类,我国耐碳青霉烯类抗生素的肺炎克雷伯菌主要以A类和B类为主。A类碳青霉烯酶,最多见的是KPC型碳青霉烯酶,我国多以KPC-2为主;B类碳青霉烯酶,即金属酶,包括 IMP、VIM、SPM、GIM、SIM、SFO 和 NDM-1 等[4],最近几年报道的超级细菌携带的NDM-1也属于B类酶;NDM-1首次在印度引起暴发感染以来,陆续在世界各地都有报道发现NDM-1引起的感染[5]。中国疾病预防控制中心传染病预防控制所2010年首次报道了在屎肠球菌中分离到3株NDM-1细菌。
本实验有2株菌Hodge试验阳性(KP7、KP36),但未检测到blaNDM或blaKPC基因。这2株菌可能产其它类型的碳青霉烯酶,例如IMP、VIM等。另外有部分菌株的抗生素敏感性试验结果对碳青霉烯类抗生素耐药,但是Hodge试验阴性,提示可能存在产酶以外的耐药机制。
PFGE可以检测菌株之间可能存在的流行病学关联性。本研究中,8株携带blaNDM-1或blaKPC-2基因的肺炎克雷伯菌,带型各不相同,表明存在不同的来源,为散发病例,提示江西虽然检出携带blaNDM-1肺炎克雷伯菌菌株,但未造成局部流行。目前全球范围内肺炎克雷伯菌播散最广的克隆是ST258,而中国报道的主要流行株是ST11[6],本研究结果显示ST-11为产KPC的肺炎克雷伯菌中主要的克隆型 (4/6),与文献[6]报道的一致。ST-11与 ST-258只有一个单基因位点变异(tonB) ,这表明他们之间密切相关。ST-11和ST-258属于同一个克隆群,CC258。而产NDM-1的肺炎克雷伯菌克隆型为ST-15、 ST-395、ST-1412、ST-1031。ST-15在全球流行非常广泛,如美国、丹麦、匈牙利、韩国、马来西亚、新加坡、中国大陆和中国台湾均有报道[7-9]。本研究检出2株ST-15,产NDM-1和产KPC-2的菌株各1株。此外,ST395属于CC258 的亚群 CC292,本研究产NDM-1肺炎克雷伯菌也有1株。此次还检测到ST-1412新的ST型,已被MLST数据库确认收录。
KPC型碳青霉烯酶是一种非金属碳青霉烯酶,可水解碳青霉烯类、青霉素类、头抱菌素类和氨曲南等抗生素。由携带KPC基因的肺炎克雷伯菌株所致的医院感染,由于耐药水平极高,并可在医院内传播扩散,对临床患者的治疗造成了极大的困难,因此医院必须加强监测和控制,以减缓耐药菌株的产生和传播。
携带NDM-1基因细菌的高耐药性已经成为威胁人类公共健康的主要危险和治疗感染性疾病的主要挑战。NDM-1金属酶较其他碳青霉烯酶水解活性强,编码该酶的基因位于质粒等移动遗传因子上,使得该类酶基因可在细菌间广泛传播,我们应该系统的监测blaNDM-1在革兰阴性的临床菌株之间的传播和扩散。
[1] Queenan AM, Bush K. Carbapenemases: the versatile beta lactamases[J]. Clin Microbiol Rev, 2007, 20(3): 440-458. DOI:10.1128/CMR.00001-07
[2] Oteo J, Delgado-Iribarren A, Vega D, et al. Emergence of imipenem resistance in clinicalEscherichiacoliduring therapy[J]. Int J Antimicrob Agents, 2008, 32(6): 534-537. DOI: 10.1016/j.ijantimicag.2008.06.012.
[3] WANG J,LI J,L CY, et al. Detection of β-lactamases and genetyping in carbapenem-resistantKlebsiellapneumoniae[J]. Chin J Clini Lab Sci. 2012, 30(3): 201-204.
王靖, 李杰, 李春英, 等. 碳青霉烯类耐药肺炎克雷伯菌 β-内酰胺酶检测和基因分型[J]. 临床检验杂志, 2012, 30(3) : 201-204.
[4] Ellington MJ, Kistler J, Livermore DM, et al. Multiplex PCR for rapid detection of genes encoding acquired metallo-b-lactamases[J]. J Antimicrob Chemother, 2007, 59(2): 321-322. DOI:10.1093/jac/dkl481
[5] Yong D, Toleman MA, Giske CG, et al. Characterization of a new metallo-β-lactamase gene,blaNDM-1, and a novel erythromycin esterase gene carried on a unique genetic structure inKlebsiellapneumoniaesequence type 14 from India[J]. Antimicrob Agents Chemother, 2009, 53(12): 5046-5054. DOI:10.1128/AAC.00774-09
[6] Qi Y, Wei Z, Ji S, et al. ST11, the dominant clone of KPC-producingKlebsiellapneumoniaein China[J]. J Antimicrob Chemother, 2011, 66(2): 307-312. DOI: 10.1093/jac/dkq431
[7] Kitchel B, Rasheed JK, Patel JB, et al. Molecular epidemiology of KPC-producingKlebsiellapneumoniaeisolates in the United States: clonal expansion of multilocussequencetype258[J]. AntimicrobAgents Chemother, 2009, 53(8): 3365-3370. DOI: 10.1128/AAC.00126-09
[8] Qi Wang, Bin Li, Alan K, et al. Genotypic analysis ofKlebsiellapneumoniaeisolates in a Beijing hospital reveals high genetic diversity and clonal population structure of drug-resistant isolates[J]. PLoS One, 2013, 8: e57091. DOI:10.1371/journal.pone.0057091
[9] Lee MY, Ko KS, Kang CI, et al. High prevalence of CTX-M-15-producingKlebsiellapneumoniaeisolates in Asian countries: diverse clones and clonal dissemination[J]. Antimicrob Agents, 2011, 38: 160-163. DOI:10.1016/j.ijantimicag.2011.03.020
Resistance genes distribution and molecular typing characteristics of carbapenem-resistantKlebsiellapneumonia
YANG Meng,WANG-Peng,XU Xiao-qian,XIONG Chang-hui,LIU Xiao-qing,YUAN Hui,PAN Huan-hong
(DepartmentofCommunicableDiseaseControlandPrevention,JiangxiProvinceCenterforDiseaseControlandPrevention,Nanchang330029,China)
To reveal the distribution of resistance genes and molecular typing characteristics of carbapenem-resistantKlebsiellapneumoniae,38 clinical carbapenem-resistantK.pneumoniawere collected and analyzed by broth dilution antibiotic susceptibility test,modified Hodge test,PCR and electrophoresis,pulsed-field gel electrophoresis typing (PFGE) and multilocus sequence typing (MLST). Results showed that 7 (18.42%),11 (28.95%),and 13 (34.21%) strains showed resistance to imipenem,meropenem,ertapenem,respectively.Of which,7 strains showed resistance to all three antibiotics,eleven strains showed positive results of modified Hodge test,and among them five and four strains carried KPC-2 and NDM-1 genes respectively. These nine strains showed eight different PFGE patterns,and five MLST types.In coclusion,antibiotic resistance amongK.pneumoniaeis serious. KPC-2 and NDM-1 genes are the main cause ofK.pneumoniaeresistant to carbapenems. There was high genetic diversity exists in these strains, suggesting there might no direct relationship of these strains.
Klebsiellapneumoniae; carbapenemase; resistance gene; molecular typing Supported by grants from the Priority Project on Infectious Disease Control and Prevention (No.2013zx10004203-002-007) and the Science and Technology Plan of Jiangxi Province (No.20123154).
Yuan Hui, Email: jxcdcyuanhui@126.com
国家科技重大专项基金项目 (No.2013zx10004203-002-007),江西省卫计委科技计划(No.20123154)联合资助
袁 辉,Email:jxcdcyuanhui@126.com
江西省疾病预防控制中心,南昌 330029
10.3969/j.issn.1002-2694.2016.012.001
R378.99
A
1002-2694(2016)12-1039-05
2016-06-29;
2016-10-19

