人线粒体转录终止因子1(hMTERF1)蛋白的生物信息学分析
人线粒体转录终止因子1(hMTERF1)蛋白的生物信息学分析
熊伟1,2,杨勇琴1,张海洋1,徐彤1,左绍远1,余敏3
(1. 大理学院基础医学院,云南 大理 671000;
2.云南省昆虫生物医药研发重点实验室,云南 大理 671000;
3.云南大学生命科学学院,昆明 650091)
摘要:为了预测和分析人线粒体转录终止因子1(human mitochondrial transcription termination factor 1, hMTERF1)蛋白的结构与功能。采用生物信息学的方法对hMTERF1进行系统的分析与研究,包括hMTERF1的理化性质、跨膜区和信号肽、亚细胞定位、二级结构功能域、蛋白质的功能分类预测、多重序列比对与系统发育树构建、三级结构建模。结果表明:hMTERF1蛋白分子量为45.78 kD,等电点为9.49,不具有信号肽和跨膜区。该蛋白定位于细胞线粒体,N端1~57个氨基酸为前导肽序列。其二级结构主要为螺旋和无规则卷曲,包含6个MTERF基序, 三级结构显示结果与二级结构预测结果相符。蛋白质多重序列比对和聚类分析显示,hMTERF1蛋白与黑猩猩、大鼠、小鼠等哺乳动物的MTERF1蛋白具有高度同源性,在系统发育树上聚为一簇。hMTERF1的生物信息学分析为进一步探索hMTERF1蛋白的功能提供参考资料和理论依据。
关键词:人类;线粒体转录终止因子1;蛋白质;结构;生物信息学
中图分类号:Q518.2 ; Q811.4文献标志码:A
收稿日期:2014-10-28;修回日期:2014-11-17.
作者简介:操利超,男,硕士研究生,研究方向:生物数据分析;E-mail:caolichao@genomics.cn.
doi:10.3969/j.issn.1672-5565.2015.01.06
Bioinformatic analysis of human mitochondrial transcription
termination factor 1 (hMTERF1)
XIONG Wei1,2, YANG Yongqin1, ZHANG Haiyang1, XU Tong1, ZUO Shaoyuan1, YU Min3
(1.SchoolofBasicMedicalSciences,DaliUniversity,DaliYunnan671000,China;
2.YunnanProvincialKeyLaboratoryofEntomologicalBiopharmaceuticalR&D,DaliYunnan671000,China;
3.SchoolofLifeSciences,YunnanUniversity,Kuming650091,China)
Abstract:To study the structure and biological function of the human mitochondrial transcription termination factor 1 (hMTERF1). Many bioinformatics methods were used to analysis physical and chemical properties, hydrophobicity, transmembrane region, signal peptide, secondary structure, functional domain and assortment, multiple alignment, phylogenetic tree and three-dimensional structures of hMTERF1. The results showed that hMTERF1 was hydrophilic protein and the calculated molecular mass was 45.78 kD and the theoretical isoelectric point was 9.49. No signal peptide and transmembrane regions were found in the protein. hMTERF1 was localized in human mitochodria, and the N-terminal 1~57 amino acids were transit peptide. Main composition of the protein secondary structure were -helix and random coil, and it contains 6 MTERF motif. The three-demensional structure prediction showed similar results with secondary structure prediction. The multiple alignment and clustering analysis showed that hMTERF1 is an orthologous protein belong to MTERF family, and has high similarity to Pongo pygmaeus, Rattus norvegicus, and Mus musculus MTERF1. The bioinformatic analysis will be helpful for the further study of the function of hMTERF1.
Keywords:Homo Sapiens; Mitochondrial transcription termination factor 1; Protein; Structure; Bioinformatics
人线粒体转录终止因子1(human mitochondrial transcription termination factor 1,hMTERF1)是MTERF蛋白家族中发现最早,研究最深入的一个蛋白质。1989年Kruse等在人类细胞中分离纯化到一个促进线粒体DNA转录终止的DNA结合蛋白,该蛋白质与线粒体DNA上16S rRNA基因与RNALeu(UUR)基因分界处的一段28bp序列特异结合,从而提前终止线粒体重链基因的转录[1]。1993年Daga等人将这一蛋白命名为线粒体转录终止因子[2]。近年来由于人们陆续在人类和其它物种中又发现了其它新的高度同源的蛋白质,如MTERF2、MTERF3和MTERF4,因此现在普遍将该蛋白称为MTERF1[3-5]。
MTERF蛋白家族共有4个成员,广泛存在于后生动物和植物中,但目前还没有在真菌中发现同源蛋白质[6-7]。随着人们对线粒体转录调控机制和人类线粒体遗传病的深入研究,线粒体转录终止因子蛋白家族的研究越来越引起人们的重视。研究表明,MTERF1存在于人类、猩猩、大鼠和小鼠等哺乳动物中,参与细胞许多重要的生理过程,如线粒体基因复制和转录调控、氧化磷酸化活性调节、细胞周期调控、细胞增殖与凋亡等,并在其中发挥重要的作用[8-12]。
目前已经确定hMTERF1基因位于人类第7号染色体7q21.2,含有5个外显子和4个内含子,全长cDNA序列为1 968 bp,编码一个由399个氨基酸组成的蛋白质[3-5]。在本研究中,我们利用生物信息学方法和工具对hMTERF1蛋白序列进行系统的预测和研究,通过美国国立生物技术信息中心(National Center of Biotechnology Information, NCBI)数据库和全球蛋白资源数据库(Unified Protein Database, Uniprot)蛋白数据库中检索到的人类和其它物种MTERF1蛋白数据,分析hMTERF1蛋白的氨基酸组成、理化性质、二级结构、功能结构域和三级结构等信息,同时对不同物种的MTERF1蛋白序列进行多重序列比对并构建分子进化树,以期为今后进一步研究该蛋白的功能提供生物信息学参考和理论依据。
1材料与方法
1.1材料
数据资料来源于国际互联网上NCBI和Uniprot蛋白质数据库中已经注册的不同物种MTERF1的核酸及其氨基酸序列(见表1)。
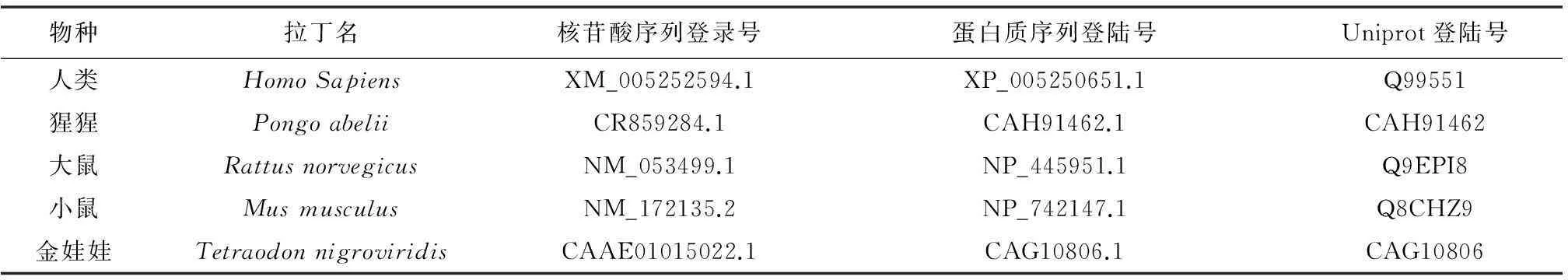
表1 系统发育分析所用的不同物种 MTERF1基因序列的登录号
1.2方法
hMTERF1蛋白的理化性质采用Protparam tool软件预测;亲水性/疏水性采用ProtScale tool软件进行预测,氨基酸分值参数选用HpHob./Kyte and Doolittle;跨膜区域使用TMHMM Server 2.0软件进行预测;信号肽采用SignalP 4.1 Server软件预测;蛋白质亚细胞定位采用TargetP软件、WoLFPSORT软件和PSORT WWW Server中的iPSORT prediction工具进行分析;蛋白质功能分类采用ProtFun 2.2 Server软件进行预测;二级结构分别采用SOPMA、GOR IV和PSIPRED软件进行分析;结构功能域采用SMART软件预测;三级结构预测采用SWISS-MODEL软件进行同源建模;多重序列比对采用Vector NTI 11.0软件进行;系统发育树构建采用MEGA 5.0软件进行。各在线分析软件的网站见表2。

表2 生物信息学研究的数据库和软件网站
2结果与分析
2.1hMTERF1蛋白的理化性质
在GenBank注册的hMTERF1基因包括1 200 bp的开放阅读框(Open reading frame, ORF),编码1个由399个氨基酸组成的蛋白质。采用Protparam tool软件预测hMTERF1蛋白的理化性质,推测该蛋白的分子式为C2 047H3 295N563O588S18,等电点为9.49,相对分子量为45.78,不稳点参数54.27,根据不稳定参数的数值在40以下才是稳定蛋白的标准,可推定hMTERF1为不稳定蛋白[13]。软件预测该蛋白在体外环境下的半衰期为30小时。通过分析该基因编码的氨基酸发现,hMTERF1蛋白由20种氨基酸组成,Leu、Ser和Asn的含量较多,其中Leu的含量高达13.30%。相对含量较少的氨基酸为His和Trp。带正电荷的氨基酸残基(Arg+Lys)总数为57个,带负电荷的氨基酸残基(Asp+Glu)为41个(见图1)。疏水性平均系数(Grand average of hydropathicity, GRAVY)为-0.201,预测该蛋白为亲水性蛋白。
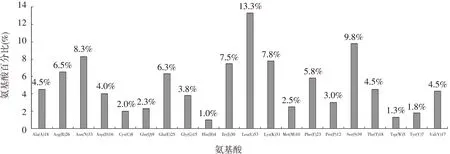
图1 hMTERF1蛋白的氨基酸组成分析
2.2hMTERF1蛋白的跨膜区域与信号肽分析
蛋白质的跨膜区域主要是膜内在蛋白和细胞膜的膜脂相结合的部位。利用TMHMM Server v2.0在线软件对hMTERF1蛋白的跨膜区域分析,结果显示,该蛋白质的跨膜螺旋数量(Number of predicted TMHs)为0,这说明hMTERF1不是跨膜蛋白质,细胞质合成后直接运输到线粒体内发挥作用,与实验报道的hMTERF1蛋白质功能相吻合[3](见图2)。
SignalP是一个信号肽及其剪切位点的预测工具,它采用一个神经网络来区分信号肽和费信号肽,另一个神经网络来识别剪切位点。C值是信号肽酶切位点分值,S值是信号肽分值,Y值是由C值和S值综合得出的剪切位点分值,用于更精确地确定信号肽酶切位点[14]。使用SignalP 4.1 Server在线软件预测mean S score值为0.162,依据mean S score>0.5才能判断为分泌蛋白质的标准,推测hMTERF1蛋白不具有信号肽,这同样说明它是一种在细胞内发挥生理作用的蛋白质(见图3)。
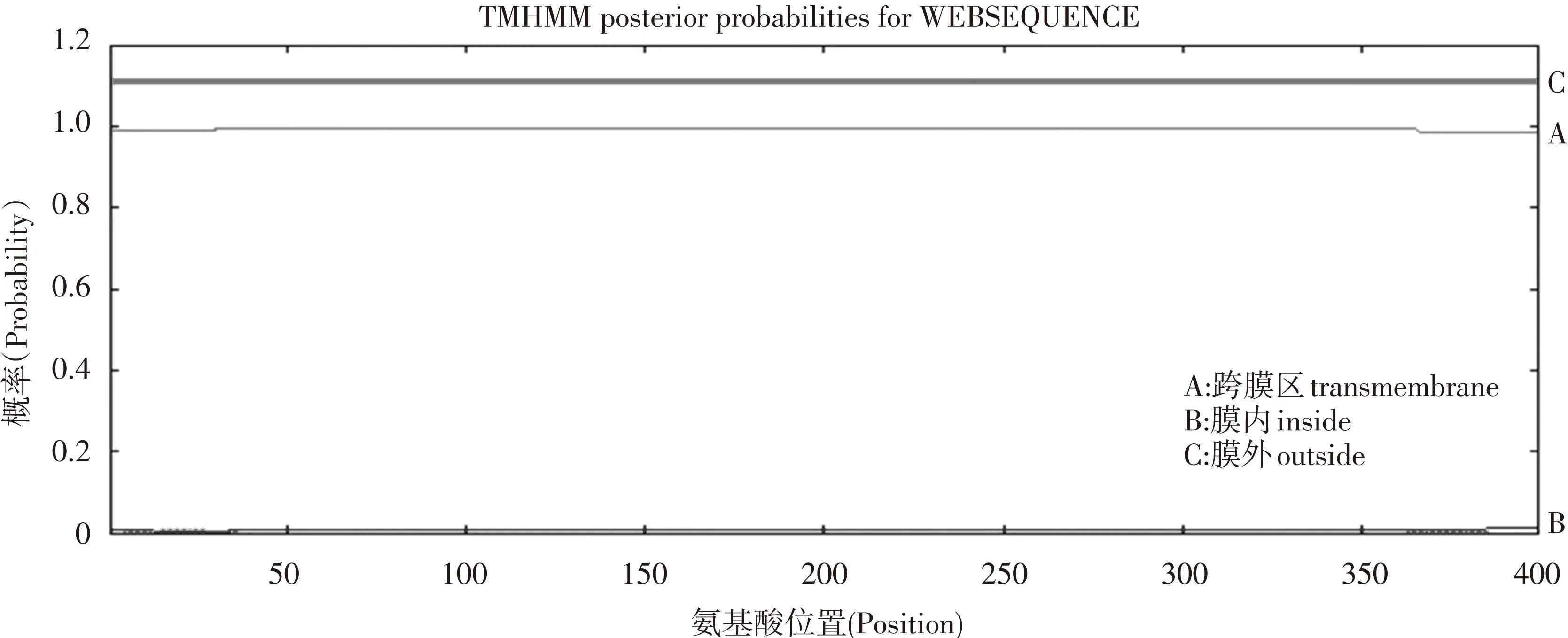
图2 hMTERF1蛋白跨膜域分析
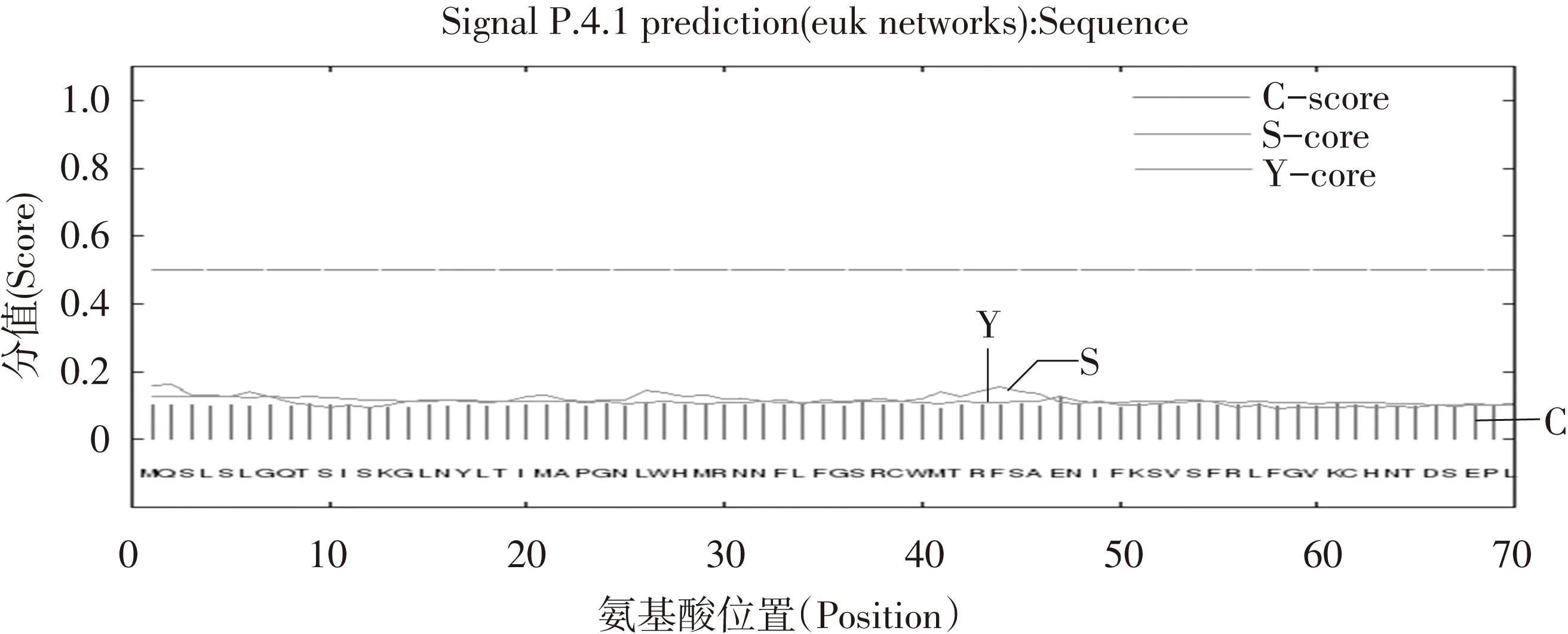
图3 hMTERF1蛋白信号肽分析
2.3hMTERF1蛋白亲水性/疏水性预测和分析
亲水性/疏水性预测和分析对于进一步预测蛋白质的二级结构和结构功能域具有重要的生物学意义,采用ProtScale tool软件进行亲水性/疏水性预测,结果表明,hMTERF1蛋白中亲水性氨基酸占69.98%,而疏水性氨基酸占25.96%,蛋白质第274位氨基酸最大分值为2.244,蛋白质第387位氨基酸最小分值为-2.322(见图4)。依据氨基酸分值越低,亲水性越强的规律,可推测hMTERF1是亲水性蛋白,从整体来看,亲水性氨基酸数量多于疏水性氨基酸,且均匀分布在整个肽链中[15]。
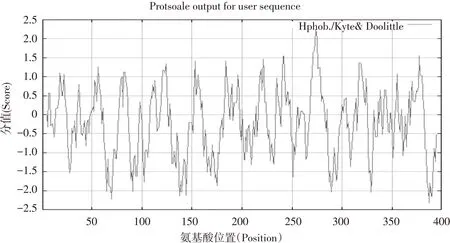
图4 hMTERF1蛋白亲水性/疏水性预测结果
注:参数选用Hphob/Kyte & Doolittle。
Notes:parameter: Hphob/Kyte & Doolittle.
2.4hMTERF1蛋白二级结构预测与分析
蛋白质二级结构主要指蛋白质分子中主链骨架原子依赖氢键排列在一维方向上具有周期性的构象,对其进行预测与分析,将有助于认识蛋白质的高级结构。分别采用SOPMA、GOR IV和PSIPRED软件进行分析,预测的结果非常相似。GOR IV和SOPMA算法是目前常用的蛋白质二级结构算法,具有较高的预测精度,2个不同方法预测hMTERF1蛋白二级结构的结果均表明,hMTERF1蛋白质由42.61%的-螺旋(alpha helix)、14.29%的延伸链(extended strand)、43.11%的无规则卷曲(random coil)构成(见图5)。图中竖线由长至短依次表示-螺旋、延伸链和无规则卷曲,可见-螺旋和无规则卷曲是该蛋白质二级结构的主要构成元件,延伸链出现在局部肽链,且没有-转角(beta-turn)出现。
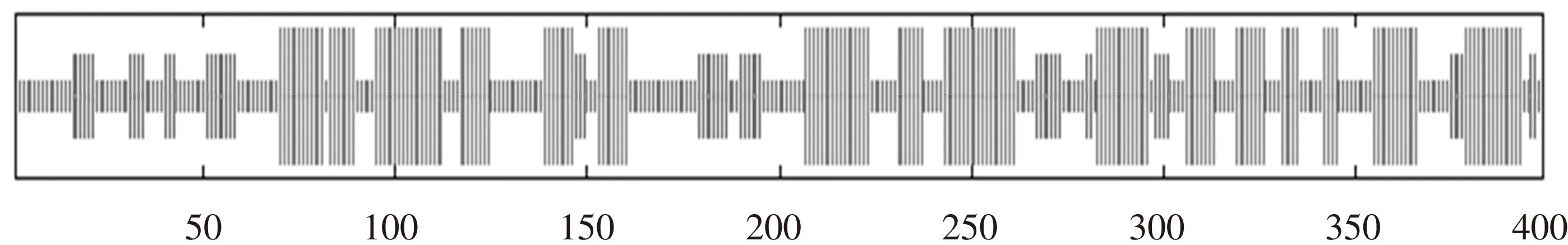
图5 hMTERF1蛋白二级结构柱状图
2.5hMTERF1蛋白结构功能域的预测
结构功能域是指生物大分子中具有特异结构与独立功能的区域,特别是指蛋白质中这样的区域。用SMART在线软件预测hMTERF1蛋白的结构功能域,结果表明,该蛋白质位于171~179,337~348的区域为2个低复杂度区域(low complexity region),在位于122~152,157~189,194~224,235~266,311~341,342~372的区域分别存在6个MTERF基序,每个基序由大约30个左右的氨基酸残基组成(见图6)。
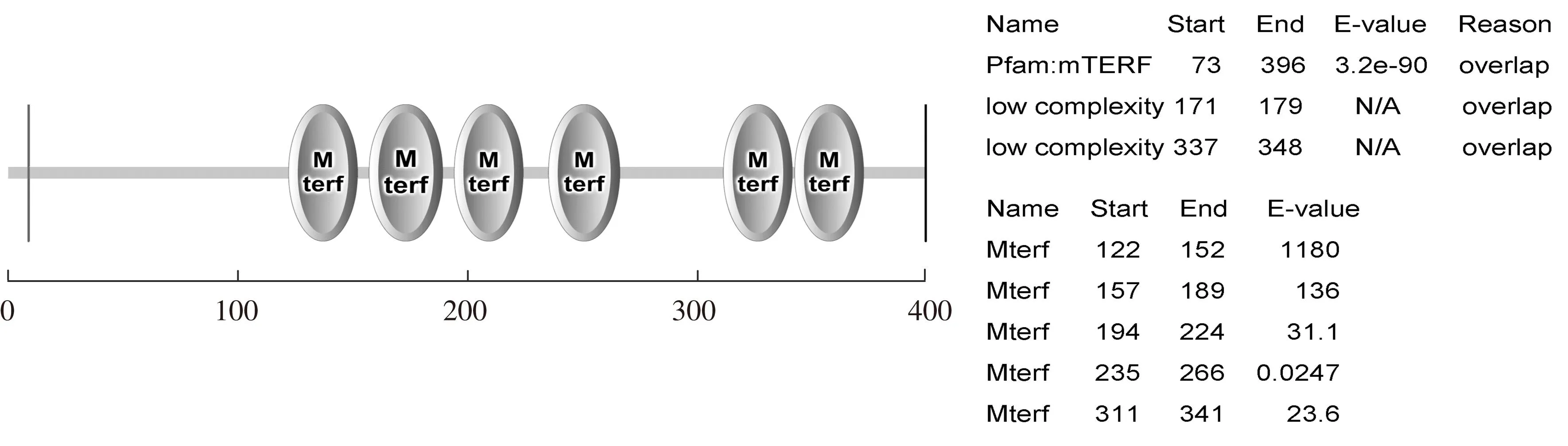
图6 hMTERF1氨基酸序列结构功能域分析
2.6hMTERF1蛋白的亚细胞定位和功能分类
分别使用WoLFPSORT软件和PSORT WWW Server中的iPSORT Prediction工具对hMTERF1蛋白质进行细胞定位,两个不同软件的分析结果均表明hMTERF1蛋白定位于细胞线粒体中,蛋白质N端的1~57个氨基酸是其进入线粒体的前导肽序列。另外,采用TargerP软件预测hMTERF1蛋白亚细胞定位,结果同样显示该蛋白质定位于线粒体。
采用ProtFun软件对hMTERF1蛋白进行功能分类,基因功能分类(Functional category)显示该蛋白可能是转运和结合蛋白质(Transport and binding), 基因本体分类(Gene Ontology category)进一步提示该蛋白是一种转录调控因子(Transcription factor),与实验研究报道的hMTERF1蛋白的功能相符[3,8]。此外,预测结果还显示hMTERF1蛋白不具有酶活性(见图7)。
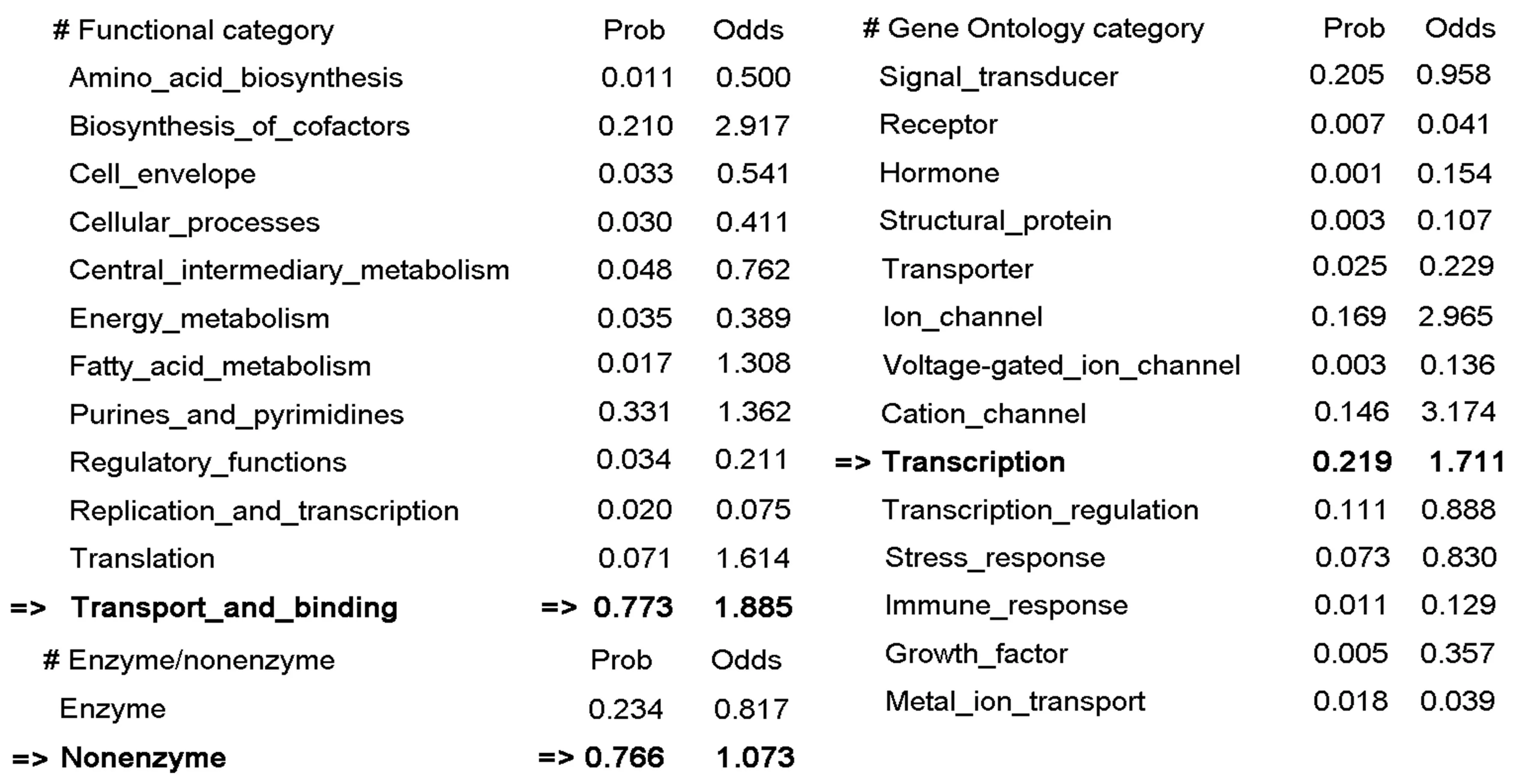
图7 hMTERF1蛋白的功能分类
2.7多重序列比对与构建系统发育树
选取人(Homo Sapiens)、猩猩(Pongoabelii)、大鼠(Rattusnorvegicus)、小鼠(Musmusculus)和金娃娃(Tetraodonnigroviridis)5个物种的直系同源MTERF1蛋白质的氨基酸序列, 通过MEGA6.05软件,分别使用邻接法(Neighbor joining method)、最大似然法(Maximum likelihood method)和最大简约法(Maximum parsimony method)构建系统发育树,结果显示3种方法构建的发育树没有大的差异,说明该蛋白质在不同物种的系统发育树构建正确(见图8)。分析发现人类与猩猩的MTERF1为一个分支类群,大鼠和小鼠的MTERF1为一个分支类群,而金娃娃MTERF1则形成另外一个分支,这与物种之间的亲缘关系和进化关系是一致的[16]。通过Vector NTI11.0软件进行多重序列比对,结果表明这些氨基酸序列具有高度同源的特点,一致性达到76.41%,尤其表现在某些高度保守结构域的氨基酸序列完全一致(见图9)。通过序列比对还发现,hMTERF1与猩猩MTERF1蛋白之间一致性达到98.75%,说明二者的亲缘关系最近。
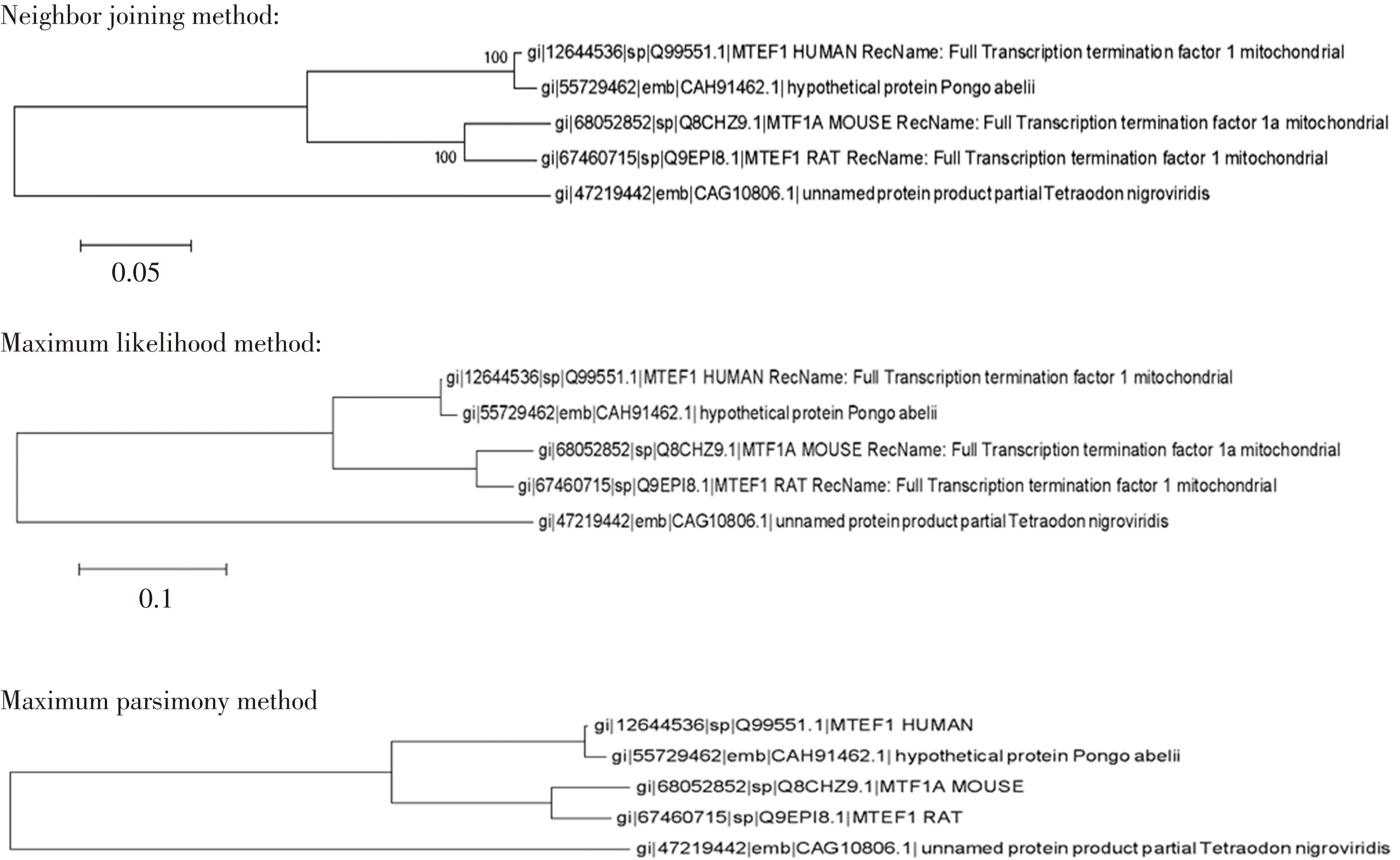
图8 MTERF1蛋白的系统发育树

图9 5个不同物种MTERF1直系同源蛋白的氨基酸序列多重比对
2.8hMTERF1蛋白三级结构预测与分析
采用SWISS-MODEL同源建模的方式得到hMTERF1蛋白的三维预测模型(见图10),经过RasMol软件分析显示该蛋白质外形呈椭球状,主要由-螺旋和无规则卷曲构成,与二级结构预测结果基本一致,说明同源建模的预测结果具有较高的精确度。
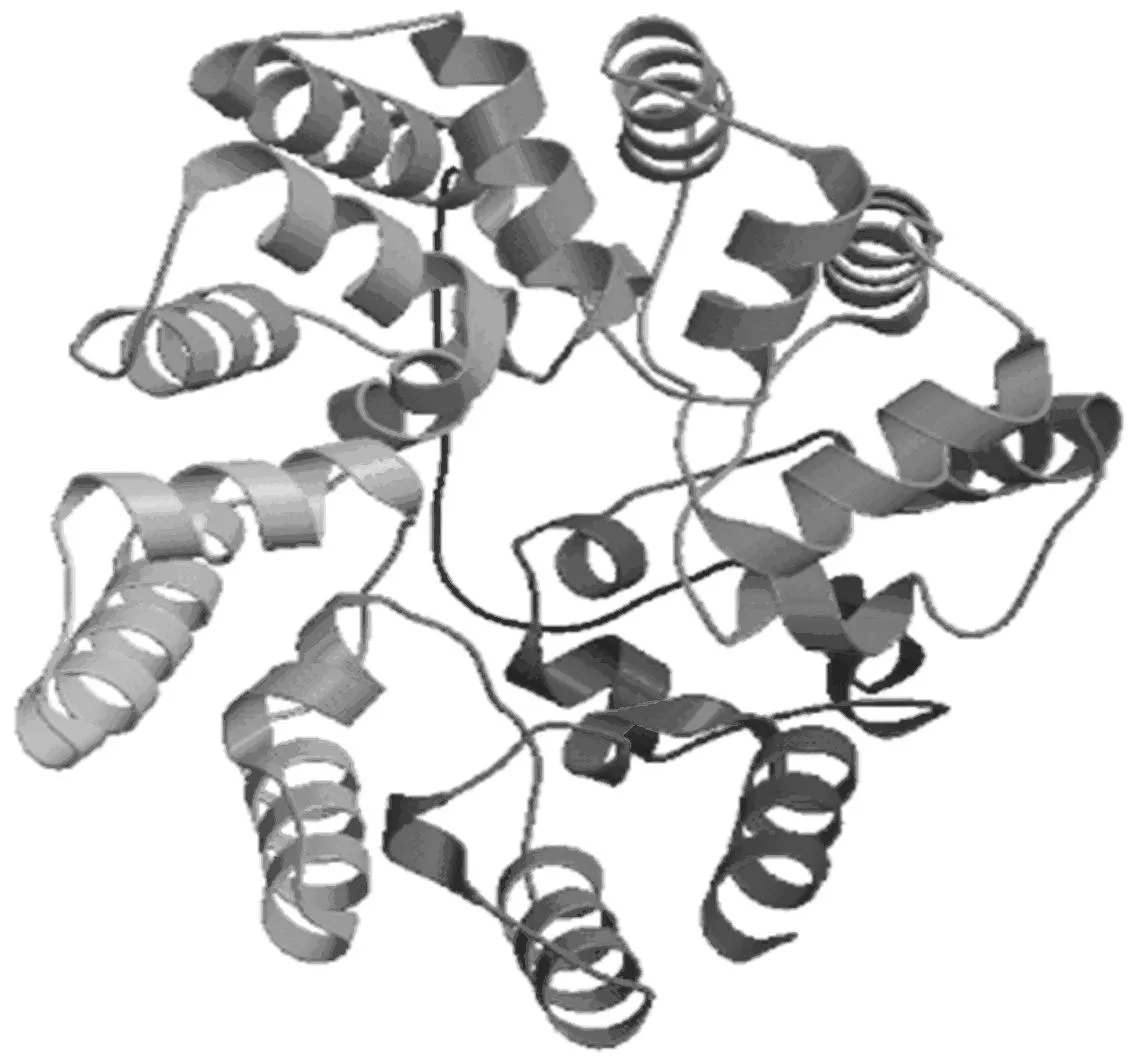
图10 hMTERF1蛋白的三维结构
2讨论
随着计算机技术和生物技术的飞速发展,通过计算机模拟的方式对蛋白质进行理化性质、序列结构和功能等方面进行预测的确信度越来越高[17]。hMTERF1是人类MTERF蛋白家族的一个重要成员,通过生物信息学分析发现该蛋白由399个氨基酸残基构成,是一个分子量为45.78 kD的亲水性蛋白质,且不具有分泌信号肽。亚细胞定位软件预测其定位于线粒体中,蛋白质N端的1~57个氨基酸为前导肽序列,线粒体内成熟的hMTERF1蛋白由342个氨基酸组成,预测结果与文献报道的实验研究结果完全一致[8,12]。通过预测蛋白质的二级结构发现hMTERF1中56.90%的结构是由螺旋和-折叠构成,其余部分都是无规则卷曲。通过SMART在线软件预测该蛋白含有6个MTERF基序,每个基序由大约30个氨基酸残基构成。对其蛋白质功能的预测显示hMTERF1蛋白不具有酶活性,但在调控线粒体基因转录过程中发挥一定的生物学功能,同样与文献报道的实验结果相符[3,8-9]。
通过hMTERF1与猩猩(Pongoabelii)、大鼠(Rattusnorvegicus)、小鼠(Musmusculus)和金娃娃(Tetraodonnigroviridis)4个不同物种MTERF1的系统发育树聚类构建分析发现,该蛋白质属于直系同源蛋白质(orthologous protein),说明MTERF1蛋白在不同物种之间来源于共同祖先的蛋白,能够很好的保留其主要序列以及结构,并且具有相似的生物学功能。由于MTERF1蛋白在不同物种中具有相似功能和共同起源,蛋白质同源家族的分析对于确立物种之间的亲缘关系和预测新蛋白质序列的功能具有重要的意义[18]。本研究为今后进一步研究MTERF1蛋白家族的生物学功能及其它物种的直系同源MTERF蛋白之间的关系奠定了基础。
参考文献(References)
[1]KRUSE B, NARASIMHAN N, ATTARDI G. Termination of transcription in human mitochondria: Identification and purification of a DNA binding protein factor that promotes termination [J]. Cell, 1989, 58(2): 391-397.
[2]DAGA A, MICOL V, HESS D, et al. Molecular characteriation of the transcription termination factor from human mitochondria [J]. J Biol Chem, 1993, 268(11): 8123-8130.
[3]CHEN G, DAI J, TAN S, et al. MTERF1 regulates the oxidative phosphorylation activity and cell proliferation in HeLa cells [J]. Acta Biochim Biophys Sin, 2014, 46(6): 512-521.
[4]TERZIOGLU M, RUZZENENTE B, HARMEL J, et al. MTERF1 binds mtDNA to prevent transcriptional interference at the light-strand promoter but is dispensable for rRNA gene transcription regulation [J]. Cell Metab, 2013, 17(4):618-626.
[5]GUSTAFSSON C M, LARSSON N G. MTERF1 gives mtDNA an unusual twist [J]. Cell Metab, 2010, 12(1): 3-4.
[6]ROBERTI M, POLOSA P L, BRUNI F, et al. The MTERF family proteins: mitochondrial transcription regulators and beyond [J]. Biochim Biophys Acta, 2009, 1787(5): 303-311.
[7]ROBERTI M, POLOSA P L, BRUNI F, et al. MTERF factors: a multifunction protein family[J]. BioMol Concepts, 2010, 1(2): 215-224.
[8]HYVATINEN A K, KUMANTO M K, MARJAVAARA S K, et al. Effect on mitochondrial transcription of manipulating mTERF protein levels in cultured human HEK239 cells [J]. BMC Mol Biol, 2010, 11(9): 72-78.
[9]ASIN-CAYUELA J, SCHWEND T, FARGE G, et al. The human mitochondrial transcription termination factor (mTERF) is fully active in vitro in the non-phosphorylated form [J]. J Biol Chem, 2005, 280(27): 25499-25505.
[10]YAKUBOVSKAYA E, MEJIA E, BYRNES J, et al. Helix unwinding and base flipping enable human MTERF1 to terminate mitochondrial transcription [J]. Cell, 2010, 141(6): 982-983.
[11]RUBINSON E H, EICHMAN B F. Nucleic acid recognition by tandem helical repeats [J]. Curr Opin Stuct Biol, 2012, 22(1): 101-109.
[12]JIMENEZ-MENENDEZ N, FERNANDEZ-MILLAN P, RUBIO-COSIALS A, et al. Human mitochondrial mTERF wraps around DNA through a left-handed superhelical tandem repeat [J]. Nat Struct Mol Biol, 2010, 17 (7): 891-893.
[13]范晶,胥成浩,张西玉,等. 番茄LeNHX3基因的生物信息学分析[J]. 湖北农业科学, 2009,48(12): 2917-2921.
FAN Jing, XU Chenghao, ZHANG Xiyu, et al. Bioinformatics analysis of LeNHX3 gene in tomato[J]. Hubei Agricultural Sciences, 2009,48(12): 2917-2921.
[14]陶隽,贾青,魏星灿,等. 猪ACACA基因及其编码蛋白质的生物信息学分析[J]. 江苏农业科学,2014, 42(5): 42-45.
TAO Juan, JIA Qing, WEI Xingcan, et al. Bioinformatics analysis of ACACA gene and protein in pig[J]. Jiangsu Agricultural Sciences, 2009,48(12): 2917-2921.
[15]钱叶雄,徐士杰,张亚男,等. 玉米精氨酸甲基转移酶蛋白家族生物信息学分析[J]. 生物技术进展,2014, 4(1): 22-29.
QIAN Yexiong, XU Shijie, ZHANG Yanan, et al. Bioinformatics analysis of PRMT protein family in maize[J]. Current Biotechnology, 2014, 4(1): 22-29.
[16]袁保梅,昝玉玺,王天云,等.人14-3-3蛋白家族的生物信息学分析[J]. 生物技术通讯, 2007, 18(4): 587-589.
YUAN Baomei, ZAN Yuxi, WANG Tianyun, et al. Bioinformatics analysis of 14-3-3 protein family of humo sapiens[J]. Letters in Biothchnology, 2007, 18(4): 587-589.
[17]李显航,刘红美. 家蝇小热休克蛋白(sHsp20.6)的生物信息学分析[J]. 生物信息学, 2013, 11(1): 65-71.
LI Xianhang, LIU Hongmei. Bioinformatics analysis of small heat shock protein(sHsp20.6) in musca domestica L[J]. Chinese Journal of Bioinformatics, 2013, 11(1): 65-71.
[18]JAYASHREE B, JONATHAN H C, PRESAD P, et al. A database of annotated tentative orthologs from eropabiotic stress transcripts[J]. Bioinformation, 2006,6(1): 225-227.
*通信作者:严志祥,男,高级工程师,博士,研究方向:生物数据分析;E-mail:yanzhixiang@genomics.cn.

