中国球状绿藻新纪录属—索囊藻属(Choricystis)*
朱 欢 宋会银, 王清华, 胡愈炘, 胡征宇 刘国祥①
(1. 中国科学院水生生物研究所中国科学院藻类生物学重点实验室 武汉 430072 2. 中国科学院水生生物研究所淡水生态与生物技术国家重点实验室 武汉 430072 3. 中国科学院大学 北京 100049)
虫绿藻属(Zoochlorella)是指同淡水原生动物、无脊椎动物共生的一类球状绿藻(coccoid green algae)(Rajević et al, 2015)。该属是 Brandt依据在多种原生动物和无脊椎动物体内分离且能够营自由生活的多株球状绿藻标本而建立, Brandt同时指出该属藻类与小球藻属(Chlorella)的主要区别在于能够与淡水原生动物和无脊椎动物共生(Letsch et al, 2009)。Beijerinck(1890)认为虫绿藻属在自由生活状态下的形态和繁殖方式均与小球藻相似而将虫绿藻属中的种均归入小球藻属。随着人们对淡水原生动物和无脊椎动物共生藻研究的展开, 越来越多的形态与“小球藻”相似的(Chlorella-like algae)种类被归入小球藻科(Chlorellaceae)或虫绿藻科(Zoochlorellaceae)(Fott et al, 1969; Lee et al, 1982; Kessler et al, 1990; Holweg et al, 1994; Pröschold et al, 2011; Rajević et al, 2015)。同时, 不断积累的形态学和植物生理学方面的证据表明虫绿藻属应该被作为小球藻属的异名处理(Fott et al, 1969; Vinayakumar et al, 1975; Kessler, 1976;Reisser, 1984; Douglas et al, 1986; Kessler et al, 1990;Kessler et al, 1992; Takeda, 1995)。1999年修订的国际植物命名法规更正了小球藻属和虫绿藻属, 认为二者是异名; 并依据优先权原则采用小球藻属和小球藻科(Greuter et al, 2000)。
分子生物学同形态学的结合使得人们对小球藻科的系统分类学有了进一步的认识(Huss et al, 1990;Henley et al, 2004; Müller et al, 2005; Darienko et al,2010; Luo et al, 2010; Bock et al, 2011; Pröschold et al,2011; Lee et al, 2012; Krienitz et al, 2015; Rajević et al,2015)。由于小球藻科中的种类形态单一, 传统的仅仅依据形态学证据而建立的种类被分子系统学和其它的分类学手段(如超微结构、病毒抗性等)而更正(Krienitz et al, 2003; Henley et al, 2004; Pröschold et al, 2007; Luo et al, 2010; Bock et al, 2011; Lee et al,2012; Leliaert et al, 2012; Krienitz et al, 2012; Krienitz et al, 2015)。近年来球状绿藻类群中新的分类学修订以及成立的新种属更多地依据分子系统学和超微结构的证据。在小球藻科中, 分子系统发育和 ITS2二级结构被认为是区分不同属种最为有效的手段(Pröschold et al, 2007; Bock et al, 2011; Pröschold et al,2011)。越来越多的分子系统发育研究表明该科模式属小球藻属是一个多系类群。之前被描述为小球藻属或虫绿藻属的种类不断被重新描述和定义, 例如,Chlorella variabilis被认为同Chlorella paramaecii异名; 微芒藻(Micractinium conductrix)与 Zoochlorella conductrix和Micractinium reisseri异名。同时, 虫绿藻属的种类被归入了多个属中, 如小球藻属(Chlorella)、胶球藻属(Coccomyxa)、微芒藻属(Micractinium)、索囊藻属(Choricystis)等(Pröschold et al, 2011; Krienitz et al, 2015; Rajević et al, 2015)。
Fott详细比较了多个胶球藻属的种的形态之后认为, 小胶球藻(Coccomyxa minor)等与模式种Coccomyxa dispar在细胞大小和似亲孢子形成过程方面不同, 建立索囊藻属(Fott, 1976)。最近的基于rDNA序列的分子系统发育和 ITS2二级结构预测研究证实索囊藻属为一个单系类群, 归属于共球藻纲胶球藻科(Darienko et al, 2015; Borowitzka, 2016)。然而基于叶绿体 rbcL序列构建的进化分析显示索囊藻属虽然为一个单系类群, 但并未与胶球藻科聚于一支(Kulichová et al, 2014)。由此可见, 索囊藻属的单系性已为多数分类学家所接受, 然而其具体的系统发育位置仍未有定论。
已有文献资料显示索囊藻模式种的分类历史最早可追溯到 Brandt(1881)描述的寄生虫绿藻(Zoochlorella parasitic)(Guiry et al, 2016)。Bejerinck(1890)将该种归入小球藻属并更名为寄生小球藻(Chlorella parasitica)。Skuja(1948)根据该种的繁殖特征将之归入胶球藻属, 并更名为小胶球藻(Coccomyxa minor)。Fott(1976)详尽地研究了胶球藻属和伪胶球藻属, 并认为小胶球藻与胶球藻属的副模式种 Coccomyxa confluens不同, 将之移出胶球藻属并以此成立索囊藻属。Pröschold等(2011)对两株与针海绵内共生的球状绿藻进行了较为详尽的分子特征鉴定, 认为小胶球藻与寄生索囊藻和小索囊藻异名, 并重新指认寄生索囊藻为该属模式种。此外, 在有记录的索囊藻属的 9个种中, 仅有其模式种(寄生索囊藻)有较为详尽地形态观察和分子特征描述(Guiry et al, 2016)。
绿色淡水海绵(green freshwater sponge)是与绿藻共生而呈现出翠绿色的淡水针海绵(Skelton et al,2013)。在形成的共生体中, 海绵细胞吞噬了大量的球状绿藻但并未消化这些藻类, 而是与之共生(Johnson,2011)。在共生体中, 海绵提供给藻类生长繁殖必须的营养, 藻类将多余的光合产物提供给海绵(Johnson,2011)。然而国内目前对淡水海绵的研究较少, 已有的研究多集中在物种的描述与生态调查方面。对绿色海绵共生藻类的研究仍旧处于空白状态。2015年, 我们在西藏错鄂的湖底和武汉紫阳湖岸边采集到了两株绿色海绵。在室内分析样品时我们发现这两株绿色海绵的共生绿藻均为球状绿藻。通过形态观察、分子系统发育分析和 ITS2二级结构比较, 我们将此两株海绵的共生藻鉴定为寄生索囊藻, 此属为在中国的首次报道。
1 材料与方法
1.1 材料采集和形态观察
两株海绵分别采集自西藏错鄂(采集人朱欢, 采集时间 2015 年 6 月 2 日, 采集地点 88°76′97″E,31°60′57″N, 标本编号为 TB15158)和湖北武汉的紫阳湖(采集人刘国祥, 采集时间2015年7月15日, 采集 地 点 114°30′42″E, 30°53′05″N, 标 本 编 号 为HB15002。采集后的标本一部分使用福尔马林加10%的甘油固定用于形态观察; 另一部分使用无水乙醇固定用于DNA提取。
在体视镜(KL1500 LCD; Zeiss, Göttingen, Germany)剥离海绵标本, 吸取浸出的藻液制作临时装片在显微镜(DM5000B, Leica, Wetzlar, Germany)下使用微分干涉(Differential Interference Contrast, DIC)模式观察形态结构, 图像获取使用Leica DFC320 CCD。
1.2 DNA提取, PCR扩增及测序
体视镜下使用磷酸盐缓冲液(PBS, pH=7.0)清洗剥离的海绵。将清洗后的植物体放入加入 1.5mL CTAB缓冲液[2%CTAB, 0.1mol/L Tris-HCl(pH=8.0),1.4mol/L NaCl, 10mmol/L EDTA, 1.5% β-巯基乙醇]和0.5mL直径0.5mm的玻璃破碎珠的2mL冻存管中。使用mini-beadbeater(Model 3110BX, Biospec Products,Bartlesville, Oklahoma, USA)振荡破碎2min。后置于65°C 温浴 30min。使用等体积的酚׃氯仿׃异戊醇(25׃24׃1)液抽提 1 次。转移上清至新的 EP 管中, 加入终浓度为10μg/mL的RNAase, 37°C温浴25min。使用氯仿׃异戊醇液(24׃1)抽提一次。转移上清, 使用2/3倍体积的异丙醇沉淀DNA。离心后弃上清, 70%的乙醇清洗沉淀 2次。常温干燥沉淀后溶于 20μL的 TE缓冲液中。
使用 Honda等(1999)中的引物和条件扩增 18S rDNA序列; 使用 Zechman(2003)中的引物 rbcLQ和rbcLB扩增rbcL序列; 使用Hayakawa等(2012)中的引物和条件扩增ITS rDNA序列。PCR产物经1.5%的琼脂糖凝胶电泳后, 在紫外透射台(CUV30A, 上海勤翔)上切取目的条带, 使用胶回收试剂盒(E.Z.N.A. Gel Extraction Kit Omega Bio-Tek, 美国)纯化回收纯化 PCR产物。纯化后的 PCR产物与pMD18-T(宝生物, 大连, 中国)载体连接, 转化到E.coli DH5α中, 以通用引物(M13)为测序引物测序(擎科生物, 武汉, 中国)。测序后的序列上传至GenBank (http://www.ncbi.nlm.nih.gov/genbank/)。
1.3 系统发育与二级结构预测分析
将本研究获取的序列从 GenBank上检索比对,并下载近缘序列。使用mafft7.2(Katoh et al, 2013)进行序列比对。使用Bioeditv7.2(Hall, 2013)修正比对后的序列。校正后的数据使用 MEGA7.0(Kumar et al,2016)和DAMBE5.0(Xia, 2013)进行数据质量评估。使用jModeltest2.1(Darriba et al, 2012)进行最适模型评估。进化树的构建采用最大似然法和贝叶斯法。最大似然法使用软件为RAXML8.0(Stamatakis, 2014), 贝叶斯法使用MrBayes3.2 (Ronquist et al, 2012)。
使用ITS2 database数据库对获取的ITS区域进行了精确注释(Ankenbrand et al, 2015)。使用数据库中已有的ITS2二级结构作为预测模型对本研究中获取的ITS2二级结构进行预测(Ankenbrand et al, 2015)。预测的模型使用4sale和VaRNA3.1软件进行比对注释(Seibel et al, 2006; Darty et al, 2009)。
2 结果与分析
2.1 形态观察
一种淡水针海绵(Spongillidae sp.)(图1a、1b, 图2a、2c)
本研究中共采集到两株海绵标本。其中西藏标本附着在湖底约4.0—7.0m处深的轮藻植物上, 海绵整体呈翠绿色, 缘管状, 缘管偶有分叉, 宽 0.5—1.2cm(图1a、1b); 硅质骨针单轴、中部纵向具一条不明显的愈合缝, 长 150—500μm, 多 250—350μm(图2a)。湖北标本附着在湖岸基质表面, 暗绿色, 整体呈垫状、无缘管状凸起; 骨针硅质, 单轴、中部纵向具一条不明显的愈合缝, 长 150—450μm, 多 300μm 左右(图 2c)。
由于标本采集后我们立即使用甲醛和无水乙醇分别进行了固定, 体视镜和显微镜下并未观察到完整的内部结构。这可能主要是因为构成淡水针海绵的几类细胞(扁平细胞、领细胞、芒状细胞、变形细胞)在甲醛固定后极易破碎, 只有硅质骨针较为完整地保持了原本的形态。因此, 这也给标本的物种鉴定带来了一定困难。这也是我们将这两株海绵仅鉴定到科的主要原因。此外, 索囊藻均共生在一个形状不规则的囊球状细胞内, 这与国外报道的淡水针海绵的变形细胞一般会内共生大量球状绿藻的描述一致。

图1 采集自错鄂的淡水海绵, 外观呈翠绿色的缘管状Fig.1 The freshwater sponge collected from Nagtsang Tso, with an olive-green tubular in morphology
索囊藻属
Choricystis (Skuja) Fott, 1976
Fott, B. (1976). Choricystis, eine neue Gattung der Chlorococcales (Chlorophyceae). Archiv für Hydrobiologie Supplement Algological Studies 49:382—388.
Holotype species: Choricystis parasitica (K.Brandt) Pröschold & Darienko 2011
细胞椭圆形、纺锤形或长卵形, 橄榄绿色; 细胞壁光滑, 无胶被; 具一个侧位片状色素体, 无蛋白核;细胞长约 1.2—2.8µm, 宽约0.8—1.5µm。无性繁殖以产生似亲孢子为主, 通常2或4个静孢子。该属内不同种的区分主要依靠营养细胞的大小和形态。
该属为中国新纪录属。
寄生索囊藻(图2c—g)
Choricystis parasitica (K. Brandt) Pröschold &Darienko 2011.
Pröschold T, Darienko T, Silva P C, Reisser W &Krienitz L (2011). The systematics of Zoochlorella revisited employing an integrative approach.Environmental Microbiology 13(2): 350—364.
Homotypic Synonyms: Zoochlorella parasitica K Brandt 1881; Chlorella parasitica (K. Brandt)Beijerinck 1890.
Heterotypic Synonyms: Coccomyxa minor Skuja 1948; Choricystis minor (Skuja) Fott 1976; Choricystis minor var. gallica (Bourrelly) Komarek 1979.
细胞椭圆形、纺锤形或长卵形, 橄榄绿色; 细胞壁光滑, 无胶被; 具一个侧位片状色素体, 无蛋白核;成熟老化的营养细胞色素体偶有不明显的分瓣; 细胞壁薄; 细胞长 1.2—2.8µm, 宽 0.8—1.5µm(图2e—g)。通常十几个至数十个内共生在一个海绵变形细胞内(图2c—d)。无性繁殖以产生两个似亲孢子为主。
该种为中国新纪录种。

图2 本研究中采集的淡水针海绵的骨针和共生索囊藻形态特征Fig.2 The morphology of spicules and Chorisystis cells
2.2 系统发育分析
本研究中获得的两条海绵的 18S rDNA在GenBank上比对后, 下载获得了包含 38条序列的矩阵, 其中 Svenzea为外类群。比对后的序列矩阵长1594bp, 其中可变位点 111个, 简约信息位点 94个,单态位点17个; 矩阵中四种碱基TCAG的平均含量分别为25.4%、20.8%、25.3%和28.5%。饱和指数分析显示序列矩阵并未饱和(Iss=0.077<Issc=0.795), 可用于进化分析。本研究中共获取了4条索囊藻的18S rDNA序列, 其中西藏标本和湖北标本各两条。下载获得了包含主要共球藻纲支系的序列约 75条, 其中以衣藻(Chlamydomonas)和团藻(Volvox)为外类群。比对校正后的序列长度为1594bp, 其中可变位点523个,简约信息位点336个, 单态位点187个; 矩阵中四种碱基TCAG的平均含量分别为25.4%、21.9%、24.4%和 28.2%。饱和指数分析显示序列矩阵并未饱和(Iss=0.054<Issc=0.791), 可用于进化分析。本研究共获得了 4条索囊藻的 rbcL克隆序列, 其中两条扩增自西藏标本两条扩增自湖北标本。比对校正后的序列矩阵共包含 60条序列, 其中以两条团藻序列为外类群。矩阵长 937bp, 可变位点 427个, 简约信息位点400个, 单态位点27个; 矩阵中四种碱基TCAG的平均含量分别为29.9%、19.5%、28.9%和21.7%。饱和指数分析显示序列矩阵并未饱和(Iss=0.194<Issc=0.739), 可用于进化分析。
本研究共得到8条索囊藻的ITS序列, 精确注释后得到的ITS2长度均为228bp, 序列与数据库中的两条寄生索囊藻(GenBank numbers: FN298929和FN298930)注释后的长度一致。在数据库中(http://its2.bioapps.biozentrum.uni-wuerzburg.de/)应用已有的胶球藻模型进行预测, 预测得到的 8条 ITS2二级结构模型均具有四条臂(Helix), 其中Helix-I长度为32bp,Helix-II长度为27bp, Helix-III长度为81bp, Helix-IV长度为8bp(图3)。本研究中预测的二级结构与数据库中的寄生索囊藻的二级结构完全一致, 基本证明本研究中得到的两株索囊藻均为寄生索囊藻。
在构建的六条进化树中, 每个矩阵运用最大似然法和贝叶斯法得到的拓扑结构基本一致。在使用18S rDNA序列构建的针海绵的进化树中, 本研究采集的两株标本与针海绵科中的其它种类聚于一起,该支的支持值和后验概率分别为100和1.00, 证实了这两株为淡水针海绵科中的种类(图 4)。但由于在针海绵科中, 各个属的分类问题较为复杂, 在将淡水针海绵科中的分类系统学问题解决之前仅通过 18S rDNA序列并不能将标本鉴定到属。然而这一问题并不是本研究中的焦点, 在此不展开描述。
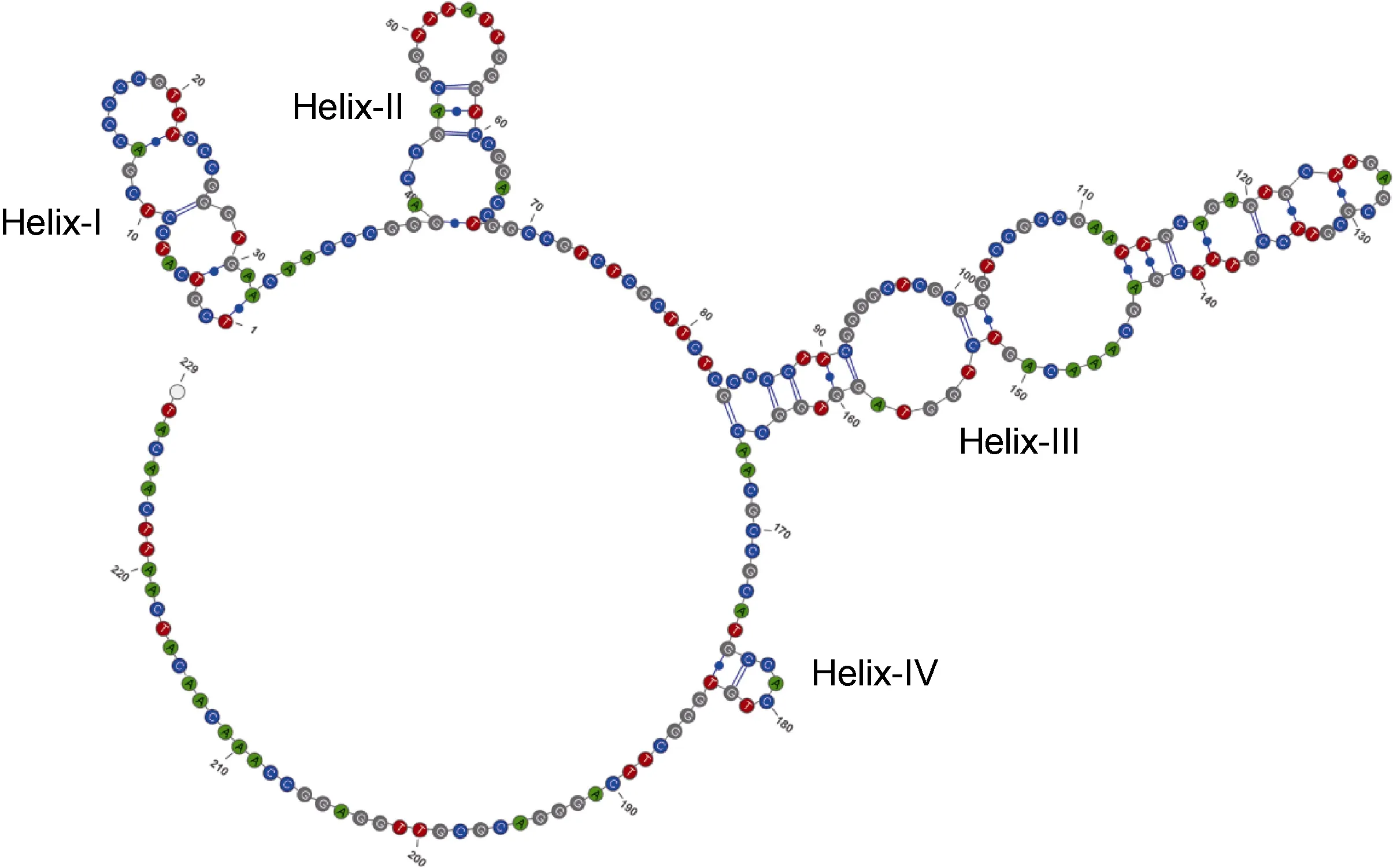
图3 基于本研究获得的8条索囊藻克隆序列预测的ITS2二级结构。共有四条臂, 分别长为32bp、27bp、81bp和8bpFig.3 The consensus secondary structure of ITS2 inferred from eight sequences clones. There are four Helix in length of 32bp, 27bp,81bp, and 8bp, respectively
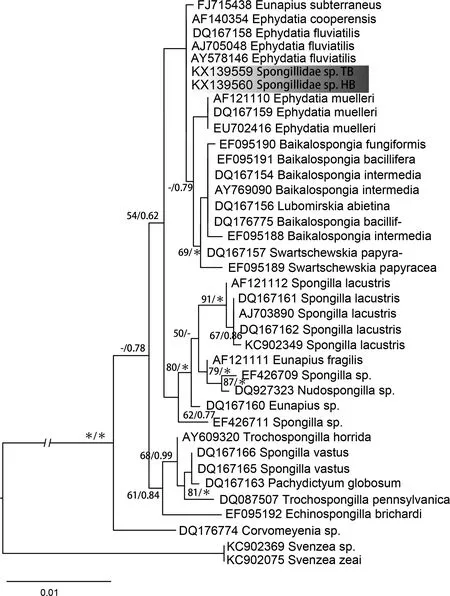
图4 基于18S rDNA序列构建的淡水针海绵的系统进化树Fig.4 The phylogram inferred from18S rDNA sequences of Spongillidae
本研究共得到索囊藻18S rDNA序列4条、rbcL cpDNA序列4条。基于18S rDNA序列构建的系统发育树显示本研究中的两株索囊藻与数据库中的索囊藻聚于一支, 与葡萄藻(Botryococcus)、胶球藻(Coccomyxa)共同构成了共球藻纲的CBC分支, 其自展支持值和后验概率分别为58和0.84。在CBC分支中, 本研究所用到的索囊藻序列均聚于一支, 其自展支持值和后验概率分别为96和1.00(图5)。本研究中,依据 rbcL序列进行的进化分析显示索囊藻均聚于一支, 其自展支持值和后验概率分别为 100和 1.00(图6)。然而, 基于rbcL的进化分析得出的共球藻纲的拓扑结构与基于18S rDNA的拓扑结构有较大差异。在rbcL序列构建的进化树中, 索囊藻单独聚于一支与其它的共球藻纲的支系(诸如小球藻目、渡边分支、共球藻目和溪菜分支等)较早地分离; 但在 18S rDNA序列构建的进化树中, 索囊藻与胶球藻和葡萄藻聚于一支。
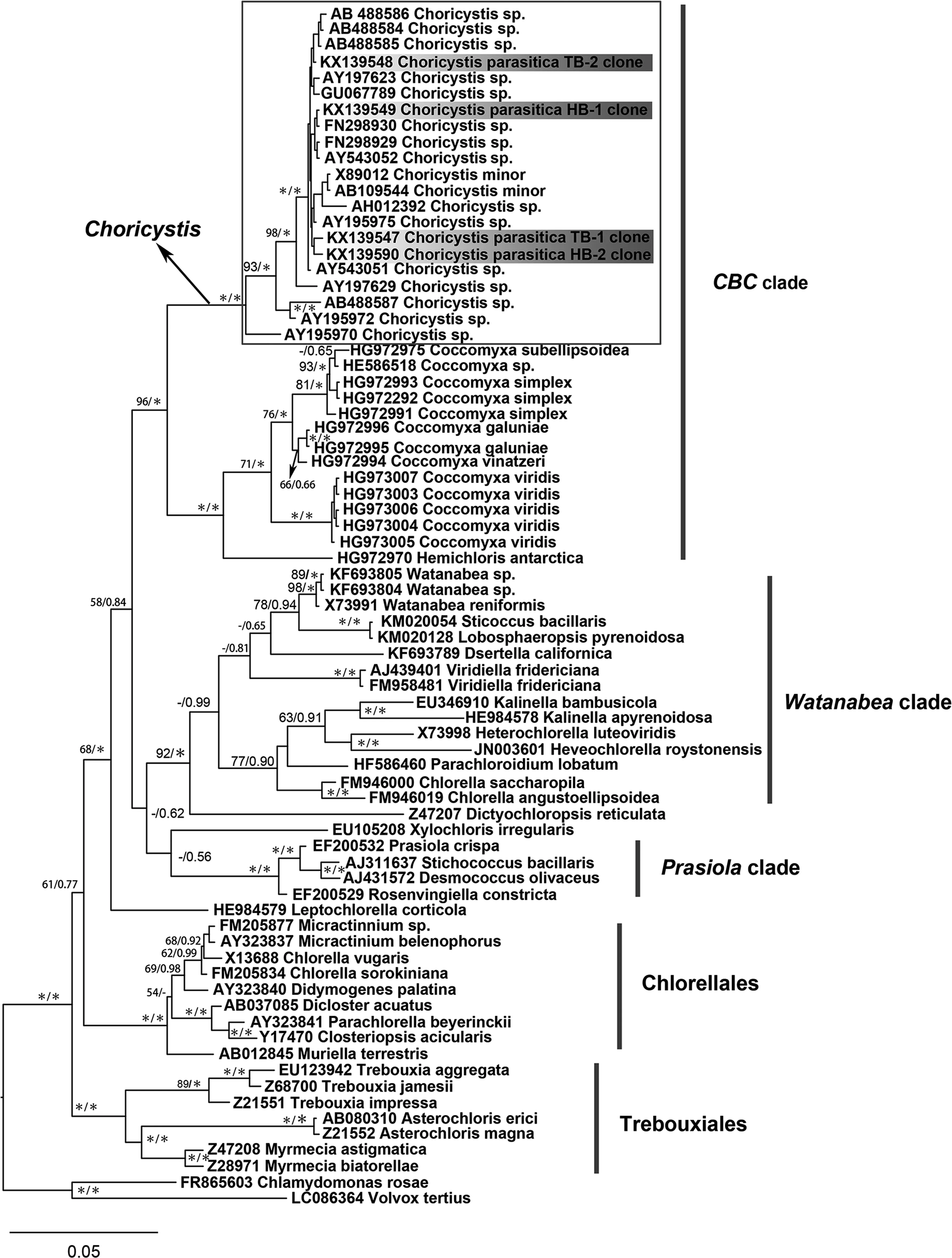
图5 基于共球藻纲18S rDNA序列构建的系统进化树Fig.5 The phylogram based on 18S rDNA sequences of Trebouxiaphyceae
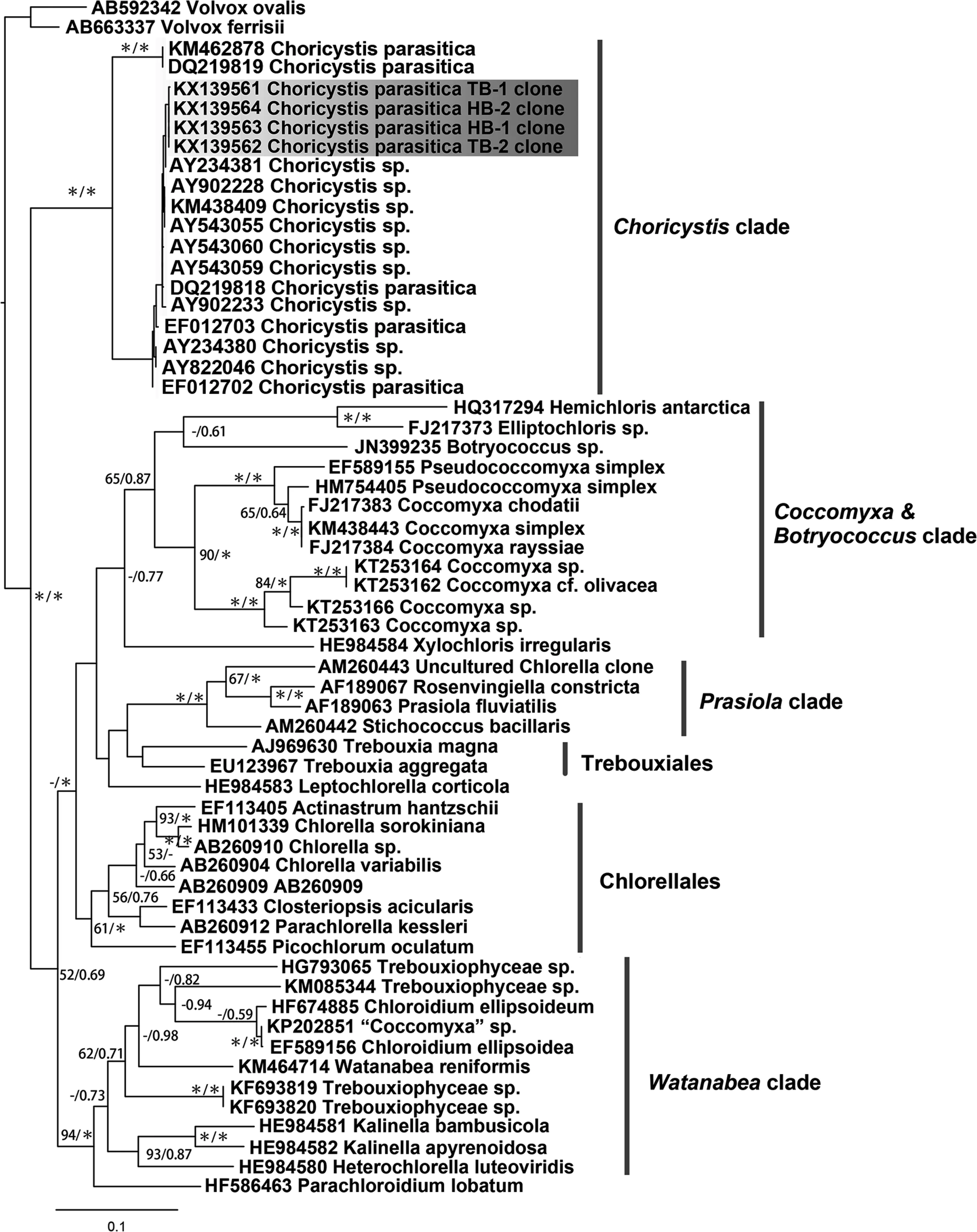
图6 基于共球藻纲rbcL cpDNA序列构建的系统进化树Fig.6 The phylogram based on rbcL rDNA sequences of Trebouxiaphyceae
3 讨论
在球状绿藻类群中, 仅仅依靠显微形态来进行属种水平上的鉴定具有很大的缺陷和局限性, ITS2二级结构对于解决种属水平的球状绿藻较为有效(Coleman, 2007; Luo et al, 2010; Buchheim et al, 2011;Pröschold et al, 2011; Friedl et al, 2012; Krienitz et al,2012; Leliaert et al, 2012)。这一观点在本研究中得到证实。由于胶球藻科中的各个属在显微形态下基本差别不大, 对该科种类的鉴定往往依靠繁殖生物学的特征和超微结构特征以及分子特征。因此, 我们认为索囊藻属中其它各种的系统发育位置仍旧需要进一步的研究。
在共球藻纲中, 各个支系的系统发育位置仍旧充满争议(Smith et al, 2011; Leliaert et al, 2012; Marin,2012; Jeong et al, 2014; Lemieux et al, 2014; Orsini et al, 2016)。同时, 部分研究显示共球藻纲的并不是一个单系类群(Fučíková et al, 2014; Sun et al, 2016)。这些看似矛盾的结论可能主要是由基于核基因分子标记得出的进化分析结论与基于细胞器(质体或线粒体)基因组(或分子标记)不一致造成的(Pröschold et al,2007; Leliaert et al, 2012)。本研究中也出现了这一现象, 即基于18S rDNA和rbcL cpDNA得出的共球藻纲的进化树具有不一致的拓扑结构。有学者认为这一问题的根源在于质体基因与核基因所面临的不同的进化压力(Smith et al, 2011; Jeong et al, 2014; Orsini et al, 2016)。总体而言, 我们推测共球藻纲的多数类群或支系其核基因进化速率与叶绿体基因的进化速率维持着相对稳定; 然而在一些特殊类群中, 如气生性的或内共生性的球状绿藻, 它们的特殊生境使得它们的叶绿体基因或核基因面临更大的进化压力, 进而表现出与二者之间有较大的进化速率。这表现在进化分析上就可能得出核基因与叶绿体基因不一致的拓扑结构。在本研究中的两株索囊藻均为内共生性的,同时本研究中所用有详尽生境描述的索囊藻序列也均为内共生性。这表明针海绵与索囊藻可能是专一性共生。同时也在一定程度上支持了上述观点, 即针海绵将索囊藻吞噬牧植后其叶绿体基因或核基因表达速率可能升高, 进而表现为与其它共球藻纲支系不同的进化速率; 在拓扑结构上(基于核基因和质体基因的进化分析)这有可能表现为索囊藻处在不同的系统发育位置。
对索囊藻属进行系统的分类学研究比较困难,这主要是因为索囊藻属内的物种采集较为困难。最近报道的索囊藻均为内共生性的种类, 且通过 ITS2二级结构预测显示均为寄生索囊藻。可以说, 目前为止经过较为系统鉴定(包含有相应分子标记如 18S 和ITS rDNA、rbcL和tufA cpDNA等)的索囊藻仅有其模式种寄生索囊藻。已有描述的索囊藻属的种类在经过分子鉴定之后其分类可能会有进一步的革新。总体来讲, 绿藻门中包括索囊藻在内的球状绿藻的分类系统正处在急速的变革中。先前根据形态性状建立的种属需要较为全面的分子系统发育验证, 进而确定其系统发育位置; 此外, 球状绿藻存在较大的隐性多样性, 大量标本的采集和多种鉴定手段结合的多相方法对于解决这个问题可能较为有效。
致谢 样品采集过程中得到了中国科学院水生生物研究所何德奎博士、崔永德博士、贾银涛博士和熊雄博士的鼎力帮助, 在此表示感谢。
Ankenbrand M J, Keller A, Wolf M et al, 2015. ITS2 database V:Twice as much. Molecular Biology and Evolution, 32(11):3030—3032
Beijerinck MW, 1890. Culturversyche mit Zoochlorellen,Lichenengonidien und anderen niederen Algen. Botanische Zeitung, 48: 725—785
Bock C, Krienitz L, Pröeschold T, 2011. Taxonomic reassessment of the genus Chlorella (Trebouxiophyceae) using molecular signatures (barcodes), including description of seven new species. Fottea, 11(2): 293—312
Borowitzka M A, 2016. Systematics, Taxonomy and Species Names: Do They Matter? In: Borowitzka M A, Beardall J,Raven J A, eds. The Physiology of Microalgae. Switzerland:Springer, 655—681
Brandt K, 1881. Ueber das Zusammenleben von Thieren und Algen. Arch Anat Physiol, 1881: 570—574
Buchheim M A, Keller A, Koetschan C et al, 2011. Internal transcribed spacer 2 (nu ITS2 rRNA) sequence-structure phylogenetics: towards an automated reconstruction of the green algal tree of life. PLoS One, 6(2): e16931
Coleman A W, 2007. Pan-eukaryote ITS2 homologies revealed by RNA secondary structure. Nucleic Acids Research, 35(10):3322—3329
Darienko T, Gustavs L, Mudimu O et al, 2010. Chloroidium, a common terrestrial coccoid green alga previously assigned to Chlorella (Trebouxiophyceae, Chlorophyta). European Journal of Phycology, 45(1): 79—95
Darienko T, Gustavs L, Eggert A et al, 2015. Evaluating the species boundaries of green microalgae (Coccomyxa,Trebouxiophyceae, Chlorophyta) using integrative taxonomy and DNA barcoding with further implications for the species identification in environmental samples. PLoS One, 10(6):e0127838
Darriba D, Taboada G L, Doallo R et al, 2012. jModelTest 2:more models, new heuristics and parallel computing. Nature Methods, 9(8): 772—772
Darty K, Denise A, Ponty Y, 2009. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics,25(15): 1974—1975
Douglas A E, Huss V A R, 1986. On the characteristics and taxonomic position of symbiotic Chlorella. Archives of Microbiology, 145 (1): 80—84
Fott B, Nováková M, 1969. A monograph of the genus Chlorella.The fresh water species. In: Fott B ed. Studies in Phycology.Praha, Czech: Academia, 10—74
Fott B, 1976. Choricystis, eine neue Gattung der Chlorococcales(Chlorophyceae). Arch Hydrobiol Supply, 17: 382—388
Friedl T, Rybalka N, 2012. Systematics of the green algae: a brief introduction to the current status. In: Lüttge U, Beyschlag W,Büdel B, eds. Progress in Botany 73. Berlin Heidelberg:Springer, 259—280
Fučíková K, Leliaert F, Cooper E D et al, 2014. New phylogenetic hypotheses for the core Chlorophyta based on chloroplast sequence data. Frontiers in Ecology and Evolution, http://dx.doi.org/10.3389/fevo.2014.00063
Greuter W, McNeill J, Barrie FR et al, 2000. International code of botanical nomenclature (Saint Louis Code). In: Saint Louis ed. International code of botanical nomenclature: St Louis, Missouri, USA: Sixteenth International Botanical Congress
Guiry MD, Guiry GM. 2016. AlgaeBase. World-wide electronic publication, National University of Ireland, Galway.http://www.algaebase.org; searched, 2016-07-26
Hall T, 2013. BioEdit v7.2.3. Biological sequence alignment editor for Win 95/98/NT/2K/XP7. California: Ibis Biosciences Carlsbad
Hayakawa Y I, Ogawa T, Yoshikawa S et al, 2012. Genetic and ecophysiological diversity of Cladophora (Cladophorales,Ulvophyceae) in various salinity regimes. Phycological Research, 60(2): 86—97
Henley W J, Hironaka J L, Guillou L et al, 2004. Phylogenetic analysis of the ‘Nannochloris-like’algae and diagnoses of Picochlorum oklahomensis gen. et sp. nov.(Trebouxiophyceae, Chlorophyta). Phycologia, 43(6):641—652
Holweg C, Huss V A R, Kessler E et al, 1994. There is an Ecological Basis for Host Symbiont Specificity in Chlorella Hydra Symbioses. Endocytobiosis and Cell Research,10(1—2): 35—46
Honda D, Yokochi T, Nakahara T et al, 1999. Molecular phylogeny of labyrinthulids and thraustochytrids based on the sequencing of 18S ribosomal RNA gene. Journal of Eukaryotic Microbiology, 46(6): 637—647
Huss V A R, Sogin M L, 1990. Phylogenetic position of some Chlorella species within the Chlorococcales based upon complete small-subunit ribosomal RNA sequences. Journal of Molecular Evolution, 31(5): 432—442
Jeong H, Lim J M, Park J et al, 2014. Plastid and mitochondrion genomic sequences from Arctic Chlorella sp. ArM0029B.BMC Genomics, 15: 286
Johnson M D, 2011. The acquisition of phototrophy: adaptive strategies of hosting endosymbionts and organelles.Photosynthesis Research, 107(1): 117—132
Katoh K, Standley D M, 2013. MAFFT multiple sequence alignment software version 7: improvements in performance and usability.Molecular Biology and Evolution, 30(4): 772—780
Kessler E, 1976. Comparative physiology, biochemistry, and the taxonomy of Chlorella (Chlorophyceae). Plant Systematics and Evolution, 125(3): 129—138
Kessler E, Huss V A R, 1990. Biochemical Taxonomy of Symbiotic Chlorella Strains from Paramecium and Acanthocystis. Plant Biology, 103(2): 140—142
Kessler E, Huss V A R, 1992. Comparative physiology and biochemistry and taxonomic assignment of the Chlorella(Chlorophyceae) strains of the culture collection of the university of Texas at Austin. Journal of Phycology, 28(4):550—553
Krienitz L, Hegewald E, Hepperle D et al, 2003. The systematics of coccoid green algae: 18S rRNA gene sequence data versus morphology. Biologia, 58: 437—446
Krienitz L, Bock C, 2012. Present state of the systematics of planktonic coccoid green algae of inland waters.Hydrobiologia, 698(1): 295—326
Krienitz L, Huss V A R, Bock C, 2015. Chlorella: 125 years of the green survivalist. Trends in Plant Science, 20(2): 67—69
Kulichová J, Škaloud P, Neustupa J, 2014. Molecular diversity of green corticolous microalgae from two sub-Mediterranean European localities. European Journal of Phycology, 49(3):345—355
Kumar S, Stecher G, Tamura K, 2016. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 33(7):1870—1874
Lee H J, Hur S B, 2012. Comparison between phylogenetic relationships based on 18S rDNA sequences and growth by salinity of Chlorella-like species (Chlorophyta). Fisheries and aquatic sciences, 15(2): 125—135
Lee J J, Reidy J, Kessler E, 1982. Symbiotic Chlorella species from larger Foraminifera. Botanica Marina, 25(4): 171—176
Leliaert F, Smith D R, Moreau H et al, 2012. Phylogeny and molecular evolution of the green algae. Critical Reviews in Plant Sciences, 31(1): 1—46
Lemieux C, Otis C, Turmel M, 2014. Chloroplast phylogenomic analysis resolves deep-level relationships within the green algal class Trebouxiophyceae. BMC Evolutionary Biology,14: 211
Letsch M R, Muller-ParkerG, Friedl T et al, 2009. Elliptochloris marina sp. nov. (Trebouxiophyceae, chlorophyta), symbiotic green alga of the temperate pacific sea Anemones Anthopleura Xanthogrammica and A. elegantissima(Anthozoa, Cnidaria). Journal of Phycology, 45(5):1127—1135
Luo W, Pröschold T, Bock C et al, 2010. Generic concept in Chlorella-related coccoid green algae (Chlorophyta,Trebouxiophyceae). Plant Biology, 12(3): 545—553
Marin B, 2012. Nested in the Chlorellales or independent class?Phylogeny and classification of the Pedinophyceae(Viridiplantae) revealed by molecular phylogenetic analyses of complete nuclear and plastid-encoded rRNA operons.Protist, 163(5): 778—805
Müller J, Friedl T, Hepperle D et al, 2005. Distinction between multiple isolates of Chlorella vulgaris (Chlorophyta,Trebouxiophyceae) and testing for conspecificity using amplified fragment length polymorphism and its rDNA sequences. Journal of Phycology, 41(6): 1236—1247
Orsini M, Cusano R, Costelli C et al, 2016. Complete genome sequence of chloroplast DNA (cpDNA) of Chlorella sorokiniana. Mitochondrial DNA Part A, 27(2): 838—839
Pröschold T, Leliaert F, 2007. Systematics of the green algae:conflict of classic and modern approaches. In: Brodie J.Lewis J, eds. Unravelling the Algae: the Past, Present, and Future of Algal Systematics. Boca Raton: CRC Press, 75:123—153
Pröschold T, Darienko T, Silva P C et al, 2011. The systematics of Zoochlorella revisited employing an integrative approach.Environmental Microbiology, 13(2): 350—364
Rajević N, Kovačević G, Kalafatić M et al, 2015. Algal endosymbionts in European Hydra strains reflect multiple origins of the zoochlorella symbiosis. Molecular Phylogenetics and Evolution, 93: 55—62
Reisser W, 1984. The taxonomy of green algae endosymbiotic in ciliates and a sponge. British Phycological Journal, 19(4):309—318
Ronquist F, Teslenko M, van der Mark P et al, 2012. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology,61(3): 539—542
Seibel P N, Müller T, Dandekar T et al, 2006. 4SALE—a tool for synchronous RNA sequence and secondary structure alignment and editing. BMC Bioinformatics, 7: 498
Skelton J, Strand M, 2013. Trophic ecology of a freshwater sponge (Spongilla lacustris) revealed by stable isotope analysis. Hydrobiologia, 709(1): 227—235
Skuja H, 1948. Taxonomie des Phytoplanktons Einiger Seen in Uppland, Schweden. Uppsala: Acta Universitatis Upsaliensis,9: 1—399
Smith D R, Burki F, Yamada T et al, 2011. The GC-rich mitochondrial and plastid genomes of the green alga Coccomyxa give insight into the evolution of organelle DNA nucleotide landscape. PLoS One, 6(8): e23624
Stamatakis A, 2014. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies.Bioinformatics, 30(9): 1312—1313
Sun L H, Fang L, Zhang Z H et al, 2016. Chloroplast Phylogenomic Inference of Green Algae Relationships.Scientific Reports, 6: 20528
Takeda H, 1995. Cell wall composition and taxonomy of symbiotic Chlorella from Paramecium and Acanthocystis.Phytochemistry, 40(2): 457—459
Vinayakumar M, Kessler E, 1975. Physiological and biochemical contributions to the taxonomy of the genus Chlorella.Archives of Microbiology, 103(1): 13—19
Xia X H, 2013. DAMBE5: A comprehensive software package for data analysis in molecular biology and evolution.Molecular Biology and Evolution, 30(7): 1720—1728
Zechman F W, 2003. Phylogeny of the dasycladales (Chlorophyta,Ulvophyceae) based on analyses of rubisco large subunit(rbcL) gene sequences. Journal of Phycology, 39(4): 819—827

