肾形肾状线虫个体间rDNA内转录间隔区的变异
肾形肾状线虫个体间rDNA内转录间隔区的变异
邓艳凤, 徐红兵, 田忠玲, 郑经武*
(浙江大学农业与生物技术学院生物技术研究所,杭州310058)
摘要利用DNA序列分析技术对我国浙江、福建和重庆3个地区的肾形肾状线虫(Rotylenchulus reniformis,RN)种内群体及群体内个体间核糖体RNA 基因(rDNA)内转录间隔区(internal transcribed spacer,ITS)序列进行PCR扩增及序列变异分析。结果显示不同的群体间均获得2种PCR产物,标记为变异类型Ⅰ(RN_VAR1)和变异类型Ⅱ(RN_VAR2)。基于每个群体内2条雌虫分别挑取各变异类型的5个克隆测序,共得到60个克隆序列。经序列比对分析,发现肾形肾状线虫rDNA基因2种类型的ITS区变异很大,其中变异类型Ⅰ序列长度为705~712 bp,鸟嘌呤和胞嘧啶(guanine and cytosine content,GC)含量为45.1%~46.7%;变异类型Ⅱ序列长度为854~860 bp,GC含量为48.4%~50.0%。2种类型的ITS相似度(包括5.8S RNA基因)仅为62.1%~65.6%,而各rDNA变异类型内部各个克隆序列也存在差异,变异类型Ⅰ内个体间相似度为89.9%~100%;变异类型Ⅱ内个体间相似度为91.4%~99.8%。系统进化分析表明2种变异序列明显分成2支,同时通过ITS序列分析无法将3个地方群体区分开来。经实时荧光定量PCR(quantitative real-time PCR,qPCR)检测发现RN_VAR1含量稍大于RN_VAR2,分别占保守18S基因的rDNA重复单位含量的56%和40%。同时,还发现肾形肾状线虫的2种rDNA-ITS变异类型与已报道的2种18S RNA基因变异类型相对应,表明肾形肾状线虫存在2种rDNA变异类型。
关键词肾形肾状线虫; rDNA; 内转录间隔区; 序列变异
中图分类号Q 933文献标志码A
基金项目:农业部公益性行业(农业)科研专项(2011030007);浙江省公益技术研究农业项目(2011C22028);浙江省“8812”计划专项(2011C12020-3)。
收稿日期(Received):2014-11-04;接受日期(Accepted):2014-12-19;网络出版日期(Published online):2015-05-19

Interindividual variability of internal transcribed spacer region of ribosomal DNA inRotylenchulusreniformis。JournalofZhejiangUniversity(Agric. &LifeSci.), 2015,41(3):252-260
DengYanfeng,XuHongbing,TianZhongling,ZhengJingwu* (Institute of Biotechnology, College of Agriculture and Biotechnology, Zhejiang University, Hangzhou 310058, China)
SummaryRibosomalRNAgene(rDNA)isconservativeanditiscomposedofnumerouscopiesoftandemlyrepeatedtranscriptionunitswithinthegenomethathasbeenrecognizedasanattractivemarkerforphylogeneticstudies.Itconsistsofthe18S, 5.8S,and28Sgenesaswellasinternaltranscribedspacer(ITS)regionandexternaltranscribedspacer(ETS)region.Asmembersofasequencefamily,themultiplecopiesoftherDNAdonotevolveindependently.Theytendtoevolveinaconcertedfashion,whichmeansthatinaspeciestherepeatsevolvetogether.Previousstudyshowsthattherearetwomajorvariantsinthe18SrRNAgeneofthesingleRotylenchulus reniformis.
ThevariationwithinITSregionofrRNAgeneamongR. reniformispopulationsfromZhejiang,FujianandChongqingofChinawasreported.Twobandsof18S-ITSsequencewithlengthsof1 250bpand1 400bp(include≈480bp18SrDNAsequence,thewholeITSsequenceand≈50bp28SrDNAsequence)wereamplifiedfromeachnematodeofthreepopulationsofR. reniformis,whichnamedasvariant1 (RN_VAR1)andvariant2 (RN_VAR2),respectively.Theamountofvariationwasassessedbysequencingfiveclonesfromeachvariantfromsixfemalereniformnematodesofthreepopulations,withatotalof60sequences.Thesesequencesweredistinguished,basedonmultiplesequencealignment(MSN),thepercentidentityandguanineandcytosinecontent(GC)contentoftheITSandclusteringinphylogenetictrees.ThelengthandGCcontentofITSregionwerecharacterizedbyRN_VAR1 (705-712bp, 45.1%-46.7%)andRN_VAR2 (854-860bp, 48.4%-50.0%),respectively.ThepercentidentitybetweenRN_VAR1andRN_VAR2wasrelativelylowwhichonlyrangedbetween62.1%and65.6%.ThepercentidentitybetweenallclonesinRN_VAR1was89.9%-100%,andthatinRN_VAR2was91.4%-99.8%.Phylogenetictreeanalysesbasedonneighbor-joiningmethodshowedthatbothRNvariantscouldbedistinguishedfromothernematodesbyITSsequencealignment,whileitwashardfordifferentiationofR. reniformisintrapopulation.Thequantitativereal-timepolymerasechainreaction(qPCR)experimentsshowedthatRN_VAR1madeup56%ofrDNAunitswithrelativelywell-conservesin18SRNAgene,whereasRN_VAR2was40%.ThetwoITSvariants,RN_VAR1andRN_VAR2,wereconsistenttothereportedtwo18SRNAgenevariants,respectively.
TheresultsconfirmthattherearetworDNAvarianttypesinreniformnematode.Thisvariationmaybeasaresultofancestralinterspecifichybridizationorgenerecombinationbetweensisterchromatids,leadingtothecoevolutionoftherDNAsequencesonthesechromosomes.
KeywordsRotylenchulus reniformis;rDNA;internaltranscribedspacerregion;sequencevariation
肾形肾状线虫是一种根部半内寄生性线虫,通常发生在热带、亚热带及一些温暖潮湿的地区,寄主范围达300种植物,是棉花、甘薯、凤梨和许多蔬菜的重要病原线虫[1]。据统计,美国平均每年因肾形肾状线虫造成的损失约为1.3亿美元[2]。我国对肾形肾状线虫的研究较少,目前陆续在广东、上海、湖北、海南、重庆、四川、广西、福建、山东等地发现有肾形肾状线虫的分布,寄主涉及常见蔬菜、热带果树及农作物,危害较大[3]。
核糖体DNA(ribosomalDNA,rDNA)是编码核糖体RNA的基因,它通常以多拷贝的串联重复形式存在于染色体DNA的1个位点或多个位点上[4]。由于rDNA总体上进化速度较慢,存在有较多的保守区,同时不同区域序列有着不同的进化水平,因此被广泛应用于不同等级线虫的分类鉴定[5-7]。然而,许多研究发现一些生物类群个体rDNA重复序列之间的内转录间隔区(internaltranscribedspacer,ITS)存在明显变异[8-13],也有一些报道称18S和28SrRNA基因也存在变异[14-15]。目前对植物寄生线虫rDNA的研究主要集中在种间分子诊断技术的应用和种内群体间遗传变异情况分析。Nyaku等[16]2013年首次报道了肾形肾状线虫个体18SrDNA序列存在2种变异类型,这对以rDNA-ITS为基础的线虫分子诊断技术提出了挑战。因此,明确生物群体内及个体间rDNA序列变异水平,对准确地解释由rDNA基因序列所提供的信息有重要的理论意义。本研究首次明确了中国3个肾形肾状线虫群体之间和个体内rDNA中ITS序列变异状况,丰富了肾形肾状线虫rDNA序列信息,并为肾形肾状线虫进化分析研究提供了重要的理论依据。
1材料与方法
1.1实验材料
供试肾形肾状线虫、南方根结线虫(Meloidogyne incongnita)、大豆孢囊线虫(Heterodera glycines)、小麦禾谷孢囊线虫(Heterodera avenae)和松材线虫(Bursaphelenchus xylophilus)均为笔者所在实验室多年纯培养和繁殖的线虫群体。其中肾形肾状线虫分别来自浙江杭州(ZJ)、福建福州(FJ)和重庆(CQ)。肾形肾状线虫培养寄主为大豆,培养条件:26 ℃,光照16h,湿度60%,光照100%。
1.2DNA的提取
单条线虫DNA提取:在灭菌的0.5-mLEppendorf管中加入10μLddH2O和8 μL 10×PCR(不含Mg2+)缓冲液,加入单条肾形肾状线虫雌虫,捣碎并加入2μL蛋白酶K溶液(600μg/mL),于-70 ℃过夜,转入PCR仪,在65 ℃下温育1h,接着95 ℃ 10min,最后在转速为12 000r/min条件下离心1min,取其上清液即可用于PCR扩增。
全基因组提取:根据Ezup柱式动物基因组DNA抽提试剂盒[生工生物工程(上海)股份有限公司]操作步骤进行。提取后的基因组DNA(genomicDNA,gDNA)溶于100μLddH2O中,测光密度D(λ),于-20 ℃保存。
1.3PCR扩增与序列测定
引物V1[17]位于18SrDNA3′端距ITS1大约190bp处,Z1(本实验室设计)位于28SrDNA5′端距ITS2约50bp处,引物序列见表1。
表1用于PCR和实时荧光定量PCR的特异性引物
Table1SpecificprimersforPCRandquantitativereal-timepolymerasechainreaction(qPCR)

引物名称Oligoname引物序列Primersequences(5'→3')方向DirectionV1TTGATTACGTCCCTGC-CCTTT正向Forward18SMFTAACGAGCGAGACTCTGGCC正向ForwardZ1TCCTCCGCTTACTGATATG反向ReverseRV1FCGACATACAAACATG-CACTG正向ForwardRV1RGCATTTGGCTACCTTA-AGAG反向ReverseRV2FATGCAGATGGCGAACT-CATCC正向ForwardRV2RGAAGCAAGACAAGGGC-CCTAC反向ReverseR18SFTGTCTGCCTTATCAACTTTC正向ForwardR18SRTTTCTCGTCACTACCTCCTC反向Reverse
分别用V1和Z1扩增肾形肾状线虫、南方根结线虫、大豆孢囊线虫、小麦禾谷孢囊线虫和松材线虫等5种线虫ITS区,用来区分不同线虫ITS区片段大小。
根据肾形肾状线虫18S rDNA保守区域设计引物18SMF,与Z1一起可以扩增得到包含480 bp左右18S rDNA序列、整个ITS区序列和部分28S rDNA序列的18S-ITS序列。所有引物均由英潍捷基(上海)贸易有限公司合成。每个群体取2条雌虫进行18S-ITS克隆,编号分别为CQ6,CQ8,FJ2,FJ9,ZJ2,ZJ4,从各样本克隆结果中选取5个长片段克隆和5个短片段克隆送上海生工测序。
PCR反应体系:10×PCR(含Mg2+)缓冲液2.5 μL、2.5 mmol/L dNTPs 2 μL、引物1 μL、5 U/μLrTaq酶0.2 μL(Takara公司),用ddH2O补足体积至25 μL。PCR扩增程序:94 ℃预变性2 min,94 ℃变性15 s,55 ℃退火30 s,68 ℃延伸1 min,20个循环,最后68 ℃延伸7 min。
1.4实时荧光定量PCR分析
通过实时荧光定量PCR(quantitative real-time polymerase chain reaction,qPCR)反应来鉴定2种类型ITS序列在基因组中的含量。根据2种ITS序列分别设计特异性引物RV1F/RV1R和RV2F/RV2R,同时设计18S上的引物R18SF/R18SR,用于总rDNA含量定量,引物见表1。
反应体系:SYBR®PremixExTaqTMⅡ (Tli RNaseH Plus)(Takara公司)混合液10 μL、引物0.8 μL、用ROX染料作参比(50×)0.4 μL、模板2 μL,用ddH2O补足体积至20 μL。PCR反应程序在荧光定量PCR仪(Applied Biosystems 7300 Fast Real Time PCR System)上进行,预扩增95 ℃,10 s;然后95 ℃,5 s,60 ℃,31 s,40个循环;PCR扩增结束后立即进行熔解曲线分析,以验证扩增的特异性。熔解曲线的程序为95 ℃,15 s;60 ℃,1 min;95 ℃,15 s。
标准品用含有目的序列的重组质粒制作,用微量紫外可见分光光度计测光密度D(260 nm)和D(280 nm),并根据摩尔定律,计算质粒中的DNA拷贝数。然后以标准DNA 10倍梯度稀释成5个梯度,以其作为模板进行荧光定量PCR。
检测样品为福建肾形肾状线虫全基因组稀释的DNA,每个反应重复3次,CT值标准误差小于0.5,且qPCR反应熔解曲线重合率较好时,才认为该反应具有代表性。
1.5数据分析
所有获得的18S-ITS序列均用DNAStar软件进行拼接并剪辑成18S rDNA和ITS 2个部分。首先用MEGA 4.0[18]软件将得到的部分18S rDNA序列与已发表的肾形肾状线虫18S变异序列进行比对,分成变异类型Ⅰ(RN_VAR1)和变异类型Ⅱ(RN_VAR2);然后用MEGA 4.0和GenDoc[19]对2种ITS变异类型进行序列比对,用邻接法(neighbor-joining,NJ)构建分子系统进化树。可能的嵌合体检测通过Bellerophon(http://comp-bio.anu.edu.au/bellerophon/bellerophon.pl)完成,其中默认设置为200 bp。
2结果与分析
2.1线虫ITS区扩增结果
肾形肾状线虫、南方根结线虫、大豆孢囊线虫、小麦禾谷孢囊线虫和松材线虫等5种线虫ITS区扩增结果见图1.肾形肾状线虫明显存在2种长度的ITS序列(1 100 bp和950 bp),说明肾形肾状线虫ITS区变异较大。
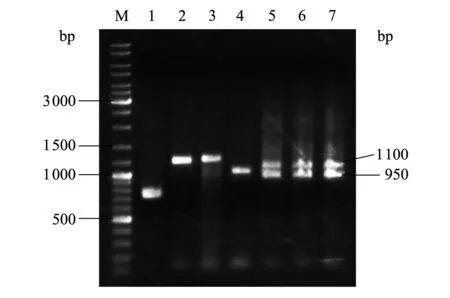
M:DNA标志物;1:南方根结线虫;2:大豆孢囊线虫;3:小麦禾谷孢囊线虫;4:松材线虫;(5~7):肾形肾状线虫. M: DNA marker; 1: Meloidogyne incongnita; 2: Heterodera glycines; 3: Heterodera avenae; 4: Bursaphelenchus xylophilus; (5-7): Rotylenchulus reniformis. 图1 5种植物线虫ITS区PCR扩增电泳图 Fig.1 Electrophoresis result of the ITS region of five kinds of tested plant nematode
2.2肾形肾状线虫rDNA中ITS区变异分析
测序结果共得到60个序列,排除18个可能的嵌合体序列,其余42个18S-ITS序列有2种不同类型,其中23个短片段(≈1 250 bp)属于变异类型Ⅰ(RN_VAR1),19个长片段(≈1 400 bp)属于变异类型Ⅱ(RN_VAR2),GenBank登录号为KP018549~KP018590。从表2中可以看出2种变异类型序列的ITS区差异很大,其中变异类型Ⅰ的ITS长度为705~712 bp,鸟嘌呤和胞嘧啶(guanine and cytosine content,GC)含量45.1%~46.7%;变异类型Ⅱ的ITS长度为854~860 bp,GC含量为48.4%~50.0%;5.8S基因序列差异较小。同时,2种类型ITS相似度(中间包含5.8S)仅为62.1%~65.6%,而各变异类型内部各个克隆序列也存在差异,类型Ⅰ相似度为89.9%~100%;类型Ⅱ相似度为91.4%~99.8%(未显示)。从GenDoc软件比对结果(图2)可以看出,在2种变异类型序列中18S和5.8S这些编码序列只有部分位点出现核苷酸变异,而ITS1和ITS2部分则有较大差异,同时同一变异类型不同克隆之间也存在差异。进化分析结果(图3)表明,2种ITS变异类型明显分成2支,且与其亚科线虫序列区分开来,根结亚科单独聚为一类,这些与Nyaku等[16]基于18S RNA基因建立的系统进化树类似,但是通过ITS序列分析无法将3个地方群体区分开来。说明肾形肾状线虫18S和ITS序列只能作为肾形肾状线虫种间分类研究依据。
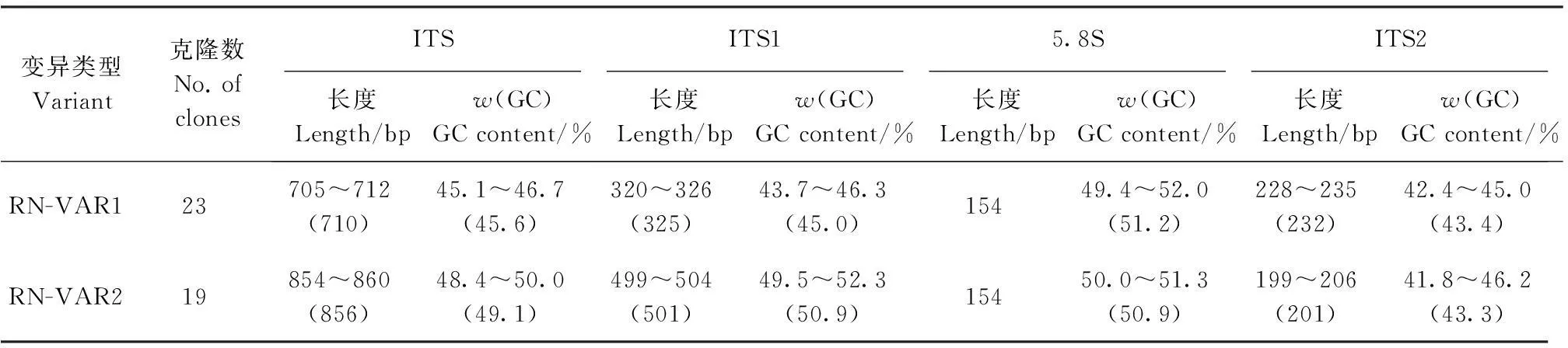
表2 肾形肾状线虫2种rDNA ITS区的长度和GC含量分析
括号内的数值为平均值.
The average values are showed in parentheses.
2.3实时荧光定量PCR结果
从ITS变异类型Ⅰ、变异类型Ⅱ和保守的18S基因熔解峰图(图4)可见,除目的基因以外,未见其他非特异性扩增熔解峰出现,说明检测引物特异性好,结果可靠。
SYBR GreenⅠ荧光定量PCR标准曲线循环阈值与模板浓度呈良好的线性关系,3条曲线中R2值均在0.99以上,说明标准曲线建立成功。
qPCR反应结果(图5)显示ITS变异类型Ⅰ(RN_VRA1)在基因组中的含量约占总rDNA单位含量的56%,而变异类型Ⅱ(RN_VAR2)为40%,变异类型Ⅰ是变异类型Ⅱ的1.4倍左右。
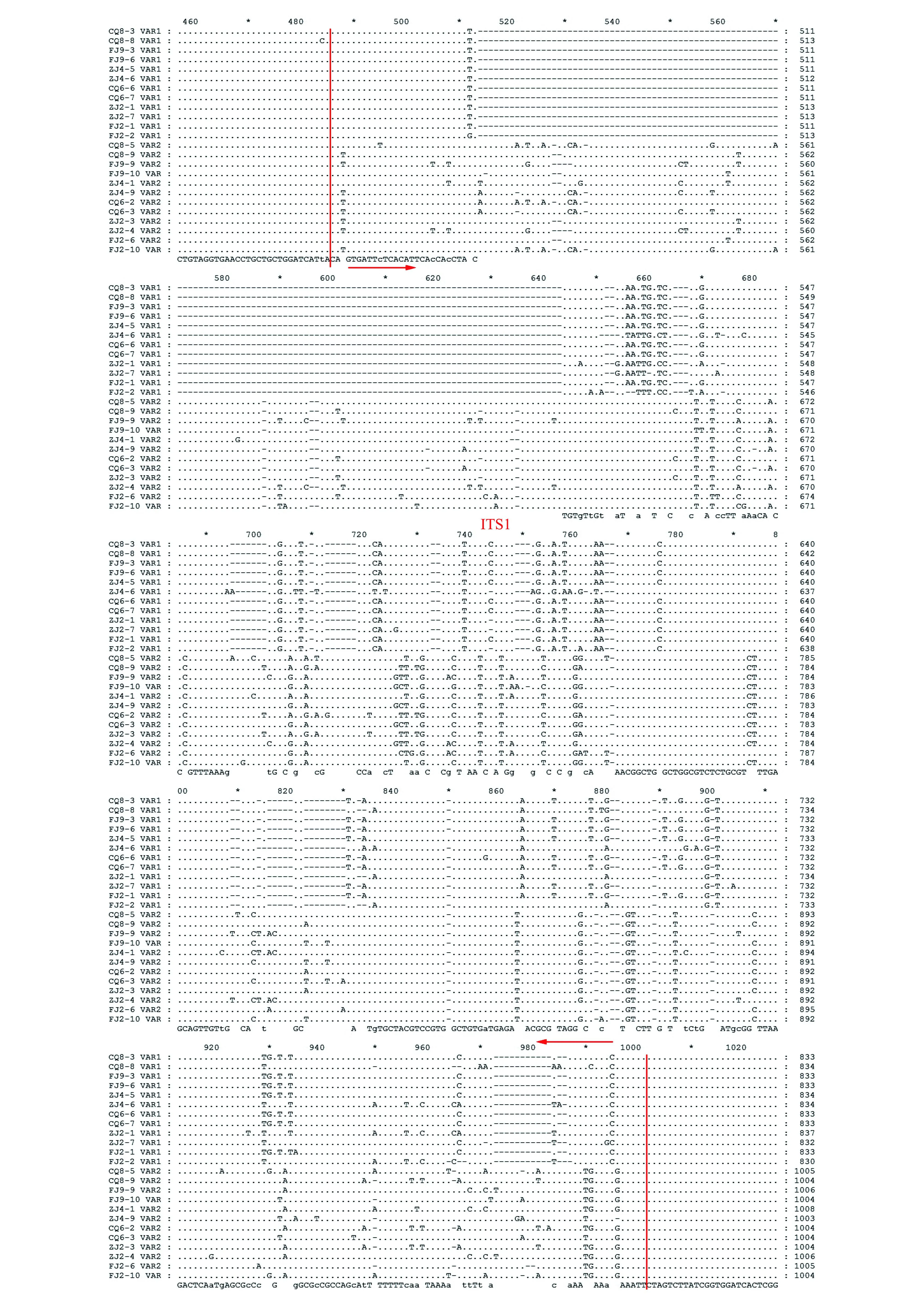

图2 用GenDoc软件对肾形肾状线虫2种类型rDNA-ITS序列进行多重序列比较 Fig.2 Multiple sequence comparison of the primary structure of the two types of rDNA-ITS of R. reniformis in RN using GenDoc
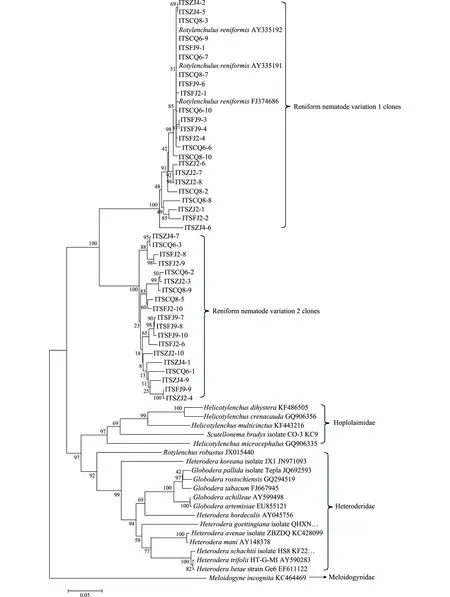
图3 42个肾形肾状线虫rDNA-ITS序列与纽带亚科、异皮亚科、肾形亚科和根结亚科线虫的系统发育树分析 Fig.3 Phylogenetic tree analyses for 42 RN rDNA-ITS clones and nematodes in the super-families, Hoplolaimidae, Heteroderidae, Rotylenchulidae, and Meloidogynidae using neighbor-joining (NJ) method
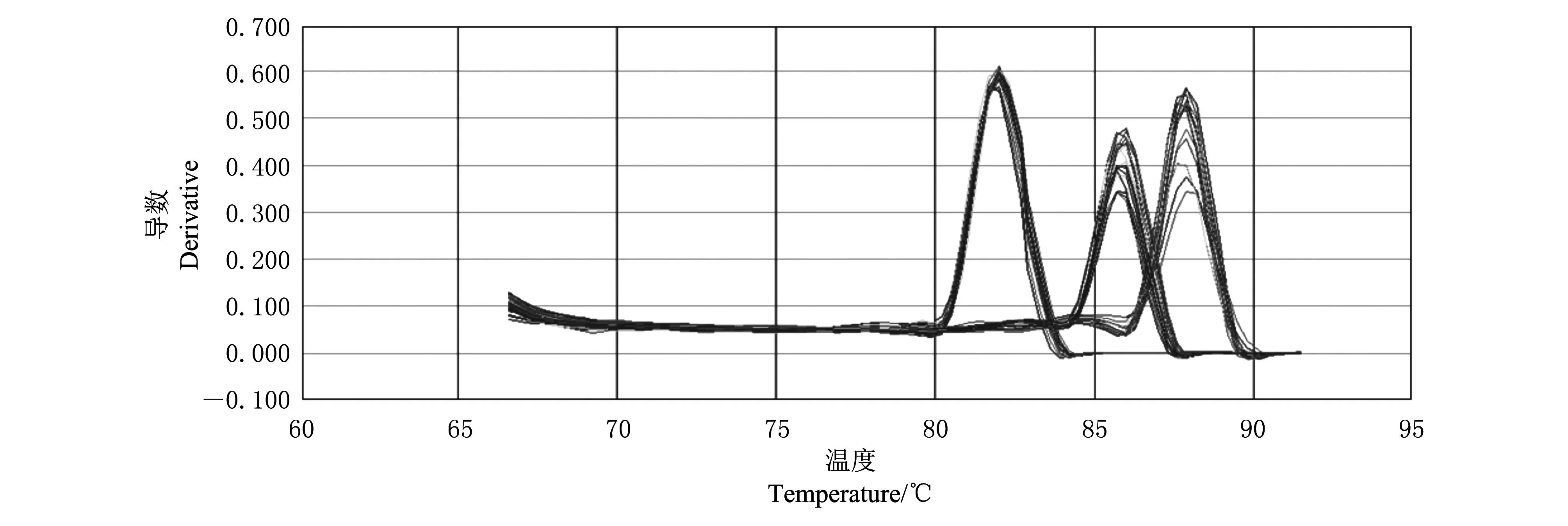
左峰:RN_VAR1;中峰:18S基因;右峰:RN_VAR2. Left peak: RN_VAR1 gene; Middle peak: 18S gene; Right peak: RN_VAR2 gene. 图4 肾形肾状线虫RN_VAR1、RN_VAR2和18S基因质粒标准品扩增产物的熔解峰 Fig.4 Dissociation curves of RN_VAR1, RN_VAR2 and 18S genes of R. reniformis after plasmid standard amplification
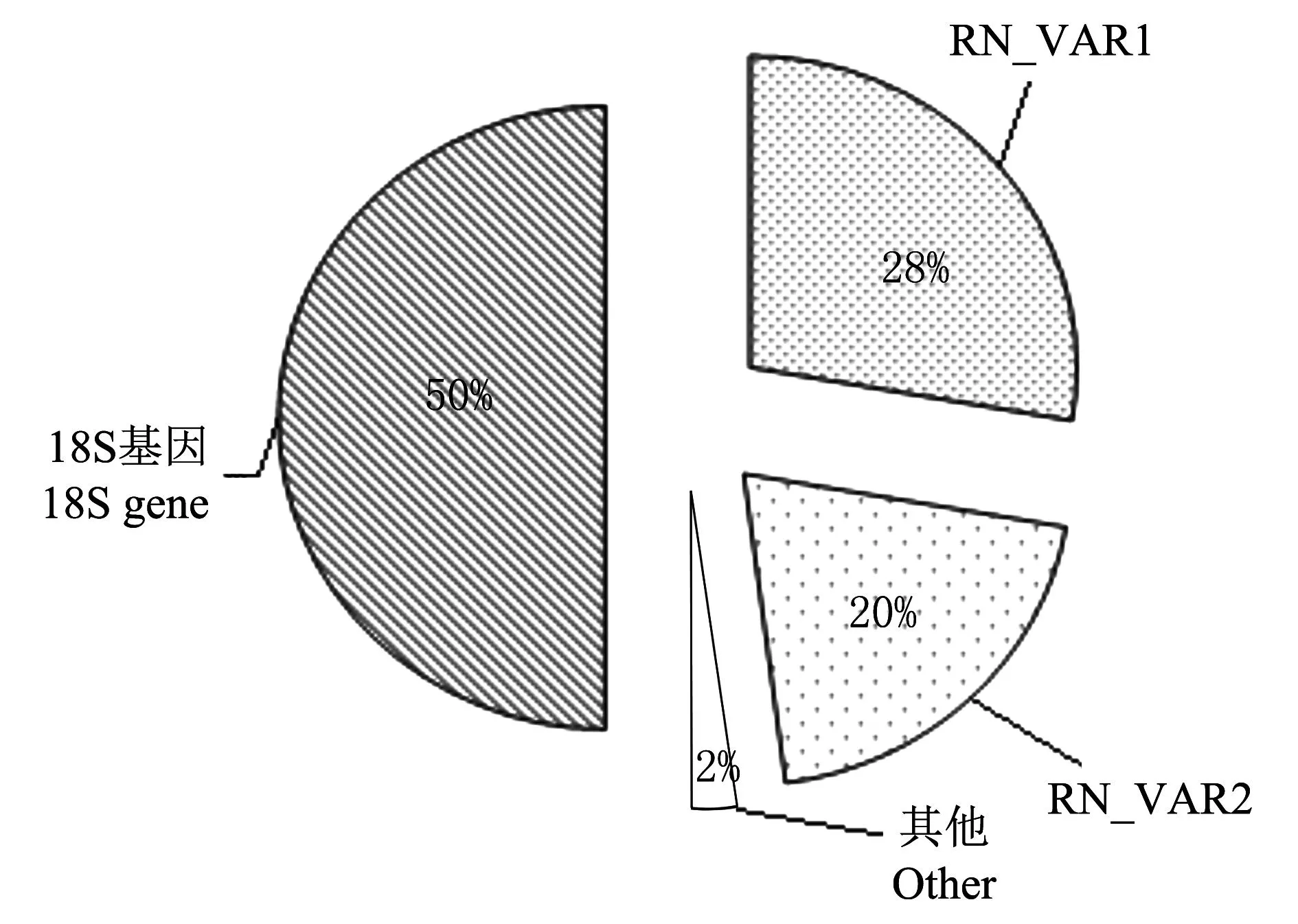
图5 肾形肾状线虫2种变异类型ITS序列在基因组中的含量与18S保守基因含量比较 Fig.5 Content comparison of RN_VAR1, RN_VAR2 and 18S genes of R. reniformis
3讨论
Nyaku等[16]研究了肾形肾状线虫18S RNA基因变异,发现肾形肾状线虫存在2种18S RNA基因变异类型.本研究结果表明肾形肾状线虫存在2种ITS变异类型,这2种变异类型分别与Nyaku报道的2种18S RNA基因变异类型相对应。同时,在这2种变异类型序列中,18S和5.8S这些编码区序列基本一致,而非编码区(ITS1和ITS2)序列存在明显差异。系统发育分析显示,这2种rDNA-ITS变异类型都能作为肾形肾状线虫与各科线虫分类的依据,而对于同一亚科各属之间的分类,由于资料有限,无法进行判断。
目前有许多生物体发现存在rDNA序列变异。Vogler等[6]等发现虎甲虫存在2种ITS1序列且这2种序列有可能存在于不同的染色体上;O’Donnell等[8]发现廉孢菌属内所有菌群均存在2种ITS2变异类型;Freire等[12]首次在双壳纲中发现了鸟蛤(Cerastodermaglaucum) rDNA中存在2种ITS变异类型,其中类型Ⅰ包含真的5.8S基因,而类型Ⅱ在5.8S基因处有部分插入缺失,影响其二级结构和功能。Keller等[20]证实山地蝗虫存在多个rDNA基因座和多种rDNA变异类型。这些研究从另一方面展现了遗传基因的多态性,为了解释这种多态性,许多研究者也提出了相应的猜测:其一认为,变异序列的产生是由于rDNA序列发生了种间杂交或基因转化重组期间出现了大的插入或缺失;其二认为,同一染色体上基因的协同进化效率远比不同染色体上的效率高[21-24],因此不同的ITS变异类型可能坐落于不同的染色体上,减少被协同进化的威胁;其三认为,有些rDNA变异序列可能不具有生物功能,因此选择压力较小,序列得到保存;其四认为,生物体内存在某些机制造成协同进化作用受到限制或者重复片段间相隔较远,协同进化作用减弱,使得同一rDNA上的不同重复片段间序列差异较大。
参考文献(References):
[1]Starr J L, Cook R, Bridge J.PlantResistancetoParasiticNematodes. Wallingford: CAB International, 2002:153-174.
[2]Starr J L, Koenning S R, Kirkpatrick T L,etal. The future of nematode management in cotton.JournalofNematology, 2007,39:283-294.
[3]张燕.中国肾形肾状线虫不同地理群体形态、分子及生物学特性研究.杭州:浙江大学,2010.
Zhang Y. Morphological, molecular and biological characteristics ofRotylenchulusreniformisoccurring in China. Hangzhou: Zhejiang University, 2010. (in Chinese with English abstract)
[4]Long E O, Dawid I B. Repeated genes in eukaryotes.AnnualReviewofBiochemistry, 1980,49:727-764.
[5]Gonzalez I L, Sylvester J E, Smith T F,etal. Ribosomal RNA gene sequences and hominoid phylogeny.MolecularBiologyandEvolution, 1990,7:203-219.
[6]Vogler A P, Desalle R. Evolution and phylogenetic information content of the ITS1 region in the tiger beetleCicindeladorsalis.MolecularBiologyandEvolution, 1994,11:393-405.
[7]Coleman A W, Vacquier V D. Exploring the phylogenetic utility of ITS sequences for animals: A test case for abalone (Haliotis).JournalofMolecularEvolution, 2002,54:246-257.
[8]O’Donnell K, Cigelnik E. Two divergent intragenomic rDNA ITS2 types within a monophyletic lineage of the fungus fusarium are non-orthologous.MolecularPhylogeneticsandEvolution, 1997,7(1):103-116.
[9]Gandolfi A, Bonilauri P, Rossi V,etal. Intraindividual and intraspecies variability of ITS1 sequences in the ancient asexualDarwinulastevensoni(Crustacea: Ostracoda).Heredity, 2001,87:449-455.
[10]Parkin E J,Butlin R K. Within- and between-individual sequence variation among ITS1 copies in the meadow grasshopperChorthippusparallelusindicates frequent intrachromosomal gene conversion.MolecularBiologyandEvolution, 2004,21:1595-1601.
[11]Simon U K, Weis M. Intragenomic variation of fungal ribosomal genes is higher than previously thought.MolecularBiologyandEvolution, 2008,25:2251-2254.
[12]Freire R, Arias A, Méndez J,etal. Sequence variation of the internal transcribed spacer (ITS) region of ribosomal DNA inCerastodermaspecies (Bivalvia: Cardiidae).JournalofMolluscanStudies, 2010,76:77-86.
[13]Hoy M S, Rodriguez R J. Intragenomic sequence variation at the ITS1-ITS2 region and at the 18S and 28S nuclear ribosomal DNA genes of the New Zealand mud snail,Potamopyrgusantipodarum(Hydrobiidae: Mollusca).JournalofMollscanStudies, 2013,79:205-217.
[14]Kreiger J, Hett A K, Fuerst P A,etal. Unusual intraindividual variation of the nuclear 18S rRNA gene is widespread within the Acipenseridae.JournalofHeredity, 2006,97:218-225.
[15]Vierna J, Gonzá lez-Tizón A M, Martínez-Lage A. Long-term evolution of 5S ribosomal DNA seems to be driven by birth-and-death processes and selection inEnsisrazor shells (Mollusca: Bivalvia).BiochemicalGenetics, 2009,47:635-644.
[16]Nyaku S T, Sripathi V R, Kantety R V,etal. Characterization of the two intra-individual sequence variants in the 18S rRNA gene in the plant parasitic nematode,Rotylenchulusreniformis.PLoSONE, 2013,8:e60891.
[17]Vrain T C, Wakarchuk D A, Lévesque A C,etal. Intraspecific rDNA restriction fragment length polymorphism in theXiphinemaamericanumgroup.FundamentalandAppliedNematology, 1992,15:563-573.
[18]Tamura K, Dudley J, Nei M,etal. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0.MolecularBiologyandEvolution, 2007,24:1596-1599.
[19]Nicholas K B, Nicholas H B, Deerfield D W. GenDoc: Analysis and visualization of genetic variation.EmbnewNews, 1997,4(1):14.
[20]Keller I, Chintauan-Marquier I C, Veltsos P,etal. Ribosomal DNA in the grasshopperPodismapedestris: Escape from concerted evolution.Genetics, 2006,174:863-874.
[21]Schlötterer C, Tautz D. Chromosomal homogeneity ofDrosophilaribosomal DNA arrays suggests intrachromosomal exchanges drive concerted evolution.CurrentBiology, 1994,4:777-783.
[22]Polanco C, González A I, de la Fuente,etal. Multigene family of ribosomal DNA inDrosophilamelanogasterreveals contrasting patterns of homogenization for IGS and ITS spacer regions: A possible mechanism to resolve this paradox.Genetics, 1998,149:243-256.
[23]Wang S, Zhang L, Zhan A,etal. Patterns of concerted evolution of the rDNA family in a natural population of Zhikong scallop,Chlamysfarreri.JournalofMolecularEvolution, 2007,65:660-667.
[24]Stage D E, Eickbush T H. Sequence variation within the rRNA gene loci of 12Drosophilaspecies.GenomeResearch, 2007,12:1888-1897。
*通信作者(Corresponding author):沈圣泉,Tel:+86-571-86971527;E-mail:shenshq@zju.edu.cn
第一作者联系方式:童杰鹏,E-mail:tongjiepeng@163.com
URL:http://www.cnki.net/kcms/detail/33.1247.s.20150519.1249.003.html

