不同脂肪源饲粮育成猪肝脏转录组差异分析
李建平秦贵信赵志辉张巧灵王大力孙博兴姜海龙∗(.吉林农业大学动物科学技术学院,长春08;2.河南牧业经济学院畜牧工程系,郑州500;.吉林大学农业试验基地,长春0062;.吉林大学动物医学学院,长春0062;)
不同脂肪源饲粮育成猪肝脏转录组差异分析
李建平1,2秦贵信1赵志辉3张巧灵4王大力3孙博兴3姜海龙1∗
(1.吉林农业大学动物科学技术学院,长春130118;2.河南牧业经济学院畜牧工程系,郑州450011;3.吉林大学农业试验基地,长春130062;4.吉林大学动物医学学院,长春130062;)
摘 要:本试验旨在肥育猪饲粮中分别添加红花籽油和椰子油,采集育成猪肝脏组织,进行高通量转录组测序,找出2种处理间的差异表达基因。利用Illumina HiSeqTM2500高通量RNA⁃seq测序技术对2种处理育成猪肝脏进行转录组测序,使用TopHat2软件将测序得到的Reads序列与猪参考基因组(Sscrofa 10.2)序列比对,找出差异表达基因,并在Nr、GO和KEGG数据库中进行功能注释、富集分析和聚类分析。结果显示:红花籽油组和椰子油组肝脏差异表达基因共有938个,与椰子油组相比,红花籽油组表达上调基因有479个,下调基因有459个;GO功能分类注释到细胞组成、生物学过程和分子功能数据库中差异表达基因数分别有773、768和729个;注释到KEGG通路中差异表达基因数346个,显著富集通路为酮体生成与降解通路和类萜骨架生物合成通路(P<0.05)。
关键词:红花籽油;椰子油;转录组;高通量RNA测序;差异表达基因;代谢通路;肝脏;育成猪
油脂脂肪酸按饱和程度可分为饱和脂肪酸、单不饱和脂肪酸和多不饱和脂肪酸,脂肪酸链长和饱和程度都会影响脂肪吸收代谢机制[1-2]。Sun等[3]报道,长链饱和脂肪酸能增加动物脂肪沉积,长链不饱和脂肪酸能调节脂肪代谢,中链饱和脂肪酸则降低动物增重效果。Vallim等[4]证实多不饱和脂肪酸能调节脂肪代谢酶、载脂蛋白、转录因子等关键基因表达,这些基因的表达水平会直接影响猪肉脂肪组织中饱和脂肪酸与不饱和脂肪酸的组成比例[5]。饲喂富含多不饱和脂肪酸饲粮动物的体脂肪沉积量要低于饲喂富含饱和脂肪酸饲粮的动物[6]。油脂类型能调节脂肪组织基因表达和脂肪酸组成[7]。红花籽油含有高品质多不饱和脂肪酸,其亚油酸含量在70%~85%,而亚油酸是过氧化物酶体增殖物激活受体(PPARs)的内源性激活剂[8-10]。椰子油饱和脂肪酸含量丰富,超过90%,其中,短、中链饱和脂肪酸能被动物直接吸收进入肝脏快速代谢[11-12]。因此,选用这2种食用油研究不同类型油脂对相关基因表达调控机制具有一定意义。随着测序技术的发展,近年来开始使用的高通量测序技术已经显示出其卓越的性能。高通量测序技术针对不同品种动物和同一品种动物不同组织间挖掘差异表达基因和显著富集代谢通路方面的研究较多。但从动物营养角度出发,饲喂不同处理饲粮,通过高通量测序技术找出组织中差异表达基因及显著富集代谢通路的研究未见报道。因此,本研究针对肥育猪饲粮中添加富含短、中链饱和脂肪酸的椰子油和富含亚油酸的红花籽油,通过使用Illumina测序技术对育成猪肝脏进行转录组测序和生物信息学分析,找出肝脏中差异表达基因和显著富集代谢通路,进一步了解脂肪代谢相关分子机制。
1 材料与方法
1.1 试验材料
1.1.1 试验动物及饲养管理
本试验从5窝相同日龄军牧一号母猪中选取18头,体重为(59.11±1.73)kg,试验地在吉林大学农业试验基地纯种猪场。试验猪随机分成2组,每组3个重复,每个重复3头猪,每栏为1个重复。猪舍每日清粪2次(04:00和13:00)。1组饲喂添加红花籽油的饲粮,2组饲喂添加椰子油的饲粮。每日05:00、11:00和16:00饲喂,自由饮水。预试期7 d,正试期60 d。
1.1.2 试验饲粮
红花籽油购于新疆红花缘科技有限公司,属于压榨一级食用油,亚油酸含量为76.66%;椰子油购于秦皇岛金海特种食用油工业有限公司,属于精炼级食品加工用油,饱和脂肪酸含量超过90%,其中,短、中链饱和脂肪酸含量接近60%。试验饲粮参照NRC(1998)肥育猪营养需要推荐营养水平进行配制。红花籽油和椰子油脂肪酸百分组成见表1。试验饲粮组成及营养水平见表2。
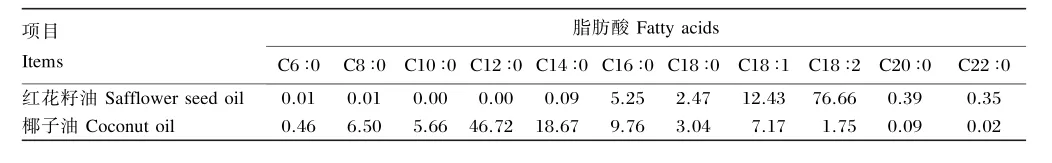
表1 红花籽油和椰子油脂肪酸百分组成Table 1 Percentage composition of fatty acids in safflower seed oil and coconut oil %
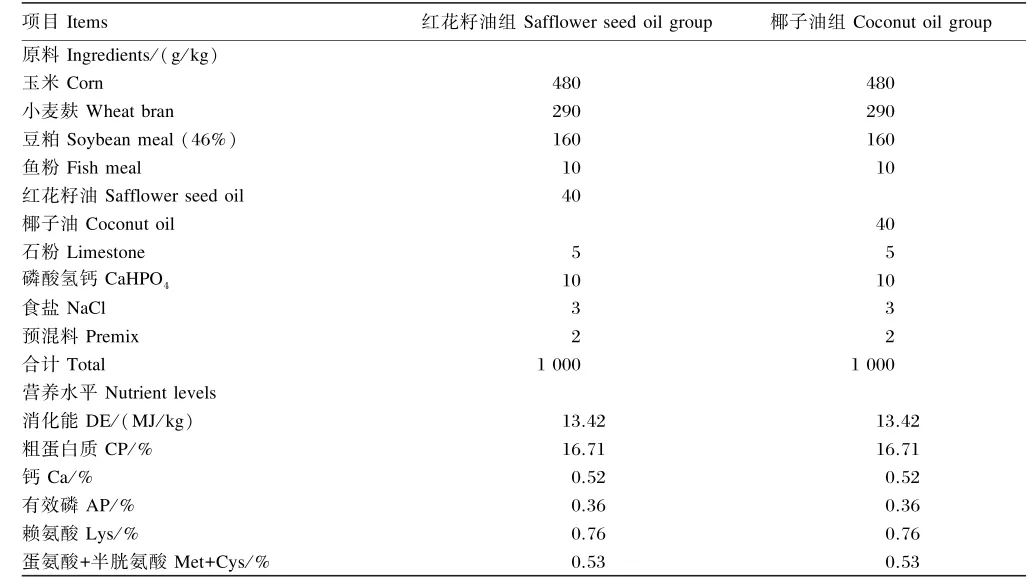
表2 试验饲粮组成及营养水平(风干基础)Table 2 Composition and nutrient levels of experimental diets(air⁃dry basis)
1.1.3 组织样品采集
试验结束后一次性屠宰,采集肝脏组织,切块,用冻存管分装,并迅速置于液氮中,然后于-80℃冰箱中冷冻保存,以备总RNA提取。
1.2 试验方法
1.2.1 总RNA提取、浓度测定和质量检测
总RNA提取前,对操作过程中用到的所有器械进行去RNA酶处理。肝脏组织总RNA提取采用Trizol法按产品说明进行。总RNA浓度用Qu⁃bit 2.0检测,纯度用Nanodrop 2000检测,完整性用Agilent 2100检测。然后干冰包装送北京百迈客生物科技有限公司进行高通量转录组测序。
1.2.2 上机文库的制备
总RNA样品检测合格后,进行文库构建,主要流程如下:1)用带有Oligo(dT)的磁珠富集真核生物mRNA;2)加入Fragmentation Buffer将mR⁃NA进行随机打断;3)以mRNA为模板,用六碱基随机引物合成第1条cDNA链,然后加入缓冲液、dNTPs、RNase H和DNA polymeraseⅠ合成第2条cDNA链,利用AMPure XP beads纯化cDNA;4)纯化的双链cDNA再进行末端修复、加A尾并连接测序接头,然后用AMPure XP beads进行片段大小选择;5)最后通过PCR富集得到cDNA文库。文库构建完成后,分别使用Qubit 2.0和Agilent 2100对文库的浓度和插入片段大小进行检测,使用qPCR方法对文库的有效浓度进行准确定量,以保证文库质量。
1.2.3 测序、数据处理和生物信息学分析
检测合格的文库在Illumina cbot上进行簇的生成,然后用Illumina HiSeqTM2500上机测序,测序读长为PE100。利用TopHat2序列比对软件[13]将测序Reads与猪参考基因组(Sscrofa10.2)进行序列比对,获得Mapped Data。基于Mapped Data,进行插入片段长度检验、随机性检验等测序文库质量评估,进行表达量分析、可变剪接分析、新基因发掘和基因结构优化等。在差异表达基因检测过程中,将差异倍数(fold change,FC)≥2且错误发现率(false discovery rate,FDR)<0.01作为筛选标准。FC表示2样品(组)间表达量的比值。FDR是通过使用公认的Benjamini⁃Hochberg校正方法对差异显著性P值进行校正得到的。根据基因在不同样品中的表达量进行差异表达分析、差异表达基因功能注释和功能富集等生物信息学分析。
1.2.4 基因表达量计算
基因表达量计算使用FPKM值法[14],FPKM计算公式为:
FPKM=cDNA Fragments/[Mapped Fragments (Millions)×Transcript Lentth(kb)]。
式中:cDNA Fragments表示比对到某一转录本上的片段数目,即双端Reads数目;Mapped Fragments(Millions)表示比对到转录本上的片段总数,以106为单位;Transcript Length(kb):转录本长度,以103个碱基为单位。
2 结果与分析
2.1 红花籽油组与椰子油组RNA⁃Seq转录组和测序基因Nr、GO、KEGG注释结果
基因需要转录为RNA和翻译为蛋白质才能发挥其功能,因此,基因的功能实际上是基因产物的功能。使用BLAST[15]软件将测序基因与Nr[16]、GO[17]、KEGG[18]数据库进行序列比对,获得已知和新发掘基因的功能注释信息。
将红花籽油组和椰子油组所有测序基因比对到Nr数据库中,得出序列相似性分布情况,相似性在80%以上的序列占到了91%(图略),可以从Nr数据库中获得较全的测序基因蛋白质功能注释信息。
GO数据库适用于各个物种,能对基因、蛋白质进行限定和描述。GO数据库包括3个一级功能数据库,分别是:细胞组分(cellular component,CC)数据库、分子功能(molecular function,MF)数据库和生物学过程(biological process,BP)数据库。对红花籽油组与椰子油组所有测序基因做GO功能分类统计,结果表明差异表达基因与全部基因的富集趋势不同(图略)。
将红花籽油组与椰子油组所有测序基因注释到KEGG数据库中,可注释到KEGG通路中所有基因数7 026个,其中差异表达基因数346个(图略)。
2.2 红花籽油组与椰子油组差异表达基因分析结果
红花籽油组与椰子油组差异表达基因统计结果显示,肝脏组织中差异表达的基因共有938个,其中,与椰子油组相比,红花籽油组表达上调基因有479个,表达下调基因有459个。差异表达基因Log2FC值范围从-6.77到8.28。
与椰子油组比较,红花籽油组上调或下调幅度前15位基因见表3。由表3可知,30个基因中有14个是本次测序发掘的新基因,有6个基因只在Ensembl数据库中能查到,没有基因名称,也属于新基因。在红花籽油组上调幅度最大前15位基因中有5个是已知基因,分别是基质金属蛋白酶8(MMP8)、毒蕈碱型胆碱受体M1(CHRM1)、早幼粒细胞白血病锌指蛋白(ZBTB16)、岩藻糖基转移酶4(FUT4)、促甲状腺激素释放激素降解酶(TRH⁃DE);下调基因中有5个是已知基因,分别是AHNAK核蛋白(AHNAK)、Toll样受体衔接分子2(TRAM2)、接触蛋白3(CNTN3)、胆固醇7α-羟化酶(CYP7A1)、胆固醇1α-羟化酶(CYP1A1)。

表3 红花籽油组与椰子油组相比上调/下调幅度最大前15位基因Table 3 The top 15 of up⁃or down⁃regulated differentially expressed genes in safflower seed oil group vs.coconut oil group

续表3
2.3 红花籽油组与椰子油组差异表达基因GO功能富集分析结果
2.3.1 红花籽油组与椰子油组差异表达基因GO-细胞组成富集分析
所有测序基因比对到CC数据库中差异表达基因数773个。由表4可知,红花籽油组与椰子油组GO-细胞组成中,显著富集在过氧化物酶体中的差异表达基因(DEG)有12个,占该细胞组成基因数(EG)的14.81%,占比对到CC数据库所有差异表达基因数的1.55%;显著富集在高密度脂蛋白(HDL)颗粒中的差异表达基因有6个,占该细胞组成基因数的30%,占比对到CC数据库所有差异表达基因数的0.78%;显著富集在质膜外侧中的差异表达基因有21个,占该细胞组成基因数的10%,占比对到CC数据库所有差异表达基因数的2.72%。
2.3.2 红花籽油组与椰子油组差异表达基因GO-生物学过程富集分析
所有测序基因比对到BP数据库中差异表达基因数768个。由表4可知,红花籽油组与椰子油组GO-生物学过程中,显著富集在细胞内锌离子动态平衡中的差异表达基因有10个,占该生物过程基因数的41.67%,占比对到BP数据库所有差异表达基因数的1.30%;显著富集在锌离子细胞应答中的差异表达基因有5个,占该生物学过程基因数的100%,占比对到BP数据库所有差异表达基因数的0.65%;显著富集在促红细胞生成素细胞应答中的差异表达基因有4个,占该生物过程基因数的100%,占比对到BP数据库所有差异表达基因数的0.52%;显著富集在铜离子解毒作用中的差异表达基因有4个,占该生物学过程基因数的80%,占比对到BP数据库所有差异表达基因数的0.52%。
2.3.3 红花籽油组与椰子油组差异表达基因GO-分子功能富集分析
所有测序基因比对到MF数据库中差异表达基因数729个。由表4可知,红花籽油组与椰子油组GO-分子功能中,显著富集在3-羟丁酸脱氢酶活性中的差异表达基因有7个,占该分子功能基因数的50%,占比对到MF数据库所有差异表达基因数的0.96%;显著富集在细胞因子受体活性中的差异表达基因有8个,占该分子功能基因数的29.63%,占比对到MF数据库所有差异表达基因数的1.10%;显著富集在蛋白质谷氨酰胺γ-谷氨酰转移酶活性中的差异表达基因有5个,占该分子功能基因数的50%,占比对到MF数据库所有差异表达基因数的0.69%。

表4 红花籽油组与椰子油组差异表达基因GO富集分析结果Table 4 Enriched analysis of differentially expressed genes in safflower seed oil group and coconut oil group in gene ontology
2.4 红花籽油组与椰子油组差异表达基因KEGG功能富集分析结果
将红花籽油组与椰子油组所有测序基因注释到KEGG数据库中,可注释到差异表达基因显著富集通路2个(P<0.05),即酮体生成与降解通路和类萜骨架生物合成通路,见表5。

表5 红花籽油组与椰子油组差异表达基因显著富集的通路Table 5 Enriched pathways of differentially expressed genes in safflower seed oil group and coconut oil group
3 讨 论
3.1 红花籽油组与椰子油组转录组数据分析
近年来,转录组测序技术已经应用于各种动物组织的差异分析,在猪的肝脏、腹部脂肪、背最长肌[19]、背部脂肪[20],以及鸡[21]、牛[22]、羊[23]、小鼠[24]、大鼠[25]等各种动物组织上都有应用。高通量测序的准确性依赖于碱基质量值和测序获得Reads的数量和质量,本研究中,红花籽油组共获得Reads总数是37 053 302个,椰子油组Reads总数是39 127 262个,2组碱基质量值Q30均在93.95%以上。将测序Reads与参考基因组序列进行比对,只有比对上的序列才能用于后续分析,所以比对效率直接决定测序序列的可利用性。本研究红花籽油组的比对效率是82.71%,椰子油组的比对效率是83.92%,符合前人的相关报道[19,26-28],说明所选参考基因组能满足后续分析的要求。之后,使用BLAST软件将测序基因分别与Nr、GO、KEGG数据库进行序列比对,获得基因的全面注释信息。
3.2 红花籽油组与椰子油组差异基因表达分析
本研究结果显示,红花籽油组与椰子油组肝脏组织中差异表达基因共有938个,与椰子油组相比,红花籽油组表达上调的基因有479个,下调的基因有459个。红花籽油组显著上调的前2个差异表达基因是Pig_newGene_6102(log2FC=8.28)和Pig_newGene_8843(log2FC=6.71),分别比对到Nr数据库中获得功能注释,Pig_newGene_6102预测功能是载脂蛋白B100,与脂肪的转运密切相关。Pig_newGene_8843预测功能是配子发育蛋白,与脂肪代谢间的联系尚不清楚。红花籽油组显著下调的第1个差异表达基因是AHNAK (log2FC=-6.77),AHNAK的研究相对较多,已发现与脂肪代谢有关联。下面主要介绍4种差异表达显著的基因。
3.2.1 载脂蛋白B100
Pig_newGene_6102是本试验中发现的新基因,其Nr蛋白数据库功能注释是载脂蛋白B100。载脂蛋白B100分子由4 563个氨基酸组成,分子质量约550 ku,分子结构现在还不清楚[29],是乳糜微粒(CM)、极低密度脂蛋白(VLDL)、中间密度脂蛋白(IDL)和低密度脂蛋白(LDL)的主要蛋白质成分,在脂蛋白代谢过程中起核心作用。载脂蛋白B100是LDL受体的配体,参与它们的组装和分泌[30-31]。LDL由脂类为核心,外层载脂蛋白B100单一肽链缠绕组成,是胆固醇的主要转运蛋白[29]。载脂蛋白B100由肝细胞分泌,其分泌受到肝脏中脂肪酸和三酰甘油的调节,比如,油酸可以增强载脂蛋白B100的分泌,二十二碳六稀酸(DHA)则抑制载脂蛋白B100的分泌,棕榈酸对载脂蛋白B100分泌的影响尚不清楚[32]。本试验中Pig_newGene_6102在椰子油中的表达量为0,在红花籽油中的表达量为10.29,与载脂蛋白B100的分泌符合。
3.2.2 T复合物蛋白1γ(T⁃complex protein 1 sub⁃unit gamma,TCP1γ)
Pig_newGene_5 912的Nr蛋白数据库功能注释是T复合物蛋白1γ。T复合物蛋白1属于热休克蛋白60家族蛋白,是一种在真核生物胞液中唯一鉴定到的伴侣蛋白,参与多种蛋白质的细胞内折叠[33]。目前,热休克蛋白70家族和90家族分子伴侣的作用已经阐明[34-35],但60家族分子伴侣的作用还在探索中,仅有少数报道。T复合物蛋白1γ在生长的四膜虫纤毛的生物合成中起重要作用[36-37]。在细胞骨架蛋白,如肌动蛋白和微管蛋白的折叠和组装过程中起关键作用[38-39]。T复合物蛋白1除了折叠和组装功能外,还参与细胞内蛋白质的迁移、复性。有研究报道,神经细胞的轴突神经丝迁移到目的组织需要花几周甚至几个月时间,T复合物蛋白1γ作为分子伴侣结合以维持轴突神经丝的结构不变[40]。T复合物蛋白1γ在红花籽油组中表达量比椰子油组显著。
3.2.3 AHNAK
AHNAK也被看作桥粒连接蛋白(desmoyok⁃in),二者具有同源性。AHNAK是一种巨大蛋白质,相对分子质量600~700 ku,包括AHNAK1和AHNAK2 2个亚型。AHNAK蛋白包括3个结构域:中间是4 400个氨基酸的中心结构域,侧翼是251个氨基酸的N端结构域和1 002个氨基酸的C端结构域。中心结构域由多个高度保守的重复单元组成,每个重复单元包括128个氨基酸残基。而128个氨基酸重复单元又呈现明显的内部重复结构,每个重复结构又由7个氨基酸残基组成[41-44]。AHNAK在多种细胞组分中被发现,如肌肉细胞、上皮细胞、T细胞、质膜[43],在细胞和生物学水平发挥重要功能,目前已知的功能包括参与细胞分化、细胞信号转导、质膜修复、胞泌作用调节、Ca2+通道调节和血脑屏障[45-46]。近年研究报道,AHNAK敲除鼠能阻碍老鼠生长和减少脂肪组织,尤其是给老鼠饲喂高脂饲粮时,AHNAK敲除鼠比正常鼠的体重和脂肪重都要低,这说明AHNAK与脂肪代谢可能相关[41]。
3.2.4 细胞色素P450家族(cytochrome P450 family)
细胞色素P450是一类含血红素蛋白的酶,催化多种疏水化合物的单加氧作用,广泛分布于动物组织、植物和微生物中,目前,已经发现有18 687个编码蛋白质的细胞色素P450基因,其中,动物中有5 442个基因,植物中超过6 800个基因,真菌中超过4 800个基因,原生动物中247个基因,真细菌中超过1 200个基因,古细菌中48个,病毒中1个。细胞色素P450有家族和亚家族之分,本研究中差异显著的基因涉及到CYP1A1和CYP7A1。有关研究已经证明,细胞色素P450酶在肾上腺皮质和肝脏中参与类固醇和脂肪酸羟基化作用[47-49]。CYP1A1在胆固醇和脂肪酸代谢中起重要作用,有助于脂肪酸和类固醇的信号分子与血管反应分子的生成,从而影响甘油三酯的浓度[50]。本研究中CYP1A1的表达与Trapnell等[26]的报道一致。CYP7A1是肝脏特异性表达基因,编码胆固醇7-α-羟化酶。7-α-羟化酶是胆汁酸生物合成途径中催化第一步反应的限速酶,通过把多余胆固醇转化成胆汁酸,来维持胆固醇水平在体内环境中的稳定[51-53]。CYP7A1活性的变化会影响血浆胆固醇浓度和肝脏胆汁酸合成量。提高CYP7A1活性能使肝细胞内胆固醇排空,胆汁酸合成增强,血浆胆固醇浓度下降;反之,降低CYP7A1活性则会使肝脏内胆固醇积累,胆汁酸合成减少,血浆胆固醇浓度升高[54]。如人体内CYP7A1基因突变导致胆固醇7-α-羟化酶活性缺失,会引起血浆低密度脂蛋白胆固醇浓度提高,胆固醇在肝脏中实质性积累,胆汁酸分泌显著下降[55]。本研究椰子油组中CYP7A1和CYP1A1表达量都高于红花籽油组。
3.3 红花籽油组与椰子油组差异表达基因GO和KEGG功能富集分析
3.3.1 差异表达基因GO功能富集分析
GO富集分析显示,红花籽油组与椰子油组差异表达基因比对到GO-细胞组成数据库中,差异表达基因显著富集在过氧化物酶体、HDL颗粒和质膜外侧。过氧化物酶体又叫微体,是一种细胞器,普遍存在于真核生物各类细胞中,在肝细胞和肾细胞中更多。过氧化物酶体含有40多种酶类,主要是氧化酶、过氧化物酶和过氧化氢酶,功能是催化脂肪酸的β氧化,将极长链脂肪酸或支链脂肪酸分解代谢,动物组织中有25%~50%的脂肪酸是在过氧化物酶体中氧化的,其他则是在线粒体中氧化。过氧化物酶体的标志酶是过氧化氢酶,它的作用主要是将过氧化氢水解。过氧化氢是氧化酶催化的氧化还原反应中产生的细胞毒性物质,氧化酶和过氧化氢酶都存在于过氧化物酶体中,从而对细胞起保护作用。红花籽油富含亚油酸,亚油酸是PPARs的激活剂,所以饲粮中添加红花籽油能使过氧化物酶体中基因的表达差异显著。HDL是血液中密度最高、颗粒最小的一种脂蛋白,主要在肝脏合成,由载脂蛋白、磷脂、胆固醇和少量脂肪酸组成。血液中多余的血脂是靠HDL来代谢的。HDL的主要功能是清除血液和细胞中过多的胆固醇和LDL,将沉积在血管壁的胆固醇、血小板颗粒剥离下来带回肝脏,转化为胆酸,最后变成胆汁,经胆道-肠道排出体外。HDL能增强血脂代谢能力,保持血管畅通,使血管更清洁,且对血管没有任何损伤,故HDL被美其名为“血管壁清洁工”。质膜是指包围在细胞表面的一层极薄的膜,主要由膜脂和膜蛋白组成,另外还有少量糖,主要以糖脂和糖蛋白的形式存在。质膜外侧常有膜糖类组分延伸与胞外蛋白形成糖-蛋白质复合物,这种复合物与大多数细胞的表面行为有关,在接受外界信息以及细胞间相互识别方面具有重要作用[56]。在GO-生物学过程数据库中显著富集在细胞内锌离子动态平衡、锌离子细胞应答、促红细胞生成素细胞应答和铜离子解毒作用。锌是许多酶的必需成分,如核醣核酸聚合酶、醇脱氢酶、碳酸酐酶和碱性磷酸酯酶等,锌缺乏会导致食欲下降,生长迟缓,免疫异常等,锌补充过量则会降低HDL的水平,因此,细胞内锌离子必须保持动态平衡。铜离子也是许多酶的重要成分,如细胞色素c氧化酶、超氧化物歧化酶和尿酸酶等,研究已经证明,饲粮中锌铜比率与心血管疾病有关联[57]。促红细胞生成素是一种糖蛋白促细胞分裂剂和细胞因子,能特异地调节红细胞先祖细胞的分化及增殖,刺激红细胞的生成。在GO-分子功能数据库中显著富集在3-羟丁酸脱氢酶活性、细胞因子受体活性和蛋白质谷氨酰胺γ-谷氨酰转移酶活性。3-羟丁酸脱氢酶是一种以烟酰胺腺嘌呤二核苷酸(NAD+)或烟酰胺腺嘌呤二核苷酸磷酸(NADP+)为受体、作用于供体CH⁃OH基团上的氧化还原酶,主要参与酮体合成与降解以及丁酸的代谢过程。细胞因子是由多种细胞产生的,具有广泛调节细胞功能作用的多肽分子。细胞因子通过结合细胞表面相应的细胞因子受体而发挥生物学作用。细胞因子与其受体结合后启动复杂的细胞内分子间的相互作用,最终引起细胞基因转录的变化。
3.3.2 差异表达基因KEGG功能富集分析
在生物体内,不同的基因产物相互协调来行使生物学功能。因此进行差异表达基因的通路注释分析有助于进一步解读基因的功能。KEGG数据库是关于通路的主要公共数据库。通过KEGG数据库富集分析,能够找出差异表达基因主要代谢通路,通过通路差异来解释表型差异的分子机制。本研究KEGG注释和聚类分析结果显示,差异表达基因显著富集的通路是酮体生成与降解通路和类萜骨架生物合成通路。酮体是脂肪酸β-氧化的中间产物,包括乙酰乙酸、β-羟丁酸和丙酮,是肝外组织中迅速氧化供能的重要原料,是肝脏输出能源的一种形式。类萜由2个或多个异戊二烯单位头尾相连组成,可以是链状,也可以是环状,其低聚物如全顺-视黄醛、β-胡萝卜素、辅酶Q、鲨烯等都是机体代谢的重要物质。视黄醛是维生素A的氧化产物;β-胡萝卜素是维生素A原;鲨烯是胆固醇的前体;辅酶Q在体内呼吸链电子传递中起重要作用,是细胞呼吸和细胞代谢的激活剂,也是重要的抗氧化剂和非特异性免疫增强剂。肝脏是脂肪代谢和脂肪酸β-氧化的主要场所,能调整外源性脂肪酸的碳链长短、饱和度及合成新的必需脂肪酸,使之接近动物组织特有的脂肪酸组成[56]。肝脏可以决定脂肪酸分解代谢过程中的各级代谢中间物在何种生理状态下该往何处去,而不做无效和无用的工作。从这个意义上看,肝脏是调控脂肪酸代谢去向最理想的器官。本试验中,椰子油所含中链脂肪酸被小肠吸收后能直接通过门静脉进入肝脏,并在肝脏中快速氧化分解成终产物,不会有脂肪酸不完全氧化的情况发生,也就不会有酮体的生成。而红花籽油中所含长链不饱和脂肪酸被小肠吸收后,形成CM,通过淋巴管汇入血液大循环,运输到脂肪组织、肌肉和肝脏进行氧化代谢。长链脂肪酸在肝脏细胞中的氧化则不很完全,氧化的产物乙酰辅酶A既可以直接进入三羧酸循环进一步分解,也可以转变成酮体以提高其在肝外组织中的利用率。肝脏中产生酮体的速度与肝外组织利用酮体的速度相关。因此,饲喂油脂类型不同,油脂在体内代谢途径不同,代谢通路也会有一定差异。
4 结 论
①高通量测序结果显示,红花籽油组与椰子油组肝脏差异表达基因共有938个,其中,上调幅度最大前15个基因和下调幅度最大前15个基因中各有10个是本次研究中新发掘的基因,在参考基因组中找不到已知基因序列,有待进一步研究。
②从差异表达基因GO功能分类注释结果看,细胞组分、分子功能和生物学过程数据库中显著富集基因都与脂类代谢相关。KEGG差异显著富集代谢通路仅有酮体生成与降解通路和类萜骨架生物合成通路。
参考文献:
[1] ROSSI R,PASTORELLI G,CANNATA S,et al.Re⁃cent advances in the use of fatty acids as supplements in pig diets:a review[J].Animal Feed Science and Technology,2010,162(1/2):1-11.
[2] RAMÍREZ M,AMATE L,GIL A.Absorption and dis⁃tribution of dietary fatty acids from different sources [J].Early Human Development,2001,65(Suppl.2):S95-S101.
[3] SUN H,JIANG T,WANG S B,et al.The effect of LXRα,ChREBP and Elovl6 in liver and white adipose tissue on medium⁃and long⁃chain fatty acid diet⁃in⁃duced insulin resistance[J].Diabetes Research and Clinical Practice,2013,102(3):183-192.
[4] VALLIM T,SALTER A M.Regulation of hepatic gene expression by saturated fatty acids[J].Prosta⁃glandins,Leukotrienes and Essential FattyAcids,2010,82(4/5/6):211-218.
[5] TAKEUCHI H,MATSUO T,TOKUYAMA K,et al.Diet⁃induced thermogenesis is lower in rats fed a lard diet than in those fed a high oleic acid safflower oil di⁃et,a safflower oil diet or a linseed oil diet[J].The Journal of Nutrition,1995,125(4):920-925.
[6] SHIMOMURA Y,TAMURA T,SUZUKI M.Lessbody fat accumulation in rats fed a safflower oil diet than in rats fed a beef tallow diet[J].The Journal of Nutrition,1990,120(11):1291-1296.
[7] IYER M N H,SARMAH B C,TAMULI M K,et al.Effect of dietary sunflower oil and coconut oil on adi⁃pose tissue gene expression,fatty acid composition and serum lipid profile of grower pigs[J].Archives of An⁃imal Nutrition,2012,66(4):271-282.
[8] JIA L H,LIU Y,LI Y Z.Determination of wholesome elements and heavy metals in safflower(Carthamus tinctorius L.)from Xinjiang and Henan by ICP⁃MS/ICP⁃AES[J].Journal of Pharmaceutical Analysis,2011,1(2):100-103.
[9] ASGARPANAH J,KAZEMIVASH N.Phytochemis⁃try,Pharmacology and medicinal properties of Cartha⁃mus tinctorius L.[J].Chinese Journal of Integrative Medicine,2013,19(2):153-159.
[10] HIGA R,KURTZ M,MAZZUCCO M B,et al.Folic acid and safflower oil supplementation interacts and protects embryos from maternal diabetes⁃induced dam⁃age[J].Molecular Human Reproduction,2012,18 (5):253-264.
[11] APPAIAH P,SUNIL L,PRASANTH KUMAR P K,et al.Composition of coconut testa,coconut kernel and its oil[J].Journal of the American Oil Chemists Soci⁃ety,2014,91(6):917-924.
[12] DEBMANDAL M,MANDAL S.Coconut(Cocos nu⁃cifera L.:Arecaceae):in health promotion and disease prevention[J].Asian Pacific Journal of Tropical Medi⁃cine,2011,4(3):241-247.
[13] KIM D,PERTEA G,TRAPNELL C,et al.TopHat2:accurate alignment of transcriptomes in the presence of insertions,deletions and gene fusions[J].Genome Bi⁃ology,2013,14(4):R36.
[14] TRAPNELL C,WILLIAMS B A,PERTEA G,et al.Transcript assembly and quantification by RNA⁃Seq reveals unannotated transcripts and isoform switching during cell differentiation[J].Nature Biotechnology,2010,28(5):511-515.
[15] ALTSCHUL S F,MADDEN T L,SCHÄFFER A A,et al.Gapped BLAST and PSI⁃BLAST:a new genera⁃tion of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
[16] DENG Y Y,LI J Q,WU S F,et al.Integrated nr data⁃base in protein annotation system and its localization [J].Computer Engineering,2006,32(5):71-73,76.
[17] ASHBURNER M,BALL C A,BLAKE J A,et al.Gene ontology:tool for the unification of biology[J].Nature Genetics,2000,25(1):25-29.
[18] KANEHISA M,GOTO S,KAWASHIMA S,et al.The KEGG resource for deciphering the genome[J].Nu⁃cleic Acids Research,2004,32(Suppl.1):D277-D280.
[19] CHEN C Y,AI H S,REN J,et al.A global view of porcine transcriptome in three tissues from a full⁃sib pair with extreme phenotypes in growth and fat depo⁃sition by paired⁃end RNA sequencing[J].BMC Ge⁃nomics,2011,12(1):448.
[20] COROMINAS J,RAMAYO⁃CALDAS Y,PUIG⁃OL⁃IVERAS A,et al.Analysis of porcine adipose tissue transcriptome reveals differences in de novo fatty acid synthesis in pigs with divergent muscle fatty acid com⁃position[J].BMC Genomics,2013,14(1):843.
[21] SEVANE N,BIALADE F,VELASCO S,et al.Dietary inulin supplementation modifies significantly the liver transcriptomic profile of broiler chickens[J].PLoS One,2014,9(6):e98942.
[22] LI R W,RINALDI M,CAPUCO A V.Characteriza⁃tion of the abomasal transcriptome for mechanisms of resistance to gastrointestinal nematodes in cattle[J].Veterinary Research,2011,42(1):114.
[23] FAN R W,XIE J S,BAI J M,et al.Skin transcriptome profiles associated with coat color in sheep[J].BMC Genomics,2013,14(1):389.
[24] MORTON N M,NELSON Y B,MICHAILIDOU Z,et al.A stratified transcriptomics analysis of polygenic fat and lean mouse adipose tissues identifies novel candidate obesity genes[J].PLoS One,2011,6(9):e23944
[25] PATEL O V,CASEY T,DOVER H,et al.Homeorhet⁃ic adaptation to lactation:comparative transcriptome a⁃nalysis of mammary,liver,and adipose tissue during the transition from pregnancy to lactation in rats[J].Functional&Integrative Genomics,2011,11(1):193-202.
[26] XING K,ZHU F,ZHAI L W,et al.The liver tran⁃scriptome of two full⁃sibling Songliao black pigs with extreme differences in backfat thickness[J].Journal of Animal Science and Biotechnology,2014,5(1):32.
[27] ESTEVE⁃CODINA A,KOFLER R,PALMIERI N,et al.Exploring the gonad transcriptome of two extreme male pigs with RNA⁃seq[J].BMC Genomics,2011,12(1):552.
[28] RAMAYO⁃CALDAS Y,MACH N,ESTEVE⁃CODI⁃NA A,et al.Liver transcriptome profile in pigs with extreme phenotypes of intramuscular fatty acid compo⁃sition[J].BMC Genomics,2012,13(1):547.
[29] CHAKRABORTY S,CAI Y,TARR M A.In vitro oxi⁃dative footprinting provides insight into apolipoprotein B⁃100 structure in low⁃density lipoprotein[J].Pro⁃teomics,2014,14(21/22):2614-2622.
[30] LI H N,YAN J R,OU W J,et al.Construction of a bi⁃otinylated cameloid⁃like antibody for lable⁃free detec⁃tion of apolipoprotein B⁃100[J].Biosensors and Bio⁃electronics,2015,64:111-118.
[31] SU Q Z,TSAI J,XU E,et al.Apolipoprotein B100 acts as a molecular link between lipid⁃induced endo⁃plasmic reticulum stress and hepatic insulin resistance [J].Hepatology,2009,50(1):77-84.
[32] CAVIGLIA J M,GAYET C,OTA T,et al.Different fatty acids inhibit apoB100 secretion by different path⁃ways:unique roles for ER stress,ceramide,and auto⁃phagy[J].Journal of Lipid Research,2011,52(9):1636-1651.
[33] BHASKAR,KUMARI N,GOYAL N.Cloning,character⁃ization and sub⁃cellular localization of gamma subunit of T⁃complex protein⁃1(chaperonin)from Leishmania donovani[J].Biochemical and Biophysical Research Communications,2012,429(1/2):70-74.
[34] FOLGUEIRA C,REQUENA J M.A postgenomic view of the heat shock proteins in kinetoplastids[J].FEMS Microbiology Reviews,2007,31(4):359-377.
[35] SHONHAI A,MAIER A G,PRZYBORSKI J M,et al.Intracellular protozoan parasites of humans:the role of molecular chaperones in development and patho⁃genesis[J].Protein&Peptide Letters,2011,18(2):143-157.
[36] CYRNE L,GUERREIRO P,CARDOSO A C,et al.The Tetrahymena chaperonin subunit CCTη gene is coexpressed with CCTγ gene during cilia biogenesis and cell sexual reproduction[J].FEBS Letters,1996,383(3):277-283.
[37] SEIXAS C,CRUTO T,TAVARES A,et al.CCTα and CCTδ chaperonin subunits are essential and required for cilia assembly and maintenance in Tetrahymena [J].PLoS One,2010,5(5):e10704.
[38] GRANTHAM J,RUDDOCK L W,ROOBOL A,et al.Eukaryotic chaperonin containing T⁃complex polypep⁃tide 1 interacts with filamentous actin and reduces the initial rate of actin polymerization in vitro[J].Cell Stress&Chaperones,2002,7(3):235-242.
[39] FRYDMAN J,NIMMESGERN E,ERDJUMENT⁃BROMAGE H,et al.Function in protein folding of TRiC,a cytosolic ring complex containing TCP⁃1 and structurally related subunits[J].EMBO Journal,1992,11(13):4767-4778.
[40] BOURKE G J,ELALAMI W,WILSON S J,et al.Slow axonal transport of the cytosolic chaperonin CCT with Hsc73 and actin in motor neurons[J].Journal of Neuroscience Research,2002,68(1):29-35.
[41] DAVIS T A,LOOS B,ENGELBRECHT A⁃M.AH⁃NAK:the giant jack of all trades[J].Cellular Signal⁃ling,2014,26(12):2683-2693.
[42] HAASE H.Ahnak,a new player in β⁃adrenergic regu⁃lation of the cardiac L⁃type Ca2+channel[J].Cardio⁃vascular Research,2007,73(1):19-25.
[43] MARG A,HAASE H,NEUMANN T,et al.AHNAK1 and AHNAK2 are costameric proteins:AHNAK1 af⁃fects transverse skeletal muscle fiber stiffness[J].Bio⁃chemical and Biophysical Research Communications,2010,401(1):143-148.
[44] DE MORRÉE A,DROOG M,GRAND MOURSEL L,et al.Self⁃regulated alternative splicing at the AH⁃NAK locus[J].The FASEB Journal,2012,26(1):93-103.
[45] HUANG Y C,LAVAL S H,VAN REMOORTERE A,et al.AHNAK,a novel component of the dysferlin protein complex,redistributes to the cytoplasm with dysferlin during skeletal muscle regeneration[J].The FASEB Journal,2007,21(3):732-742.
[46] ALVAREZ J L,PETZHOLD D,PANKONIEN I,et al.Ahnak1 modulates L⁃type Ca2+channel inactivation of rodent cardiomyocytes[J].Pflügers Archiv⁃Europe⁃an Journal of Physiology,2010,460(4):719-730.
[47] NEBERT D W,WIKVALL K,MILLER W L.Human cytochromes P450 in health and disease[J].Philo⁃sophical Transactions of the Royal Society of London Series B⁃Biological Sciences,2013,368(1612):20120431.
[48] COOPER D Y,ESTABROOK R W,ROSENTHAL O.The stoichiometry of C21 hydroxylation of steroids by adrenocortical microsomes[J].Journal of Biologi⁃cal Chemistry,1963,238:1320-1323.
[49] ORRENIUS S.Some aspects on the hydroxylation of drugs,steroid hormones and fatty acids(omega⁃oxida⁃tion)in rat liver microsomes[J].Hoppe⁃Seylers Zeitschrift für Physiologische Chemie,1968,349 (11):1619-1621.
[50] BAILÓN⁃SOTO C E,GALAVIZ⁃HERMÁNDEZ C,LAZALDE⁃RAMOS B P,et al.Influence of CYP1A1∗2C on high triglyceride levels in female Mexican indigenous tarahumaras[J].Archives of Medical Research,2014,45(5):409-416.
[51] RUSSELL D W.The enzymes,regulation,and genetics of bile acid synthesis[J].Annual Review of Biochem⁃istry,2003,72(1):137-174.
[52] NORLIN M,WIKVALL K.Enzymes in the conver⁃sion of cholesterol into bile acids[J].Current Molecu⁃lar Medicine,2007,7(2):199-218.
[53] MAST N,GRAHAM S E,ANDERSSON U,et al.Cholesterol binding to cytochrome P450 7A1,a key enzyme in bile acid biosynthesis[J].Biochemistry,2005,44(9):3259-3271.
[54] ABRAHAMSSON A,KRAPIVNER S,GUSTAFS⁃SON U,et al.Common polymorphisms in the CYP7A1 gene do not contribute to variation in rates of bile acid synthesis and plasma LDL cholesterol concentration[J].Atherosclerosis,2005,182(1):37-45.
[55] BARCELOS A L V,CHIES R,ALMEIDA S E M,et al.Association of CYP7A1⁃278A>C polymorphism and the response of plasma triglyceride after dietary intervention in dyslipidemic patients[J].Brazilian Journal of Medical and Biological Research,2009,42 (6):487-493.
[56] 王镜岩,朱圣庚,徐长法.生物化学[M].3版.北京:高等教育出版社,2002.
[57] GOLDHABER S B.Trace element risk assessment:es⁃sentiality vs.toxicity[J].Regulatory Toxicology and Pharmacology,2003,38(2):232-242.
Differentially Analysis of Liver Transcriptomes of Finishing Pigs Fed Different Dietary Fats Sources
LI Jianping
1,2
QIN Guixin
1
ZHAO Zhihui
3
ZHANG Qiaoling
4
WANG Dali
3
SUN Boxing
3
JIANG Hailong
1∗
(责任编辑 武海龙)
(1.College of Animal Science and Technology,Jilin Agricultural University,Changchun 130118,China;2.Department of Animal Husbandry,Henan University of Animal Husbandry and Economy,Zhengzhou 450011,China;3.Agriculture Test Station,Jilin University,Changchun 130062,China;4.College of Animal Science and Veterinary Medicine,Jilin University,Changchun 130062,China)
Abstract:The study was to detect differentially expressed genes in liver of finishing pigs which were divided into two groups and separately fed safflower seed oil and coconut oil.The chosen pigs were slaughtered and liv⁃er tissue were isolated and their mRNA were respectively extracted.The liver transcriptomes were analyzed by Illumina HiSeq TM 2500 high⁃throughput RNA sequencing system.All RNA⁃Seq reads were mapped on the reference pig genome(Sscrofa10.2)using TopHat2 software.All of differentially expressed genes were anno⁃tated using Nr,GO and KEGG databases.The results showed that there were 938 differentially expressed genes in liver between two groups.479 genes were up⁃regulated and 459 genes were down⁃regulated in safflower seed oil group vs.coconut oil group.There were 773 differentially expressed genes annotated into database of cellu⁃lar component,768 into biological process,729 into molecular function in GO databases.There were 346 dif⁃ferentially expressed genes annotated into KEGG databases,and clustered pathways of differentially expressed genes included synthesis and degradation of ketone bodies and terpenoid backbone biosynthesis(P<0.05).[Chinese Journal of Animal Nutrition,2015,27(7):2128⁃2134]
Key words:safflower seed oil;coconut oil;transcriptome;high⁃throughput RNA sequencing;differentially expressed gene;metabolism pathway;liver;finishing pig
Corresponding author∗,associate professor,E⁃mail:hljiang@jlau.edu.cn
通信作者:∗姜海龙,副教授,硕士生导师,E⁃mail:hljiang@jlau.edu.cn
作者简介:李建平(1976—),男,山西兴县人,讲师,博士,研究方向为动物营养与饲料科学,E⁃mail:sxljp2004@126.com
基金项目:吉林省安全优质肉猪现代生产关键技术示范(20140301013ny);国家科技支撑计划课题生猪健康养殖模式构建与示范(2012bad39b03)
收稿日期:2015-01-06
doi:10.3969/j.issn.1006⁃267x.2015.07.018
文章编号:1006⁃267X(2015)07⁃2128⁃12
文献标识码:A
中图分类号:S828
——头发

