基于SRAP的松茸特异性分子标记*
兰 西,吴松权,金美玉,全雪丽
(延边大学农学院,吉林 延吉 133000)
基于SRAP的松茸特异性分子标记*
兰 西,吴松权,金美玉,全雪丽**
(延边大学农学院,吉林 延吉 133000)
为了准确鉴定松茸,确保消费者品尝到货真价实的松茸,该研究基于SRAP(sequence-related amplified polymorphism分子标记,从300对引物组合中分别筛选出1对松茸特异性引物(ME10、EM6),并将这对引物扩增的特异条带进行克隆、测序和设计引物后,成功地构建了松茸特异性分子标记(304 bp)。松茸在特异性分子标记(304 bp)均能扩增出条带,该标记在分子水平上可准确、快速地鉴定出松茸,并可有效地保证松茸的品质。
松茸;假松茸;SRAP;特异性分子标记;特异引物
松茸(Tricholoma matsutake) 属于口蘑属 (Tricholoma)真菌[1-2],菌株与针叶树根部形成外生菌根[3]。被誉为“菌中之王”[4]。除了可食用外,松茸还具有很高的药用价值。由于难以合成具有树木根系活力的营养条件和生物环境,驯化、栽培十分困难,目前还不能实现人工栽培[5-6]。松茸营养丰富,现代医学研究证明松茸可治疗糖尿病,并具有抗癌作用[7]。现已经被列为国家重点二级保护物种[8]。
假松茸 (Tricholoma bakamatsutake)即假松口蘑,因其外观、香味、口感与松茸相似[9],常被拿来冒充松茸销售。常规鉴别松茸与假松茸的方法主要通过目测和显微镜观察子实体形态特点,但这些常规方法仍存在较大误差,易出现判断失误的情况。
前人对该领域的研究大多数是针对多态性条带加以分析,该方法具有不稳定、差异性大等特点,随着人们对松茸的不懈探索,近几年更多分子水平上的不同方法陆续应用于鉴别松茸和假松茸,如:基于 ITS(internal transcribed spacers)[10]、基于RAPD(polymerase chain reaction) 的 SCAR(sequence characterized amplified regions) 标记[11]。但还没有基于SRAP(sequence-related amplified polymorphism)的方法区分松茸和假松茸的报道。SRAP是一种新型的分子技术,具有更高的共显性、高重复性、多态性、信息量。除此之外,其引物设计简单,具有在基因组中分布均匀的特点。因此,SRAP广泛应用于种质资源遗传评价、遗传图谱构建、绘制基因转录图谱[12-14]。本研究将基于SRAP分子标记构建松茸的特异性分子标记,以探索出一种准确、快速鉴定松茸的方法。
1 材料与方法
1.1材料
分别从黑龙江省牡丹江、吉林省安图县、吉林省龙井县采集松茸子实体,经过专家傅伟杰教授鉴定以后,在-75℃的超低温冰箱中保存。
1.2基因组总DNA提取
采用改进的CTAB(chexadecyltrimethylammonium bromide)法提取松茸与假松茸的DNA[15]。
1.3SRAP分析
松茸、假松茸基因池的建立采用了BSA(bovine serum albumin)方法[16],选取5个松茸子实体和5个假松茸子实体。每个松茸子实体各取500 ng混合组成松茸池;每个假松茸子实体各取500 ng混合组成假松茸基因池。松茸与假松茸来源见表1。

表1 松茸、假松茸的来源Tab.1 Source of T.matsutake and T.bakamatsutake in this study
1.3.1反应体系
总体积为20 μL,包括模版5 ng·μL-1DNA 2 μL,2 μmol·L-1上游、下游引物各2 μL,2 mmol·L-1dNTP 2 μL,2.5 U·μL-1B Taq DNA聚合酶0.4 μL(Toyobo公司),10×B Taq buffer 2 μL,剩余使用双重蒸馏水补足。
1.3.2PCR扩增程序
94℃预变性5 min,94℃变性1 min,35℃退火1 min,72℃延伸1 min,5个循环;94℃变性1 min,50℃退火1 min,72℃延伸1 min,36个循环;最后72℃延伸5 min。
1.3.3PCR产物检测
PCR产物放在1%的琼脂糖凝胶上电泳,用0.5 μg·mL-1的溴化乙锭染色。电泳缓冲液为1×TAE(pH 8.0),在85 V恒压下电泳约40 min左右,用Dolphin凝胶成像仪拍照保存。
1.3.4特异性片段的回收、克隆和测序
将目的DNA片段切下后,装进1.5 mL的离心管中,按照康为世纪公司胶回收试剂盒说明书将目的片段回收。将回收的片段与pMD18-T载体连接后,转化到大肠杆菌JM109上,转化后的片段经克隆过夜摇菌培养,提取质粒DNA后,采用M13引物进行鉴定,其鉴定体系总体积为20 μL,包括质粒5 ng·μL-1DNA 2 μL,2 μmol·L-1上游、下游引物各2 μL,2 mmol·L-1dNTP 2 μL,2.5 U·μL-1B Taq DNA聚合酶0.4 μL(Toyobo公司),10×B Taq buffer 2 μL,剩余用双重蒸馏水补足。鉴定的阳性克隆委托上海英潍捷基贸易有限公司进行测序。
1.4松茸特异引物设计和PCR扩增
根据测序结果和引物设计原则用Primer primer 5设计松茸特异性上游引物Tm-1(ACGGAGGGAG GAGGATAACG) 和下游引物Tm-2(ATCTTGCC CTTTGTCCAGCC);引物由上海英潍捷基贸易有限公司合成。PCR反应总体积为20 μL,除上游、下游引物(2 μmol·L-1)各2 μL,其余成分同1.3.2 PCR反应体系相同。
松茸特异性PCR程序为:94℃预变性5 min,94℃变性30 s,60℃退火30 s,72℃延伸20 s,循环30次;最后72℃延伸5 min。
2 结果与分析
2.1SRAP标记
以松茸、假松茸基因池DNA为模板,从300对引物组合中(ME1~ME10和EM1~EM30),筛选出1对松茸具有特异性的引物ME10(TGGGGACAACC CGGCTT)和EM6(GACTGCGTACGAATTGCA)。采用这对引物对构成松茸基因池每个单株的DNA进行扩增(图1),在松茸单株中均能够扩增出一条650 bp的条带,而在假松茸单株中则没有出现650 bp的条带。

图1 引物ME10、EM6对松茸、假松茸的单株PCR扩增图谱Fig.1 PCR results of individuals of T.matsutake and T. bakamatsutake with primers of ME10 and EM6

图2 松茸目的片段的碱基序列Fig.2 Sequence of target fragment of T.matsutake
2.2松茸特异引物性标记的建立
根据测序的结果,设计了1对松茸特异引物Tm-1和Tm-2(图2双下滑线部分),对松茸和假松茸基因池DNA的每个单株重新扩增。在Tm-1和 Tm-2的引物扩增中,松茸均能扩增出304 bp的特征带,但是假松茸未能扩增出任何条带,结果与预测的相似,见图3。结果表现出特异标记的准确性和高效。

图3 引物Tm-1和Tm-2对松茸、假松茸基因池DNA每个单株的PCR扩增图谱Fig.3 PCR results of individuals of T.matsutake and T. bakamatsutake with primers of Tm-1 andTm-2
2.3对SRAP特异分子标记的验证结果
为了进一步验证松茸标记的准确性,用松茸特异性引物(Tm-1、Tm-2)对源自吉林省龙井市、云南省昆明市、日本、云南省宝山镇等不同地区松茸和源自云南省丽江市、吉林省安图县、吉林省龙井市等不同地区的假松茸进行了PCR扩增结果见图4。
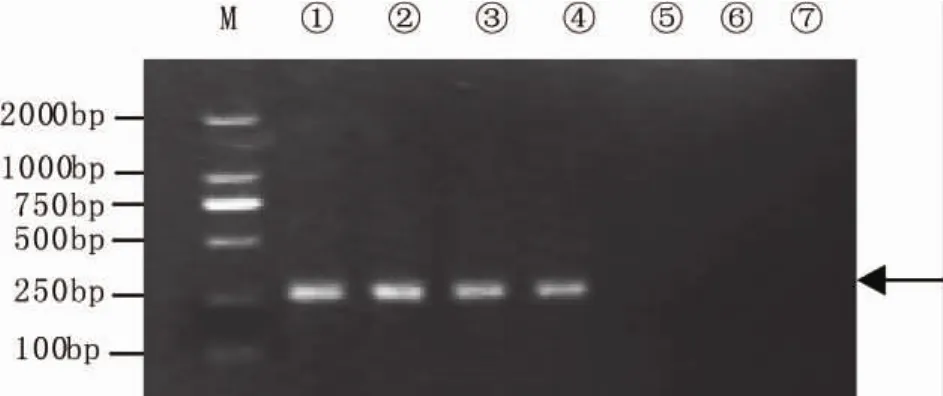
图4 引物Tm-1和Tm-2对松茸和假松茸PCR扩增图谱Fig.4 PCR results of T.matsutake and T.bakamatsutake with primers of Tm-1 and Tm-2
图4表明在来源不同的松茸中,松茸特异性引物均扩增出304 bp特异条带,假松茸中则未扩增出任何条带。
2.4结论
本研究基于SRAP建立松茸特异性分子标记即304 bp,通过该标记可以在分子水平上快速准确地鉴定出松茸与假松茸。基于松茸特异序列构建的分子标记可确保消费者购买和品尝到货真价实的松茸。
3 讨论
假松茸目前也属于中国珍稀的食用菌,因其肉嫩味美,营养物质丰富,而深受人们喜爱。从形态上很难鉴定出松茸和假松茸,即使是松茸方面专家有时也不能区分开来。松茸与假松茸相比,其营养价值更高。加之松茸由于种种原因不能人工栽培,目前出售的松茸均为野生松茸,其价格是假松茸价格的10倍以上。因此常有不法商贩将假松茸混入松茸中出售,严重影响了松茸品质。基于SRAP建立松茸特异性分子标记可以快速、准确地鉴别出松茸与假松茸,不但维护消费者利益,同时也为以采摘松茸为生的菇农创造更加公平的条件。
鉴定物种的真伪常用分子标记方法,该方法是在生物DNA层面进行鉴定,可准确反应出该个体或种群的本质特征[17],其鉴定结果准确,而且成本低,实验操作方便,可以在实验室操作而不需要等待生物生长周期,也不受外界环境条件的影响[18-19]。目前分子标记方法很多,如RAPD、ITS、SRAP、SCAR等。吴松权等[11]在RAPD分子标记的基础上建立松茸特异性SCAR标记。SRAP分子标记方法相对RAPD能扩增出更多的条带,可更加准确地鉴定出松茸与假松茸。沙涛等[10]利用松茸与假松茸存在的核糖体基因间隔区(ITS)长度大小不一,而将松茸与假松茸区别开。而本实验是基于特异基因序列的有无来鉴别松茸,实验结果更可靠。本研究在SRAP的分子标记基础上,根据基因序列的有无,分别发现了松茸与假松茸的特异序列;同时根据其特异序列分别设计了1对特异引物,经PCR建立了松茸的特异性的分子标记,此分子标记具有高效、准确、可重复等特点,进一步表明了SRAP分子标记在物种真伪鉴别中的实用性。之前我们曾在SRAP分子标记基础上克隆了松茸特异性序列(422 bp) 并建立了其特异性分子标记(243 bp)[20]。但该标记仍然不能满足开发松茸特异性试剂盒所需的DNA序列及其分子标记的要求。本研究所克隆的松茸特异性DNA序列长度为650 bp,与之前422 bp DNA序列的相似性仅为50.6%,属于不同的基因或DNA片段;同时所构建的松茸特异性分子标记长度(304 bp)与之前也不同,这些松茸特异性分子标记的建立,为松茸特异试剂盒的开发奠定了基础。
将本实验所得的序列在NCBI上比对分析未发现任何结果,原因可能是已报到松茸和假松茸基因序列太少,也可能是所得的序列只是基因标记序列,其验证工作正在进行中。
[1]李长田,刁盈盈,毛欣欣,等.松茸液态发酵菌丝生长量因子的研究[J].菌物学报,2012,31(2):229-234.
[2]方明,李玉,姚方杰,等.松茸研究概况[J].中国食用菌,2005,24(6):12-15.
[3]Takashi Yamanaka,Miki Konno,Masataka Kawai,et al.The host ranges of conifer-associated Tricholoma matsutake,Fagaceae-associated Tricholoma bakamatsutake and Tricholoma fulvocastaneum are wider in vitro than in nature[J].Mycologia, 2014,106(3):397-406.
[4]张微思,罗孝坤,张丽英,等.松茸菌丝体的纯培养及其鉴定[J].中国食用菌,2010,29(3):34-36.
[5]吴松权,全雪丽,傅伟杰,等.RAPD标记在松口蘑菌丝体鉴定中的应用[J].中国食用菌,2006,25(4):41-43.
[6]Yamada Akiyoshi,Endo Naoki,Murata Hitoshi,et al.Tricholoma matsutake Y1 strain associated with Pinus densiflora shows a gradient of in vitro ectomycorrhizal specificity with Pinaceae and oak hosts[J].Mycoscience,2014,55(1):27-34.
[7]傅伟杰,许广波,梁运江,等.松茸、姬松茸生产关键技百问百答[M].北京:中国农业出版社,2005:1-8.
[8]刘培贵,袁明生,王向华,等.松茸群生物资源及其合理利用与有效保护[J].自然资源学报,1999,14(3):245-252.
[9]史刚荣.姬松茸研究的现状和展望[J].江苏食用菌,1995,16(4):21-22.
[10]沙涛,丁骅孙,李觅,等.松口蘑与假松口蘑ITS序列测定和分析比较[J].菌物学报,2005,24(1):48-52.
[11]吴松权,管清杰,全雪丽,等.松茸SCAR标记的建立[J].林业科学,2007,43(10):150-153.
[12]徐操,赵宝华.SRAP分子标记的研究进展及其应用[J].生命科学仪器,2009,7(4):24-27.
[13]应正河,吴小平,谢宝贵,等.香菇SRAP反应体系的优化[J].食用菌学报,2006,13(4):1-5.
[14]许美燕,唐传红,张劲松,等.利用SRAP和ISSR建立快速鉴定灵芝属菌株的SCAR标记[J].菌物学报,2008,27(5):707-717.
[15]曾东方,罗信昌,傅伟杰.松口蘑菌丝体的分离和RAPD-PCR分析[J].微生物报,2001,41(3):278-286.
[16]邹喻苹,葛颂,王晓东.系统与进化植物学中的分子标记[J].北京:科学出版社,2001:30-51.
[17]方宣钧,刘思衡,江树业.品种纯度和真伪的DNA分子标定及其用[J].农业生物技术学报,2000,8(2):106-110.
[18]张婷,王元忠,张晓东,等.分子标记在食(药)用真菌遗传多样性研究中的应用[J].生命科学,2013,25(10):1000-1007.
[19]曹辉,刘玉萍.分子标记技术在药品种鉴别中的应用[J].中国药学杂志,1998,33(5):269-273.
[20]兰西,全雪丽,金美玉,等.基于SRAP的松茸与假松茸特异性分子标记[J].食用菌学报,2015,22(1):21-25.
Specific Molecular Markers of Tricholoma matsutake Based on SRAP
LAN Xi,WU Song-quan,JIN Mei-yu,QUAN Xue-li
(College of Agriculture,Yanbian University,Yanji 133000,China)
In order to accurately identify between Tricholoma matsutake and T.bakamatsutake,and to ensure that consumers enjoy real T.matsutake,we screened a pair of primer that was specific for T.matsutake from 300 pairs of primers based on SRAP(sequence-related amplified polymorphism molecular markers.Then a SCAR(sequence characterized amplified regionsmarker(304 bp)for T.matsutake was successfully developed with cloning,sequencing and designing specific primers.This SCAR marker only presented in the individuals of T.matsutake,and the marker might be used quickly and accurately identify T.matsutake on the molecular level and effectively guaranteed the quality of the T.matsutake.
Tricholoma matsutake;Tricholoma bakamatsutake;SRAP;molecular markers of specific;specific primer
S646.9A1003-8310(2015)05-0041-05
10.13629/j.cnki.53-1054.2015.05.011
吉林省自然科学基金(20130101146JC)。
兰西(1989-),女,在读硕士研究生,主要从事生物技术方面研究。E-mail:1006322610@qq.com
**通信作者:全雪丽(1973-),女,博士,教授,主要从事植物学方面研究。E-mail:quanxueli2000@yahoo.com.cn
2015-06-28

